INTRODUCCIÓN
Se considera la Salmonella como un género variado de bacilos gramnegativos y un importante patógeno alimentario, responsable de una alta morbilidad y mortalidad en un rango amplio de huéspedes, entre ellos, humanos, aves y mamíferos. Esta bacteria causa en humanos la enfermedad conocida como salmonelosis, que se presenta con diarrea leve, aunque puede llegar a ser mortal dependiendo del estado inmune del individuo, de la dosis de infección y la cepa infectante. Es de distribución cosmopolita y afecta a todos los grupos de edades, tanto en los países desarrollados como en los que están en vía de desarrollo, constituyéndose en un importante problema de salud pública, con impacto a nivel económico tanto en humanos como animales 1.
La detección e identificación rápida y confiable de este patógeno en alimentos y demás ambientes es la clave para salvaguardar el suministro de alimentos y evitar su contaminación 2. Entre las patologías más importantes se presenta la salmonelosis no tifoidea, una de las enfermedades bacterianas transmitida por los alimentos más frecuente. Los miembros del género Salmonella son considerados el segundo patógeno bacteriano más prevalente, y ocasionan alrededor de 94 625 casos en personas, confirmados en 2015 en Europa, y representan el 22.5 % de los brotes reportados por contaminación en alimentos. En los Estados Unidos, Salmonella causa aproximadamente 1.1 millones de casos de enfermedades transmitidas por alimentos cada año 3.
A nivel Colombia, se tiene un subregistro acerca del verdadero problema de Salmonella, tanto en identificación de reservorios como en infecciones, morbilidad y mortalidad asociada; además, el estudio más reciente sobre Salmonella aislada de alimentos se realizó en la Costa Atlántica y demostró la presencia de diversos serotipos circulantes 4.
El género Salmonella consta de dos especies: Salmonella entérica y Salmonella bongori (V), que incluye más de 2500 serotipos. Por su parte, S. entérica se subdivide en seis subespecies: entérica (I), salamae (II), arizonae (IIIa), diarizonae (IIIb), houtenae (IV) e indica (VI) 5.
La identificación de los serogrupos y serotipos de Salmonella se ha realizado a través del uso de antisueros (serotipificación); no obstante, el aislamiento microbiológico, las pruebas bioquímicas y la serotipificación llevan un tiempo considerable, son costosas y han mostrado limitaciones, en algunos casos han sido incapaces de establecer dichos serotipos 6. Estas dificultades han hecho que se desarrollen técnicas moleculares más precisas para la identificación, como por ejemplo, el uso de la reacción en cadena de la polimerasa (PCR) y sus variantes, para la amplificación de genes como invasina A (invA), siendo un ensayo selectivo y robusto 7.
Considerando la importancia del diagnóstico con certeza del género Salmonella y sus serotipos, se propone esta revisión con el objetivo de describir cuáles son los marcadores moleculares y las diferentes técnicas utilizadas para la determinación de este patógeno.
METODOLOGÍA
Para la elaboración de esta revisión de tema no sistemática se realizó una búsqueda bibliográfica en las siguientes bases de datos: Pubmed, Science Direct, Elsevier, Proquest y Ovid, utilizando los siguientes descriptores: Alimento (D005502), Molecular (D002850), Patógeno (D009676), PCR (D016133) y Salmonella (D012475), los cuales se combinaron de diferentes maneras para finalmente obtener 50 artículos. Los criterios de selección de los artículos fueron: [1] publicaciones en revistas indexadas; [2] publicaciones de los últimos quince años; [3] publicaciones que mencionaran análisis y técnicas moleculares. Los artículos seleccionados fueron tanto en inglés como en español, publicados entre 2005 y 2020, años en los que se encontró información indispensable para la construcción de esta revisión.
MÉTODOS MOLECULARES PARA LA DETERMINACIÓN DE SALMONELLA
La detección de patógenos en alimentos por métodos de diagnóstico convencional implica varias etapas, y en muchos casos son eficaces y con bajos costos, sin embargo, presentan la desventaja de aportar resultados en días o semanas, ser laboriosos en algunos casos y solo permiten la identificación de género y en pocos casos la especie 8.
A través de la historia se tiene registro que los principales avances en los ensayos de detección de patógenos en alimentos, basados en ácidos nucleicos, se produjeron a partir de 1980 9. Considerando lo mencionado, los primeros métodos de identificación molecular que se emplearon fueron la hibridación ADN-ADN, el análisis de secuencias del ADNr 16S, la hibridación con sonda específica y el análisis de Polimorfismos de longitud de fragmentos de restricción (RFLP) o ribotipificación 9.
Con el pasar de los años, descubrimientos como la PCR, la clonación, la secuenciación y la tecnología de detección por fluorescencia, así como la accesibilidad a información en bases de datos, han aportado al desarrollo de nuevas herramientas moleculares, mostrando un incremento en su uso 8,9.
Se han desarrollado diferentes protocolos basados en métodos moleculares para la detección de Salmonella a partir de muestras clínicas y alimentos 10. Recientemente se ha dado un paso hacia las plataformas moleculares más sofisticadas para la identificación de microrganismos patógenos, incluyendo sistemas de amplificación in vitro en tiempo real, biosensores y microarreglos, los cuales han sido desarrollados o se están desarrollando para su uso como métodos rápidos en la detección de patógenos en alimentos 9,11.
Considerando lo referido, se observa que a través de los años se han venido desarrollando técnicas dependiendo de las necesidades que se requieran en cuanto a investigación, por eso se muestra la evolución con respecto a estos métodos. (figura 1)
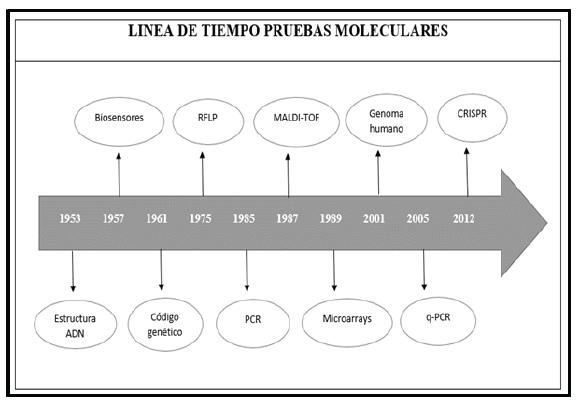
Fuente: elaboración propia.
Figura 1 Línea de tiempo de las pruebas moleculares más relevantes a través de la historia. 1953: se realiza el descubrimiento de la estructura de ADN. 1957: se utilizan por primera vez los biosensores. 1961: se descifra el código genético. 1975: se da inicio a ensayos con la técnica RFLP (Polimorfismos de fragmentos de restricción). 1985: Kary Mullis inventa la técnica de biología molecular PCR (Reacción en Cadena de la Polimerasa). 1987: Se desarrolla la técnica MALDI-TOF en Japón. 1989: se realizan los primeros ensayos con Microarrays. 2001: se descubre el 99 % del genoma humano. 2005 se avanza en variantes de la PCR como la qPCR (Reacción en Cadena de la Polimerasa en tiempo real). 2012: desarrollo de la técnica CRISPR.
TÉCNICAS PARA DETERMINAR PATÓGENOS EN ALIMENTOS
Como ya se ha mencionado, para la determinación de Salmonella se han estandarizado una gran variedad de métodos, como los inmunosensores, pruebas bioquímicas 12, estudio microbiológico de control de alimentos 13, reacciones de amplificación de ADN basadas en PCR 14, repeticiones palindrómicas cortas, agrupadas y regularmente interespaciadas por su acrónimo en inglés, CRISPR 15, microarreglos e inmunoensayos 16, cada uno con sus características y campos de acción determinados.
En la actualidad las técnicas moleculares superan las limitaciones de las técnicas tradicionales y serológicas para la detección correcta de patógenos. Las pruebas moleculares permiten una identificación más eficiente y de mayor fiabilidad, además de ser muy variadas y aplicables en casi cualquier diagnóstico asociado con agentes biológicos y en cualquier campo de acción 17. Al respecto, entre las técnicas moleculares que se han utilizado para genotipificar microorganismos está la PCR. Este método es más rápido, más simple y más económico que otros métodos de tipificación genómica mencionados anteriormente 18.
Los procedimientos de tipificación molecular como electroforesis en gel de campo pulsado (PFGE), rep-PCR (Repetitive Element Palindromic PCR) y tipificación de la secuencia multilocus (MLST) se han empleado de manera efectiva en investigaciones epidemiológicas y filogenéticas de los diferentes patógenos, especialmente Salmonella19.
Existen otras técnicas más recientes, como la espectrometría de masas MALDI-TOF, que se posiciona como una metodología relevante para la identificación de microorganismos; esta se fundamenta en el análisis de proteínas, a través de la creación de un espectro de masas específico de género y especie 20.
Otro método de diagnóstico que se utiliza con frecuencia y que permite obtener resultados rápidos de detección de Salmonella y demás patógenos es la técnica inmunocromatográfica. Se trata de una prueba cualitativa en la cual la muestra reacciona con los conjugados coloreados (anticuerpos anti-Salmonella)12.
Los inmunosensores electroquímicos son biosensores de ligando de afinidad basados en dispositivos de estado sólido en los cuales ocurren reacciones inmunoquímicas en la superficie del transductor para generar una señal electroquímica. El concepto de la metodología del inmunosensor es similar a la prueba de ELISA convencional. La tecnología permite la determinación altamente sensible del inmunocomplejo (anticuerpo-antígeno) utilizando diferentes métodos 21,22.
El inmunodiagnóstico se aplica para la rápida detección de patógenos que no pueden identificarse fácilmente por otros métodos convencionales. El desarrollo del ensayo se considera como un hito importante en el avance del diagnóstico serológico de enfermedades causadas por virus 23. Sin embargo, cambios importantes en el ADN y las tecnologías de secuenciación están enfrentando un desafío en la detección e identificación de subtipos de Salmonella24.
Las secuencias CRISPR han sido reportadas como posibles secuencias diana para el desarrollo de ensayos PCR específicos encaminados a la subtipificación de aislados de Salmonella. A pesar de los resultados no concluyentes reportados en diversos estudios, otros han hallado que estas consecuencias pueden ser prometedoras para la identificación de aislados de Salmonella. Estas regiones CRISPR están compuestas por una serie de repeticiones directas con secuencias de 21 a 37 nucleótidos, separadas por secuencias espaciadoras únicas de 20 a 40 nucleótidos específicamente 25.
La mayoría de las técnicas aplicadas para la determinación de patógenos se caracterizan por ser sencillas, rápidas, sensibles y versátiles 26,27, pero no todas cumplen estos requisitos, algunas son de alto costo y de un manejo complejo, como por ejemplo, la espectrometría de masas MALDI-TOF 28.
En virtud de las técnicas moleculares, se ha podido identificar que la contaminación de los alimentos, del medio ambiente (agua, roedores, insectos, aves) y de los productos de origen animal tienen un desempeño clave en el ciclo infeccioso de Salmonella por medio de los biomarcadores 29.
MARCADORES MOLECULARES DEL GÉNERO SALMONELLA
La identificación del serotipo y posterior clasificación de los aislados de Salmonella se han basado históricamente en estudios serológicos, reacciones de anticuerpos específicos con características del antígeno del aislado particular 5.
Considerando lo mencionado previamente, para determinar los diferentes biomarcadores que ayudan en la detección de los serotipos del género Salmonella, existen diferentes técnicas, las cuales se pueden clasificar en las siguientes categorías: métodos basados en ácidos nucleicos como la PCR, métodos inmunológicos (ELISA) y métodos basados en biosensores, dando así un amplio campo para detectar y convertirse en una gran ayuda diagnóstica 30,31.
La posibilidad de confirmar los resultados obtenidos aplicando un método molecular mediante el cultivo no es adecuado, y esto podría deberse a reacciones inespecíficas que ocurren en los diferentes cultivos o caldos de enriquecimiento utilizados, ya que se puede presentar una reacción cruzada del ADN investigado con similares microorganismos taxonómicos 32.
Para llegar a la determinación efectiva de los biomarcadores del género Salmonella se han empleado diferentes métodos moleculares como principal herramienta diagnóstica 33, hasta llegar a un punto muy importante, los linajes de serogrupo / serovar, que se basan en la fórmula de Kauffmann-White (figura 2), cada categoría (en recuadro) es detectada por un marcador genético 34.
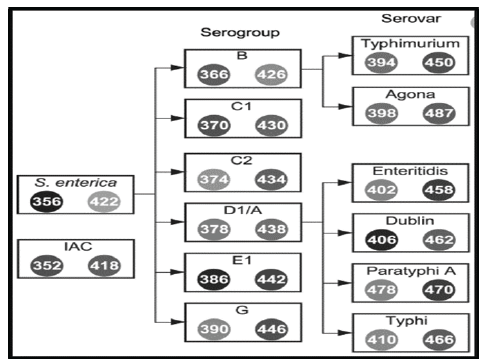
Fuente: Figura 2: Richmond G, Khine H, Zhou T, Ryan D, Brand D, McBride M, Killeen K. Mass-Code Liquid Arrays as a Tool for Multiplexed High-Throughput Genetic Profiling. PLOS ONE. 2011; 4 (6): 1-14.
Figura 2: Linajes de serogrupo / serovar basados en fórmulas de Kauffmann-White; esta clasificación se basa en un sistema que clasifica el género Salmonella en serotipos, basados en antígenos de superficie 51.
La clasificación por serotipos es el primer paso esencial en la caracterización de los aislamientos de Salmonella y es importante para la vigilancia, seguimiento de la fuente y detección de brotes en pro de frenar las ETA 35. Para mejorar la detección y reducir la carga de salmonelosis, se han desarrollado métodos moleculares de serotipos de Salmonella de alto rendimiento y a bajo costo 36.
Los serotipos S. Enteritidis y S. Paratyphi son los más prevalentes en los aislamientos de alimentos, los cuales también se identifican correctamente a través de PCR múltiple diseñados para la amplificación de genes, que permiten la identificación de serogrupo y serotipo, respectivamente 37. En los productos de PCR convencional la manera como se detecta si ocurrió la amplificación es por medio de electroforesis en gel de agarosa además, al incluir marcadores de peso molecular es posible determinar el peso del segmento amplificado, y así corroborar si realmente corresponde al tamaño del gen que se quiere identificar 38.
Algunos biomarcadores del género Salmonella permiten la detección directa de los genes que codifican para los antígenos somáticos O y flagelares H de Salmonella y posibilitan de manera rápida la determinación de los posibles serovares implicados en las enfermedades transmitidas por alimentos 39.
DISCUSIÓN
La salmonelosis es una enfermedad infecciosa comúnmente encontrada en personas alrededor del mundo 40. El género Salmonella es considerado uno de los causantes más frecuentes de diarrea aguda en las personas a través de los alimentos y la incidencia parece estar aumentando en varios países a nivel mundial; además, Salmonella Typhimurium es el serotipo más común identificado y considerado como uno de los más virulentos que afectan a las personas de todas las edades 41.
La detección de Salmonella en los alimentos es esencial desde la perspectiva clínica, epidemiológica y de control de infecciones 42; también la PCR es considerado un método rápido para la detección e identificación de patógenos como Salmonella43. Estos métodos moleculares, como la PCR en tiempo real, son altamente sensibles, muy específicos, rápidos y reproducibles en todos los campos de acción que se desempeñen 44,45.
La virulencia de los diferentes serovares existentes de Salmonella probablemente no sea el resultado de un solo cambio genético, sino, más bien, de una serie de alteraciones en una gran cantidad de genes asociados a la virulencia 46.
Para llegar a este punto se pasó por varios métodos tanto moleculares como convencionales. El método de cultivo convencional es conocido por ser el estándar para la detección de bacterias en alimentos, pero generalmente lleva más tiempo y podría ser menos sensible en comparación con las técnicas de base molecular. Por lo tanto, el uso del método de PCR específico es un procedimiento útil para la identificación rápida de Salmonella en alimentos, ya sea en rutina pruebas o esfuerzos de investigación 47.
El ensayo de PCR y demás técnicas moleculares constituyen una herramienta que agiliza y fortalece el diagnóstico de Salmonella en diferentes alimentos, lo cual permite liberar los productos alimenticios en un menor tiempo 48. La variación en los datos observados de los diferentes estudios realizados para la detección de Salmonella en alimentos está relacionada con la implementación de normas estrictas de bioseguridad en diferentes lugares de producción y fabricación de productos alimenticios y un adecuado manejo de estos productos durante los últimos años 49.
La mayoría de técnicas tienen limitaciones son relativamente complicadas; necesitan experticia y utilizan productos químicos peligrosos, por lo que el análisis rutinario de muchas muestras resulta poco práctico.
Los biomarcadores se consideran propiedades cuantificadas que pueden establecer una patología o, en este caso, una característica del microorganismo, siendo estos una herramienta diagnóstica, ya que pueden ser específicos de células, enzimas, genes y hormonas y por medio de las técnicas moleculares se llega al punto de reconocer estos biomarcadores. Para Salmonella, un ejemplo de biomarcadores son los antígenos O somático y H flagelar, los cuales tienen funciones diferentes y se encuentran en la capa lipídica y los flagelos de la bacteria; además de poder clasificar los serotipos y serovares por medio de ellos, se encuentra una característica única y es la medición de la virulencia de este patógeno en los humanos 39.
Las infecciones bacterianas del género Salmonella son responsables de una gran cantidad de enfermedades alrededor del mundo. Estas enfermedades causan pérdidas económicas significativas a los empresarios y demás productores de alimentos. Además, en los diferentes alimentos, especialmente productos cárnicos y avícolas, son los principales reservorios de Salmonella que pueden transmitirse a los humanos a través de la cadena alimentaria 50.
CONCLUSIONES
Las diferentes técnicas de diagnóstico molecular representan en la actualidad una alternativa prometedora en el campo de la investigación y en el control de calidad de los alimentos, debido a su rapidez, alta sensibilidad y eficiencia para la detección temprana de microorganismos patógenos. Por esto mismo, el número de métodos moleculares con gran utilidad en el área de microbiología de alimentos se ha ido aumentando y diversificando, cada uno con sus respectivas fortalezas y debilidades, las cuales deben tenerse en cuenta en el momento de cumplir con los objetivos planteados en los diferentes campos de acción.
Los biomarcadores moleculares se consideran una herramienta de referencia para la identificación de serovares de Salmonella y su serotipificación, a través de técnicas moleculares, con un alto nivel de especificidad y sensibilidad en los resultados, un tiempo menor en los procesos, rendimiento en el costo-beneficio, además de obtener datos específicos los cuales son de importancia epidemiológica.
La PCR destaca entre todas las técnicas como el método de diagnóstico molecular más aplicado en el área de alimentos y, recientemente, variaciones de este, como la PCR en tiempo real, han sumado ventajas adicionales a esta técnica, entre las que se destaca una mayor velocidad en la obtención de resultados, especialmente en el aislamiento de Salmonella y la determinación de sus biomarcadores.
La detección de microorganismos patógenos en muestras de alimentos, o en materias primas utilizadas para la fabricación de los mismos, es uno de los principales retos en la industria alimenticia actual; por ello mismo es de gran ayuda y de apoyo las técnicas moleculares, para así obtener un gran control de calidad, evitando la contaminación de patógenos como Salmonella.















