Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO  Similars in Google
Similars in Google
Share
Revista colombiana de Gastroenterología
Print version ISSN 0120-9957On-line version ISSN 2500-7440
Rev Col Gastroenterol vol.21 no.2 Bogotá June 2006
Polimorfismo 399C>T en el exón 3 del gen MSH2 en individuos con cáncer colorrectal
Polimorphism 399 C>T in exon 3 of the MSH2 gene in individuals with colorectal cancer.
Jenny Montenegro Medina,1 Luis Fernando Isaza Jiménez ,2 Gabriel Bedoya Berrío ,3 José Luis Ramírez Castro,1 Juan Ricardo Márquez Velásquez ,2 Carlos Mario Muñetón Peña .1
1. Unidad de Genética Médica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia
2. Departamento de Cirugía, Universidad de Antioquia, HUSVP, Medellín, Colombia.
3. Grupo Genética Molecular, Universidad de Antioquia, Medellín, Colombia
Fecha Recibido: 28-08-05 / Fecha Aceptado: 17-05-06
Resumen
Introducción
El cáncer colorrectal (CCR) es una de las entidades malignas más frecuentes en nuestro medio y ocupa el cuarto lugar después del cáncer de cérvix, seno y estómago; esta enfermedad continúa siendo diagnosticada en la mayoría de los casos en estadios avanzados. En Antioquia, la incidencia en menores de 40 años es de 20,9%, siendo esta tasa la más alta y el doble de lo informado a escala mundial para este grupo de edad.
Propósito
Caracterización molecular del polimorfismo 399 C>T en el exón 3 del gen MSH2 en pacientes con cáncer colorrectal hereditario (HNPCC) y esporádico.
Pacientes y métodosSe analizaron seis pacientes con cáncer colorrectal hereditario no polipoideo (CCHNP), seis con cáncer colorrectal esporádico y seis individuos sanos. La extracción del DNA se realizó a partir de sangre periférica. Las muestras se amplificaron por PCR; el polimorfismo se detectó por secuenciamiento directo.
Resultados
En todos los individuos estudiados con cáncer colorrectal hereditario y esporádico, así como en los individuos sanos se detectó el polimorfismo 399 C>T en el exón 3 del gen MSH2.
Conclusiones
El cambio molecular detectado no varía el aminoácido que se codifica, por lo que se considera como una mutación neutra no identificada previamente en otras poblaciones. Varios polimorfismos se han asociado con la susceptibilidad para desarrollar cáncer colorrectal. Por lo tanto, futuros estudios son necesarios para determinar si este polimorfismo está relacionado con el desarrollo del cáncer colorrectal en la población colombiana.
Palabras clave: Cáncer colorrectal, gen MSH2, síndrome de Lynch (HNPCC), polimorfismo, genes de reparación de bases mal apareadas.
Abstract
Introduction
Colorrectal cancer (CCR) is one of the most common neoplasias in our country, occupying the fourth place after cervix, breast and stomach cancers; being diagnosed most of the cases in advanced stages. In Antioquia the incidence of young people (less of 40 years) affected by this disease is 20,9%, being this rate the highest and doubling the one reported world-wide for this group of age.
Aims
Molecular characterization of a novel polymorphism 399 C >T in MSH2 exon 3 in sporadic and hereditary colorectal cancer patients.
Patients and methods
We have analyzed 6 patients with hereditary non polypoid colorectal cancer (HNPCC), 6 sporadic colorectal cancer patients and 6 healthy individuals. Genomic DNA was taken from each patients peripheral blood lymphocytes. All samples were amplified by PCR and its products were directly sequenced in a genetic analyzer.
Results
In all colorectal cancer and healthy individuals evaluated a novel polymorphism 399C>T in the MSH2 gene was detected.
Conclusions
The identified molecular variant does not change the protein sequence. Therefore, this variant could be considered a neutral mutation, still not identified in other populations. The relationship between 399C>T polymorphism and colorectal cancer is unknown. Further analysis is required in order to determine the role of this polymorphism in Colombian CRC families.
Key Words: Colorectal cancer, MSH2 gene; Lynch Syndrome (HNPCC), Polimorphism; Mismatch repair genes.
INTRODUCCION
El cáncer colorrectal (CCR) es una de las neoplasias más comunes en la población mundial. Así mismo, presenta una alta tasa de mortalidad en los países desarrollados (1). En Colombia, la incidencia del CCR ocupa el cuarto lugar tanto en hombres como en mujeres. En 2002 se registraron 4.295 nuevos casos de CCR, con una cifra mayor en mujeres (2).
La etiología del CCR es multifactorial, en cerca del 80% de los pacientes es de tipo esporádico, mientras que el 20% restante presenta una historia familiar, lo cual sugiere un componente genético (3). Entre los tipos de CCR hereditario se encuentran el cáncer colorrectal hereditario sin poliposis ((CCHNP) o síndrome de Lynch y la poliposis adenomatosa colónica familiar (FAP) (4).
El CCR fue el primer tipo de cáncer en el cual se estableció un modelo de tumorogénesis que comprende la secuencia de una serie de eventos que explica el desarrollo de esta neoplasia (5). Lo anterior lleva a la acumulación de mutaciones en diversos genes, que conducen a la inactivación de genes supresores de tumores (GST): APC, P53 y DCC, genes de reparación MLH1 y MSH2 o a la amplificación de oncogenes: k-Ras, COX-2 (4, 6). Las mutaciones descritas se presentan durante el proceso de transformación desde el epitelio del colon normal hasta el adenocarcinoma. Específicamente en el CCR se requieren entre 5 y 7 alteraciones genéticas diferentes (4, 5). Existen dos vías genéticas independientes que conducen a la ocurrencia del CCR, dependiendo de cual GST inicialmente se inactiva. La inactivación del gen APC se presenta en cerca del 85% de los CCR esporádicos; y la otra es la inactivación de genes del sistema de reparación de bases mal apareadas o "mismatch" (MMR) que representa el resto de todos los CCR (3, 4, 7).
Particularmente el (CCHNP) se presenta entre el 5 al 6% de todos los CCR; exhibe un patrón de herencia autosómico dominante, comúnmente afecta individuos menores de 45 años, se asocia con otros tipos de neoplasias extracolónicas (endometrio, ovario, gástrico) (4, 8). Genéticamente el HNPCC se origina por mutaciones en células germinales de uno de los ocho genes MMR (MLH1, MSH2, MLH3, MSH3, MSH6, MSH3, PMS1 o PMS2). En los genes MLH1 y MLH2 ocurren cerca del 60% de las mutaciones informadas (9-13). Las proteínas codificadas por estos genes reconocen y reparan errores en la cadena de DNA durante la replicación. La pérdida de la función de los genes MMR permite la acumulación de errores en la replicación y por lo tanto, conduce hacia la inestabilidad microsatelital (MSI), la cual acelera el proceso de carcinogénesis (14, 15).
Por otra parte, recientes estudios informan la identificación de nuevos polimorfismos en genes relacionados con el CCR (16, 17). Sin embargo, no está bien definido el papel de estos polimorfismos con el desarrollo del CCR.
Por todo lo expuesto anteriormente, la caracterización molecular de alteraciones en los genes relacionados con el CCR, tiene gran utilidad para el diagnóstico y pronóstico de los pacientes.
En un estudio multicéntrico previo nuestro grupo identificó el polimorfismo C399T en el exón 3 del gen MSH2 en pacientes con CCHNP (datos no publicados). Con el anterior hallazgo, el propósito de este estudio fue el de caracterizar molecularmente dicho polimorfismo en una muestra de la población colombiana conformada por individuos con cáncer colorrectal esporádico (CCRE) y por individuos sanos.
PACIENTES Y METODOSSe incluyeron seis pacientes con (CCHNP), seis pacientes con CCRE y seis individuos sanos sin antecedentes familiares de cáncer, para un total de 18 casos procedentes de diferentes regiones del país. Este trabajo fue evaluado y aprobado por el comité de Bioética de la Corporación Académica para las Enfermedades Tropicales (CAET), de la Universidad de Antioquia. Previo consentimiento informado se extrajeron 10 cc de sangre periférica de cada paciente. Se obtuvo información adicional de las historias clínicas y exámenes de anatomopatológicos. En todos los casos de (CCHNP) se elaboraron las correspondientes genealogías.
Extracción de ADN: Se realizó a partir de linfocitos de sangre periférica con el kit comercial Wizard de Promega y Chelex de Sigma siguiendo las instrucciones del fabricante.
Amplificación del DNA: La amplificación se realizó mediante PCR, utilizando cebadores marcados con fluorescencia. Las condiciones de las reacciones se realizaron según los protocolos previamente descritos (18).
Secuenciamiento: Los amplicones se purificaron con el kit Wizard DNA Purification (Promega), siguiendo instrucciones del proveedor. Se secuenciaron las dos cadenas de ADN, utilizando el método de Sanger por terminación de cadena acoplado a PCR. Las reacciones de secuencia se resolvieron por medio de una electroforesis capilar en un analizador genético ABI 310 (Applied Biosystems, CA), mediante el programa Navegator. Las secuencias se editaron empleando los programas Chromas y Vised. Finalmente se realizó una comparación por alineamiento con las secuencias informadas por GenBank para cada exón.
RESULTADOS
Del total de los doce individuos con CCR estudiados (6 CCRE y 6 CCHNP), la mayoría (75%) cumplía con los criterios de Bethesda y uno solo con los de Ámsterdam.
El promedio de edad al momento del diagnóstico fue de 37,2 años, con un rango entre 20-70 años (tabla 1). Nueve de los doce individuos eran menores de 50 años. Los criterios de Ámsterdam y Bethesda tienen en cuenta esta edad para la identificación de individuos con CCHNP, que generalmente se presenta antes de los 45 años.
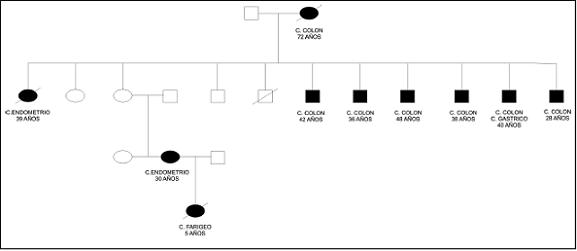
El análisis realizado a las genealogías de los seis individuos con CCHNP revela que estas familias presentan un patrón de herencia autosómico dominante, con penetrancia incompleta; estos resultados concuerdan con lo establecido para individuos con CCHNP (figura 1). Adicionalmente, en estas mismas familias se encontraron otros tipos de cánceres, tales como el de endometrio, ovario, hígado, esófago, gástrico, mama y garganta. El cáncer de endometrio fue el más frecuente en todas las familias analizadas, similar a lo informado en familias con sospecha de CCHNP.
En cuanto a la variedad histológica se encontró que el adenocarcinoma bien diferenciado fue el más común (10 casos) (tabla 1); un caso con adenocarcinoma mal diferenciado y otro con células en anillo. La mayoría de estos casos cumplían con los criterios de Bethesda.
Se encontró que en la región ascendente del colon se localizó la mayor parte de los casos de CCR (7/12), seguido del colon transverso (3 casos); un solo caso se localizó en el recto (tabla 1). En los individuos estudiados los diferentes estadios Duke´s mostraron una distribución igual de cuatro casos para Duke´s B, Duke´s C y Duke´s D (tabla 1). Estos hallazgos son similares a los informados en otros estudios (16).
Previamente nuestro grupo identifico el polimorfismo 399 C >T en el exón 3 del codón 133 (GAT>GAC—Asp) en el gen MSH2 en una muestra de pacientes con CCHNP. Posteriormente decidimos secuenciar la misma región en otros pacientes con CCRE e individuos sanos de la misma población, con el fin de descartar un posible desequilibrio de ligamiento en otra región del gen que pudiese estar influyendo en la expresión de la enfermedad; se anota que este polimorfismo hasta el presente no se había informado en otras poblaciones.
En nuestro estudio se detectó el polimorfismo 399 C>T en el exón 3 del codón 133 (GAT>GAC) del gen MSH2 en todos los 18 individuos analizados (seis pacientes con CCHNP, seis pacientes con CCRE y en seis individuos sanos) (figura 2). En conclusión, este polimorfismo fue identificado tanto en pacientes con CCR de tipo hereditario (HNPCC) como de tipo esporádico.
La transición C por T en el exón 3 del codón 133 (GAT>GAC) codifica para el ácido aspártico, este cambio molecular no varía el aminoácido codificado, en consecuencia constituye una mutación neutra o silenciosa. Por lo tanto, es de gran importancia realizar otros estudios con el fin de determinar el papel de este polimorfismo en el desarrollo del CCR en familias colombianas. Es decir, si el polimorfismo está asociado con la susceptibilidad para el desarrollo del cáncer de colon.
Tabla 1. Características clínicas e histopatológicas de los doce individuos con cáncer colorrectal esporádico y hereditario estudiados.
| Caso | Edad | Sexo | Diagnóstico histopatológico | Localización | Criterio | Duke´s | Anteced. familiares |
| 1 | 40 | M | Adenocarcinoma bien diferenciado | Colon transverso | Amsterdam | C | Si |
| 2 | 30 | M | Adenocarcinoma bien diferenciado | Recto | Bethesda | D | Si |
| 3 | 57 | M | Adenocarcinoma bien diferenciado | Colon derecho | No | B | Si |
| 4 | 24 | F | Adenocarcinoma bien diferenciado | Colon derecho | No | B | Si |
| 5 | 55 | M | Adenocarcinoma bien diferenciado | Colon derecho | Bethesda | C | Si |
| 6 | 28 | F | Adenocarcinoma bien diferenciado | Colon derecho | Bethesda | B | Si |
| 7 | 70 | F | Adenocarcinoma bien diferenciado | Colon derecho | Bethesda | D | No |
| 8 | 38 | F | Adenocarcinoma bien diferenciado | Colon derecho | Bethesda | D | No |
| 9 | 32 | F | Adenocarcinoma bien diferenciado | Colon transverso | Bethesda | C | No |
| 10 | 20 | M | Adenocarcinoma mal diferenciado | Colon derecho | Bethesda | D | No |
| 11 | 33 | M | Células en anillo | Colon izquierdo | Bethesda | C | No |
| 12 | 20 | F | Adenocarcinoma bien diferenciado | Colon transverso | Bethesda | B | No |
Figura 1. Genealogía perteneciente a un individuo con HNPCC. Se observa un patrón de herencia autosómico dominante, con penetrancia incompleta. En esta familia el cáncer de endometrio fue común en las mujeres.
DISCUSION
En este estudio se realizó una caracterización molecular del polimorfismo C399T en el exón 3 del codón 133 (GAT>GAC→Asp) del gen MSH2 en pacientes con CCRE, (CCHNP) e individuos sanos. En Colombia existen muy pocos estudios sobre la genética molecular del CCR de tipo hereditario (CCHNP) y esporádico.
La mayoría de individuos con CCR estudiados estaban en los estadios Duke´s C y D. Los hallazgos histopatológicos son de gran ayuda para el diagnóstico y pronóstico de los pacientes con CCR, puesto que se ha observado que individuos en estadio Duke´s A presentan una tasa de sobrevida a 5 años superior del 90%, mientras que en individuos con estadio Duke´s D la tasa de sobrevida cae al 5% (18). En este estudio, ocho de los doce pacientes, al momento del diagnóstico presentaban estadios Duke´s C y D. Es decir, cursaban con un estado avanzado del CCR y por lo tanto tienen un mal pronóstico. Por tal razón, los métodos de diagnóstico temprano en el cáncer son de gran importancia y tienen como fin disminuir la tasa de mortalidad por cáncer.
La relación entre el polimorfismo 399 C>T y el desarrollo del CCR en los individuos analizados aún no está determinada, por tal razón se requieren otros estudios para esclarecer esta posible relación, o si esta variante identificada hace parte de la estructura genética de la población colombiana, sin relación alguna con la patogénesis del CCR.
En los últimos años se ha identificado un alto número de polimorfismos en diversos genes implicados en el desarrollo del CCR (16, 19, 20). En la literatura se plantea una controversia en cuanto si realmente existe una diferencia funcionalmente significativa entre polimorfismo y mutación. Estudios recientes en individuos con CCHNP muestran que ciertos polimorfismos disminuyen las interacciones proteína-proteína entre las proteínas reparadoras codificadas por los genes MMR, como en el caso del complejo proteico PMS2-MLH1 (17, 21), como consecuencia se afecta la eficiencia del sistema de reparación. Estos hallazgos sugieren que los polimorfismos podrían jugar un papel importante en el desarrollo del CCHNP y resaltan la importancia de los estudios de función de proteínas reparadoras para el entendimiento del significado funcional de los polimorfismos en el CCR.
Generalmente, los polimorfismos se consideran alteraciones genéticas que ocurren con una frecuencia > 1% en la población general, que contribuyen en cerca del 90% de la variación del genoma humano y que no afectan el fenotipo (22). Varios trabajos informan que los polimorfismos podrían estar involucrados en la susceptibilidad para desarrollar determinadas enfermedades humanas, específicamente en el CCR se han informado polimorfismos en diversos genes que se asocian con un alto riesgo de desarrollar CCR (ej: p53, PIN3; MTHFR C667T; COX2) (16, 17, 19, 23). Estos hallazgos apoyan la hipótesis de la asociación entre polimorfismos y cáncer.
En resumen, nuestro trabajo conduce a plantear estudios adicionales que incluyan metodologías de tamizaje molecular más específicas con el fin de determinar la presencia de polimorfismos o alteraciones genéticas, como también, establecer la relación del polimorfismo identificado con el CCR en nuestra población. De igual manera, emplear técnicas complementarias de inmunohistoquímica y genómicas para detectar otros tipos de alteraciones en individuos con CCR, dada la gran heterogeneidad genética que presenta este tipo de cáncer.
Finalmente, este tipo de estudios es importante para establecer un programa de diagnóstico molecular para el CCR, que incluya un adecuado asesoramiento genético e implementar medidas de prevención en familias con alto riego de desarrollar CCR; como es el caso de la colonoscopia para el seguimiento de estos pacientes.
AGRADECIMIENTOS
Agradecemos la participación de los pacientes en este trabajo. A los cirujanos, enfermeras y jefes de los departamentos de cirugía y patología de las instituciones hospitalarias que participaron por su valiosa colaboración (Hospital San Vicente de Paúl, Medellín; Hospital Hernando Moncaleano Perdomo, Neiva; Hospital Militar Central, Bogotá). Este trabajo fue financiado por la Universidad de Antioquia, proyectos CODI-CPT-0018 y 0211.
REFERENCIAS
1. Jemal A, Thomas A, Murray T, Thun M. Cancer statistics 2002. CA Cancer J Clin 2002; 52: 181-2. [ Links ]
2. GLOBOCAN 2002. http://www-depdb.iarc.fr/globocan/GLOBOframe.htm. [ Links ]
3. De la Chapelle A. Genetic predisposition to colorectal cancer. Nature 2004; 4: 769-780. [ Links ]
4. Lynch T, De la Chapelle A. Hereditary colorectal cancer. N Engl J Med 2003; 348(10): 919-32. [ Links ]
5. Fearon ER, Vogelstein B. A genetic model for colorectal tumorigenesis. Cell 1990; 61: 759-767. [ Links ]
6. Montenegro Y, Ramírez A, Muñetón C. Cáncer colorrectal heredado. Rev Colomb Cir 2002; 17(1): 30-6. [ Links ]
7. Tomlinson I. Different pathways of colorectal carcinogenesis and their clinical pictures. Annals N.Y Acad Science 2000; 9(10):10-20. [ Links ]
8. Potter JD. Colorectal cancer: molecules and populations. J. Natl Cancer Inst 1999; 91: 916-932. [ Links ]
9. PeltomakI P, Vasen HF. Mutations predisposing to hereditary nonpolyposis colorectal cancer: database and results of a collaborative study. The International Collaborative Group on Hereditary Nonpolyposis Colorectal Cancer. Gastroenterology 1997; 113: 1146-58. [ Links ]
10. PeltomakI P. Deficient DNA mismatch repair: a common etiologic factor for colon cancer. Hum Mol Genet 2001; 10(7): 735-40. [ Links ]
11. Abdel-Rahman WM, Ollikainen M, Kariola R, Jarvinen HJ, Mecklin JP, Nystrom- Lahti M, Knuutila S, Peltomaki P. Comprehensive characterization of HNPCC-related colorectal cancers reveals striking molecular features in families with no germ line mismatch repair gene mutations. Oncogene 2005; 24 (9): 1542-51. [ Links ]
12. Wagner A, Barrows A, Wijnen JT, van der Klift H, Franken PF, Verkuijlen P, Nakagawa H, Geugien M, Jaghmohan-Changur S, et al. Molecular analysis of hereditary nonpolyposis colorectal cancer in the United States: high mutation detection rate among clinically selected families and characterization of an American founder genomic deletion of the MSH2 gene. Am J Hum Genet 2003; 72 (5): 1088-100. [ Links ]
13. Casey G, Lindor NM, Papadopoulos N, Thibodeau SN, Moskow J, Steelman S, Buzin CH, Sommer SS, Collins CE, et al. Colon Cancer Family Registry. Conversion analysis for mutation detection in MLH1 and MSH2 in patients with colorectal cancer. JAMA 2005; 293: 799-809. [ Links ]
14. Lengauer C, Kinzler KW, Volgelstein B. Genetic instability in colorectal Cancers. Nature 1997; 386: 623-627. [ Links ]
15. Raut CP, Pawlik TM, Rodriguez-Bigas MA. Clinicopathologic features in colorectal cancer patients with microsatellite instability. Mutat Res 2004; 568(2): 275-82. [ Links ]
16. Houlston RS, Tomlinson IP. Polymorphisms and colorectal tumor risk. Gastroenterology 2001; 121(2): 282-301. [ Links ]
17. Yuan ZQ, Gottlieb B, Beitel L K, Wong N, Gordon P H, Wang Q, Puisieux A, Foulkes W D, Trifiro M. Polymorphisms and HNPCC: PMS2-MLH1 protein interactions diminished by single nucleotide polymorphisms. Hum Mutat 2002; 19: 108-113. [ Links ]
18. Wijnen J, Vasen H, Meera K P, Menko FH, Van der Klift H, Van Leeuwe NC, Van Den Broek, et al. Seven new mutations in MSH2 an HNPCC gene, identified by denaturing gradient gel electrophoresis. Am J Hum Genet 1995; 56: 1060-66. [ Links ]
19. De Jong MM, Nolte IM, te Meerman GJ, van der Graaf WT, de Vries EG, Sijmons RH, Hofstra RM, Kleibeuker JH. Low-penetrance genes and their involvement in colorectal cancer susceptibility. Cancer Epidemiol Biomarkers Prev 2002; 11(11): 1332-52. [ Links ]
20. Isidro G, Matos S, Goncalves V, Cavaleiro C, Antunes O, Marinho C, Soares J, and Boavida M. G. Novel MLH1 mutations and novel MSH2 polymorphism identified by SSCP and DHPLC in portuguese HNPCC families. Inst Nal de Saúde, Aug 2003 1-5. Hum Mutat 2003; 22(5): 419-20. [ Links ]
21. Plevova P, Sedlakova E, Zapletalova J, Krepelova A, Skypalova P, Kolar Z. Expression of the hMLH1 and hMSH2 proteins in normal tissues: relationship to cancer predisposition in hereditary non-polyposis colon cancer.Virchows Arch 2005; 446: 112-9. [ Links ]
22. Collins FS, Brooks LD, Chakravarti A. A DNA polymorphism discovery resource for research on human genetic variation. Genome Res 1998; 8(12): 1229-31. [ Links ]
23. Gemignani F, Moreno V, Landi S, Moullan N, Chabrier A, Gutierrez-Enriquez S, Hall J, Guino E, Peinado MA, Capella G, Canzian F. A TP53 polymorphism is associated with increased risk of colorectal cancer and with reduced levels of TP53 mRNA. Oncogene 2004; 23(10): 1954-6. [ Links ]