Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista colombiana de Gastroenterología
versión impresa ISSN 0120-9957versión On-line ISSN 2500-7440
Rev Col Gastroenterol v.23 n.4 Bogotá oct./dic. 2008
Análisis de las aneuploidías del cromosoma 17 y deleción del gen TP53 en tumores gastrointestinales por FISH-bicolor
Analyses of numerical aberrations of chromosome 17 And TP53 gene deletion in gastrointestinal tumors by dual-color fluorescence in situ hybridization (FISH)
Gloria Cecilia Ramírez Gaviria (1), Juan Carlos Herrera Patiño (1), Carlos Mario Muñetón Peña (1), Juan Ricardo Márquez Velásquez (2) , Luís Fernando Isaza Jiménez (3),
(1) Unidad de Genética Médica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
(2) Hospital Pablo Tobón Uribe. Medellín, Colombia.
(3) Departamento de Cirugía, Universidad de Antioquia, HUSVP, Medellín, Colombia.
Resumen
Introducción. Las alteraciones cromosómicas numéricas y estructurales son comunes en las neoplasias gastrointestinales; estas alteraciones se originan por la inestabilidad cromosómica que ocurre durante el desarrollo del cáncer, afectando la expresión de diversos genes como protooncógenes, genes supresores de tumores y genes de reparación.
Objetivo. Evaluar las aneuploidías del cromosoma 17 y deleción del gen TP53 en tumores gastrointestinales primarios, mediante la técnica del FISH bicolor.
Muestras y métodos. Se analizaron 15 muestras de tumores gastrointestinales primarios, se disociaron mecánica y enzimáticamente con colagenasa para obtener núcleos interfásicos. El FISH bicolor se realizó con sondas marcadas con fluorocromos para el centrómero del cromosoma 17 y para el locus 17p13.1 del gen TP53. Se analizaron 100 núcleos por cada muestra.
Resultados. Se encontró que el 33,3% (5/15) de las muestras tenía aneuploidías para el cromosoma 17; la monosomía fue detectada en todos los casos (5/5). La mayoría de las muestras tenía subpoblaciones de núcleos heterogéneos (mosómicos, disómicos, trisómicos y tetrasómicos). El 93,3% (14/15) de los tumores tenían deleción del gen TP53. El estudio histopatológico mostró que 14 de las 15 muestras presentaban un estado avanzado del cáncer.
Conclusiones. Se demostró un imbalance entre las señales del centrómero del cromosoma 17 y del gen TP53 por núcleo mediante el FISH bicolor. Aneuploidías del cromosoma 17 y deleciones en el locus 17p13.1 del gen TP53 son alteraciones muy frecuentes en tumores gastrointestinales. El FISH bicolor permite evidenciar la heterogeneidad genética intratumoral que se presenta en el desarrollo del cáncer.
Palabras clave
Cáncer gastrointestinal, Gen TP53, FISH-bicolor, aneuploidía, deleción, heterogeneidad genética.
Summary
Introduction. Numerical and structural chromosome aberrations are common in gastrointestinal cancers; these are originated by chromosomal Instability that happens during development of cancer. Thus, the expression of many genes is affected, such as protooncogene, tumor suppressor genes and repair genes.
Aims. To evaluate aneuploidy of chromosome 17 and TP53 gene deletions in primary gastrointestinal tumors by dual- color fluorescence in situ hybridization (FISH).
Samples and methods. 15 primary gastrointestinal tumor samples were analyzed, which were mechanically and enzimatically disaggregated with 0.2% collagenase in order to obtain interphase nuclei. Dual-color FISH assays was performed using direct fluorescent labeling probes for the chromosome 17 centromere and TP53 gene (17p13.1). Hybridized signals were counted in 100 interphase nuclei by each case.
Results. Aneuploidy of chromosome 17 was found in 33.3% (5/15) of the samples. Monosomy was detected in 100% (5/5) of cases with aneuploidies. Most of tumor samples exhibited heterogeneous clones that were monosomic, disomic, trisomic and occasionally tetrasomic. The TP53 deletion was found in 93.3% (14/15) of the analyzed samples. The histopathological study showed that 14 out of 15 tumors samples displayed an advance stage of tumorigenesis.
Conclusions. We found an imbalance of signals for chromosome 17 and TP53 per nucleus. Aneuploidy of chromosome 17 and TP53 gene deletion are very frequent aberrations in gastrointestinal tumors. Dual-color FISH analysis allow detect intratumoral genetic heterogeneity that had occurred in development of cancer.
Key Words
Gastrointestinal cancer, TP53 gene, Dual-FISH, aneuploidy, deletion, genetic heterogeneity.
Fecha recibido: 03-10-07 Fecha aceptado: 02-10-08
Introducción
El cáncer gastrointestinal (CGI) es uno de los más frecuentes en la población mundial, especialmente el de estómago y colon, los cuales presentan altas tasas de mortalidad en diferentes países (1). Por tal razón, el CGI constituye uno de los principales problemas de salud pública en muchos países. En Colombia, el cáncer de estómago presenta una de las mayores tasas de incidencia y de mortalidad en ambos sexos (2).
En la etiología del CGI están involucrados factores ambientales, genéticos e infecciosos (específicamente asociados con la presencia de Helicobacter pylori), como también, la alimentación, el consumo de alcohol, el tabaquismo, el estrés, factores socioeconómicos, entre otros (2, 3). Por su parte, los factores genéticos juegan un papel importante en la aparición y desarrollo del CGI, debido a la acumulación de diversos tipos de alteraciones genéticas, algunas de éstas detectadas en lesiones precancerosas (3, 4). Diferentes estudios concluyen que el CGI se caracteriza porque presenta una gran heterogeneidad genética (5); sin embargo, los mecanismos moleculares del CGI no están bien elucidados.
Las alteraciones genéticas más comunes en el CGI son las mutaciones, amplificaciones génicas, pérdida de heterocigocidad (LOH), inactivación génica y alteraciones cromosómicas numéricas y estructurales, tales como las aneuploidías que afectan la estabilidad cromosómica, así como la expresión de genes importantes como son los protooncógenes, genes supresores de tumores, genes que controlan el ciclo celular y genes de reparación (4, 6, 7). Las alteraciones mencionadas inducen a su vez otros cambios genéticos, los cuales conducen finalmente a la célula hacia un estado de inestabilidad genética (8). El CGI se asocia con alteraciones en los genes APC, MCC, C-MYC, E-CADERINA, MET, K-RAS, RB, TP53, DCC, TGF-βR II, MLH1 y MSH2, localizados en diferentes cromosomas (4, 9, 10). A diferencia del cαncer de estómago, la genética molecular del cáncer de colon ha sido ampliamente estudiada y se ha establecido como un modelo de la secuencia de eventos genéticos que ocurren en la carcinogénesis de este tipo de cáncer (11).
El gen TP53 se considera como uno de los genes más importantes dentro del desarrollo del CGI. Es un gen supresor de tumores, localizado en el brazo corto del cromosoma 17 (17p13.1); codifica para una fosfoproteína nuclear de 53 KD, que actúa como un factor de transcripción (12) y juega un papel fundamental en el control del ciclo celular, la apoptosis, respuesta al daño y reparación del DNA (13). Por lo anterior, también se le denomina como gen guardián del genoma humano. El gen TP53 se encuentra alterado en cerca del 50% de todos los cánceres estudiados, incluido el CGI (14). Adicionalmente, este gen se propone como un marcador genético para el diagnóstico y pronóstico de diversos tipos de tumores sólidos (10, 15).
Con el desarrollo de la hibridización in situ con fluorescencia (FISH), una técnica de citogenética molecular, se revolucionó el análisis cromosómicos en la genética del cáncer, porque permite detectar simultáneamente cambios cromosómicos numéricos (aneuploidías) y estructurales (deleciones y translocaciones) en un locus específico de protooncógenes o genes supresores de tumores, localizados en un mismo cromosoma o en cromosomas diferentes (16). La principal ventaja del FISH es que se puede realizar en núcleos interfásicos sin tener que establecer cultivos celulares. Los cultivos de tumores sólidos presentan muchas dificultades especialmente con la proliferación celular in vitro, lo que afecta notoriamente el éxito de los estudios cromosómicos convencionales (17). Por lo tanto, el FISH permite realizar un análisis citogenético molecular rápido, con una alta especificidad y sensibilidad en tejidos tumorales.
El objetivo del presente trabajo fue estudiar las aneuploidías del cromosoma 17 y deleción del gen TP 53 en 15 tumores gastrointestinales primarios, mediante la técnica del FISH bicolor.
Materiales y métodos
Muestras
Se analizaron 15 muestras de tumores gastrointestinales primarios (5 de estómago, 2 de esófago, 6 de colon, 1 de duodeno y 1 de recto) provenientes de 6 mujeres y 9 hombres, con un rango de edad entre los 23 y 76 años. Previo consentimiento informado por escrito y luego de su aprobación, las muestras se obtuvieron mediante resección quirúrgica o biopsia del tumor primario de los pacientes procedentes de los servicios de cirugía y endoscopia del Hospital Universitario San Vicente de Paúl de Medellín. Se envió una porción de cada una de las muestras al departamento de Patología de la Universidad de Antioquia para el estudio histopatológico. Ninguno de los pacientes había recibido quimioterapia y/o radioterapia. Este estudio fue aprobado por el Comité de Bioética de la Universidad de Antioquia.
Procesamiento de las muestras
Las muestras analizadas se procesaron para obtener núcleos interfásicos. Las muestras de tejidos tumorales fueron recolectadas y transportadas en medio de cultivo RPMI-1640 Sigma; suplementado con antibióticos y antimicóticos; los tejidos se lavaron dos veces con PBS, luego se cortaron en pequeños fragmentos y se disociaron enzimáticamente con colagenasa Tipo I (Sigma) al 0,2% e incubadas a 37ºC entre 1 a 16 h. Posteriormente, se centrifugó a 800 rpm por 10 min; las células se resuspendieron en una solución hipotónica de KCL al 0,075M por 15 min a 37ºC y se fijaron en metanol/ácido acético frío (3:1, vol/vol). Finalmente para cada muestra se goteo la suspensión celular en 3 láminas de vidrio. Las placas con los extendidos de núcleos interfásicos se almacenaron en un congelador a - 20ºC, hasta el momento de realizar la técnica del FISH bicolor. Muestras de sangre periférica y tejido de colon de individuos sanos se utilizaron como control.
FISH
El FISH bicolor se realizó con sondas marcadas directamente con diferentes fluorocromos, para el centrómero del cromosoma 17 (CEP 17; señal verde; VYSIS) y para el locus específico del gen TP53 (LSI 17p13.1; señal naranja; VYSIS). La hibridización bicolor y detección en núcleos interfásicos se realizó siguiendo las instrucciones y recomendaciones del proveedor comercial (Vysis-Abbott). Las placas con núcleos interfásicos se desnaturalizaron en formamida al 70% a 73ºC por 5 min. Luego las placas se deshidrataron en una serie de etanoles fríos al 70%, 85% y 100%. En cada placa se adicionó 10 µl de cada una de las sondas CEP 17 y LSI TP53 en el área de hibridización seleccionada, sobre la cual se puso un cubreobjetos de 22x22 mm; la hibridización se realizó dentro de una cámara húmeda a 37ºC por 16 h. Luego, se realizaron dos lavados poshibridización. Finalmente las placas con núcleos interfásicos se colorearon con DAPI. Las señales de hibridización se observaron en un microscopio de epifluorescencia (Axyoskop 2 plus Zeiss, Alemania) dotado con un filtro triple simultáneo (DAPI/ORANGE/GREEN) y con una cámara CCD AxioCam MRc (Zeiss). En cada caso se analizaron 100 núcleos interfásicos; núcleos sobrelapados o con señales difusas se excluyeron para el análisis. En los casos normales (controles) el patrón de hibridización fue de dos señales de color verde (cromosoma 17) y dos señales de color naranja (locus 17p13.1 del gen TP53), para un total de cuatro señales de hibridización (2V2N).
Por otra parte, con base en los análisis previos realizados en las muestras control (datos no mostrados), se establecieron los siguientes puntos de corte para el análisis del FISH bicolor: aneuploidía cuando se tiene más del 9% de los núcleos con un número menor o mayor de 2 señales de hibridización para el centrómero del cromosoma 17; es decir, que una muestra es normal (disómica) si tiene un porcentaje igual o mayor del 91% de los núcleos con dos señales para el centrómero del cromosoma 17; mientras que para la nulisomía, monosomía y trisomía el punto de corte fue del 1%, 7% y 4% respectivamente; La tetrasomía no fue observada en los núcleos normales, por tanto, se definió arbitrariamente el 1% como punto de corte. En todos los casos los puntos de corte se calcularon con dos desviaciones estándar. En los casos donde se detecten subpoblaciones de núcleos con clones heterogéneos (mosómicos/ disómicos/ trisómicos/), se estableció que el resultado final se realizará con base en el clon mayor, es decir, el que presente el mayor porcentaje.
De otro lado, la deleción en el locus 17p.13.1 del gen TP53 se determinó por la relación entre el número total de señales de hibridización del gen TP53 observadas, dividido por el número total de señales del centrómero del cromosoma 17 (Nº de señales LSI TP53/Nº de señales CEP 17). Se definió que una muestra tenía deleción del gen TP53 cuando se obtiene un valor ≤0,94.
Resultados
Se analizaron 15 muestras de tumores gastrointestinales primarios mediante el FISH bicolor. Las muestras procedían de pacientes del departamento de Antioquia.
En la tabla 1 se presenta la información general de cada uno de los casos estudiados, la edad, el sexo, los hábitos en cuanto al consumo de tabaco y alcohol, la localización del tumor primario, el diagnóstico histopatológico, así como los resultados de las señales de hibridización del FISH bicolor.
Tabla 1. Relación entre edad, sexo, hábitos, localización del tumor primario y diagnóstico histopatológico con los perfiles de hibridización y el resultado del FISH bicolor en 15 pacientes con tumores sólidos
El estudio histopatológico mostró que la mayoría de las muestras analizadas (14/15) tenían un estado histopatológico avanzado, excepto el caso Nº 12 (un lipoma de colon). En estas muestras el adenocarcinoma bien diferenciado fue el estadio predominante; el caso Nº 8, consistió en una poliposis adenomatosa colónica la cual se considera como un estado premaligno (tabla 1).
En este estudio se encontró que el 33,3% (5/15) de los tumores gastrointestinales tenía aneuploidías del cromosoma 17 (tabla 1), mientras que el 66,7% (10/15) era normal para el número de copias del cromosoma 17. Los 5 casos con aneuploidías (casos 1, 2, 8, 9 y 15) presentaron monosomía del cromosoma 17 (figura 1). Los casos 1 y 9 mostraron los mayores porcentajes de núcleos monosómicos. Además, el caso 9 mostró un alto porcentaje (10%) de núcleos con cuatro señales para el cromosoma 17 (tetrasomía). En la mayoría de los casos analizados con el FISH bicolor, se detectó la presencia simultánea de subpoblaciones de núcleos con clones disómicos, monosómicos, trisómicos y en menor proporción tetrasómicos. En ningún caso se detectaron núcleos con pérdida de las dos copias del cromosoma 17 (nulisomía). El caso Nº 8, un estado premaligno, mostró un patrón de señales de hibridización anormal para el número de copias del cromosoma 17 (monosomía). En general, se obtuvo un valor promedio de dos copias del cromosoma 17 (89,4) en el grupo de los casos, significativamente menor que el promedio del grupo control (95) (tabla 1).
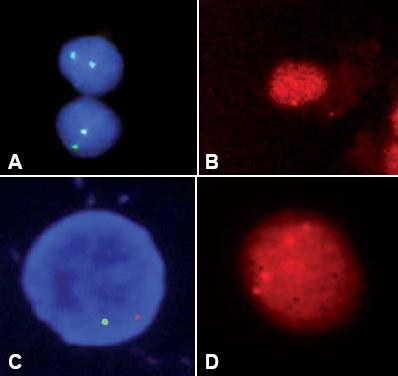
Figura 1. Microfotografías de núcleos interfásicos obtenidos de tumores gastrointestinales, luego del FISH y coloreados con DAPI. Señales de hibridización verdes corresponden a la sonda del cromosoma 17; señales de hibridización naranjas corresponden a la sonda del gen TP53. A. Tumor de estómago que muestra dos señales para el cromosoma 17 y sin señales para el gen TP53 (deleción bialélica). B. Tumor de estómago con una señal del gen TP53 (deleción monoalélica). C. Tumor de colon que muestra una señal para el cromosoma 17 (monosomía) y una señal para el gen TP53 (deleción). D. Adenocarcinoma de estómago con 4 señales para el gen TP53.
En cuanto a la deleción en el locus 17p13.1 del gen TP53, se encontró que el 93,3% (14/15) de las muestras examinadas tenía la deleción en este locus (tabla 1 y figura 1). Solo el caso Nº 12 (lipoma de colon) no presentó dicha deleción (1,02). El caso 6 (tumor de esófago) presentó un valor significativamente bajo para la deleción del gen TP53 (0,12), seguido de los casos 7 (tumor de esófago) y 4 (tumor de estómago). El caso 13 (tumor de colon) mostró un alto porcentaje (71%) de núcleos con una señal para el gen TP53 (deleción monoalélica), seguido de los casos 1 (tumor de estómago) y 9 (colon). Por su parte, el caso 6 presentó un alto porcentaje (86%) de núcleos sin señales para el gen TP53 (deleción bialélica), seguido de los casos 7 y 4. El caso Nº 9 fue el único que mostró un mayor porcentaje (8%) de núcleos con cuatro copias del gen TP53 (figura 1).
En general, se encontró un menor número de señales de hibridización del gen TP53, en relación con el número de señales del cromosoma 17. En esta relación se determinó un valor promedio para los casos de 0,70, mientras que para el grupo control fue de 0,97 (tabla 1). Estos valores presentan una diferencia muy significativa. Por otra parte, a diferencia de lo que se observó para el número de copias del cromosoma 17, en el grupo de los casos fue muy frecuente (13/15) la pérdida de las dos copias del gen TP53, con un rango entre 3 al 86% y un promedio de 19,5% (tabla 1). Este valor es muy alto comparado con el promedio del control (1,6%).
Los resultados obtenidos de las señales de hibridización para el gen TP53, muestran un comportamiento similar a los que se hallaron para la aneuploidía del cromosoma 17, con respecto a la detección de subpoblaciones de núcleos heterogéneos sin señales de hibridización y con una señal o copia del gen TP53. También se detectó un bajo porcentaje de núcleos con tres y cuatro señales.
Por otra parte, todos los casos con aneuploidía del cromosoma 17, también presentaron deleción del gen TP53 (tabla 1). De igual forma 9 de los 10 casos normales para el número de copias del cromosoma 17, tenían deleción del gen TP53 (tabla 1).
De las 15 muestras analizadas, el único caso normal fue el Nº 12 (lipoma de colon), tanto para el número de copias del cromosoma 17 (95%), como para las dos copias del gen TP53 (1,02) (tabla 1).
Discusión
En el presente estudio se evaluaron las aneuploidías del cromosoma 17 y deleción del gen TP53, en una muestra de tumores gastrointestinales primarios (TGI), mediante la técnica del FISH bicolor. En la población colombiana se han realizado muy pocos estudios sobre los aspectos genéticos del TGI. Por tal razón, la información es limitada con respecto a las alteraciones genéticas que ocurren en este tipo de tumores.
En este trabajo, todas las muestras analizadas de los pacientes con TGI, excepto la Nº 12 (lipoma de colon), presentaron un estado histopatológico avanzado del cáncer. El adenocarcinoma bien diferenciado fue el estadio más frecuente. Cabe anotar que el diagnóstico de ciertos tipos de TGI se realiza en estadios avanzados, lo cual podría explicarse por qué ciertos tipos de cánceres son asintomáticos durante las etapas iniciales del desarrollo y por lo tanto pasan inadvertidos en los individuos o por qué existen fallas en los sistemas de salud para el diagnóstico y tamizaje para algunas neoplasias, lo que ocasiona que mucha de éstas no se detectan temprana y oportunamente.
En este estudio se encontró una frecuencia de aneuploidías del cromosoma 17 del 33,3%, la monosomía se detectó en estos casos. Aunque esta frecuencia de aneuploidía (monosomía) es baja, estos hallazgos son similares a los informados por otros autores en TGI y están dentro del rango de porcentajes de alteraciones descritos en la literatura (18, 19). En este trabajo se confirma que la aneuploidía del cromosoma 17 es una alteración cromosómica común durante el desarrollo del TGI. Sin embargo, Risio et al (20) encontraron un porcentaje muy bajo, tanto de aneuploidía como de monosomía del cromosoma 17 en 20 muestras de cáncer colorrectal; mientras que otros estudios informan una alta frecuencia de trisomía del cromosoma 17 (19, 21). Lo anterior indica, específicamente, que para el cáncer colorrectal se observa la presencia de dos patrones de alteraciones cromosómicas: uno monosómico (como el este trabajo) otro trisómico, los cuales se asocian con un estado avanzado de malignidad (20, 22). En conclusión, la inestabilidad cromosómica es un evento común en el desarrollo del TGI y en general en el cáncer. Además, la aneuploidía podría considerarse como una alteración genética importante en los estados iniciales de la carcinogénesis.
De acuerdo con la teoría de aneuploidía y cáncer, Duesberg sustenta que el cáncer se origina por aneuploidías (pérdida o ganancia de cromosomas) al azar, que se originan espontáneamente o inducidas por carcinógenos (23). De esta manera, se establece un imbalance genético caracterizado por la inestabilidad cromosómica y genómica que afecta la expresión de diversos genes, responsables de la segregación cromosómica, control del ciclo celular, síntesis y reparación del ADN. Dichos cambios genéticos serían una consecuencia directa de la aneuploidía. Así mismo, esta teoría propone que la inestabilidad cromosómica es proporcional al grado de aneuploidía y al número de cromosomas afectados (24). En resumen, la teoría de Duesberg sustenta que las alteraciones cromosómicas juegan un papel fundamental en la iniciación y progresión del cáncer.
De otro lado, debe tenerse en cuenta que en el cromosoma 17 se localizan genes importantes involucrados en diversos mecanismos biológicos, como protooncógenes, genes supresores de tumores, como el ya mencionado TP53, genes que controlan el ciclo el ciclo celular y genes de reparación, entre muchos otros. Ejemplos de estos genes son el ERBB2, BRCA1, FLCN, NME2, CRK, THRA, HER2/neu (4, 6, 11). Varios de estos genes están involucrados en el control de la proliferación de la mucosa gástrica. (4, 21). Por tal razón, la pérdida o ganancia en el número de copias del cromosoma 17 afecta notablemente la expresión de dichos genes. De esta forma, se establece una vía relacionada con la génesis del TGI; así como para otros tipos de tumores como el de mama, ovario, vejiga, cabeza y cuello (13, 15, 25).
En este trabajo fue común el hallazgo de diferentes patrones de señales de hibridización por FISH en un mismo tejido. Es decir, que las muestras tenían clones con núcleos heterogéneos, los cuales eran monosómicos, disómicos, trisómicos y en menor frecuencia tetrasómicos. Esta condición se conoce como heterogeneidad genética intratumoral. Particularmente en el caso 1 (tumor de estómago) se observaron clones con diferente número de copias para el cromosoma 17. El 8% de los núcleos eran trisómicos, 1% tetrasómicos y 25% monosómicos. De acuerdo con los criterios establecidos para todos los casos, este último clon se define como el clon mayor, a partir del cual se determina el resultado final del FISH, en este caso una monosomía del cromosoma 17. En la biología del cáncer, el fenómeno de heterogeneidad genética es considerado como un evento común, que básicamente se origina por la inestabilidad cromosómica y por mutaciones en los genes mencionados (5, 8, 26). La heterogeneidad genética y su expansión clonal se evidencia ampliamente en diversos tipos de tumores sólidos, la cual se relaciona con el fenotipo tumoral, la progresión, metástasis y resistencia a ciertas drogas antineoplásicas (11, 24, 27).
En el presente estudio se demostró que el 93,3% de las muestras de TGI tenían deleción del gen TP53. Este porcentaje encontrado es muy alto comparado con otros estudios en TGI, pero está dentro del rango informado y es consistente con los hallazgos previos que indican, que este gen se encuentra alterado en más del 50% de todos los tipos de tumores (18, 19, 21, 26). Se considera que la pérdida del gen TP53 es una alteración grave que afecta el genoma, por su importancia de evitar la proliferación de células con lesiones, que por lo general presentan una ventaja selectiva sobre las células normales y en consecuencia se altera el control del ciclo celular (12-14).
La deleción del gen TP53 en este estudio se asocia con un importante imbalance entre el número de señales para este gen y las señales del centrómero del cromosoma 17 en núcleos interfásicos. Estos resultados sugieren que este imbalance se genera por las aneuploidías del cromosoma 17 y la deleción del locus 17p13.1 del gen TP53, las cuales ocurren durante el proceso del desarrollo del TGI. Adicionalmente, se ha informado que deleciones en la región 17p están presentes con mayor frecuencia en carcinomas avanzados (20, 26). De igual manera, se observa que deleciones del gen TP53 son más comunes en los carcinomas pobremente y bien diferenciados, tal como se encontró en el presente trabajo (27). En síntesis, de este trabajo puede afirmarse que el alto porcentaje de muestras con deleción del gen TP53, se relaciona con el estado avanzado de malignidad que presentaron dichas muestras. Por lo anterior, deleciones en la región 17p13.1 podrían jugar un papel clave durante la transición de un estadio temprano (benigno) hacia un estadio avanzado (carcinoma) del cáncer. No obstante, la significancia clínica y biológica de la deleción del gen TP53 en una variedad de tumores, incluido el TGI, aún no están bien definidas.
Similar a lo encontrado para las señales de hibridización del centrómero del cromosoma 17, varios patrones de hibridización se observaron para el gen TP53 en un mismo tejido. Es decir, se detectaron clones heterogéneos predominantemente con una señal para este gen (deleción monoalélica) y con pérdida de las dos copias del gen (deleción biálelica); núcleos con tres y cuatro señales se observaron en menor proporción. Estos hallazgos se explican por la heterogeneidad genética antes discutida.
En este trabajo se encontró que 5 casos (3, 4, 6, 7, 10) tenían un alto porcentaje de núcleos con pérdida de las dos copias del gen TP53 (deleción bialélica), especialmente el caso Nº 6 (tumor de esófago) que presentó un alto porcentaje (86%) de núcleos sin las dos copias del gen TP53 y tan solo el 9% de los núcleos analizados tenía las dos copias del gen. En estos casos podría afirmarse que la función del gen TP53 se encuentra inactiva. Por lo tanto, el ciclo celular de estas células está notablemente afectado. De otra parte, se ha observado que en los casos que presentan deleción en la región 17p de uno de los dos cromosomas, con frecuencia ocurren mutaciones en el gen TP53 localizado en el cromosoma 17 homólogo, lo que conduce a la pérdida de la función de este gen (28). Por tal razón, la pérdida de las dos copias de este gen se asocia con la progresión de la transformación maligna del TGI y con un mal pronóstico (20, 27). Además, cuando en la célula se presenta lo anterior, por los mecanismos descritos, tiene como consecuencia no sólo la pérdida del control del ciclo celular, sino también, la acumulación de diferentes tipos de alteraciones genéticas en diversos genes (14, 15).
Por otra parte, debe mencionarse que 9 casos no presentaron aneuploidías del cromosoma 17. Sin embargo, todos los casos tenían deleción del gen TP53. Estos hallazgos sugieren que estas muestran tienen una alteración cromosómica de tipo estructural. En conclusión, todas las muestras de TGI examinadas, excepto el caso Nº 12, tenían alteraciones genéticas, ya sea aneuploidías del cromosoma 17 y/o deleción de la región 17p13.1. Estos resultados llaman la atención sobre la frecuencia de alteraciones genéticas en el TGI y resaltan la importancia de los estudios genéticos en el cáncer en nuestra población.
Con respecto al caso Nº 12 (lipoma de colon), el único caso normal en este estudio para el número de copias tanto del cromosoma 17 como del gen TP53, podría mencionarse que esta muestra se encontraba al momento del estudio en un estado temprano de la tumorogénesis; es decir, un estadio benigno, en el cual las alteraciones en el gen TP53 no son muy frecuentes. Por el contrario, se observa dentro de la secuencia de eventos genéticos del cáncer de colón, que alteraciones en el gen TP53 ocurren en estadios avanzados (carcinoma de colon) (11). Sin embargo, no podría descartase que este caso presente alteraciones genéticas en otros cromosomas o genes diferentes a los evaluados en este trabajo.
El presente estudio es el primero en nuestro medio que informa el hallazgo de aneuploidías del cromosoma 17 y deleción del gen TP53 en tumores gastrointestinales primarios, mediante la técnica del FISH-bicolor. Esta técnica tiene muchas ventajas dada su alta especificidad y sensibilidad. Adicionalmente, permite realizar un análisis en corto tiempo para detectar simultáneamente alteraciones cromosómicas numéricas y estructurales en núcleos interfásicos de genes relacionados con el cáncer (19, 29). Así mismo, con el FISH puede demostrarse la heterogeneidad genética intratumoral que ocurre durante el desarrollo del cáncer (27, 30). Por estas ventajes el FISH se considera una técnica "gold standard" para los estudios cromosómicos en tumores sólidos y hematológicos. No obstante, se recomienda que este tipo de estudios se complementen con otras técnicas, tales como, la inmunohistoquímica y de biología molecular como el secuenciamiento directo del ADN o PCR en tiempo real, con el propósito de lograr un mayor conocimiento de la genética molecular del cáncer.
En conclusión, con base en los resultados obtenidos en este trabajo, la monosomía del cromosoma 17 y la deleción del locus 17p13.1 del gen TP53 son alteraciones comunes en TGI, especialmente en estados avanzados de malignidad, lo que sugiere que dichas alteraciones están entre las anomalías citogenéticas más frecuentes en los carcinomas gastrointestinales. Por lo tanto, se ha propuesto que estas dos alteraciones se consideren como marcadores cromosómicos recurrentes en el desarrollo del TGI, lo cual podría ser de gran utilidad para el diagnóstico y el pronóstico de este tipo de cáncer (4, 7, 20).
Finalmente, en las muestras analizadas en este estudio, no se descarta la presencia de otros tipos de alteraciones en cromosomas y genes diferentes a los evaluados. Varios estudios en TGI informan aneuploidías en los cromosomas 1, 7, 8, 13 y 18, como también mutaciones en los genes K-RAS, DCC, APC y C-MYC, entre otros (4, 6, 10, 11, 13, 20, 31). Cabe anotar que la técnica del FISH no detecta mutaciones puntuales. Adicionalmente, no puede afirmarse categóricamente que las aneuploidías del cromosoma 17 y las deleciones del gen TP53 son la causa del proceso neoplásico; más bien podría sugerirse que las alteraciones numéricas y estructurales del cromosoma 17 juegan un papel importante en el desarrollo y la progresión del TGI. Por tal razón, futuras investigaciones son necesarias para identificar alteraciones en otros cromosomas y genes involucrados en el proceso de carcinogénesis gastrointestinal en la población colombiana.
Agradecimientos
Los autores agradecen la participación de los pacientes en este trabajo. A los cirujanos, enfermeras y jefes de los departamentos de cirugía y patología de la Universidad de Antioquia y del Hospital Universitario San Vicente de Paúl, de la ciudad de Medellín por su valiosa colaboración. Este trabajo fue financiado por la Universidad de Antioquia, proyecto CODI-CPT-0312.
Referencias
1. Jemal A, Siegel R, Ward E, Murray T, Xu J, Thun MJ. Cancer statistics, 2007. Cancer J Clin 2007; 57(1): 43-66. [ Links ]
2. Zambrano A, Camacho L. Estrategias para la prevención del cáncer en Colombia. Acta Médica Colombiana 2002; 27(4): 221-234. [ Links ]
3. Smith MG, Hold GL, Tahara E, El-Omar EM. Cellular and molecular aspects of gastric cancer. World J Gastroenterol 2006; 12(19): 2979-90. [ Links ]
4. Tahara E. Genetic alterations in human gastrointestinal cancers. The application to molecular diagnosis. Cancer 1995; 75: 1410-7. [ Links ]
5. Lengauer C, Kinzler KW, Vogelstein B. Genetic instabilities in human cancers. Nature 1998; 396: 643-649. [ Links ]
6. Vauhkonen M, Vauhkonen H, Sipponen P. Pathology and molecular biology of gastric cancer. Best Pract Res Clin Gastroenterol 2006; 20(4): 651-74. [ Links ]
7. Rajagopalan H, Lengauer C. Aneuploidy and cancer. Nature 2004; 432: 338-41. [ Links ]
8. Tang R, Changchien CR, Wu MC, Fan CW, LIu KW, Chien JS, Hsieh LL. Colorectal cancer without high microsatellite instability and chromosomal instability an alternative genetic pathway to human colorectal cancer. Carcinogenesis 2004; 25: 5. [ Links ]
9. Michor F, Iwasa Y, Lengauer C, Nowak MA. Dynamics of colorectal cancer. Semin Cancer Biol 2005; 15(6): 484-93. [ Links ]
10. Conlin A, Smith G, Carey FA, Wolf CR, Steele RJ. The prognostic significance of K-ras, p53, and APC mutations in colorectal carcinoma. Gut 2005; 54(9): 1283-6. [ Links ]
11. Lengauer C, Kinzler KW and Vogelstein B. Genetic instability in colorectal cancers. Nature 1997; 386: 623-627. [ Links ]
12. Strano S, DellOrso S, Di Agostino S, Fontemaggi G, Sacchi A, Blandino G. Mutant p53: an oncogenic transcription factor. Oncogene 2007; 26(15): 2212-9. [ Links ]
13. Herrera JC, Vásquez G, Ramírez JL, Muñetón CM. Papel del gen TP53 en la oncogénesis. Salud UIS 2004; 26: 88-99. [ Links ]
14. Soussi T. p53 alterations in human cancer: more questions than answers. Oncogene 2007; 26(15): 2145-56. [ Links ]
15. Petitjean A, Achatz MI, Borresen-Dale AL, Hainaut P, Olivier M. TP53 mutations in human cancers: functional selection and impact on cancer prognosis and outcomes. Oncogene 2007; 26(15): 2157-65. [ Links ]
16. Levsky JM, Singer RH. Fluorescence in situ hybridization: past, present and future. J Cell Sc 2003; 116(14): 2833-37. [ Links ]
17. Muñetón CM, Vásquez EM, Durango NE, Martínez J, Ramírez JL. Cytogenetic analyses of eight solid tumors and correlation with histopathologic findings. Patología (México) 2004; 42 (2):65-72. [ Links ]
18. Rao PH, Mathew S, Lauwers G, Rodríguez E, Kelsen DP, Chaganti RS. Interphase cytogenetics of gastric and esophageal adenocarcinomas. Diagn Mol Pathol 1993; 2(4): 264-8. [ Links ]
19. Takahasi Y, Nagata T, Asai S, Shintaku K, Eguchi T, Ishi Y, Fujii M, Ishikawa K. Detection of aberrations on 17p and gene in gastrointestinal cancers by dual (two-color) fluorescence in situ hybridization and GeneChip p53 assay. Cancer Genet Cytogenet 2000; 121: 38-43. [ Links ]
20. Risio M, Casorzoa L, Chiecchioa L, De Rosaa G, Rossinib F. Deletions of 17p are associated with transition from early to advanced colorectal cancer. Cancer Genet Cytogenet 2003; 147: 44-49. [ Links ]
21. Cesar AC, Borim AA, Caetano A, Cury PM, Silva AE. Aneuploidies, deletion, and over expression of TP53 gene in intestinal metaplasia of patients without gastric cancer. Cancer Genet Cytogenet 2004; 153: 127-32. [ Links ]
22. García J, Durán A, Tabernero MD, Garcia Plaza A, Flores Corral T, Nájera ML, Gómez-Alonso A, Orfao A. Numerical abnormalities of chromosomes 17 and 18 in sporadic colorectal cancer: Incidence and correlation with clinical and biological findings and the prognosis of the disease. Cytometry B Clin Cytom 2003; 51(1): 14-20. [ Links ]
23. Duesberg P, Li R, Rasnick D, Rausch C, Willer A, Kraemer A, Yerganian G, Hehlmann R. Aneuploidy precedes and segregates with chemical carcinogenesis. Cancer Genet Cytogenet 2000; 119(2): 83-93. [ Links ]
24. Duesberg P, Rausch C, Rasnick D, Hehlmann R. Genetic instability of cancer cells is proportional to their degree of aneuploidy. Proc Natl Acad Sci USA 1998; 95(23): 13692-7. [ Links ]
25. Risio M, Casorzo L, Redana S, Montemurro F. HER2 gene-amplified breast cancers with monosomy of chromosome 17 are poorly responsive to trastuzumab-based treatment. Oncol Rep 2005; 13(2): 305. [ Links ]
26. Gomyo Y, Osaki M, Kaibara N, Ito H. Numerical aberrations and point mutation of p53 gene in human gastric intestinal metaplasia and well-differentiated adenocarcinoma: analysis by fluorescence in situ hybridization (FISH) and PCR-SSCP. Int J Cancer 1996; 66: 594-9. [ Links ]
27. Fringes B, Mayhew TM, Reith A, Gates J, Ward DC. Numerical aberrations of chromosomes 1 and 17 correlate with tumor site in human gastric carcinoma of the diffuse and intestinal types. Fluorescence in situ hybridization analysis on gastric biopsies. Lab Invest 2000; 80(10): 1501-8. [ Links ]
28. Hoff C, Seranski P, Mollenhauer J, Korn B, Detzel T, Reinhardt R, Ramser J, Poustka A. Physical and transcriptional mapping of the 17p13.3 region that is frequently deleted in human cancer. Genomics 2000; 70(1): 26-33. [ Links ]
29. Muñetón CM. Citogenética molecular del cáncer. Med Lab 2000; 9: 11-26. [ Links ]
30. Furuya T, Uchiyama T, Murakami T, Adachi A, Kawauchi S, Oga A, Hirano T, Sasaki K. Relationship between chromosomal instability and intratumoral regional DNAploidyheterogeneity in primary gastric cancers. Clin Cancer Res 2000; 6(7): 2815-20. [ Links ]
31. Assumpcao PP, Ishak G, Chen ES, Takeno SS, Leal MF, Guimaraes AC, Calcagno DQ, Khayat AS, Demachki S, Smith Mde A, Burbano RR. Numerical aberrations of chromosome 8 detected by conventional cytogenetics and fluorescence in situ hybridization in individuals from northern Brazil with gastric adenocarcinoma. Cancer Genet Cytogenet 2006; 169(1): 45-9. [ Links ]