Introducción
Helicobacter pylori es una bacteria gram-negativa, de forma espiral y microaerofílica que infecta a más del 50% de la población mundial 1. La infección se ha asociado con enfermedades como gastritis, úlcera péptica, linfoma asociado con mucosa (MALT) y adenocarcinoma gástrico 2. Los factores de virulencia más importantes de H. pylori son el gen asociado con la citotoxina A (CagA) y la citotoxina vacuolizante A (VacA) 3. Según la presencia del gen cagA, las cepas se clasifican en positivas o tipo I, asociadas con una mayor virulencia debido al daño gástrico visible inducido; y negativas o de tipo II, asociadas con menor virulencia, actuando más como bacterias comensales que como patógenas 4.
La proteína VacA causa una serie de alteraciones en las células gástricas humanas, incluidas la formación de vacuolas citoplasmáticas 5,6, permeabilización de la membrana plasmática 6, secreción mitocondrial del citocromo c 7, fragmentación mitocondrial 8, activación de quinasas activadas por mitógenos 9 e inducción de autofagia 6; y ha sido clasificada como proteína inmunomoduladora 6,10. El gen vacA tiene 3 regiones polimórficas llamadas región señal (s), región intermedia (i) y región media (m), involucradas en el desarrollo de la enfermedad 2,3,6,11,12,13.
La región s está implicada en la eficiencia de la formación de canales y la región media (m) afecta el tropismo hacia las células huésped 3. La región intermedia (i) se describió recientemente y se ha observado que la actividad vacuolizante de la variante VacA i1 es más fuerte que la del subtipo VacA i2 3,6. Algunos análisis de la secuencia de nucleótidos y aminoácidos de este gen han revelado polimorfismos agrupados en los clústeres A, B y C, de los cuales los aminoácidos en los clústeres B y C son los responsables de la actividad vacuolizante de la proteína 11.
Para clasificar una cepa como i1, i2 o i3 se compara la secuencia de aminoácidos con las secuencias de las cepas de referencia (60190 para i1 y Tx30a para i2). La variante vacA i3 incluye cepas, cuyo clúster B es similar al i1 y el clúster C al i211. Se han descrito diferentes combinaciones de las 3 regiones: las cepas vacA s1/m1 con actividad vacuolizante y asociadas con un mayor riesgo de atrofia gástrica y adenocarcinoma en comparación con las cepas vacA s2/m2 menos virulentas 11,12. Asimismo, se ha descrito que las cepas vacA i1 están asociadas con adenocarcinoma gástrico 2,11,12.
Recientemente, estudios epidemiológicos han sugerido que la interacción entre los factores de virulencia cagA y vacA6, la susceptibilidad del huésped como diferencias entre hombres y mujeres 14,15,16 y factores ambientales modulan el desarrollo de la enfermedad 4,12.
Se han publicado varios estudios de genotipificación de aislamientos de H. pylori de diferentes poblaciones colombianas. Teniendo en cuenta que la región intermedia puede cambiar el comportamiento de la toxina, este estudio complementa los datos sobre la genotipificación de estas 2 toxinas, incluye la caracterización de esta región de vacA en aislamientos en una población andina colombiana con enfermedades gástricas de diferente severidad y evalúa las correlaciones entre el genotipo y el desarrollo de la enfermedad gástrica.
Materiales y métodos
Pacientes y población de estudio
Se incluyeron 62 pacientes mayores de 18 años infectados por H. pylori en Bogotá (Colombia) con sintomatologías gástricas e indicación de endoscopia. Se excluyeron pacientes que hubieran ingerido inhibidores de la bomba de protones (IBP) en los últimos 15 días, antiácidos 12 horas antes del procedimiento o antibióticos en el mes anterior; que padecieran enfermedades cardiovasculares y respiratorias; pacientes con cáncer, sometidos a radioterapia o quimioterapia en los 6 meses previos al procedimiento; y pacientes con coagulopatías o amiloidosis.
Cepas y purificación del ADN
Las biopsias se maceraron y se sembró una dilución 10-1 en agar GC (Oxoid, Alemania) suplementado con colesterol (1X, Gibco) y DENT (Oxoid, Alemania) 17. Se incubaron a 37 °C en una atmósfera controlada con 10% de dióxido de carbono (CO2) durante 4 a 10 días. Para la extracción de ADN se usó el kit Quick-gDNA Miniprep (Zymo Research, California, Estados Unidos) según las instrucciones del fabricante. Las cepas se seleccionaron por cultivo y se confirmaron como H. pylori mediante reacción en cadena de la polimerasa (PCR) del gen 23S con los cebadores HPY-S y HPY-A reportados previamente 18.
Detección de cagA
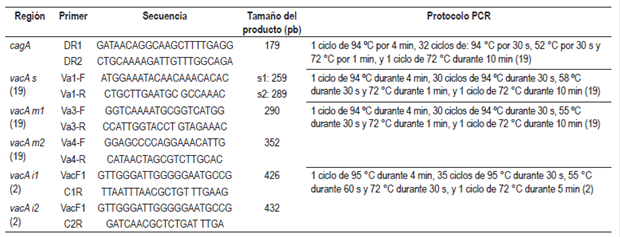
Se realizó una PCR con los cebadores diseñados en este estudio y el protocolo previamente reportado 19 modificado y descrito en la Tabla 1. La PCR tuvo un volumen final de 25 µL, con GoTaq Green Master Mix 1X (Promega, Wisconsin, Estados Unidos) y 0,25 mM de cada cebador. La visualización se realizó en geles de agarosa al 1,5%.
Genotipificación de vacA
Las 3 regiones polimórficas del gen vacA se caracterizaron por medio de PCR con un volumen final de 25 μL, que contenía GoTaq Green Master Mix 1X (Promega, Wisconsin, Estados Unidos) y 0,25 mM de los cebadores correspondientes para cada región, según los protocolos previamente reportados y modificados en este estudio (Tabla 1).
Para la visualización de los productos se usaron geles de agarosa al 2% (regiones m, i) y poliacrilamida al 10% (29: 1) (región s). Los geles de policacrilamida se tiñeron con una solución de GelRed 3X (15 μL de Gelred 10 000X y 5 mL de cloruro de sodio ENT#091;NaClENT#093; 1M) durante 1 hora bajo agitación constante a temperatura ambiente.
Se secuenciaron las muestras que tuvieron un resultado positivo para ambos subtipos (vacA i1 e i2) (Macrogen, Seúl, Corea). El análisis de secuencias se realizó con el software CLC Genomics Workbench 10 y se comparó la secuencia de aminoácidos con las secuencias de referencia 60190 (GenBank No. U05676) y Tx30a (GenBank No. U29401). Se definieron las cepas como i3 cuando el clúster B fue similar a la cepa de referencia i1 y el clúster C fue similar a la cepa de referencia i211.
Análisis estadísticos
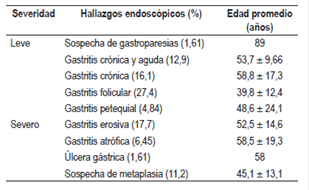
Se utilizó una prueba de Fisher para determinar posibles asociaciones entre el estado cagA y el genotipo vacA de las cepas, y se utilizó un modelo lineal generalizado para determinar con cuál de las variables había una asociación significativa. El mismo análisis estadístico se realizó entre las regiones polimórficas del gen vacA y el estatus cagA; y entre el estatus cagA, los subtipos del gen vacA y la gravedad de los hallazgos endoscópicos. Se utilizó un chi cuadrado (χ2) para determinar la posible asociación entre el sexo del paciente y el estatus cagA, y el sexo del paciente y la gravedad de los hallazgos endoscópicos. Los hallazgos endoscópicos se separaron en 2 grupos: enfermedad gástrica leve y enfermedad gástrica severa (Tabla 2). Para el análisis se utilizó el software R 20 y se consideró significativo un valor p <0,05.
Resultados
Pacientes y aislamiento de H. pylori
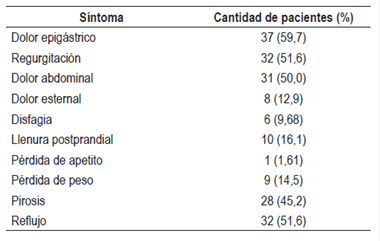
Se analizaron 124 cepas identificadas como H. pylori provenientes de 62 pacientes con una edad promedio de 50,2 ± 16,2 años con diferentes síntomas (Tabla 3) y cuya relación hombre-mujer fue de 1,48/1.
Genotipificación de cagA y vacA
La presencia del gen cagA se pudo establecer mediante PCR con los cebadores correspondientes y 52 (48,1%) de las 124 cepas analizadas fueron positivas para cagA. Sin embargo, no se encontró una asociación significativa entre la severidad de los hallazgos endoscópicos y la presencia de cagA (χ2, p >0,05) (Tabla 4). Tampoco se encontró una asociación significativa entre el sexo de los pacientes y la presencia de cagA (χ2, p >0,05) ni entre la gravedad de los hallazgos endoscópicos y el sexo (prueba exacta de Fisher, p >0,05) o la presencia de cagA (χ2, p >0,05) (Tabla 4).
Tabla 4 Asociación entre la severidad de los hallazgos endoscópicos, el estatus cagA y las regiones polimórficas del gen vacA*
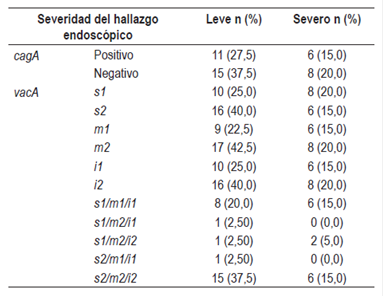
*No se encontró una relación significativa entre la severidad de los hallazgos endoscópicos y estatus cagA-vacA. Se excluyeron a los pacientes con genotipos diferentes entre antro y cuerpo. Prueba exacta de Fisher y modelo lineal generalizado binomial (p >0,05).
En el caso de vacA, se descartaron 16 cepas (n = 8 pacientes) en las que no fue posible determinar el genotipo para la región s: n = 3 (37,5%), la región m: n = 3 (37,5%), las regiones s e i: n = 1 (12,5%) o ninguna de las regiones: n = 1 (12,5%). De las 108 cepas restantes, 50 cepas fueron s1 y 58 s2, 41 cepas fueron m1 y 67 m2, y 34 cepas fueron i1 y 55 i2. Se secuenciaron 19 muestras (17,6%) que mostraron una amplificación para ambos subtipos vacA i1 e i2 (GenBank No. MF457450 a MF457477). De estas, 4 fueron clasificadas como i1, 3 como i2, 3 como i3 y 9 como i1i2; estas últimas no se tuvieron en cuenta para los análisis posteriores. No se encontró una asociación significativa entre los subtipos vacA o el genotipo completo de vacA y la gravedad de los hallazgos endoscópicos χ2, p >0,05) (Tabla 4).
Para evaluar la relación entre los 2 genes se descartaron 28 cepas (22,6%) debido a que tenían un genotipo diferente para cagA y vacA entre antro y cuerpo. No se encontró un tropismo o correlación significativa entre el genotipo y la ubicación dentro del estómago de estas cepas. Sin embargo, se encontró una asociación significativa entre el estado cagA y los subtipos del gen vacA por separado (prueba exacta de Fisher, p <0,05) (Figura 1) y combinados así: s1/m1/i1-cagA positivo (32,3%), s1/m2/i1-cagA positivo (2,02%) y s1/m2/i2 (7,07%); de los cuales el 71,4% fue cagA positivo, s1/m1/i3-cagA positivo (2,02%), s1/m2/i3-cagA positivo (1,01%), s2/m1/i1-cagA positivo (2,02%) y s2/m2/i2-cagA negativo (48,5%) (prueba exacta de Fisher, p <0,001; modelo lineal generalizado binomial p <0,05). Además, todas las cepas vacA i3 fueron cagA positivas.
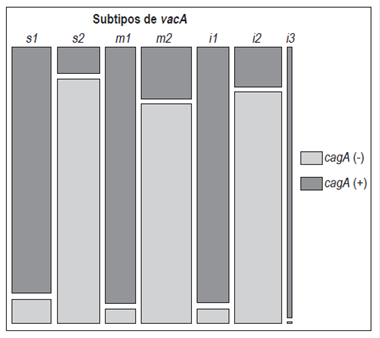
Figura 1 Asociación entre el estado cagA y los subtipos vacA. Existe una asociación significativa entre el estado cagA y los subtipos de vacA. Se excluyeron pacientes con estatus cagA o genotipos de vacA diferentes entre el antro y el cuerpo. Prueba exacta de Fisher y modelo lineal generalizado binomial (p <0,001).
Discusión
Este estudio caracteriza la diversidad de los genes de virulencia cagA y vacA en pacientes colombianos para determinar las posibles asociaciones entre estos 2 genes y la severidad de los hallazgos endoscópicos considerando todos los genotipos reportados para el gen vacA (s, m e i). Durante la infección por H. pylori, su variabilidad genética junto con factores del hospedero y del medio ambiente juegan un papel fundamental en el desarrollo de las patologías 3,4,6,12,21,22,23. De acuerdo con esto, se ha reportado que los pacientes infectados con cepas vacA s1/m1/i1- cagA (+) se encuentran en mayor riesgo de desarrollar carcinomas gástricos que aquellos pacientes infectados con cepas menos virulentas vacA s2/m2/i2-cagA (-) 2,4,11,12,13,19,24.
En la población colombiana estudiada, se encontró una prevalencia del 48,1% de los pacientes infectados con cepas cagA positivo, inferior a lo previamente reportado en estudios realizados en Bogotá, Colombia, que alcanzaban una prevalencia del 73% 24. Sin embargo, es similar a la reportada en pacientes provenientes de Tolima, Colombia, con un 43% de cepas cagA positivo 25. La prevalencia de este grupo estudiado difiere también de lo encontrado en otras regiones del mundo como Senegal (73,3%) 26, Medio Oriente (100%), Estados Unidos (80%), Europa Occidental y América Latina (60%-70%) 11. Esto probablemente se debe a que la población seleccionada era de clase socioeconómica media y alta, pues se ha visto que la prevalencia de la infección es mayor en los niveles socioeconómicos bajos, en los que existen pobres condiciones sanitarias 27,28.
Este estudio muestra también que el 22,6% de los pacientes presentaba infecciones por cepas con un genotipo diferente para cagA y vacA entre las 2 locaciones del estómago estudiadas (antro y cuerpo) sin ningún tropismo. Es posible que estos pacientes hayan sido infectados en ocasiones diferentes por diferentes cepas de H. pylori29, debido a que la prevalencia de infecciones múltiples parece ser mayor en lugares donde el riesgo de infección por H. pylori es alto, como en Colombia 30. La presencia de este tipo de infecciones reafirma la necesidad de toma de biopsias en cuerpo y antro de forma rutinaria.
La relación entre la presencia de cagA y del genotipo vacA s1 ha sido descrita por diferentes autores 31,32. En este estudio, el 68,1% de las cepas de cagA positivo fue vacA s1/m1/i1 y solo el 6,4% fue vacA s2/m2/i2. De este modo, el genotipo vacA s1/m1/i1 está significativamente asociado con la presencia del gen cagA. En el caso de las cepas vacA s1/m2, se ha observado que hay una mayor prevalencia de cepas tipo i1 o i2 dependiendo de la región geográfica 2,26. Se encontró una baja prevalencia de la combinación vacA s2/m1, lo que concuerda con lo descrito previamente, que sugiere que la baja prevalencia de este genotipo podría explicarse por una selección negativa de este subtipo por ser desfavorable para los microorganismos 26.
Además, se describe la región intermedia del gen vacA en las cepas circulantes en una población colombiana. Casi el total de cepas vacA s1/m1 (94,4%) fueron i1 y todas la cepas vacA s2/m2 fueron i2, en concordancia con lo reportado en la primera descripción de la región intermedia 2. La mayoría de las cepas analizadas fueron vacA i2 y se encontraron 3 pacientes infectados por cepas vacA i3 definidas como aquellas con el clúster B similar a i1 y el clúster C similar al tipo i211. La variante i3 podría reflejar un proceso de recombinación entre las cepas de tipo i1 e i26,11, pero la importancia clínica o patológica de esta variante sigue siendo desconocida y en nuestro estudio no se encontró ninguna asociación significativa con los hallazgos endoscópicos. Sin embargo, todas las cepas vacA i3 fueron cagA positivo. Sería necesaria una muestra más grande de pacientes infectados por cepas i3 para entender mejor la naturaleza de este genotipo y el desarrollo de la enfermedad en estos pacientes.
Contrario a la asociación significativa entre el genotipo vacA s1/m1/i1-cagA (+) y algunas enfermedades digestivas severas como la gastritis atrófica, úlceras y adenocarcinoma gástrico observada en otros estudios 33; no se encontró ninguna asociación significativa entre el estatus cagA-vacA de las cepas y la severidad de los hallazgos endoscópicos, a pesar de que la mayor parte de las cepas aisladas fueron vacA s2/m2/i2- cagA (-) provenientes de pacientes que en su mayoría presentaban gastritis folicular (Figura 2). Lo anterior pudo ser el resultado del tamaño reducido de la muestra para cada una de las categorías de la severidad de los hallazgos endoscópicos (leve o severo) y de la alta prevalencia de cepas tipo II en los pacientes. Por este motivo, el genotipo de vacA no puede utilizarse para identificar pacientes de alto riesgo en la población estudiada.
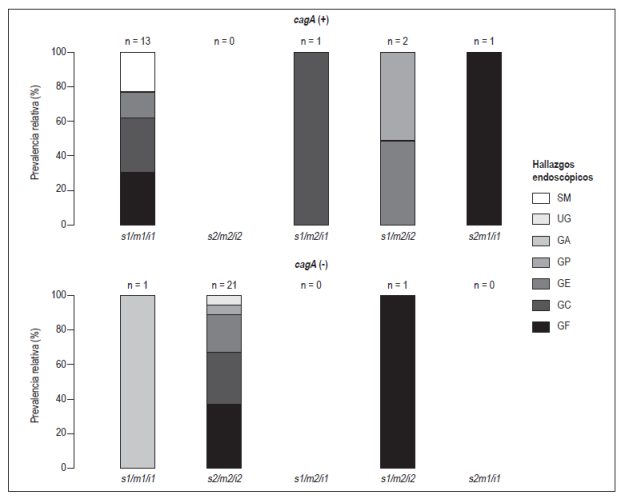
Figura 2 Prevalencia relativa de los hallazgos endoscópicos según el genotipo de las cepas de H. pylori aisladas en estos pacientes. El 52,5% de pacientes estaba infectado por cepas de H. pylori vacA s2/m2/i2-cagA negativo relacionadas en su mayoría con gastritis folicular. Se excluyeron pacientes con estatus cagA o genotipos de vacA diferentes entre el antro y el cuerpo. Sospecha de metaplasia (SM), úlcera gástrica (UG), gastritis atrófica (GA), gastritis petequial (GP), gastritis erosiva (GE), gastritis crónica (GC) y gastritis folicular (GF).
En conclusión, este estudio brinda información nueva sobre la prevalencia de los factores de virulencia cagA y vacA, y su asociación con los hallazgos endoscópicos, debido a que estudios realizados en la costa occidental y en el sur de los Andes difieren de lo encontrado en la presente investigación 34,35. Lo anterior recalca la necesidad de incrementar los estudios comparativos entre diferentes regiones y poblaciones colombianas, porque los datos publicados en Colombia muestran una alta variabilidad en la presencia de marcadores genéticos de patogenicidad y su correlación con la severidad de patologías gástricas.
Por su parte, algunos estudios recientes indican una evolución independiente de las cepas de H. pylori en la población colombiana originándose en la línea europea (HpEurope) 36. Es probable que los típicos mecanismos moleculares asociados con enfermedades gástricas severas asociadas con la presencia de esta bacteria se comporten diferente en la población colombiana gracias a las diferentes ancestrías presentes 37. Si se considera la relación bacteria-huésped, es posible que la falta de correlación entre cagA y vacA, y la patología de la infección en comparación con otros estudios se pueda investigar a fondo si se considera en futuros estudios la distribución ancestral presente en las regiones de Colombia.











 text in
text in