Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Medicas UIS
versão impressa ISSN 0121-0319
Medicas UIS vol.29 no.2 Bucaramanga maio/ago. 2016
https://doi.org/10.18273/revmed.v29n2-2016002
DOI: http://dx.doi.org/10.18273/revmed.v29n2-2016002
Resistencia a aminoglucósidos y quinolonas en
cepas de Klebsiella pneumoniae aisladas en dos
unidades de cuidados intensivos del Hospital
Universitario de Los Andes Mérida, Venezuela,
entre 2007 y 2009
Ana González*
Beatriz Nieves**
* Licenciada en Bioanálisis. Magister Sciantiae en Microbiología Clínica. Doctorado en Ciencias Médicas Fundamentales (Mención Microbiología). Laboratorio de investigaciones en Bacteriología "Roberto Gabaldón". Departamento de Microbiología y Parasitología. Facultad de Farmacia y Bioanálisis. Universidad de Los Andes. Mérida. Venezuela.
** Licenciada en Bioanálisis. Magister Scientiarum. Doctorado en Philosophus Scientiarum. Laboratorio de investigaciones en Bacteriología "Roberto Gabaldón". Departamento de Microbiología y Parasitología. Facultad de Farmacia y Bioanálisis. Universidad de Los Andes. Mérida. Venezuela.
Correspondencia: Dra. Ana Carolina González Romero. Laboratorio de investigaciones en Bacteriología "Roberto Gabaldón" Departamento de Microbiología y Parasitología. Facultad de Farmacia y Bioanálisis. Universidad de Los Andes. Sector Campo de Oro. Edificio Gonzalo González Mérida 5101, Mérida, Venezuela. Teléfono: 0058-274-2403515. Correo electrónico: caro60005@hotmail.com
Artículo recibido el 17 de septiembre de 2015 y aceptado para publicación el 30 de marzo de 2016.
RESUMEN
Introducción: el incremento en la resistencia a aminoglucósidos y quinolonas es un grave problema para el tratamiento de las infecciones a nivel mundial. Sin duda uno de los factores que ha contribuido a este incremento, es la gran plasticidad que poseen para adquirir genes que le confieren resistencia a los antimicrobianos. Objetivo: determinar los patrones y genes de resistencia a aminoglucósidos y quinolonas en cepas de K. pneumoniae aisladas de dos unidades del Hospital Universitario de los Andes Mérida. Venezuela en 2007 y 2009. Materiales y Método: evaluaron 39 cepas de K. pneumoniae aisladas en la unidad de alto riesgo neonatal y unidad de cuidados intensivos de adultos. La sensibilidad antimicrobiana se determinó mediante la prueba de difusión del disco y por E-test. La detección de los genes aac(3)-IIa, aac(6)-Ib, arma, rmt, qnrA, qnrB y qnrS se realizó por PCR y posterior secuenciación. Resultados: el 100% de las cepas aisladas en 2007 portaban el gen aac(6)-Ib, mientras que el 57% de las cepas aisladas de la misma unidad y de la unidad de cuidados intensivos de adultos en el año 2009 portaban los genes de resistencia aac(3)-IIa, aac(6)-Ib. La resistencia a quinolonas se observó en el 88,8% de las cepas aisladas de la y portaban el gen qnrB. Conclusiones: la resistencia a los aminoglucósidos en las cepas aisladas en la unidad de alto riesgo neonatal estuvo mediada por los genes genes aac(3)-IIa y aac(6)-Ib. Mientras que la resistencia a quinolonas se presento en las cepas aisladas de la unidad de cuidados intensivos de adultos mediadas por el gen qnrB. MÉD.UIS. 2016;29(2):21-30.
Palabras clave: Klebsiella pneumoniae. Factores R. Quinolonas. Resistencia a Medicamentos.
Aminoglycoside and quinolone resistance in strains of Klebsiella pneumonia isolated from
two units of intensive care unit of the University of the Andes Hospital, Merida, Venezuela
between 2007 and 2009
ABSTRACT
Introduction: aminoglycoside and quinolone resistance is serious problem for the treatment of infections worldwide.Without doubtone of the factors that has contributed to the increase in the resistance in enterobacteria, is the great plasticity that possess the bacteria of this family to acquire genes that confers resistance to antimicrobials. Objective: to determine the patterns of resistance to aminoglycosides and quinolones in strains of Klebsiella pneumoniae isolated from two intensive care units of the Universidad de los Andes, Merida, Venezuela. Methods: weevalvated with 39 K. pneumoniae strains isolated in neonatal high risk unit and adult intensive care unit. Antibiotic susceptibility was tested by the disk diffusion method and by the E-test method. Detection of aac (3)-IIa, aac (6)-Ib, arma, rmt, qnrA, qnrS qnrB genes was carried out by PCR and subsequently sequenced. Results: 100 % of the strains isolated in 2007 were carriers of the gene aac(6)-Ib , while 57% of the strains isolated from the same unit and the in the year 2009 were carriers of resistance genes aac(3)-IIa, aac(6)-Ib. The resistance to quinolones were observed in the 88.8% of the strains of Klebsiella isolated from the AICU and were carriers the qnrB gene. Conclusions: the resistance to aminoglycosides in strains isolated in the neonatal high risk unit was mediated by genes aac(3)-IIa and aac(6)-Ib. While the resistance to quinolones was presented in the strains isolated from the adult intensive care unit mediated by the qnrB gene. MÉD.UIS. 2016;29(2):21-30.
Keywords: Klebsiella pneumoniae. R Factors. Quinolones. Drug Resistance.
¿Cómo citar este artículo?: Gonzáles A, Nieves B. Resistencia a aminoglucósidos y quinolonas en cepas de Klebsiella pneumoniae aisladas en dos unidades de cuidados intensivos del Hospital Universitario de Los Andes Mérida, Venezuela, entre 2007 y 2009. MÉD.UIS. 2016;29(2):21-30.
INTRODUCCIÓN
El uso continuo e indiscriminado de los antimicrobianos en los hospitales y la falta de control para su uso por la población en general, ha llevado a la selección de microorganismos resistentes y por tanto, al incremento en la dificultad en el tratamiento de las infecciones por estos1. La resistencia a los aminoglucósidos ha sido reportada frecuentemente ligada a la de los antibióticos β-lactámicos, ya que las enzimas modificadoras suelen estar codificadas en plásmidos que portan genes codificadores de β-lactamasas2,3. La gentamicina es un aminoglucósido ampliamente utilizado para el tratamiento de las infecciones nosocomiales, como lo reportan varios estudios, los cuales describen brotes epidémicos causados por cepas de Klebsiella pneumoniae resistente a este fármaco, con capacidad de diseminación horizontal mediante plásmidos conjugativos que pueden portar otros determinantes de resistencia4-6.
La resistencia a los aminoglucósidos puede presentarse por tres mecanismos principales: resistencia ribosómica, transporte insuficiente y modificación enzimática del antibiótico, siendo este último el mecanismo principal. Dicha resistencia es producida por diversas enzimas como acetilasas, adenilasas y fosfatasas, las cuales modifican los grupos hidroxilos o aminos en los residuos amiciclitol del antibiótico, originando un compuesto que tiene baja afinidad por el ribosoma bacteriano7. También se han descrito distintos genes responsables de la metilación postranscripcional del ARN ribosómico (arma, rmt o npmA) con una prevalencia moderada y geográficamente dependiente. En este estudio, la resistencia observada suele ser de alto nivel a todos los aminoglucósidos, excepto neomicina8.
Por otra parte, se ha observado que la incidencia de resistencia a las fluoroquinolonas también se ha incrementado en los últimos años en todas las especies bacterianas. Dicha resistencia se ha descrito, principalmente, en aislamientos nosocomiales. Desde 1998 se empezaron a describir mecanismos de resistencia de origen plasmídico, como la protección de la diana por proteínas Qnr, la modificación de las quinolonas por la acetiltransferasa aac(6')- Ib-cr, o las bombas de expulsión activa QepA y OqxAB9-12. La resistencia de los aminoglucósidos y quinolonas en las enterobacterias se ha convertido en un grave problema para el tratamiento de las infecciones causadas por estas bacterias en las unidades de cuidados intensivos en los hospitales a nivel mundial13-15. Debido a la escasa información en Venezuela acerca de la presencia de estos determinantes en cepas hospitalarias de enterobacterias, se propuso el presente estudio para determinar los diferentes patrones de resistencia a los aminoglucósidos y quinolonas, así como los genes que los codifican en las cepas de K. pneumoniae resistentes aisladas de dos unidades de cuidados intensivos del Hospital Universitario de los Andes.
MATERIALES Y MÉTODOS
Se analizaron 39 cepas de K. pneumoniae multirresistente aisladas de pacientes hospitalizados en las unidades de Unidad de Alto Riesgo Neonatal (UARN) y del medio ambiente durante los periodos de febrero a junio de 2007 y junio a noviembre de 2009, así como cepas de K. pneumoniae aisladas en la Unidad de Cuidados Intensivos de Adultos (UCIA), durante el periodo comprendido entre junio y noviembre de 2009 en el Hospital Universitario de los Andes. Como criterio de inclusión se consideró la selección de todas las cepas de K. pneumoniae con sensibilidad disminuida a cefalosporinas de tercera generación, aminoglucósidos y quinolonas aisladas de ambos periodos en las diferentes unidades de cuidados intensivos y se excluyeron aquellas que presentaron un perfil de sensibilidad.
Se incluyeron cinco cepas de K. pneumoniae aisladas de neonatos hospitalizados en la UARN durante los meses de febrero y marzo de 2007 a consecuencia de un brote, así como siete cepas obtenidas durante el muestreo microbiológico que se realizó en la unidad para determinar la posible fuente de infección6. También se incluyeron cuatro cepas de K. pneumoniae aisladas posteriormente durante el mismo periodo.
Dos años después, de junio a noviembre de 2009, se realizó un nuevo muestreo en la UARN con los mismos criterios de selección para evaluar la evolución del brote, aislándose 14 cepas de K. pneumoniae de la sangre de los neonatos hospitalizados. Así mismo, con la finalidad de detectar la posible diseminación de cepas de K. pneumoniae multirresistente a otras unidades del Instituto Autónomo Hospital Universitario de los Andes, también se incluyeron en este estudio nueve cepas con el mismo patrón de resistencia aisladas de pacientes ingresados en la UCIA de junio a noviembre de 200916.
El procesamiento de las muestras y el estudio microbiológico se llevó a cabo en el Laboratorio de Bacteriología Anaeróbica "Dr. Roberto Gabaldón", del Departamento de Microbiología y Parasitología de la Facultad de Farmacia; y el estudio Molecular en el Laboratorio de Biomedicina Experimental (LABIOMEX) de la Facultad de Ciencias de la Universidad de los Andes.
La sensibilidad a los antimicrobianos se corroboró mediante la prueba de difusión de disco-placa en agar Mueller-Hinton. Los antibióticos probados fueron los siguientes: a) Quinolonas: ácido nalidíxico (30 μg BD BBL TM), ciprofloxacina (5μg BD BBL TM); b) Aminoglucósidos: gentamicina (10μg. BD BBL TM), amikacina (30μg. BD BBL TM), tobramicina (30μg. BD BBL TM), se utilizó como cepa control a Escherichia coli ATCC 25922. La concentración mínima inhibitoria se determinó por E-test (BioMérieux, Francia), siguiendo las instrucciones de la casa comercial; la sensibilidad de las cepas en estudio se interpretó mediante la aplicación de los puntos de corte del Clinical Laboratory Standards Institute (2013)17.
CARACTERIZACIÓN DE LOS GENES QUE CODIFICAN RESISTENCIA A LOS AMINOGLUCÓSIDOS Y QUINOLONAS
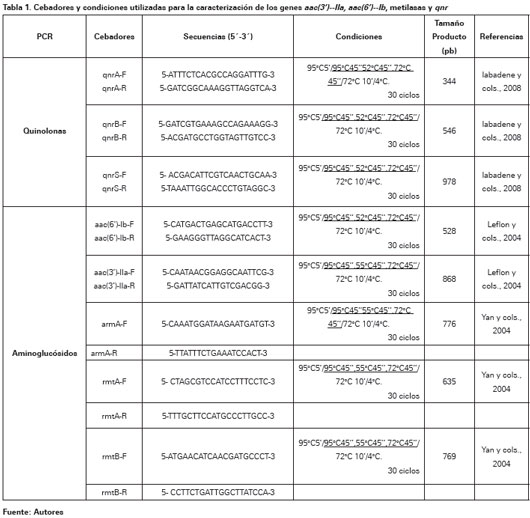
El aislamiento de ADN se realizó resuspendiendo en un tubo eppendorf una colonia de K. pneumoniae proveniente de un cultivo fresco en agar MH (no mayor de 18 horas) en 500 μl de agua destilada estéril. Se mezcló bien la solución y se colocó en baño María a ebullición por diez minutos. Luego se centrifugó a 12 000 rpm por diez minutos a temperatura ambiente. Se transfirió el sobrenadante a un tubo eppendorf de un mL y se almacenó a -20°C para su uso. Las mezclas de reacción de PCR para la caracterización de los genes de resistencia a aminoglucósidos aac(3')- IIa, aac(6')-Ib y metilasas (armA, rmtA, rmtB) y los de resistencia a quinolonas (qnrA, qnrB y qnrS), se realizó en un volumen final de 25μL, que contuvo 13μL de agua destilada estéril, nueve microlitros de EconotaqR PLUS (Master mix), un μL de cada primer (un micromol) y un microlitros de ADN. Se utilizaron como controles positivos cepas previamente caracterizadas y como control negativo el empleo de una mezcla de los componentes de la reacción sin ADN molde, utilizando agua para completar el volumen. La amplificación de los distintos genes se llevó a cabo con los cebadores específicos y las siguientes condiciones (Ver Tabla 1).
Para detectar los productos de la amplificación se realizó una corrida electroforética en un gel de agarosa (Agarose for Routine Use, Sigma-Aldrich, USA) al 1% durante 60 minutos, a 100 voltios. Posteriormente, el gel fue teñido con bromuro de etidio (un μg/mL) en agua destilada. Para estimar el tamaño de los fragmentos de ADN resultante, se utilizó un marcador de peso molecular con bandas de concentración definida Gene RulerTM100bp DNA Ladder plus (MBI Fermentas). Los patrones resultantes se visualizaron por tansiluminación utilizando el Gel Doc EZ Imager con el software ImageLab (Bio- Rad) que permite visualizar las bandas en un ordenador y documentarlo fotográficamente.
SECUENCIACIÓN
La secuenciación se realizó en un secuenciador ABI3730XL Analyzer (AB Applied Biosystems) previa purificación del ADN amplificado obtenido en la PCR. Se utilizaron los cebadores de cada PCR a una concentración de diez micromol. Las secuencias obtenidas fueron analizadas mediante el programa SeqMan analyse software DNA y la página web BLAST® (Basic Local Alignment Search Tool)21.
ANÁLISIS ESTADÍSTICO
Se realizó un análisis estadístico descriptivo por medio del programa estadístico SPSS para Windows versión 19, efectuando tablas de distribución de frecuencias y porcentajes simples de las variables objeto de investigación.
RESULTADOS
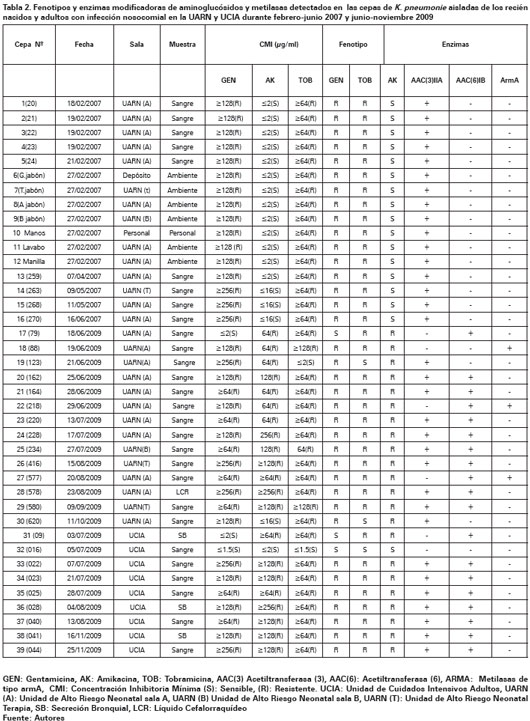
En este estudio se trabajó con un total de 39 cepas de K. pneumoniae multirresistentes aisladas de neonatos y medio ambiente de la UARN así como también de pacientes hospitalizados en la UCIA en diferentes periodos de tiempo. El 100% de cepas aisladas de la UARN en 2007, se mostraron resistentes a los antibióticos betalactámicos, especialmente a las cefalosporinas de tercera generación y monobactámicos con fenotipo de βLEE (+) además, se mostraron resistentes a los aminoglucósidos; antibióticos utilizados en el tratamiento empírico en la unidad. El 100% de las cepas aisladas en el 2007 eran resistentes a gentamicina y tobramicina con concentración mínima inhibitoria elevadas entre 128 y 64 μg/ml, respectivamente, siendo estas cepas portadoras del gen aac(3)-IIa (Ver Tabla 2).
Las 14 cepas de K. pneumoniae multirresistentes aisladas de la sangre de los neonatos durante el 2009 mostraron antibiotipos diferentes a las cepas aisladas en el 2007 y en su mayoría con fenotipo de βLEE (+). También se mostraron resistentes a los aminoglucósidos, donde el 57% era resistente a gentamicina, amikacina y tobramicina, siendo portadoras de los genes aac(6')-Ib y aac(3)-IIa. Además, es importante señalar que tres cepas eran portadoras del gen armA, siendo dos de estas, a su vez portadoras del gen aac(6')-Ib (Ver Tabla 2).
Por otro lado, el 88,8% de las cepas de K. pneumoniae multirresistentes aisladas de pacientes con sepsis y neumonía hospitalizados en la UCIA eran productoras de βLEE. Adicionalmente, se observó que el 77,7% de las cepas eran resistentes a los tres aminoglucósidos probados, portando los genes aac(6')-Ib y aac(3)-IIa (Ver Tabla 2).
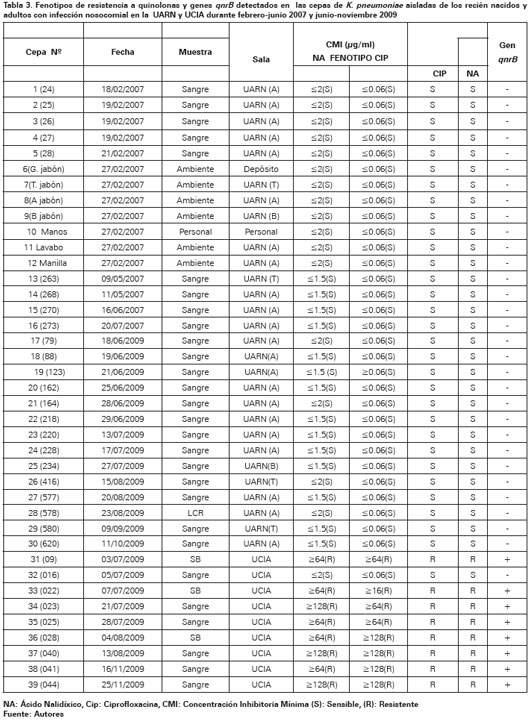
En relación a la resistencia observada en ambas unidades a las quinolonas, el 100% de las cepas aisladas de la UARN en los dos periodos 2007 y 2009 se mostraron sensibles; mientras que el 88,8% cepas de Klebsiella aisladas de la UCIA se mostraron resistentes a las quinolonas con concentración mínima inhibitoria elevadas para ácido nalidíxico y ciprofloxacina; y portaban el gen qnrB. (Ver Tabla 3). Es importante destacar que ninguna de las cepas aisladas de ambas unidades en los dos periodos de tiempo portaba los genes de resistencia qnrA, qnrS, rmtA y rmtB.
DISCUSIÓN
Numerosos estudios han explorado la relación entre el consumo de un antibiótico y la resistencia a este. Sin embargo, la existencia simultánea de distintos mecanismos de resistencia en las llamadas bacterias multirresistentes podría promover el fenómeno de co-selección, mediante el cual el uso de un antimicrobiano de alto consumo en el hospital seleccionaría subpoblaciones de bacterias resistentes a otros fármacos de estructura química diferente22. Los hospitales venezolanos no han estado ajenos a esta situación, por lo que el conocimiento de los mecanismos de resistencia, así como la ubicación de los genes implicados en dicha resistencia, proporcionan antecedentes importantes para entender la situación actual de la resistencia en el país, especialmente, si se consideran antibióticos de frecuente uso a nivel hospitalario como son las quinolonas, aminoglucósidos y los β-lactámicos.
El principal mecanismo de resistencia a aminoglucósidos es la producción de enzimas modificantes (acetiltransferasas, nucleotidiltransferasas y fosfotransferasas), las cuales han generando diferentes patrones de resistencia cada una. La coexistencia de más de una de estas genera patrones de resistencia a aminoglucósidos que dificultan la interpretación del antibiograma23,24. También se han descrito alteraciones en la dianas a nivel ribosómico o por metilación del 16s ARN, mediados en este último caso, por genes que codifican las metilasas de tipo arm o rmt25,26.
En este estudio se detectó resistencia a los aminoglucósidos, grupo de antibióticos utilizados en el tratamiento empírico en las unidades. Las cepas aisladas en el 2007 en la UARN mostraron el fenotipo de resistencia GENR, TOBR, siendo portadoras del gen aac(3)-IIa6. Por otro lado, el 57% las cepas aisladas de la misma unidad en el año 2009 mostraron el fenotipo de GENR, TOBR, AKR y portaban los genes de resistencia aac(6)Ib y aac(3)IIa, al igual que el 77,7% de las cepas de Klebsiella aisladas de la UCIA. Resultado que coincide con lo reportado en otros estudios como el realizado por Guzmán y cols4 donde reportan una frecuencia del 72% para el fenotipo de resistencia (amikacina, tobramicina, kanamicina y gentamicina); este fenotipo es poco usual y su presencia puede sugerir que en las cepas existía una combinación de las enzimas AAC(6) I Y AAC(3)I.
Neonakis y cols.,27 reportan que cuando existe esta asociación de enzimas se produce una combinación de amplio espectro capaz de inactivar a casi todos los aminoglucósidos disponibles, lo que coincide con los resultados de este estudio. Esta asociación es posible puesto que estas enzimas se pueden encontrar en plásmidos, asociadas a transposones e integrones y, ocasionalmente, pueden estar codificadas en el cromosoma bacteriano.
En otros estudios realizados en el Hospital Universitario de los Andes se han reportado que la resistencia de bacilos gramnegativos no fermentadores como Acinetobacter sp. a los aminoglucosidos, se ha ubicado en porcentajes que van de 25 a 100% 28. En un trabajo llevado a cabo en el mismo centro hospitalario, se identificó un brote epidémico causado por Acinetobacter 13TU:RUH 1139, que resultaron portadores del gen que codifica para la enzima aph -(3')-VIa, lo cual explicó, en gran parte la resistencia observada ante amikacina, gentamicina y kanamicina29.
En el presente trabajo, es importante destacar que tres cepas aisladas de la UARN durante el año 2009 eran portadoras del gen armA, dos de estas portaban también el gen aac(6')Ib y presentaron el fenotipo de resistencia GENR, TOBR, AMKR. Otros reportes describen la asociación entre metiltransferasas y βLEE, siendo especialmente notable el caso de ArmA y RmtB con CTX-M-330. En este estudio se detectó la asociación de ArmA con β-lactamasas CTX-M-15 y CTX-M-2. La frecuencia de genes que codifican metiltransferasas en K. pneumoniae fue baja (7,6%) en este trabajo, lo que coincide con estudios reportados en Taiwan y Shanghai, donde se han registrado prevalencias de 0,4 % y 3,4 % respectivamente31.
En América Latina se han reportado varios estudios donde se han detectado estas enzimas en miembros de la familia Enterobacteriaceae, en Brasil se describe la enzima RmtB en cepas de E. coli y Proteus mirabilis, también se ha reportado la enzima RmtD en Chile en aislados de Enterobacter cloacae y K. pneumoniae y en Argentina, en cepas de K. pneumoniae32. En relación a la resistencia observada en ambas unidades a las quinolonas se puede decir que las cepas aisladas de la UARN en los dos periodos 2007 y 2009 se mostraron sensibles. Mientras que el 88,8% de las cepas de K. pneumoniae aisladas de la UCIA eran resistentes y presentaron concentración mínima inhibitorias (entre 64-128 μg/mL) elevada a las quinolonas probadas, ácido nalidixíco y ciprofloxacina.
Entre los DPRQ descritos en enterobacterias a nivel mundial y en América Latina, se encuentran las proteínas Qnr (A, S, B, C y D), las bombas de expulsión QepA y OqxAB y la variante de la aminoglucósido acetil transferasa AAC(6')-Ib, denominada AAC(6')- Ib-cr, capaz de acetilar ciprofloxacino y norfloxacino, además de amikacina, tobramicina y kanamicina33-39. Los determinantes QnrA, QnrB y QnrS han sido identificados mundialmente en varios miembros de la familia Enterobacteriaceae y son frecuentemente relacionados a βLEEs, MBLs y a enzimas AmpC mediadas por plásmidos40-42. En este estudio, el 88,8% de las cepas de Klebsiella aisladas de la UCIA eran portadoras del gen qnrB y a su vez, portaban genes que codificaban β-lactamasas de tipo CTX-M-2, CTX-M-15 y SHV-12.
Este es el segundo estudio realizado en el país donde se reportan la presencia de estos genes. El primero realizado por González y cols.43, quienes identifican los genes qnrB en el 4,7% de las cepas de Salmonella aisladas de heces de pacientes pediátricos y muestras de carne de pollo, y el presente estudio que reportaba presencia de estos genes en cepas nosocomiales de K. pneumoniae aisladas en la UCIA del Hospital Universitario de los Andes.
Con la proliferación de cepas multirresistentes, el problema de las infecciones intrahospitalarias causadas por K. pneumoniae es aún mayor, ya que al ser más difíciles de tratar, dan lugar a incrementos de las tasas de mortalidad, de las estancias hospitalarias y de los costos de atención. De allí la importancia de este estudio que se basa en dar a conocer la existencia simultánea de distintos mecanismos de resistencia en bacterias intrahospitalarias lo que promueve el fenómeno de co-selección, mediante el cual el uso de un antimicrobiano de alto consumo en el hospital seleccionaría subpoblaciones de bacterias resistentes a otros fármacos de estructura química diferente. Los hospitales venezolanos no han estado ajenos a esta situación, por lo que el conocimiento de los mecanismos de resistencia, así como la ubicación de los genes implicados en dicha resistencia, proporcionan antecedentes importantes para entender la situación actual de la resistencia en el país, especialmente, si se consideran antibióticos de frecuente uso a nivel hospitalario como son las quinolonas, aminoglucósidos y los β-lactámicos.
CONCLUSIONES
Debido a la resistencia observada ante los aminoglucósidos y quinolonas en cepas de K. pneumoniae productoras de betalactamasas de espectro extendido aisladas de dos unidades de cuidados intensivos del Hospital Universitario de los Andes mediadas por los genes aac(3)-IIa, aac(6)-Ib, amrA y qnrB respectivamente se concluye que es indispensable vigilar continuamente la diseminación genética de dicha resistencia en las unidades de alto riesgo del Hospital Universitario de los Andes. Por lo que se recomienda realizar estudios de vigilancia epidemiológica, con el fin de racionalizar el uso de antimicrobianos de amplio espectro basados en la epidemiología local y en los mecanismos de resistencia y realizar el seguimiento del comportamiento de la resistencia bacteriana a betalactámicos, aminoglucósidos y quinolonas en las diferentes áreas de atención de la salud del Hospital para así establecer medidas de prevención de la propagación de dichos mecanismos.
CONFLICTO DE INTERESES
Los autores declaran no tener conflictos de intereses.
CONSIDERACIONES ÉTICAS
Este estudio cumple con las normas de Bioética y Bioseguridad del Ministerio de Ciencia y Tecnología y las normas para la investigación biomédica con humanos del Fondo Nacional de Ciencia y Tecnología.
FINANCIAMIENTO
Al Fondo Nacional de Ciencia y Tecnología en el marco del Programa de Estimulo a la Innovación e Investigación (PEII) N° 2012001234 por la financiación del estudio.
Al consejo de desarrollo científico humanístico y tecnológico (CDCHT) de la universidad de Los Andes. Proyecto código (FA-471-10-03-A).
REFERENCIAS BIBLIOGRÁFICAS
1. Alonso G, Narvaez P, Toba F, Gomes C, Pedroza R, Rodríguez V. Caracterización de plásmidos de resistencia a antibióticos en aislados nosocomiales del Hospital Universitario de Caracas. Rev. Soc. Ven. Microbiol. 2005; 25(1) [ Links ]
2. Lipsitch M, Matthew H. Antimicrobial Use and Antimicrobial Resistance: A population perspective. Emer Infect Dis. 2002;8(4):347-54 [ Links ]
3. Machado E, Coque T, Cantón R, Novais A, Sousa J, Baquero F, et al. High Diversity of Extended-Spectrum β-lactamases Among Clinical Isolates of Enterobacteriaceae from Portugal. J. antimicrob. chemother. 2007; 60(6): 1370-1374. [ Links ]
4. Guzmán M, Alonso G. Caracterización de β-lactamasas de espectro extendido (βLEE) en cepas nosocomiales de K. pneumoniae. Invest Clín. 2009; 50(4):419 - 431. [ Links ]
5. Araque M, Rivera I. Simultaneus presence of blaTEM and blaSHV genes on a large conjugative plasmid carried by extended espectrum β- lactamase-producing Klebsiella pneumoniae. Am J Med Sci. 2004; 327(3):118-22. [ Links ]
6. González A, Gil F, Solórzano M, Cruz J, Puig J, Suárez M, Nieves B. Brote por Klebsiella pneumoniae multiresistente y productora de β-lactamasa de espectro extendido en una unidad de alto riesgo neonatal. Rev Chil Infect. 2011; 28 (1): 28-34. [ Links ]
7. Tolmasky M, Bonomo R. Enzyme-mediated resistance to antibiotics: mechanisms, dissemination, and prospects for inhibition. ASM Press; Washington, DC: 2007. [ Links ]
8. Navarro F, Calvo R, Canton R, Fernandez F, Mirellis B. Phenotypic detection of resistance mechanisms in microorganisms. Enferm Infecc Microbiol Clin. 2011; 29 (7):524-534. [ Links ]
9. Jacoby GA. Mechanisms of Resistance to Quinolones. Clin Infect Dis. 2005; 41(2):120-126. [ Links ]
10. Martinez JL. Antibiotics and antibiotic resistance genes in natural environments. Sciencer. 2008; 321(5887):365-7. [ Links ]
11. Strahilevitz J, Jacoby GA, Hooper DC, Robicsek A. Plasmid. Mediated quinolone resistance: a multifaceted threat. Clin Microbiol Rev. 2009; 22(4):664-89. [ Links ]
12. Giamarellou H. Aminoglycosides plus beta-lactams against gram-negative organisms. Evaluation of in vitro synergy and chemical interactions. Am J Med. 1986;80(6B): 126-137. [ Links ]
13. Abreu S, Varela Y, Millán B, Araque M. Klebsiella pneumoniae y Escherichia coli productoras de beta-lactamasas de espectro extendido, aisladas en pacientes con infección asociada a los cuidados de la salud en un hospital universitario. Enf Inf Microbiol. 2014;34(3):92-9. [ Links ]
14. Espinal P, Mantilla J, Saavedra C, Leal A, Alpuche C, Valenzuela E. Epidemiología molecular de infección nosocomial por Klebsiella pneumoniae productora de betalactamasa de espectro extendido. Biomédica. 2004;24(3):252-61 [ Links ]
15. Torres L, Gagliotta V, Torres O, Benítez M, Domínguez M, Pedroza R. Extended-Spectrum β-Lactamases in Enterobacteriaceae isolated in Health Centres of Caracas. Rev Soc Ven Microbiol. 2006;26,80-8. [ Links ]
16. González A, Nieves B, Solórzano M, Cruz J, Puig J, Moreno M. Characterization of extended-spectrum β-lactamases-producing Klebsiella pneumoniae isolates of two intensive care units. Rev Chil Infect. 2013;30(4):374-80. [ Links ]
17. Clinical and Laboratory Standards Institute. (CLSI). Performance standards for antimicrobial susceptibility testing, 21th informational supplement. Document M100-S23. 2013;31(1). [ Links ]
18. Leflon-Guibout V, Jurand C, Bonacorsi S, Espinasse F, Guelfi MC, Duportail F, et al. Emergence and Spread of Three Clonally Related Virulent Isolates of CTX-M-15-Producing Escherichia coli with Variable Resistance to Aminoglycosides and Tetracycline in a French Geriatric Hospital. Antimicrob Agents Chemother. 2004;48(10):3736-42. [ Links ]
19. Yan JJ, Wu JJ, Ko WC, Tsai SH, Chuang CL, Wu HM, et al. Plasmid mediated 16S rRNA methylases conferring high-level aminoglycoside resistance in Escherichia coli and Klebsiella pneumoniae isolates from two Taiwanese hospitals. J Antimicrob Chemother. 2004;54(6):1007-12. [ Links ]
20. Iabadene H, Messai Y, Ammari H, Ramdani-Bouguessa N, Lounes S, Bakour R, et.al. Dissemination of ESBL and Qnr determinants in Enterobacter cloacae in Algeria. J Antimicrob Chemother. 2008;62(1):133-6. [ Links ]
21. Oteo J, Navarro C, Cercenado E, Delgado-Iribarren A, Wilhelmi I, Orden B, et al. Spread of Escherichia coli strains with high-level cefotaxime and ceftazidime resistance between the community, long-term care facilities, and hospital institutions. J Clin Microbiol. 2006;44(7):2359-66. [ Links ]
22. Ángel-Díaz M, Ramón-Hernández J, Martínez-Martínez L, Rodriguez-Baño J, Pascual A. Escherichia coli y Klebsiella pneumoniae productoras de betalactamasas de espectro extendido en hospitales espanoles: segundo estudio multicéntrico (Proyecto GEIH-BLEE 2006). Enferm Infecc Microbiol Clin. 2009;27(9):503-10. [ Links ]
23. Kotra LP, Haddad J, Mobashery S. Aminoglycosides: perspectives on mechanisms of action and resistance and strategies to counter resistance. Antimicrob Agent Chemother. 2000;44(12):3249-56. [ Links ]
24. Houghton JL, Green KD, Chen W, Garneau-Tsodikova S. The Future of Aminoglycosides: The End or Renaissance. Chembiochem. 2010;11(7):880-902. [ Links ]
25. Doi Y, Yokoyama K, Yamane K, Wachino J, Shibata N, Yagi T, et al. Plasmid Mediated 16S RNA Methylase in Serratia marcescens conferring high-level resistance aminoglycosides. Antimicrob Agent Chemother. 2004;48(2):491-6. [ Links ]
26. Galimand M, Courvalin P, Lambert T. Plasmid-mediated high- level resistance to aminoglycosides in Enterobacteriacea due to 16S RrRNA methylation. Antimicrob Agent Chemother. 2003;47(8):2565-71. [ Links ]
27. Neonakis A, Scoulica E, Manios A, Georgiladakis A, Tselentis Y. Evolution of aminoglycoside resistance phenotypes of four Gram negative bacteria: an 8-year survey in a University Hospital in Greece. Int J Antimicrob Agents. 2003;22(5):526-31. [ Links ]
28. Briceño I, Suarez M. Resistencia bacteriana en la unidad de cuidados intensivos del Hospital Universitario de Los Andes. MEDICRIT. 2006;3:30-42. [ Links ]
29. Salazar E, Nieves B, Guzmán M, Albarado L, Láreza I, González D. Determinación del gen que codifica la enzima APH-(3')-VIa en aislamientos de Acinetobacter 13TU:RUH1139 de origen hospitalario. Rev Soc Ven Microbiol. 2013;33:6-12. [ Links ]
30. Isturiz R. Global resistance trends and the potential impact on empirical therapy. Int J Antimicrob Agents. 2008;32(4):201-6. [ Links ]
31. Wu JJ, Ko WC, Tsai SH, Yan JJ. Prevalence of plasmid-mediated quinolone resistance determinants QnrA, QnrB, and QnrS among clinical isolates of Enterobacter cloacae in a Taiwanese hospital. Antimicrob Agents Chemother. 2007;51(4):1223-7. [ Links ]
32. Fritsche TR, Castanheira M, Miller GH, Jones RN, Armstrong ES. Detection of Methyltransferases Conferring High-Level Resistance to Aminoglycosides in Enterobacteriaceae from Europe, North America, and Latin America. Antimicrob. Agent Chemother. 2008;52(5):1843-5. [ Links ]
33. Jacoby GA, Walsh KE, Mills DM, Walker VJ, Oh H, Robicsek A, et al. qnrB, another plasmid-mediated gene for quinolone resistance. Antimicrob Agents Chemother. 2006;50(4):1178- 82. [ Links ]
34. Poirel L, Leviandier C, Nordmann P. Prevalence and genetic analysis of plasmid-mediated quinolone resistance determinants QnrA and QnrS in Enterobacteriaceae isolates from French university hospital. Antimicrob Agents Chemother. 2006;50(12):3992-7. [ Links ]
35. Jiang Y, Zhou Z, Qian Y, Wei Z, Yu Y, Hu S, et al. Plasmid-mediated quinolone resistance determinants qnr and aac(6')Ib-cr in extended -spectrum β-lactamase producing Escherichia coli and Klebsiella pneumoniae in China. J Antimicrob Chemother. 2008;61(5):1003-6. [ Links ]
36. Cambau E, Lascols C, Sougakoff V, Bébéar C, Bonnet R, Cavallo JD, et al. Occurrence of qnrA-positive clinical isolates in French teaching hospitals during 2002-2005. Clin Microbiol Infect. 2006;12(10):1013-20. [ Links ]
37. Martínez-Martínez L, Cano M, Rodríguez-Martínez J, Calvo J, Pascual A. Plasmid-mediated quinolone resistance. Expert Rev Anti Infect Ther. 2008;6(5):685-711. [ Links ]
38. Castanheira M, Pereira A, Nicoletti A, Pignatari A, Barth A, Gales A. First report of plasmid-mediated qnrA1 in a ciprofloxacin-resistant Escherichia coli strain in Latin America. Antimicrob Agents Chemother. 2007;51(4):1527-9. [ Links ]
39. Rodríguez-Martínez J, Conejoa M, Díaz de Albab P, López- Cererob L, Fernández Echaurib P, Pascuala A. Asociación en un mismo plásmido de blaVIM-1 y qnrS2 en Klebsiella pneumoniae y Klebsiella oxytoca aisladas en Sevilla. Enferm Infecc Microbiol Clin. 2012;30(5):246-8. [ Links ]
40. Cattoir V, Poirel L, Rotimi V, Soussy C, Nordmann P. Multiplex PCR for detection of plasmid-mediated quinolone resistance qnr genes in ESBL-producing enterobacterial isolates. J Antimicrob Chemother. 2007;60(2):394-7. [ Links ]
41. Hooper DC. Emerging mechanisms of fluoroquinolone resistance. Emerg Infect Dis 2001;7(2):337-41. [ Links ]
42. Hata M, Suzuki M, Matsumoto M, Takahashi M, Sato K, Sakae K. Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri. Antimicrob Agents Chemother. 2005;49(2):801-3. [ Links ]
43. González F, Pallecchi L, Rossolini GM, Araque M. Plasmid-mediated quinolone resistance determinant qnrB19 in non-typhoidal Salmonella enteric strains isolated in Venezuela. J Infect Dev Ctries. 2012; 6(5):462-4. [ Links ]