Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Iatreia
versão impressa ISSN 0121-0793
Iatreia vol.15 no.2 Medellín abr./jun. 2002
INVESTIGACIÓN ORIGINAL
Revisión de tema Citogenética de tumores sólidos
CYTOGENETICS OF SOLID TUMORS
CARLOS MARIO MUÑETÓN PEÑA1; JOSÉ LUIS RAMÍREZ CASTRO
1 Biólogo. MSc, JOSÉ LUIS RAMÍREZ CASTRO, Médico, MS. Profesores, Unidad de Genética Médica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia. Financiación: CODI-U. de A. CIM 9956.
EL ANÁLISIS CITOGENÉTICO DE TUMORES ha proporcionado valiosa información sobre la biología del cáncer. Se ha establecido que más de la mitad de los tumores sólidos presentan alteraciones cromosómicas; por lo tanto, el análisis citogenético es de gran utilidad para el diagnóstico y el pronóstico. La identificación de cambios cromosómicos específicos recurrentes en numerosos tumores se considera un indicador de importancia clínica. Los estudios en este campo han revelado cerca de 100.000 alteraciones cromosómicas en más de 30.000 neoplasias humanas. Sin embargo, los tumores sólidos son los menos caracterizados citogenéticamente, sólo una tercera parte del total de ellos, debido a problemas técnicos en los cultivos celulares. La citogenética convencional ha sido muy útil para la posterior caracterización molecular de nuevos oncogenes y genes supresores de tumores involucrados en la génesis tumoral. En esta revisión se presentan algunos aspectos relacionados con el cáncer de etiología cromosómica, tipos de alteraciones cromosómicas, origen, frecuencia, técnicas modernas para su estudio, así como el impacto en el diagnóstico y el pronóstico del cáncer.
PALABRAS CLAVE
ALTERACIONES CROMOSÓMICAS, CITOGENÉTICA, TUMORES SÓLIDOS, CARIOTIPO, CÁNCER
SUMMARY
CYTOGENETIC ANALYSIS OF TUMORS has provided valuable information on the biology of cancer. It has been established that more than half of solid tumors show chromosomal anomalies; therefore, cytogenetic analysis is of great usefulness for diagnostic and prognostic purposes. Identification of recurrent chromosomal anomalies in numerous tumors has been considered as an indicador of clinical importance. Cytogenetic studies in tissue tumors have revealed near 100,000 clonal chromosome abnormalities belonging to more that 30,000 human neoplasms. However, due to technical difficulties in cell cultures, only one third of solid tumors have been cytogenetically characterized. Conventional cytogenetics has been very useful for molecular characterization of new oncogenes and tumor-suppressor genes involved in human tumorigenesis. In this review, some important issues related with tumors of chromosomal etiology, the diverse types of chromosomal anomalies with their frequencies, modern diagnostic techniques as well as their impact on the diagnosis and prognosis of cancer are presented.
INTRODUCCIÓN
DESDE HACE MÁS DE TRES DÉCADAS se han acumulado evidencias del papel fundamental de las alteraciones cromosómicas en el proceso de transformación de una célula normal hacia un estado maligno (1,2). En 1890 el patólogo David Hansemann (3) hizo el primer estudio de tejidos tumorales, y observó la presencia de mitosis aberrantes en sus células, sugiriendo que este fenómeno podría utilizarse como un criterio para el diagnóstico de un estado maligno. Posteriormente, Theodor Boveri en 1914 (4) propuso la teoría de la mutación somática, y el origen clonal de las neoplasias. Según dicha teoría, el cáncer se desarrolla a partir de una célula individual que presenta cambios genéticos, en especial un desequilibrio en la dotación cromosómica. Sin embargo, esta teoría permaneció por largo tiempo sin evaluar críticamente por falta de técnicas apropiadas para su estudio. Con los avances en las técnicas de cultivo para la obtención de cromosomas metafásicos, rápidamente se confirmó la hipótesis de Boveri, la cual constituye en la actualidad un punto clave en la patogénesis del cáncer.
En 1960 Nowell y Hungerford (5) informaron la primera alteración cromosómica asociada con un tipo de cáncer en humanos, la leucemia mieloide crónica (LMC); el cromosoma marcador se denominó cromosoma Filadelfia (Ph1). Este descubrimiento de los años 60 fue de gran impacto en la citogenética del cáncer, pues corroboró las observaciones previas de Hansemann y Boveri, y además generó un notable interés por parte de numerosos investigadores. Pocos años después Levan y Van Steenis (6,7) en dos estudios independientes, encontraron una clara evidencia de que los cromosomas mostraban una tendencia a aumentar o disminuir en número en diferentes tipos de tumores sólidos, principalmente carcinomas mamario, gástrico, uterino y ovárico.
Debe anotarse que en épocas pasadas los estudios de tumores mostraban cromosomas con una morfología de baja calidad, lo cual impedía el reconocimiento de los mismos y de sus regiones específicas. Además, existían dificultades técnicas en cuanto al cultivo y procesamiento de los tejidos tumorales (8).
Los problemas técnicos antes mencionados, que además estaban relacionados con la citogenética humana en general, se solucionaron en gran parte en 1970, con el desarrollo de las técnicas de bandeo cromosómico por Caspersson y colaboradores (9).
Gracias a las mismas ocurrió un avance notable en los análisis cromosómicos de diferentes tipos de muestras y fue posible la identificación y clasificación de cada cromosoma según los patrones de bandeo. En 1973 Rowley (10) observó que el cromosoma Filadelfia se originaba de una translocación que comprometía los cromosomas 9 y 22, t(9;22) (q34;11); más tarde se determinó que en la región de ruptura se alteraban la estructura y la función de un oncogén.
A partir de dichos avances se logró un progreso significativo en la identificación de numerosas alteraciones cromosómicas en diferentes tipos de tumores (11-13). Actualmente es posible determinar sitios de ruptura en un gran número de translocaciones cromosómicas y además correlacionar ciertas regiones y bandas con procesos de transformación maligna (14). Estos avances no sólo son importantes en el campo de la citogenética del cáncer, sino también, en otras áreas como la hematología, la oncología y la patología.
CONCEPTOS GENERALES
EN LA ACTUALIDAD NUMEROSOS ESTUDIOS en humanos establecen una clara asociación de las alteraciones cromosómicas con los tumores sólidos y blandos de diferente origen histológico. Muchas de ellas se consideran marcadores específicos para un gran número de neoplasias (15-17). Lo anterior no sólo es importante para un mejor diagnóstico clínico, sino también para el pronóstico y el tratamiento de determinada entidad (18,19). Entre los diferentes mecanismos en la etiología del cáncer, las alteraciones cromosómicas juegan un papel importante, puesto que la mayoría de los tumores exhiben un cariotipo anormal (8, 11,20-23).
La citogenética del cáncer es un área que se ha especializado en la identificación de los cambios numéricos y estructurales de los cromosomas en diferentes neoplasias humanas. Hasta el presente las leucemias son las más estudiadas (18,21), pero también se han identificado numerosas alteraciones cromosómicas en linfomas, tumores sólidos y blandos, tanto malignos como benignos, la mayoría de ellas muy complejas (24).
En las últimas décadas se ha concluido que las alteraciones cromosómicas tienen un origen clonal, es decir, la formación de una población de células que proviene de una misma célula progenitora y con una dotación cromosómica anormal, la cual no ocurre al azar (4,21,25). Hallazgos recientes muestran cómo el funcionamiento anormal del telómero favorece la aparición de alteraciones cromosómicas complejas en tumores sólidos, concluyendo que la disfunción del mismo promueve la inestabilidad genómica como un evento temprano en la génesis tumoral (26).
Periódicamente se informan en la literatura mundial nuevas alteraciones cromosómicas identificadas en las neoplasias humanas. Este tipo de información se sistematizó y se recopiló en una base de datos Catalog of Chromosome Aberrations in Cancer. La primera edición de 1987 registraba alteraciones cromosómicas en más de 8.000 neoplasias humanas analizadas con técnicas de bandeo (27). La sexta edición del mismo catálogo (1998) contiene información sobre más de 30.000 alteraciones, recopilada de cerca de 7.000 artículos publicados hasta entonces en la literatura (28). Estas observaciones aportan una información valiosa acerca de la naturaleza cromosómica de numerosos tumores.
Mitelman y Levan en 1981 (14), en una revisión de 1.871 casos de tumores, concluyeron que por lo menos 15 de los 24 cromosomas humanos, presentan alteraciones directamente comprometidas con la génesis de diferentes neoplasias humanas. Cinco años después Mitelman (29) en un estudio de 5,345 casos informó 77 alteraciones cromosómicas diferentes observadas en todos los cromosomas humanos, excepto los sexuales. De estos casos se concluyó que determinados segmentos cromosómicos contienen genes que activan los mecanismos iniciales del tumor. Dichos genes se conocen como protooncogenes, genes supresores de tumores y genes reguladores del ciclo celular; todos ellos se encuentran distribuidos en los diferentes cromosomas humanos y tienen una relación directa con el cáncer (23,30).
A mediados de 1990 se habían informado estudios citogenéticos de 14.141 casos de cáncer (31); la mayoría de éstos (68%) correspondían a trastornos hematológicos, 21% a tumores sólidos y 11% a linfomas. El bajo porcentaje de tumores sólidos es explicable por las limitaciones técnicas y el poco éxito obtenido en el cultivo de células de los mismos (entre 15-30%).
ALTERACIONES CROMOSÓMICAS RECURRENTES Y CLASIFICACIÓN DE TUMORES SÓLIDOS
EN LA ACTUALIDAD CERCA DE 600 ALTERACIONES cromosómicas recurrentes se asocian con tumores de determinado de origen histológico; por ejemplo, en el carcinoma mamario los cambios cromosómicos ocurren con mayor frecuencia en los cromosomas 1,3,6,13,17 y en el cáncer de colon en los cromosomas 5, 17 y 18 (8,13,17,25).
Los tumores sólidos se clasifican según su origen histológico, en cinco categorías (8,15):
- Epitelial: colon, recto, mama, útero, ovario, pulmón, estómago, riñón, vejiga y próstata.
- Mesénquima: lipomas, liposarcomas, sarcoma sinovial, rabdomiosarcoma alveolar.
- Neurogénico: meningiomas, gliomas, neuroblastomas, retinoblastoma y neuroepitelioma
- Células germinales: teratomas de testículo, ovario y seminomas.
- Tumores de origen histológico desconocido: sarcoma de Ewing, melanoma.
De los tumores epiteliales humanos hay una mayor probabilidad de obtener cultivos exitosos, son los mejor caracterizados citogenéticamente, y el 47% de los mismos tienen un cariotipo con alteraciones específicas en los cromosomas 3,8 y 12 (8). En general, se han estudiado tanto tumores sólidos benignos (lipomas, leiomiomas, adenomas) como malignos (carcinoma mamario, de pulmón, de ovario, de próstata, de colon, sarcomas); en los benignos se observan cambios cromosómicos simples, mientras que en los malignos se identifican cariotipos complejos con múltiples alteraciones (16,17). Esto último tiene interés puesto que dichos tumores tienen mal pronóstico y poca respuesta a los tratamientos (18,19,32).
TIPOS DE ALTERACIONES CROMOSÓMICAS
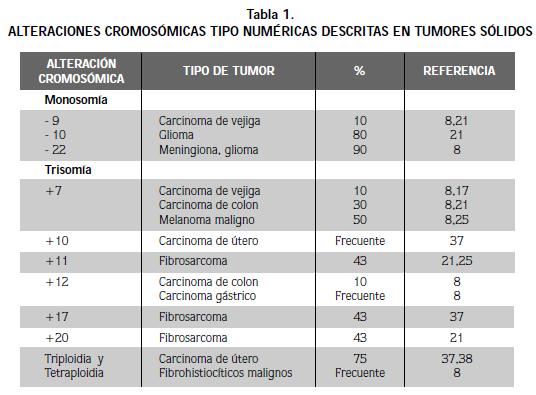
LAS ALTERACIONES CROMOSÓMICAS NUMÉRICAS y estructurales identificadas hasta el presente en tumores sólidos y blandos son muy variables, es decir, citogenéticamente presentan una gran heterogeneidad (15,17,33-36) y se observan con notable frecuencia; las estructurales se observan mayor proporción. Ambos tipos juegan inicialmente un papel importante en el origen de los tumores. Entre las alteraciones numéricas son comunes la pérdida (hipodiploidias, nulisomias, monosomia) o ganancia de cromosomas (trisomias, triploidias, tetraploidias), es decir, hiperdiploidias (36) (tabla 1). En estas últimas se observan con frecuencia cromosomas marcadores o supernumerarios de origen desconocido difíciles de clasificar por los métodos citogenéticos usuales.
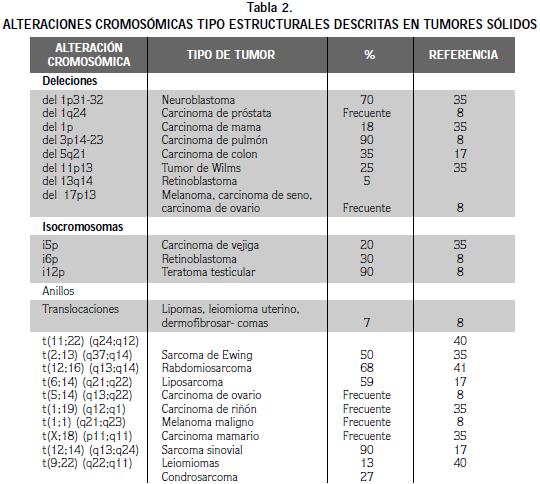
Las alteraciones cromosómicas estructurales representan el mayor porcentaje de anomalías observadas en tumores. En la tabla 2 se indican las más comunes. Las deleciones, pérdida de un segmento cromosómico (terminal o intersticial) pueden comprometer solamente una o pocas bandas o un segmento cromosómico de mayor tamaño (8,13,32). Los isocromosomas de brazos largos o cortos también son frecuentes en varios tipos de tumores (31). Igualmente, las deleciones que ocurren en ambos extremos de un cromosoma y la posterior fusión de los mismos (cromosomas en anillo) se presentan como alteraciones recurrentes en varios tumores (39) (Tabla 2).
Tanto en los tumores sólidos como en los blandos, los intercambios de material cromosómico (translocaciones balanceadas o no balanceadas) constituyen un grupo especial entre las alteraciones cromosómicas, que alcanza el mayor porcentaje del total de las alteraciones estructurales observadas. Además, un gran de número de ellas se consideran marcadores recurrentes en diversos tipos de tumores, por lo cual brindan gran ayuda para un diagnóstico preciso (8,17, 35, 39, 40, 41) (Tabla 2).
RELACIÓN ENTRE LAS ALTERACIONES CROMOSÓMICAS CON GENES SUPRESORES DE TUMORES Y PROTOONCOGENES
LOS CAMBIOS CROMOSÓMICOS y el posterior desarrollo de tumores tienen una relación directa con muchos protooncogenes, genes supresores de tumor y genes reguladores del ciclo celular (30,42,43). Las alteraciones cromosómicas antes descritas influyen notablemente en los mecanismos genéticos de tales genes; cuando se presenta una deleción, ésta puede contener genes supresores de tumores o protooncogenes, dando como resultado una haploinsuficiencia o desequilibrio en la expresión de estos genes; por esta vía se activan los mecanismos iniciales de la génesis tumoral. Ejemplos de lo anterior ocurren con la deleción intersticial 11p13 que contiene el gen WT1 supresor del tumor de Wilms; la deleción de la banda 13q14, en la cual se localiza el gen RB responsable del retinoblastoma en niños y la deleción 17q13 en la cual se localiza el gen PT53 relacionado con los carcinomas gástrico, mamario, melanoma, de pulmón, de ovario y de colon (8,23,25,35,42). Por lo tanto, el gen TP53 está bien reconocido como el guardián del genoma humano. Cabe anotar que las deleciones que comprometan los loci para los genes p53 y RB desencadenan la aparición de diferentes tipos de tumores en más del 50% de los casos (23,36,44).
Por otra parte, las translocaciones conducen a la transactivación génica anormal de los tipos de genes antes mencionados, como ocurre en los casos del L-myc (1p32) en carcinoma de pulmón, el N-ras (1p11) en neuroblastoma, APC (5q21) en poliposis del colon, BRCA1 (1p36 y 17q21) en carcinomas de mama y ovario (29,40,41,45) y la sobreexpresión del oncogen Her-2/neu en carcinoma de mama (46). También, las translocaciones afectan la estructura y función de otros genes, como aquéllos que codifican factores de crecimiento (22). De igual manera, originan la aparición de genes híbridos (genes fusionados) cuyos productos, proteínas quiméricas, aumentan notoriamente la proliferación celular y activan mecanismos de la génesis tumoral y sus posteriores metástasis (40,47). En síntesis, cualquier cambio en el número o estructura de los cromosomas humanos que comprometa el locus o loci de protooncogenes y/o genes supresores de tumores, activa los mecanismos para la aparición de neoplasias en humanos.
CITOGENÉTICA MOLECULAR
EN LA ACTUALIDAD SE HAN DESARROLLADO MÉTODOS muy especializados de citogenética molecular que hacen posible un rápido análisis de los tumores. Se destacan la hibridización in situ por fluorescencia (FISH) y sus variantes, la hibridización genómica comparativa (CGH), el cariotipo espectral (SKY-FISH) y el multiplex FISH (M-FISH) (48-53); son técnicas más precisas y confiables para detectar en forma rápida alteraciones cromosómicas numéricas y estructurales en núcleos interfásicos. Muchas de estas alteraciones no pueden definirse por la citogenética convencional, cuando ésta, en algunos casos, da resultados negativos o confusos. Por lo tanto, las técnicas mencionadas tienen la gran ventaja de identificar translocaciones crípticas y reordenamientos cromosómicos complejos, y definir la procedencia de segmentos cromosómicos involucrados en un gran número de reordenamientos, así como para el análisis de la actividad de genes supresores de tumores y oncogenes en núcleos interfásicos de tejidos tumorales (54-57).
Con los avances en las técnicas de citogenética molecular ha sido posible clonar genes localizados en numerosos sitios de ruptura en diferentes tipos de translocaciones cromosómicas presentes en neoplasias (40). Además, empleando técnicas de biología molecular se han identificado más de 100 genes fusionados (genes híbridos), como resultado de translocaciones, en trastornos hematológicos y en una variedad de tumores sólidos (58).
Otros estudios más específicos que se efectúan en los tumores son los análisis de ADN utilizando PCRSSCP y secuenciamiento, lo cual permite identificar mutaciones puntuales en genes asociados con el cáncer (TP 53,RB, Myc, entre muchos otros) (59,60). Una de las mayores ventajas que presentan estas técnicas moleculares es la posibilidad de hacer análisis retrospectivos en muestras incluidas en parafina, fijadas en formol o almacenadas a -70° C. Por lo tanto, las técnicas descritas se consideran de gran ayuda para el diagnóstico molecular, la clasificación, el pronóstico y la monitorización de diferentes tumores.
Finalmente, en la actualidad los estudios citogenéticos de tejidos neoplásicos cobran un gran interés por sus grandes aportes al Cancer Genome Anatomy Project (CGAP) (61), un proyecto colaborativo internacional creado en 1997 por el Instituto Nacional de Cáncer de Estados Unidos, dirigido a completar el catálogo de cambios genéticos relacionados con el cáncer. Este ambicioso proyecto tiene como misión descifrar la anatomía molecular del cáncer, en cuanto a los cambios genéticos que ocurren durante su formación y progresión. Dentro de este proyecto también se incluye el Cancer Chromosome Aberration Project. En ambos proyectos se están empleando técnicas muy refinadas para acelerar los estudios que permitan un mejor entendimiento de los mecanismos moleculares del cáncer. Información sobre estos proyectos es de libre acceso al público en internet (62).
Como conclusión, debe tenerse en cuenta la importancia de los estudios cromosómicos en cualquier tipo de neoplasia (leucemia, linfoma, tumores sólidos y blandos) con el fin de obtener una mejor información acerca del espectro de alteraciones y la identificación de anomalías cromosómicas recurrentes específicas durante el proceso de transformación maligna. Además, la posibilidad de correlacionar estos estudios con los hallazgos histopatológicos, puesto que se tendría una información complementaria que ayudaría en el diagnóstico y pronóstico de los pacientes con diferentes tumores. Por otra parte, estimular investigaciones multidisciplinarias en este campo que aporten nuevos conocimientos acerca de la biología del cáncer.
BIBLIOGRAFÍA
1. Sandberg AA, Yamada K. Chromosomes and cancer. CA 1965; 15: 58-74. [ Links ]
2. Yunis JJ. The chromosomal basis of human neoplasia. Science 1983; 221: 227- 236. [ Links ]
3. Hansemann D. Ueber asymmetrische Zelltheilung in Epithelkrebsen und deren biologische Bedeuntung. Virchows Arch Anat 1890; 119: 299-326. [ Links ]
4. Boveri T. Zur frage der Entstehung maligner Tumoren. Jena: Gustar Fischer; 1914: 1-64. [ Links ]
5. Nowell PC, Hungerford DA. A minute chromosome in human chronic granulocytic leukemia. Science 1960; 132: 1.497-1.498. [ Links ]
6. Levan A. Non-random representation of chromosome types in human tumor stemlines. Hereditas 1966; 55: 28-38. [ Links ]
7. Van Steenis H. Chromosome and cancer. Nature 1966; 209: 819-821. [ Links ]
8. Heim S, Mitelman F. Cancer Cytogenetics, 2ª ed. New York: Alan R. Liss. Inc.1995; 60: 315-319. [ Links ]
9. Caspersson T, Zech L, Johansson C. Differential binding of alkylating fluorochromes in human chromosomes. Exp Cell Res 1970; 60: 315-319. [ Links ]
10. Rowley JD. A new consistent chromosomal abnormality in chronic myelogenous leukemia identified by quinacrine fluorescence and Giemsa staining. Nature 1973; 234: 290-293. [ Links ]
11. Yunis JJ. New chromosome techniques in the study of human neoplasia. Hum Pathol 1981; 12: 540-549. [ Links ]
12. Gibas L, Gibas Z, Sandberg AA. Technical aspects of cytogenetic analysis of human solid tumors. Karyogram 1984; 10: 25-27. [ Links ]
13. Steinarsdóttir M, Pétursdóttir I, Snorradóttir S, Eyfjord E, Ogmundsdóttir HM. Cytogenetic studies of breast carcinomas: Different karyotypic profiles deteced by direct harvesting and short-term culture. Genes Chromosom Cancer 1995; 13: 239-248. [ Links ]
14. Miltelman F, Levan G. Clustering of aberrations to specific chromosomes in human neoplams. IV. A survey of 1,871 cases. Hereditas 1981; 95:79-139. [ Links ]
15. Sandberg AA, Turc-Carel C. The cytogenetics of solid tumors. Relations to diagnosis, classification and pathology. Cancer 1987; 59: 387-395. [ Links ]
16. Del Cin P, Van Den Berghe H. Ten years of the cytogenetics of soft tissue tumors. Cancer Genet Cytogenet 1997; 95: 59-66. [ Links ]
17. Sreekantaiah C. The cytogenetic and molecular characterization of benign and malignant soft tissue tumors. Cytogenet Cell Genet 1998; 82: 13-29. [ Links ]
18. Classman AB. Cytogenetics. An evolving role in the diagnosis and treatment of cancer. Clin Lab Med 1997;17: 21-37. [ Links ]
19. Mitelman F, Johanssen B, Mandani N, Mertens F. Clinical significance of cytogenetic findings in solid tumors. Cancer Genet Cytogenet 1997; 95: 1-8. [ Links ]
20. Dellarco V, Voytek P, Hollander A. Aneuploidy: Etiology and mechanisms. New York: Plenum; 1985. [ Links ]
21. Sandberg AA. The Chromosome in Human Cancer and Leukemia. 2ª ed. New York; Elsevier; 1990. [ Links ]
22. Shaeknev SE, Shaeknev IV. Common patterns of genetic evolution in human solid tumors. Cytometry 1997; 29: 1-27. [ Links ]
23. Bertram JS. The molecular biology of cancer. Molecular aspects of Medicine 2000; 6: 167-223. [ Links ]
24. Dal Cin P, Vanni R, Pelite P, Van den Berghe H. Diagnostic impact of cytogenetics for solid tumors. Pathologica 1998; 90: 337-342. [ Links ]
25. Verma RS, Babu A. Human Chromosome. Manual of BasicTechniques. 2ª ed. New York: Oxford; 1995: 28-30. [ Links ]
26. Gisselsson D, Jonson T, Perterson A, Strömbeck B, Dal Cin P, Höglund M, et al. Telomere dysfunction triggers extensive DNA fragmentation and evolution of complex chromosome abnormalities in human malignant tumors. Proc Natl Acad Sci 2001; 98: 12.683-12.688. [ Links ]
27. Mitelman F. Catalog of Chromosome Aberrations in Cancer. 4ª ed. New York: Wiley-Liss. 1987. [ Links ]
28. Mitelman F. Catalog of Chromosome Aberrations in Cancer. '98. CD-ROM. Version 1, New Yok; Wiley-Liss; 1998. [ Links ]
29. Mitelman F. Clustering of breakpoints to specific chromosomal regions in human neoplasia. A survey of 5.345 cases. Hereditas 1986; 104: 113-119. [ Links ]
30. Knudson AG. Antioncogenes and human cancer. Proc Natl Acad Sci 1993; 90: 10.914-10.921. [ Links ]
31. Rooney DE, Czepulskoski BH. Human Cytogenetics: A Practical Approach. 2ª ed. New York: McGraw Hill; 1995: p 274. [ Links ]
32. Smoralek TA, Blough RS, Ulbrigh TM, Palmer CG, Heerema NA. Cytogenetic analyses of 85 testicular germ cell tumors: comparison of postchemotherapy and untreated tumors. Cancer Genet Cytogenet 1999; 108: 57-69. [ Links ]
33. Turc-Carel C, Pedeutour F, Durieux E. Characteristic chromosome abnormalities and karyotype profiles in soft tissue tumors. Currents topics in Pathology 1995; 89:73-94. [ Links ]
34. Gisselsson D, Pettersson L, Höglund M, Heidenblad M, Gorunova L, Wiegant J, et al. Chromosomal breakage-fusion-bridge events cause genetic intratumor heterogeneity. Proc Natl Acad Sci 2000; 97: 5.357-5.362. [ Links ]
35. Sreekantaiah C, Landanyi M, Rodriguez E, Chaganti SK. Chromosomal aberrations in soft tissue tumors. Relevance to diagnosis, classification, and molecular mechanisms. Am J Pathol 1994; 44: 1.121- 1.134. [ Links ]
36. Sandberg AA. Cancer cytogenetics for clinicians. Cancer J Clin 1994; 44 :136-159. [ Links ]
37. Dooner LR. Cytogenetics of tumors of soft tissue and bone. Implications for pathology. Cancer Genet Cytogenet 1994; 78: 115-126. [ Links ]
38. Quade BJ. Pathology, cytogenetics and molecular biology of uterine leiomyomas and others smooth muscle lesions. Current Opinion Oncol 1995; 7: 333-339. [ Links ]
39. Bhattaonanee MB, Armstrong DD, Vogel H, Coolev LD. Cytogenetic analysis of 120 primary pediatric brain tumors and literature review. Cancer Genet Cytogenet 1997; 97: 39-53. [ Links ]
40. Mitelman F. Recurrent chromosome aberrations in cancer. Mutat Res 2000; 462: 247-253. [ Links ]
41. Rabbitts TH. Chromosomal translocations in human cancer. Nature 1994; 72: 143-149. [ Links ]
42. Gunter C. The molecular genetics of cancer: Down the rabbit hole. Hum Mol Genet 2001; 10: 655-656. [ Links ]
43. Vogelstein B, Kinzler W. Achilles' heel of cancer? Nature 2001; 412: 865-866. [ Links ]
44. Hollstein M, Sidransky D, Vogelstein B, Harris C. p53 mutations in human cancers. Science 1991; 253: 49-53. [ Links ]
45. Scwab M. Oncogene amplification in solid tumors. Semin Cancer Biol 1999; 9: 303-318. [ Links ]
46. Ross JS , Fletcher JA. The HER2/neu oncogene in breast cancer: Prognostic factor, predictive factor and target for therapy. Oncologist 1998; 3: 237-252. [ Links ]
47. Aman P. Fusion genes in solid tumors. Semin Cancer Biol 1999; 9: 303-318. [ Links ]
48. Muñetón CM. Citogenética molecular del cáncer. Med & Lab 2000; 9: 11-26. [ Links ]
49. Virmani AK, Tonk VS, Gazdar AF. Comparison between fluorescence in situ hybridization and classical cytogenetics in human tumors. Anticancer Res 1998;18: 1.351-1.356. [ Links ]
50. Patel AS, Hawkins AL, Griffin CA. Cytogenetics and cancer. Curr Opin Oncol 2000; 12: 62-67. [ Links ]
51. Theillet C, Orsetti B, Rendon R, Manoir SD. Genomic profiling: from molecular cytogenetics to DNA arrays. Bull Cancer 2001; 88: 261-268. [ Links ]
52. Lichter P, Joos S, Bentz M, Lampel S. Comparative genomic hybridization: uses and limitations. Semin Hematol 2000; 37: 348-357. [ Links ]
53. Schrock E, Pasilla-Nash H. Spectral karyotyping and multicolor fluorescence in situ hybridization reveal new tumor-specific chromosomal aberrations. Semin Hematol 2000; 37: 334-347. [ Links ]
54. Galteland E, Holte H, Stokke T. C-MYC, Rb-1, TP53 and centromere 8 and 17 copy number in B-cell non-Hodgkin's lymphomas assessed by dual-color fluorescence in situ hybridization. Cytometry 1999; 38: 53-60. [ Links ]
55. Takahashi Y, Nagata T, Asai S, Shintaku K, Eguchi T, Ishiiy, et al. Detection of aberrations of 17p and p53 gene in gastrointestinal cancers by dual (two-color) fluorescence in situ hybridization and GeneChip p53 assay. Cancer Genet Cytogenet 2000; 121: 38-43. [ Links ]
56. Obara K, Yokoyama M, Asano G, Tanaka S. Evaluation of myc and chromosome 8 copy number in colorectal cancer using interphase cytogenetics. Int J Oncol 2001; 18: 233-239. [ Links ]
57. Lehr HA, Jacobs TW, Yaziji H, Schnitt S, Gown A. Quantitative evaluation of HER2/neu status in breast cancer by fluorescence in situ hybridization and by inmunohistochemistry with image analysis. Am J Clin Pathology 2000; 115; 1-12. [ Links ]
58. Mitelman F, Mertens F, Johansson B. Breakpoint map of recurrent cytogenetic aberrations in human neoplasia '99, NCI Cancer Genome Anatomy Project, http://www.ncbi.nlm.nih.gov/CGAP. [ Links ]
59. Furuwatari C, Yagi A, Yamagami O, Ishikawa M, Hidaka E, Ueno I, et al. A comprehensive system to explore p53 mutations. Am J Clin Pathol 1998; 110: 368-373. [ Links ]
60. Shou Y, Martelli M, Gabrea A, Qi Y, Brents L, Roschke A, et al. Diverse karyotypic abnormalities of the c-myc locus associated with c-myc dysregulation and tumor progression in multiple myeloma. Proc Natl Acad Sci 2000; 97: 228-233. [ Links ]
61. Riggins G, Strausberg L. Genome and genetic resources from the cancer genome anatomy proyect. Hum Mol Genet 2001; 10: 663-667. [ Links ]
62. http://cgap.nci.nih.gov/ [ Links ]