Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Iatreia
Print version ISSN 0121-0793
Iatreia vol.21 no.3 Medellín July/Sept. 2008
ARTÍCULO DE REVISIÓN
Células madre: generalidades, eventos biológicos y moleculares
Stem cells: general aspects, biological and molecular events
Paola Andrea Acevedo Toro1; Mónica María Cortés Márquez2
1.Docente de Hematología, Escuela de Microbiología. Integrante del grupo HEMO (Hematopatología molecular). Universidad de Antioquia, Medellín, Colombia. Candidata a magíster en Ciencias Básicas Biomédicas.
2.Bacterióloga y Laboratorista clínica, Universidad de Antioquia. Docente de Hematología. Escuela de Microbiología, Universidad de Antioquia, Medellín, Colombia. Candidata a magíster en Educación.
Dirección electrónica de contacto: micropao@hotmail.com
RESUMEN
Las autorrenovación y la diferenciación son características de las células madre que varían entre los diferentes tipos celulares según el tejido en el que se encuentren y el microambiente que las rodee. En ambos procesos intervienen inhibidores del ciclo celular, genes implicados en rearreglos cromosómicos, proteínas del desarrollo esencial y vías de señalización específicas. La autorrenovación está regulada por diversos mecanismos, entre los cuales se destacan las vías Wnt, Notch y Hedgehog, y los factores BMI–1, p16Ink4a, ARF, NANOG, OCT3/4, SOX2, HOXB4 y sus páralogos. Los adelantos en el conocimiento de la biología de las células madre y de los mecanismos moleculares que regulan la autorrenovación y la diferenciación han convertido a estas células en una importante promesa para la investigación básica y aplicada.
Palabras clave: Autorrenovación, Células madre, Diferenciación, Factores de transcripción HOXB4. Gen BMI–1, Gen NANOG, Genes OCT3/4, Genes p16Ink4a, Gen SOX2, Microambiente, Proteína ARF 1, Proteína Hedgehog, Proteínas Wnt, Receptores Notch
Summary
Self–renewal capacity and differentiation are features of stem cells that vary among the different cellular types according to the tissue in which they reside and the surrounding microenvironment. Cellular cycle inhibitors, genes implied in chromosomal rearrangements, essential development proteins and specific signaling pathways intervene in these processes. Self–renewal is regulated by different mechanisms, the most important of which are the Wnt, Notch and Hedgehog pathways, and the factors BMI–1, p16Ink4a, ARF, NANOG, OCT3/4, SOX2, HOXB4 and their paralogs. Advances in the knowledge of stem cells biology and of the molecular mechanisms that influence their selfrenewal and differentiation have made these cells an important promise for both basic and applied research.
Key words: ARF 1 protein, BMI–1 gene, Differentiation, Microenviroment, NANOG gene, Notch receptors, OCT3/4 genes, p16Ink4a genes, Self–renewal, SOX2 gene, Stem cells, Transcription factors.Wnt proteins.
DEFINICIÓN Y CARACTERÍSTICAS
El estudio de la biología de las células madre nació en 1916 cuando Danchakoff describió la presencia en la médula ósea de ciertas células precursoras de otras; posteriormente Maximow confirmó sus hallazgos.1 En general se define una célula madre, también denominada célula troncal, como la que tiene capacidad de autorreplicarse indefinidamente por división celular (autorrenovación) y que, cuando se encuentra bajo condiciones microambientales adecuadas, puede diferenciarse a otros tipos de células especializadas, no solo morfológica sino también funcionalmente; es así como una población pequeña de células madre puede en unos meses proliferar hasta dar origen a millones de ejemplares con las mismas características de sus predecesoras.2
En años recientes la transdiferenciación o plasticidad, característica especial de dichas células, las ha convertido en un recurso extraordinariamente prometedor tanto para la investigación básica como para las aplicaciones clínicas. El término plasticidad corresponde a la capacidad de estas células de generar grupos celulares diferentes de los de su tejido de origen; tal es el caso de las células madre hematopoyéticas (CMH) (hematopoietic stem cells, HSC) y mesenquimales, que bajo condiciones experimentales e incluso in vivo pueden diferenciarse hacia diversos linajes celulares: las CMH puedan dar lugar a células hepáticas, nerviosas y musculares, mientras que las células mesenquimales han demostrado, cuando se las expone a los estímulos apropiados, la capacidad de transdiferenciarse hacia osteoblastos y adipocitos.3,4
TIPOS DE CÉLULAS MADRE
Hay dos maneras de clasificar las células madre: 1) de acuerdo con su potencial de diferenciación en células totipotenciales, pluripotenciales y multipotenciales. 2) Según el tejido de origen, en células madre embrionarias y del adulto.
Son totipotenciales las células capaces de generar un embrión completo, es decir, de dar origen a tejidos embrionarios y extraembrionarios; tal es el caso de la fecundación, proceso por el cual se genera una célula, el zigoto, paradigma de célula totipotencial, que dará lugar a todas las células del embrión y posteriormente a las del adulto; por lo tanto, este tipo de células solo se puede obtener durante las primeras fases de formación del embrión, pues a medida que este evoluciona dichas células se especializan y transforman en otras, las pluripotenciales, capaces de generar cualquier linaje celular, pero no un embrión completo. Esta diferenciación resulta de cambios en la expresión génica; por ello las células extraídas y cultivadas entre los 7 y 14 días posfecundación, cuando el embrión se encuentra en la fase de blastocisto, nunca darán lugar a un embrión completo sino a estirpes celulares específicas determinadas por los genes que se expresen en el momento.5,6
Una vez que el embrión se implanta en el útero y continúa su desarrollo, sus células se van especializando cada vez más, transformándose en células madre comprometidas, las multipotenciales, con mayor grado de diferenciación puesto que llevan la marca de un tejido concreto, es decir, solo pueden dar lugar a tipos celulares del tejido al que pertenecen; un ejemplo de ellas son las CMH que dan origen a todas las células sanguíneas: glóbulos rojos, glóbulos blancos y plaquetas.3
De acuerdo con el origen, las células madre pueden proceder de tejido embrionario o de un organismo adulto; de ahí que se las denomine Células madre embrionarias (CME) (Embryonic stem cells, ESC) y Células madre adultas (CMA), respectivamente. Las primeras corresponden a precursores totipotenciales con capacidad de proliferar indefinidamente in vitro, por lo que gran parte de las investigaciones se han centrado en la producción de distintos tipos de tejidos e incluso de órganos simples, así como en la incorporación de genes foráneos, lo que convierte a este tipo de células en un vehículo idóneo para la terapia génica paliativa y en una firme promesa para la medicina futura.7
Las células madre adultas (CMA), por su parte, corresponden a progenitores multipotenciales, que pueden renovarse constantemente y dar lugar a células especializadas capaces de reparar lesiones tisulares y de renovar células seniles; así mismo presentan una gran versatilidad biológica, fundamentada en su capacidad de alterar drásticamente el fenotipo en respuesta a los cambios del microambiente en donde se desarrollan.7
CÉLULAS MADRE PRESENTES EN LA MÉDULA ÓSEA
La médula ósea ha demostrado ser la mejor fuente de células madre del organismo adulto; entre las más estudiadas se destacan las células madre hematopoyéticas (CMH) que se caracterizan por su capacidad de proliferación y diferenciación en progenitores hematopoyéticos comprometidos; se pueden hallar tanto en la médula ósea (1–3% de los mononucleares), como en la sangre del cordón umbilical (0,2–1%) y en la sangre periférica (0,001–0,025%).8 Cabe resaltar que la proporción de células madre entre los mononucleares del cordón umbilical disminuye con la edad gestacional, así: de 11% a las 17 semanas desciende a 1% a las 38 semanas; sin embargo, presentan una ventaja competitiva sobre las CMH de la médula ósea, pues se injertan 10 a 50 veces mejor en huéspedes xenógenos.
Los estudios sobre la ontogenia de las CMH indican que aparecen en el embrión entre la tercera y cuarta semanas de la gestación; luego migran a través de la circulación fetal, primero hacia el saco vitelino, luego al bazo y al hígado, para finalizar en el órgano hematopoyético por excelencia del adulto: la médula ósea.9
En la médula ósea se pueden reconocer dos tipos de CMH: en primer lugar las de largo plazo (CMH–LP) (long–term hematopoietic stem cells, LT–HSC), que participan en el mantenimiento del sistema hematopoyético durante toda la vida; así, en los modelos murinos, las CMH–LP cumplen un papel importante en el éxito del trasplante hematopoyético. El otro tipo de CMH son las denominadas de corto plazo (CMH–CP) (short term hematopoietic stem cells, STHSC), que originan los progenitores comprometidos en el proceso de la hematopoyesis; en conjunto, las CMH se han convertido en la base biológica de los trasplantes de médula ósea para las personas que padecen leucemia o aplasia medular; además, se usan terapéuticamente en pacientes con enfermedades no hematológicas como los infartos e isquemias del miocardio.10
Se reconocen en la médula ósea otros tipos de células madre, a saber: células madre mesenquimales, células progenitoras multipotenciales adultas (CPMA) (multipotent adult progenitor cells, MAPC) y las denominadas células madre SP (side population cells). Las primeras, también conocidas como células estromales, son capaces de diferenciarse a tejidos mesodérmicos funcionales, como osteoblastos, condroblastos, adipocitos y mioblastos; su aislamiento se basa en ciertos marcadores que han hecho posible identificarlas: SH2, SH3, CD29, CD44, CD71 y CD90; sin embargo, no expresan antígenos de superficie típicos de las CMH como CD34 y CD45.8 Por su parte, las CPMA poseen capacidades de proliferación y diferenciación similares a las de las células madre embrionarias, porque presentan gran actividad de la telomerasa durante el tiempo de cultivo, lo que les permite dividirse más de 120 veces sin envejecimiento aparente; incluso de manera similar a las células del embrión se ha detectado activación de los factores de transcripción Oct–4 Nanog y Rex–1, que son necesarios para mantener el estado indiferenciado y proliferativo de la célula. A diferencia de la mayoría de las células madre, las CPMA no expresan el CD34, pero sí, aunque en niveles muy bajos, Flk–1, Sca–1, Thy–1, y en niveles elevados marcadores como CD13, SSEA–1 (ratón/rata) y SSEA–4 (humano).11
Por último, se encuentran las células madre SP observadas por primera vez en la médula ósea del ratón utilizando un nuevo método para identificar CMH, basado en el análisis por citometría de flujo de doble longitud de onda, utilizando el colorante Hoechst que emite fluorescencia azul a 450 nm y un color rojo a 650 nm; mediante esta técnica dual se reconoció un pequeño subgrupo de células (menos del 0,1%) que revelaban fluorescencias roja y azul bajas; posteriormente se demostró que estas células expresaban el Sca–1, antígeno encontrado en las CMH, pero no se teñían con el cóctel de anticuerpos dirigidos contra otros marcadores encontrados en las CMH maduras, lo cual demostró una gran actividad de repoblación hematopoyética a largo plazo en la médula ósea del ratón. Hasta el momento se sabe que estas células son capaces de diferenciarse en CMH tanto en humanos como en roedores; este es un campo poco explorado pero promisorio para diversas aplicaciones terapéuticas.12,13
EL MICROAMBIENTE MEDULAR
El desarrollo de las células madre está determinado por un espacio específico en la médula, denominado 'nicho', en el cual confluyen elementos del microambiente como sustancias químicas, entre ellas hormonas, y diversos tipos celulares (del endotelio, adipocitos, linfocitos T, macrófagos y fibroblastos), que intervienen en el proceso de diferenciación celular y ofrecen a las células el soporte físico y el punto de adherencia necesarios para sobrevivir. En el caso de las CMH, la interacción con el microambiente incluye los factores de crecimiento y la matriz extracelular; se ha considerado que el control derivado de este ambiente medular es de mayor importancia para el compartimiento de las células madre que para el de las células más diferenciadas.14,15 El cultivo de células madre in vitro ha sido posible gracias a la reproducción del microambiente; el uso de una capa de soporte de fibroblastos mitóticamente inactivos permite mantener líneas celulares indiferenciadas para propósitos de investigación; sin embargo, ocurre con facilidad la diferenciación espontánea de estos linajes,15 situación que plantea la necesidad de conocer los mecanismos bioquímicos y genéticos reguladores de los procesos de autorrenovación y diferenciación en ese compartimiento específico; en esa forma se daría respuesta a los interrogantes que rodean a las células madre en relación con los eventos que las definen en términos moleculares o en cuanto a las señales que controlan su diferenciación y reprogramación.
AUTORRENOVACIÓN Y DIFERENCIACIÓN
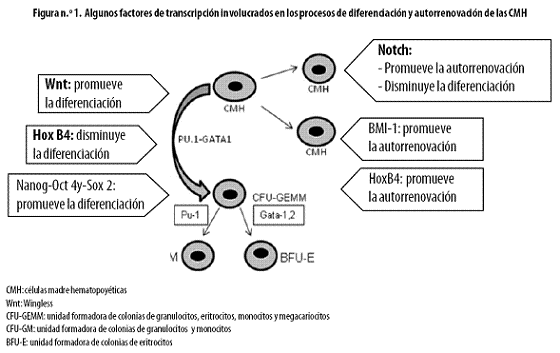
Las capacidades de autorrenovación y diferenciación son propias de los diferentes tipos de células madre, según el microambiente y el tejido en que se encuentren. Diversas investigaciones han tratado de hallar la razón por la que estas células siguen uno u otro camino, pero aún quedan muchas preguntas sin resolver. Recientemente se ha podido avanzar en el entendimiento de la biología básica de los mecanismos de autorrenovación, diferenciación y proliferación celulares, así como en dilucidar las diferentes vías de señalización que participan en estos eventos. (Figura n.º 1).16–19
En términos generales los diferentes nichos pueden modificar sus propiedades reguladoras en respuesta a las necesidades particulares del tejido; sin embargo, independientemente del nicho en cuestión, hay mecanismos comunes de señalización que es muy importante conocer y que están mejor caracterizados en el sistema hematopoyético; pero no ha sido sencillo el estudio de los mecanismos moleculares que controlan la hematopoyesis, porque no se puede mantener a las CMH in vitro por largos períodos; además, se presenta la dificultad de controlar la autorrenovación frente a la diferenciación.20,21
Específicamente en el proceso de autorrenovación intervienen inhibidores del ciclo celular, genes implicados en rearreglos cromosómicos, proteínas esenciales para el desarrollo y factores específicos como el Notch 1, Shh (Sonic hedgehog), el factor de transcripción Hox B4, el inhibidor de la quinasa dependiente de la ciclina P21/waf 1, las proteínas Wnt, entre otros. Para mantener con éxito el estado indiferenciado se requiere la integración de diferentes vías de señalización intrínsecas con las señales extrínsecas emitidas desde el microambiente. Es fundamental comprender los mecanismos que regulan el estado de indiferenciación, por su importancia para entender la biología de las células madre y el desarrollo del cáncer.22–24
VÍA WINGLESS (Wnt)
Esta vía es una cascada de señales que dirige los eventos proliferativos y de diferenciación en el desarrollo embrionario y en el adulto. Sus proteínas son hidrofóbicas, debido al palmilato unido a la cisteína en la posición C77 y que es vital para su funcionamiento. Esta amplia familia de proteínas ha sido estudiada especialmente en modelos murinos: en la médula ósea de ratones se ha descrito la expresión de Wnt 2B, Wnt 3A y Wnt 10B. El factor Wnt 5A, además de ejercer su actividad en la médula, cumple un papel preferencial en el mantenimiento de las células del estroma y, junto con el Wnt 10B, en células de hígado fetal. Todos estos factores de transcripción propios de las células madre interactúan con los producidos por las células del nicho con el fin de encaminar a las primeras hacia la autorrenovación o la diferenciación.25
En general las proteínas Wnt, secretadas al medio extracelular, actúan como ligandos que se unen a receptores específicos de la membrana celular de las células productoras y de células adyacentes. Existe una variedad de receptores que pueden interactuar con estos ligandos, como los pertenecientes a la familia Frizzled (Fz) y la Proteína relacionada con el receptor de lipoproteína de baja densidad (low density lipoprotein receptor protein, LRP); cuando el ligando se une a LRP puede formar un complejo tetramérico con Fz. De igual manera, las proteínas relacionadas con Frizzled (FRP) pueden antagonizar la acción de las Wnt, porque se unen directamente a ellas y bloquean su acción.26
Aunque se han propuesto diversas vías de señalización intracelular para Wnt, la mayoría de ellas se han estudiado principalmente en el ámbito hematopoyético, donde tienen un papel importante en la producción celular y del estroma medular. Se ha estudiado la capacidad de ratones irradiados letalmente para reconstituir este sistema; la presencia del factor Wnt 5A, junto con el estroma y las CMH murinas, promueve la expansión de progenitores hematopoyéticos indiferenciados, y también participa indirectamente en la regulación del microambiente, porque influye en la producción de osteoblastos que a su vez son importantes reguladores del nicho de estas células.16
Los estudios de los mecanismos de autorrenovación de las CMH están restringidos principalmente a la vía de la β–catenina, cuyos eventos finales son la translocación nuclear y la unión física de la β–catenina para activar el factor de transcripción TCF/LEF (Tcell–specific transcription factor/lymphoid enhancer binding factor–1) y prevenir la diferenciación celular, colaborando así en el mantenimiento de la pluripotencialidad.27
La β–catenina actúa en el núcleo como un coactivador transcripcional de la familia TCF/LEF, reemplazando a los correpresores de la familia del gen Groucho (GRG), y recluta proteínas como CBP (Ciclic AMP binding protein), BRG1 (gen relacionado con brama–1) y p300, que se unen a regiones promotoras presentes en los genes que intervienen en la proliferación celular para activarlos. Así mismo, cuando esta vía no se activa, la β–catenina en baja concentración se une a correpresores transcripcionales del mismo complejo TCF/LEF para reprimir los genes blanco de Wnt. Todo este complejo mecanismo conduce a inducir la proliferación de células progenitoras inmaduras, incluyendo las CMH.26,28,29
Esta vía también presenta una retroalimentación negativa que desestabiliza la β–catenina por unión de los productos del gen supresor de tumor axina y del gen APC (Adenomatous polyposis coli), después de lo cual, la quinasa caseína 1 (CK1) y la quinasa glicógeno sintasa–3B axina (GSK3B) fosforilan secuencialmente la β–catenina en el extremo aminoterminal; específicamente, la CK1 fosforila en el aminoácido Ser 45, dando paso a GSK3B para fosforilar en otros 3 aminoácidos (Thr 41, Ser 37 y Ser 33) y finalmente dar lugar al complejo E3–ubiquitinaligasa que marca la β–catenina para que sea degradada por el proteosoma. La translocación nuclear es un suceso contrario a los anteriores pasos, en el cual se transfiere la proteína citoplasmática Dishevelled (DVL) a la membrana celular cuando los ligandos Wnt se unen con su receptor; este paso lleva a la disociación del complejo GSK3B y axina evitando la fosforilación de la β–catenina y su posterior degradación; de esta manera se acumula la β–catenina y es translocada al núcleo.30,31 (Figura n.º 2)
Esta cascada de señalización también cumple un papel importante durante el desarrollo embrionario, la morfogénesis y la migración, proliferación y diferenciación celulares. Estudios recientes la implican además en la proliferación de las CMH en células madre embrionarias y neuronales; incluso se ha demostrado que el tratamiento in vitro de células madre epidérmicas con la proteína Wnt3a estimula la proliferación e inhibe la diferenciación celulares. 32–34
VÍA NOTCH
Esta vía la conforman principalmente cuatro proteínas llamadas Notch 1, 2, 3 y 4, que interactúan en vertebrados con diversos ligandos (Delta, Delta like, Jagged 1 y Jagged 2).35 Estructuralmente las proteínas Notch son del tipo transmembrana que poseen un dominio extracelular encargado de la unión al ligando y la activación de la vía; estos receptores requieren una serie de clivajes por parte de los miembros de la familia de proteasas ADAM (A disintegrin and metalloproteinase protein), así como un clivaje transmembrana llevado a cabo por la β–secretasa o presenilina que da lugar a la translocación nuclear del dominio intracelular de Notch (DICN). El complejo ligando–receptor favorece la liberación del DICN, que contiene señales de localización nuclear y secuencias OPA (opacity associated adhesin proteins) ricas en glutamina que funcionan como activadores de la transcripción. 36–39 Una vez que se presenta la liberación del DICN se activa la molécula CSL (abreviatura de Chisel) para mediar la transducción de señales hacia el núcleo y el complejo se une a secuencias promotoras en el ADN para regular la expresión de algunos genes blanco como en el caso del gen HES (Hairy/enhancer of split) que regula negativamente la expresión génica específica de linaje.37
In vitro se ha demostrado que la sobreexpresión de Notch 4 inhibe la diferenciación de células epiteliales normales de la glándula mamaria; in vivo, esta misma proteína cumple una función importante en el desarrollo y la carcinogénesis mamarios.40 Otros estudios sustentan el papel de las proteínas Notch en la autorrenovación de las CMH; usando genes Notch reporteros se demostró que esta vía se encuentra activa en dichas células madre y que al irse diferenciando las células hematopoyéticas la expresión del gen reportero presenta una baja regulación y solo una pequeña fracción de las células maduras muestra actividad reportera in vivo; lo anterior sugiere la importancia de esta vía para el mantenimiento del estado indiferenciado de las CMH.41 Puede concluirse que en cualquier tipo de célula madre la activación de Notch conduce a la supresión transcripcional de los genes específicos de linaje, de tal manera que inhibe la diferenciación y favorece la autorrenovación.42–44
VIA HEDGEHOG
Esta vía de señalización fue identificada primero en Drosophila y hasta el momento se sabe que tres de sus ligandos son relevantes en mamíferos: Sonic hedgehog (Shh), Desert hedgehog (Dhh) e Indian hedgehog (Ihh), los cuales se unen a la proteína de interacción Hedgehog 1 (Hip1) y a la proteína Patched (Ptch), receptores transmembrana importantes para la activación de la cascada.45
En ausencia del ligando, Ptch se une a la proteína Smo (Smoothened) e inhibe su función; una vez que se presenta la interacción ligando–receptor, Smo puede activar la transcripción de los factores Gli 1, Gli 2 y Gli 3 que posteriormente se translocan al núcleo para controlar la transcripción de genes blanco importantes en el control de la proliferación celular, tales como los que codifican para las ciclinas D y E y para la proteína Myc, así como también componentes de la vía del factor de crecimiento epidérmico y la angiogénesis, como es el caso del Factor de crecimiento derivado de plaquetas y el Factor de crecimiento endotelial vascular.46
La presencia del ligando también actúa en el citoplasma, en donde las quinasas transfieren grupos fosfato a regiones ricas en serina y treonina de la proteína quinasa Fused (Fu), el supresor de Fused (Su–Fu) y la proteína Costal 2 (Cos 2). A su vez, estas proteínas forman un complejo de alto peso molecular
anclado a los microtúbulos del citoesqueleto. En ausencia del ligando, la proteína quinasa A (PKA) fosforila al factor de transcripción Ci (Ci155) que posteriormente es destruido por el proteosoma; este paso permite la liberación de un péptido presente en la región amino–terminal del mismo factor (Ci75) que funciona como represor de la transcripción. Todo lo contrario sucede en presencia del ligando: el complejo citoplasmático se disocia de su anclaje (los microtúbulos) y Ci155 se transloca al núcleo para activar la expresión de genes como ptc, dpp (Decapentaplegic), wg (Wingless), produciendo una respuesta final de inhibición de la vía; de esta manera presenta una retroalimentación negativa.28,47
Estudios recientes han demostrado que la vía Hedgehog es necesaria para el mantenimiento de las células madre hematopoyéticas y neuronales; además, dicha vía promueve la proliferación y supervivencia de estas células in vivo. En Drosophila esta cascada favorece la autorrenovación de las células madre de ovario, aumentando su proliferación.48–50
La importancia de esta vía radica también en el desarrollo embrionario y la morfogénesis; la ausencia de Ptch1 y Gli 2 produce defectos durante la etapa embrionaria e hiperplasia mamaria en los seres humanos; el uso de Shh recombinante estimula la formación de tejido mamario, que puede ser inhibido a su vez por acción de la ciclopamina, un inhibidor de Smo; estos estudios sugieren el papel de Hedgehog en la autorrenovación de las células madre de mama.51–53
Así mismo, la señalización ejercida por esta vía está implicada en la carcinogénesis: la presencia de mutaciones en algunos oncogenes que intervienen en la cascada, tales como Smo, Shh, Gli 1 y Gli 2, se ha asociado con el desarrollo de algunos tipos de cáncer entre ellos el meduloblastoma, carcinomas basales de la piel y cáncer de mama.54–58
OTROS FACTORES INVOLUCRADOS EN EL PROCESO DE AUTORRENOVACIÓN BMI–1, p16 Ink4a y ARF
El factor BMI–1 regula la expresión de p16Ink4a y ARF, genes supresores de tumores identificados como biomarcadores del envejecimiento celular; la expresión anormal de ambos se asocia con interferencia de la autorrenovación en CMH murinas, puesto que detiene la proliferación y lleva a la apoptosis; sin embargo, el papel de estos dos genes en el control del ciclo en las células madre parece depender de su tipo. En general, actúan como reguladores de las proteínas retinoblastoma (Rb) y p53, respectivamente, controlando la diferenciación, senescencia y supervivencia.
Estudios recientes han demostrado que mediante retroalimentación negativa este factor regula la expresión de p16 Ink4a y ARF que se encuentran sobrerregulados en CMH y células madre neuronales (CMN) de ratones deficientes en BMI–1.59,60
BMI–1 hace parte del grupo Polycombo (PcG) de modificadores epigenéticos de la cromatina; se lo identificó originalmente como un protooncogén necesario para el mantenimiento y control de la diferenciación de las CMH murinas. Los genes PcG intervienen en el silenciamiento de otros genes y ejercen su función formando complejos proteicos multiméricos con actividad enzimática; la identificación de diversas histonas deacetilasas y metilasas en dichos complejos sugiere que el PcG genera cambios epigenéticos que contribuyen al silenciamiento de genes involucrados en la diferenciación celular, de tal manera que su función es favorecer el proceso de autorrenovación.61
La deficiencia de BMI–1 resulta en una pérdida progresiva de CMH y en defectos en las células madre neuronales (CMN) (Neuronal stem cells, NSC), puesto que también se ha demostrado que es necesario para llevar a cabo eficientemente la autorrenovación de dichas células en los sistemas nerviosos central y periférico. Los estudios de repoblación competitiva muestran que diez semanas después del trasplante de médula ósea en ratones Bmi–1 –/–, se presenta una disminución de las CMH, debido a su incapacidad para autorrenovarse.62 También se ha demostrado que se requiere BMI–1 para la proliferación de las células madre leucémicas y su desregulación se ha asociado con la aparición de ciertos tipos de cáncer en humanos: meduloblastoma, neuroblastoma y linfoma difuso de células B, lo cual abre la posibilidad de estudiar blancos terapéuticos dirigidos a controlar la actividad de este factor.63,64
HOXB4 y proteínas parálogas
Los genes Hox codifican para factores de transcripción que regulan la embriogénesis y la hematopoyesis. Esta gran familia de proteínas está compuesta por 39 miembros que poseen una secuencia de 60 aminoácidos altamente conservada que actúa como un dominio de unión al ADN.65,66.
De todos los genes Hox, el factor de transcripción HoxB4 fue el primero en ser asociado con el mecanismo de autorrenovación; es así como la sobreexpresión de este factor en la médula ósea se relaciona con expansión de las CMH, in vivo e in vitro, lo cual ha demostrado su función en la autorrenovación de dichas células; además solo se encuentra una alta expresión en las células hematopoyéticas primitivas que declina al ocurrir la diferenciación específica de linaje.67–69 De manera similar Daga y colaboradores mostraron que las células CD34 positivas expresan el factor de transcripción HoxC4 y que su sobreexpresión induce la proliferación de progenitores multilinaje; los datos reportados hasta el momento indican que la combinación de HoxB4 y HoxC4 se asocia con la expansión de las CMH.70
La activación de HoxB4 está a cargo del factor de transcripción nuclear trimérico Y (NF–Y) en cooperación con el factor USF1/2. Los estudios realizados en ratones deficientes en HoxB4 muestran un desarrollo hematopóyetico normal; estos resultados aportan evidencia de que los parálogos de los genes Hox4 son capaces de compensar la pérdida de función del HoxB4, pues poseen una estructura proteica idéntica a la de este factor y además contienen secuencias HxRE1 y HxRE2, que son sitios de unión para NF–Y y USF1/2.71
NANOG, OCT3/4 Y SOX2
Nanog es un factor de transcripción que contiene un dominio homeobox y su actividad es esencial para el mantenimiento in vitro e in vivo de las CMH y de las células madre embrionarias (CME); además, durante la diferenciación se presenta una baja regulación de este factor cuya ausencia en ratones resulta en una diferenciación primitiva del endodermo. 72–74
En su conjunto los factores Nanog, Oct3/4 y SOX2 regulan la autorrenovación y la pluripotencia especialmente en las CME; sin embargo, hasta el presente no se han dilucidado por completo los mecanismos de acción de estos factores ni cómo se controla su expresión; algunos informes sugieren la intervención de Oct3/4 y SOX2 como reguladores de Nanog, pues poseen sitios de unión a la región promotora de este factor; sin embargo, el gen Nanog no es el único regulado por el complejo Oct–Sox, sino que se han identificado sitios de unión para estos dos factores en las regiones promotoras de los genes Fgf–4, Utf–1 y Fbx–15.75–79
Oct 3/4 regula los genes 'corriente abajo' mediante la unión a secuencias de repetición AGTCAAAT presentes en las regiones promotoras de algunos genes; este factor actúa junto con SOX 2, un miembro de la familia SOX de los factores de transcripción HMG box; ambos factores ejercen un papel esencial en la autorrenovación y presentan una alta expresión en la mayoría de las líneas de células madre embrionarias.80–82 El incremento en la expresión de Oct 3/4 promueve la formación de mesodermo y endodermo y la baja regulación resulta en diferenciación hacia trofoectodermo; por su parte, la pérdida de SOX2 también contribuye al desarrollo de endodermo extraembrionario.83 Oct 3/4, al igual que otros factores que intervienen en el proceso de autorrenovación, se encuentra implicado en la tumorogénesis, específicamente en las células germinales adultas; la expresión anómala de este factor en ratones adultos conduce a la aparición de lesiones displásicas en la piel y el intestino. Estos hallazgos abren un nuevo panorama a la comunidad científica y el esclarecimiento de estos mecanismos de regulación en el proceso de autorrenovación se ha convertido en un punto clave para entender la carcinogénesis y sus alternativas terapéuticas.84
DIFERENCIACIÓN
Al igual que las propiedades de autorrenovación, se ha estudiado ampliamente el proceso de diferenciación de las células madre en especial mediante ensayos con células madre hematopoyéticas. El proceso de diferenciación está regulado por un conjunto de factores transcripcionales; la señalización ejercida por estos factores activa una serie de genes específicos directores de un linaje determinado; el mejor ejemplo de este mecanismo son las CMH, en las que se destacan dos categorías: la primera incluye el factor de transcripción de la célula madre de leucemia (stem cell leukemia), el factor de transcripción GATA–2 y el factor–1 de transcripción de leucemia mieloide aguda (AML–1) que influyen directamente en la diferenciación de todos los linajes hematopoyéticos; la segunda comprende los reguladores del desarrollo específico de linaje como el GATA–1 (por la secuencia guanina–adenina–timina–adenina) y el PU–1; sin embargo, la diferenciación no es exitosa si no intervienen los factores estimulantes y las citoquinas que pueden variar dependiendo del linaje (eritroide, mieloide, linfoide, monocítico o megacariopoyético).85,86
Se han propuesto varias teorías para explicar el proceso de diferenciación de las células madre; entre ellas está su irreversibilidad, basada en análisis de redes aleatorias (random networks); también se ha descrito la diferenciación celular como un fenómeno adaptativo, cuyo modelo involucra cambios en la expresión génica de estas células.19 Estos aportes han sido de gran utilidad para investigaciones posteriores que se han enfocado en las vías genéticas implicadas en el proceso de diferenciación: los factores GATA 1 y PU–1 han sido considerados mediadores importantes en la diferenciación de las células madre hematopoyéticas; así mismo, se ha considerado que la relación dinámica entre los genes OCT4, SOX2 y Nanog es de gran relevancia en el proceso de diferenciación de las células madre embrionarias. En el caso específico de la diferenciación de las células madre hematopoyéticas, el microambiente medular y las vías de señalización celular juegan un papel relevante; se han hecho numerosos ensayos in vitro para estudiar el papel de las interleuquinas (IL) en este proceso, en particular la IL–3, la IL–6 y la IL–11.87,88
En conclusión, el conocimiento generado a partir del comportamiento biológico y las vías implicadas en los procesos de autorrenovación y diferenciación de las células madre, permite la apertura de un panorama alentador en la investigación básica y aplicada; así mismo, las propiedades exclusivas de estas células las hacen ver como muy promisorias en la terapia de diferentes enfermedades; sin embargo, la mayoría de los resultados obtenidos bajo condiciones experimentales in vitro todavía no han sido confirmados in vivo; por otra parte, es importante resaltar que algunos de los mecanismos moleculares estudiados requieren mayor profundización en el campo experimental.
REFERENCIAS BIBLIOGRÁFICAS
1. Maximow AA. Relation of blood cells to connective tissues and endothelium. Physiol Rev 1924; 4: 533–563. [ Links ]
2. Jesper B, David AH, Jan AN. Recent advances in hematopoietic stem cell biology. Curr Opin Hematol 2004; 11: 392–398. [ Links ]
3. Piscaglia AC, Zocco MA, Di Campli C, Sparano L, Rutella S, Monego G, et al. How does human stem cell therapy influence gene expression after liver injury. Microarray evaluation on a rat model. Dig Liver Dis 2005; 37: 952–963. [ Links ]
4. Preston SL, Alison M, Forbes S, Direkze C, Poulsom R, Wright N. The new stem cell biology: something for everyone. J Clin Pathol Mol Pathol 2003; 96: 56–86. [ Links ]
5. Rodríguez V. Células madre: conceptos generales y perspectivas de investigación. Universitas Scientiarum 2005; 10: 5–14. [ Links ]
6. Giraldo J, Madero J, Ávila M, Cuneo S, López C, Escobar M, et al. Células madre. Rev Col Obstetr Ginecol 2003; 54: 87–96. [ Links ]
7. Sanchez JM. Terapia celular en enfermedades cardiovasculares y renales. Iatreia 2006; 19: 164 –171. [ Links ]
8. Rodríguez VM, Cuéllar A, Cuspoca LM, Contreras CL, Mercado M, Gómez A. Determinación fenotípica de subpoblaciones de células madre derivadas de sangre de cordón umbilical. Biomédica 2006; 26: 51–60. [ Links ]
9. Holyoake TL, Nicolini FE, Eaves CJ. Functional differences between transplantable human hematopoietic stem cells from fetal liver, cord blood, and adult marrow. Exp Hematol 1999; 27: 1418–1427. [ Links ]
10. Lapidos K, Chen Y, Earley J, Heydemann A, Huber J, Chien M, et al. Transplanted hematopoietic stem cells demonstrate impaired sarcoglycan expression after engraftment into cardiac and skeletal muscle. J Clin Invest 2004; 114: 1577–1585. [ Links ]
11. Jiang Y, Vaessen B, Lenvik T, Blackstad M, Reyes M, Verfaillie CM. Multipotent progenitor cells can be isolated from postnatal murine bone marrow, muscle, and brain. Exp Hematol 2002; 30: 896–904. [ Links ]
12. Prosper F, Verfaillie CM. Células madre adultas. An Sist Sanit Navar 2003; 26: 345–356. [ Links ]
13. Eaker S, Hawley T, Ramezani A, Hawley R. Detection and enrichment of hematopoietic stem cells by side population phenotype. Methods Mol Biol: Flow Cytometry Protocols. 2a ed. Totowa, NJ: Humana Press; 2002; pp. 161–180. [ Links ]
14. Florensa L. Hematopoyesis, morfología de los elementos formes de la sangre y órganos hematopoyéticos. Hematología clínica de Sans-Sabrafen, 4 ed. Madrid: Harcourt; 2001; pp. 1-33. [ Links ]
15. Hoffman R, Benz E. Hematology: basic principles and practice, 4ª ed. Philadelphia: Elsevier Churchill Livingstone; 2005. [ Links ]
16. Rattis F, Voermans C, Reya T. Wnt signaling in the stem cell niche. Curr Opin Hematol 2004; 11: 88–94. [ Links ]
17. Ronzoni L, Bonara P, Rusconi D, Frugoni C, Libani I, Domenica C. Erythroid differentiation and maturation from peripheral CD34+ cells in liquid culture: Cellular and molecular characterization. Blood Cells Mol Dis 2008; 40: 148–155. [ Links ]
18. Bray SJ. Notch signaling: a simple pathway becomes complex. Nat Rev Mol Cell Biol 2006; 7: 678–689. [ Links ]
19. Qu K, Ortoleva P. Understanding stem cell differentiation through self–organization theory. J Theor Biol 2007; 250: 606–620. [ Links ]
20. Roeder I, Glauche I. Towards an understanding of lineage specification in hematopoietic stem cells: a mathematical model for the interaction of transcription factors GATA–1 and PU.1. J Theor Biol 2006; 241: 852–865. [ Links ]
21. Laricchia–Robbio L, Nucifora G. Significant increase of self–renewal in hematopoietic cells after forced expression of EVI1. Blood Cells Mol Dis 2008; 40: 141–147. [ Links ]
22. Jordan CT, Guzman ML, Noble M. Cancer stem cells. N Engl J Med 2006; 12: 1253–1261. [ Links ]
23. Walkley CR, Fero ML, Chien WM, Purton LE, McArthur GA. Negative cell cycle regulators cooperatively regulate self–renewal and differentiation of haematopoietic stem cells. Nat Cell Biol 2005; 7: 172–178. [ Links ]
24. Yu H, Yuan Y, Shen H, Cheng T. Hematopoietic stem cell exhaustion impacted by p18 INK4C and p21 Cip1/Waf1 in opposite manners. Blood 2006; 107: 1200–1206. [ Links ]
25. Van Den Berg D, Sharma AK, Bruno E, Hoffman R. Role of members of the Wnt gene family in human hematopoyesis. Blood 1998; 92: 3189–3202. [ Links ]
26. Nusse R. Wnts and Hedgehogs: lipid–modified proteins and similarities in signaling mechanisms at the cell surface. Development 2003; 130: 5297–5305. [ Links ]
27. Staal FJT, Clevers HC. WNT signalling and haematopoiesis: a WNT–WNT situation. Nat Rev Immunol 2005; 5: 21–30. [ Links ]
28. Taipale J, Beachy P. The Hedgehog and Wnt signaling pathways in cancer. Nature 2001; 411: 349–354. [ Links ]
29. Willert K, Brown JD, Danenberg E, Duncan AW, Weissman IL, Reya T, et al. Wnt proteins are lipidmodified and can act as stem cell growth factors. Nature 2003; 423: 448–452. [ Links ]
30. Takemaru K, Yamaguchi S, Lee YS, Zhang Y, Carthew RW, Moon RT. Chibby, a nuclear B–catenin associated antagonist of the Wnt/Wingless pathway. Nature 2003; 422: 905–909. [ Links ]
31. Reya T, Duncan AW, Ailles L, Domen J, Scherer DC, Willert K, et al. A role for Wnt signaling in self–renewal of haematopoietic stem cells. Nature 2003; 423: 409–414. [ Links ]
32. Jia L, Zhou S, Peng S, Li J, Cao Y, Duan E. Effects of Wnt3a on proliferation and differentiation of human epidermal stem cells. Biochem Biophys Res Commun 2008; 368: 483–488. [ Links ]
33. Owens D, Watt F. Contribution of stem cells and differentiated cells to epidermal tumours. Nat Rev Cancer 2003; 3: 444–451. [ Links ]
34. Dravid A, Ye H, Hammond G, Chen AP, Donovan P, Cheng Y. Defining the role of Wnt/b–catenin signaling in the survival, proliferation and self–renewal of human embryonic stem cells. Stem Cells 2005; 23: 1489–1501. [ Links ]
35. Mumm JS, Kopan R. Notch signaling: from the outside in. Dev Biol 2000; 228: 151–165. [ Links ]
36. Artavanis–Tsakonas S, Rand MD, Lake RJ. Notch signaling: cell fate control and signal integration in development. Science 1999; 284: 770–776. [ Links ]
37. Milner LA, Bigas A. Notch as a mediator of cell fate determination in hematopiesis: evidence and speculation. Blood 1999; 93: 2431–2448. [ Links ]
38. Aster JC, Pear WS. Notch signaling in leukemia. Curr Opin Hematol 2001; 8: 237–244. [ Links ]
39. Bigas A, Martin DI, Milner LA. Notch1 and Notch2 inhibit myeloid differentiation in response to different cytokines. Mol Cell Biol 1998; 18: 2324–2333. [ Links ]
40. Liu S, Dontu G, Wicha M. Mammary stem cells, selfrenewal pathways, and carcinogenesis. Breast Cancer Res 2005; 7: 86–95. [ Links ]
41. Duncan AW, Rattis FM, DiMascio LN, Congdon KL, Pazianos G, Zhao C, et al. Integration of Notch and Wnt signaling in hematopoietic stem cell maintenance. Nat Immunol 2005; 6: 314–322. [ Links ]
42. Brennan K, Gardner P. Notching up another pathway. Bioassays 2002; 24: 405–410. [ Links ]
43. Iso T, Kedes L, Hamamori Y. HES and HERP families: Multiple effectors of the Notch signaling pathway. J Cell Physiol 2003; 194: 237–255. [ Links ]
44. Wu L, Aster JC, Blacklow SC, Lake R, Artavanis–Tsakonas S, Griffin JD. MAML1, a human homologue of Drosophila mastermind, is a transcriptional co–activator for NOTCH receptors. Nat Genet 2000; 26: 484–489. [ Links ]
45. Kalderon D. Transducing the Hedgehog signal. Cell 2000; 103: 371–374. [ Links ]
46. Hahn H, Christiansen J, Wicking C, Zaphiropoulos PG, Chidambaram A, Gerrard B, et al. A mammalian patched homolog is expressed in target tissues of sonic hedgehog and maps to a region associated with developmental abnormalities. J Biol Chem 1996; 271: 12125–12128. [ Links ]
47. Ingham PW. Transducing hedgehog: the story so far. EMBO J 1998; 17: 3505–3511. [ Links ]
48. Bhardwaj G, Murdoch B, Wu D, Baker DP, Williams KP, Chadwick K, et al. Sonic hedgehog induces the proliferation of primitive human hematopoietic cells via BMP regulation. Nat Immunol 2001; 2: 172–180. [ Links ]
49. Dahmane N, Ruiz A. Sonic hedgehog regulates the growth and patterning of the cerebellum. Development 1999; 126: 3089–3100. [ Links ]
50. Palma V, Lim DA, Dahmane N, Sanchez P, Brionne TC, Herzberg C D, et al. Sonic hedgehog controls stem cell behavior in the postnatal and adult brain. Development 2005; 132: 335–344. [ Links ]
51. Chen JK, Taipale J, Cooper MK, Beachy PA. Inhibition of Hedgehog signaling by direct binding of cyclopamine to Smoothened. Genes Dev 2002;16: 2743–2748. [ Links ]
52. Romer JT, Kimura H, Magdaleno S, Sasai K, Fuller C, Baines H, et al. Suppression of the Shh pathway using a small molecule inhibitor eliminates medulloblastoma in Ptc1+/– p53–/– mice. Cancer Cell 2004; 6: 229–240. [ Links ]
53. Kubo M, Nakamura M, Tasaki A, Yamanaka N, Nakashima H, Nomura M, et al. Hedgehog signaling pathway is a new therapeutic target for patients with breast cancer. Cancer Res 2004; 64: 6071–6074. [ Links ]
54. Lewis MT. Hedgehog signaling in mouse mammary gland development and neoplasia. J Mammary Gland Biol Neoplasia 2001; 6: 53–66. [ Links ]
55. Lewis MT, Veltmaat JM. Next stop, the twilight zone: hedgehog network regulation of mammary gland development. J Mammary Gland Biol Neoplasia 2004; 9: 165–181. [ Links ]
56. Pasca di Magliano M, Hebrok M. Hedgehog signalling in cancer formation and maintenance. Nat Rev Cancer 2003; 3: 903–911. [ Links ]
57. Olsen CL, Hsu PP, Glienke J, Rubanyi GM, Brooks AR. Hedgehog– interacting protein is highly expressed in endothelial cells but down–regulated during angiogenesis and in several human tumors. BMC Cancer 2004; 4: 43–54. [ Links ]
58. Xie J, Johnson RL, Zhang X, Bare JW, Waldman FM, Cogen PH, et al. Mutations of the PATCHED gene in several types of sporadic extracutaneous tumors. Cancer Res 1997; 57: 2369–2372. [ Links ]
59. Kiyono T, Foster SA, Koop JI, McDougall JK, Galloway DA, Klingelhutz AJ. Both Rb/p16INK4a inactivation and telomerase activity are required to immortalize human epithelial cells. Nature 1998; 396: 84–88. [ Links ]
60. Grinstein P W. Cellular signaling in normal and cancerous stem cells. Cell Signal 2007; 19: 2428–2433. [ Links ]
61. Raaphorst FM. Self–renewal of hematopoietic and leukemic stem cells: a central role for the Polycomb–group gene Bmi–1. Trends Immunol 2003; 24: 522–524. [ Links ]
62. Park IK, Qian D, Kiel M, Becker MW, Pihalja M, Weissman IL, et al. Bmi–1 is required for maintenance of adult self renewing haematopoietic stem cells. Nature 2003; 423: 302–305. [ Links ]
63. Lessard J, Sauvageau G. Bmi–1 determines the proliferative capacity of normal and leukaemic stem cells. Nature 2003; 423: 255–260. [ Links ]
64. Van der Lugt NM, Domen J, Linders K, Van Roon M, Robanus Maandag E, Riele TH, et al. Posterior transformation, neurological abnormalities, and severe hematopoietic defects in mice with a targeted deletion of the bmi–1 proto–oncogene. Genes Dev 1994; 8: 757–769. [ Links ]
65. McGinnis W, Krumlauf R. Homeobox genes and axial patterning. Cell 1992; 68: 283–302. [ Links ]
66. Levine M, Hoey T. Homeobox proteins as sequencespecific transcription factors. Cell 1988; 55: 537–540. [ Links ]
67. Sauvageau G, Thornsteinsdottir U, Eaves CJ, Lawrence HJ, Largman C, Landsdorp PM, et al. Overexpression of HOXB4 in hematopoietic cells causes the selective expansion of more primitive populations in vitro and in vivo. Genes Dev 1995; 9: 1753–1765. [ Links ]
68. Thorsteinsdottir U, Sauvageau G, Humphries RK. Enhanced in vivo regenerative potential of HOXB4–transduced hematopoietic stem cells with regulation of their pool size. Blood 1999; 94: 2605–2612. [ Links ]
69. Brun AC, Bjornsson JM, Magnusson M, Larsson N, Leveen P, Ehinger M, et al. Hoxb4–deficient mice undergo normal hematopoietic development but exhibit a mild proliferation defect in hematopoietic stem cells. Blood 2004; 103: 4126–4133. [ Links ]
70. Daga A, Podesta M, Capra MC, Piaggio G, Frassoni F, Corte G. The retroviral transduction of HOXC4 into human CD34–cells induces an in vitro expansion of clonogenic and early progenitors. Exp Hematol 2000; 28: 569–574. [ Links ]
71. Zhu J, Giannola DM, Zhang Y, Rivera AJ, Emerson SG. NF–Y cooperates with USF 1/2 to induce the hematopoietic expression of HOXB4. Blood 2003; 102: 2420–2427. [ Links ]
72. Mitsui K, Tokuzawa Y, Itoh H, Segawa K, Murakami M, Takahashi K, et al. The homeoprotein Nanog is required for maintenance of pluripotency in mouse epiblast and ES cells. Cell 2003; 113: 631–642. [ Links ]
73. Palmieri SL, Peter W, Hess H, Scholer HR. Oct–4 transcription factor is differentially expressed in the mouse embryo during establishment of the first two extraembryonic cell lineages involved in implantation. Dev Biol 1994; 166: 259–267. [ Links ]
74. Bhattacharya B, Miura T, Brandenberger R, Mejido J, Luo Y, Yang AX, et al. Gene expression in human embryonic stem cell lines: unique molecular signature. Blood 2004; 103: 2956–2964. [ Links ]
75. Ambrosetti DC, Scholer HR, Dailey L, Basilico C. Modulation of the activity of multiple transcriptional activation domains by the DNA binding domains mediates the synergistic action of Sox2 and Oct–3 on the fibroblast growth factor–4 enhancer. J Biol Chem 2000; 275: 23387–23397. [ Links ]
76. Yuan H, Corbi N, Basilico C, Dailey L. Developmentalspecific activity of the FGF–4 enhancer requires the synergistic action of Sox2 and Oct–3. Genes Dev 1995; 9: 2635–2645. [ Links ]
77. Nishimoto M, Fukushima A, Okuda A, Muramatsu M. The gene for the embryonic stem cell coactivator UTF1 carries a regulatory element which selectively interacts with a complex composed of Oct–3/4 and Sox–2. Mol Cell Biol 1999; 19: 5453–5465. [ Links ]
78. Kuroda T, Tada M, Kubota H, Kimura H, Hatano SY, Suemori H, et al. Octamer and Sox elements are required for transcriptional cis regulation of Nanog gene expression. Mol Cell Biol 2005; 25: 2475–2485. [ Links ]
79. Rodda DJ, Chew JL, Lim LH, Loh YH, Wang B, Ng HH, et al. Transcriptional regulation of nanog by OCT4 and SOX2. J Biol Chem 2005; 280: 24731–24737. [ Links ]
80. Pesce M, Scholer HR. Oct–4: gatekeeper in the beginnings of mammalian development. Stem Cells 2001; 19: 271–278. [ Links ]
81. Pan GJ, Chang ZY, Scholer HR, Pei D. Stem cell pluripotency and transcription factor Oct4. Cell Res 2002; 12: 321–329. [ Links ]
82. Niwa H. Molecular mechanism to maintain stem cell renewal of ES cells. Cell Struct Funct 2001; 26: 137–148. [ Links ]
83. Avilion AA, Nicolis SK, Pevny LH, Perez L, Vivian N, Lovell–Badge R. Multipotent cell lineages in early mouse development depend on SOX2 function. Genes Dev 2003; 17: 126–140. [ Links ]
84. Hochedlinger K, Yamada Y, Beard C, Jaenisch R. Ectopic expression of Oct–4 blocks progenitor–cell differentiation and causes dysplasia in epithelial tissues. Cell 2005; 121: 465–477. [ Links ]
85. Baron MH. Embryonic origins of mammalian hematopoiesis. Exp Hematol 2003; 31: 1160–1169. [ Links ]
86. Akashi K, Traver D, Miyamoto T, Weissman IL. A clonogenic common myeloid progenitor that gives rise to all myeloid lineages. Nature 2000; 404: 193–197. [ Links ]
87. Blank U, Karlsson G, Karlsson S. Signaling pathways governing stem–cell fat. Blood 2008; 111: 492–503. [ Links ]
88. Galan I, DeLeon JA, Diaz L, Hong JS, Muñoz MA, et al. Effect of a bone marrow microenvironment on the exvivo expansion of umbilical cord blood progenitor cells. Int J Lab Hematol 2007; 29: 58–63. [ Links ]
Recibido: abril 14 de 2008
Aprobado: mayo 05 de 2008