Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Iatreia
versión impresa ISSN 0121-0793
Iatreia v.21 n.3 Medellín jul./sep. 2008
ARTÍCULO DE REVISIÓN
Nuevos métodos para el diagnóstico de la tuberculosis
New methods for the diagnosis of tuberculosis
Carlos Andrés Agudelo1,2,3; Luz Natalia Builes1; Mauricio Hernández1,4; Jaime Robledo1,4
1. Escuela de Ciencias de la Salud, Universidad Pontificia Bolivariana, Medellín, Colombia.
2. Hospital Pablo Tobón Uribe, Medellín, Colombia.
3. Clínica Universitaria Bolivariana, Medellín, Colombia.
4. Corporación para Investigaciones Biológicas, CIB, Medellín, Colombia.
Correspondencia: Luz Natalia Builes natibu@une.net.co
RESUMEN
La magnitud del problema mundial de la tuberculosis y su potencial de incremento han conducido a la necesidad de mejorar los métodos de diagnóstico como una de las estrategias conducentes al control de la enfermedad. Los nuevos métodos incluyen pruebas moleculares para la detección directa, medios de cultivo rápido y novedosos procedimientos de identificación. Para la detección directa de micobacterias se dispone hoy de pruebas basadas en la amplificación de los ácidos nucleicos, cuya sensibilidad supera a la de la baciloscopia y es cercana a la de los cultivos, pero con mayores costos que los procedimientos tradicionales.
La automatización para detectar el crecimiento en medios de cultivo produjo un cambio radical en la micobacteriología, pues con ella se logró disminuir considerablemente el tiempo requerido para aislar el M. tuberculosis (MTB); la combinación de métodos de detección rápida permite en la actualidad mejores tasas de aislamiento y la demostración más precoz del crecimiento. Las nuevas alternativas han logrado aumentar tanto la sensibilidad como la especificidad y la rapidez diagnósticas; sin embargo, es necesario mejorar aún más los métodos de diagnóstico de esta enfermedad de modo que se combinen el mejor desempeño y la simplicidad de los procedimientos con la disminución de los costos.
Palabras clave: Cultivos para micobacterias, Detección directa de micobacterias, Diagnóstico de la tuberculosis, Identificación de micobacterias
Summary
The magnitude of the global tuberculosis problem and its potential for increase have led to efforts to improve the diagnostic methods as one of the leading control strategies. New diagnostic tools have been developed, including direct detection tests, rapid culture media and identification methods. For the direct detection of mycobacteria, tests based on nucleic acid amplification have been implemented, with higher sensitivity than the direct smear, close to that of the cultures; however, they are still expensive methods. Availability of the automated cultures resulted in a radical change in mycobacteriology, reducing the time required for the isolation of Mycobacterium tuberculosis. The combination of rapid methods has increased the rates of isolation and decreased the time required for the detection of growth. Different tools for identifying mycobacteria have also been developed, aimed at improving the performance of traditional biochemical methods and achieving species identification in a few hours. The new diagnostic alternatives have succeeded in improving the sensitivity, specificity and speed in the diagnosis of tuberculosis; however, the challenge remains to reduce their cost and to make them more accessible in countries where the problem with this disease is still serious.
Key words: Diagnosis of tuberculosis, Direct detection of mycobacteria, Identification of mycobacteria, Mycobacterial cultures
INTRODUCCIÓN
El problema de la tuberculosis (TB), lejos de tener una solución, ha continuado creciendo alrededor del planeta; la Organización Mundial de la Salud estimó en 8,8 millones los casos de TB para el año 2003;1 se calcula que la enfermedad produce 2 millones de muertes anuales y que entre 19 y 43% de la población mundial está infectada por Mycobacterium tuberculosis (MTB).2,3
Tradicionalmente el diagnóstico de TB se ha basado en el examen clínico, las radiografías de tórax y el examen directo del esputo, el cual detecta solo el 50– 80% de los casos de TB con cultivos positivos.4 El fenómeno creciente del complejo M. aviumintracellulare en muchas regiones ha producido una disminución en la especificidad del examen directo para la detección de tuberculosis.5 Además, pacientes con baciloscopias negativas transmiten el 17% de los casos de TB.6
Los cultivos del microorganismo en medios tradicionales, como Lowenstein–Jensen, Ogawa y Middlebrook, son necesarios para detectar los casos negativos para bacilos ácido–alcohol resistentes (BAAR), identificar el microorganismo y hacer las pruebas de sensibilidad; sin embargo, el crecimiento del MTB en estos medios puede tardar hasta ocho semanas. Se ha descrito que hasta 10–20% de las muestras clínicas no se pueden cultivar satisfactoriamente.7
El retardo en el diagnóstico (mayor de 7 días) se ha asociado con aumento de la tasa de mortalidad en todos los grupos de edad, tanto en pacientes VIH positivos como negativos, así como con el ingreso tardío (mayor de 7 días) a la unidad de cuidados intensivos en pacientes hospitalizados. El diagnóstico tardío de TB se relaciona con la ausencia de tos y expectoración, el estado VIH positivo, la ausencia de cavernas en las radiografías de tórax, la edad avanzada y los exámenes directos o baciloscopias negativas.8 El retraso en el inicio del tratamiento mayor de un mes luego de tomada la muestra se relacionó con la muerte de 11 de 13 pacientes VIH positivos en la ciudad de Nueva York.9 El desarrollo de TB multirresistente también se ha asociado con el retardo en el diagnóstico.10
De acuerdo con lo anterior, los Centros para el Control y la Prevención de Enfermedades (CDC, del inglés Centers for Disease Control and Prevention) han recomendado el uso de cultivos en medios líquidos y sólidos asociados a un método rápido de identificación para obtener un informe definitivo en menos de 21 días.11 Esto ha generado un estímulo al desarrollo de nuevas técnicas para el diagnóstico de TB, muchas de las cuales buscan disminuir el tiempo necesario para el aislamiento e identificación de las micobacterias con mejoría en la sensibilidad y la especificidad. Se presenta seguidamente una revisión de estos nuevos métodos.
DETECCIÓN DIRECTA DE MICOBACTERIAS
El desarrollo de las técnicas de amplificación de ácidos nucleicos ha revolucionado el diagnóstico microbiológico del MTB, permitiendo detectar en menos de ocho horas secuencias específicas de ADN o ARN, con una sonda de secuencias complementarias, usando un microscopio de luz o métodos colorimétricos.12 La mayoría de estas pruebas se basan en técnicas de reacción en cadena de la polimerasa (PCR) y las puede desarrollar el mismo laboratorio que hace el diagnóstico o adquirirlas comercialmente.
La FDA (Food and Drug Administration) ha aprobado dos pruebas comerciales de este tipo para la detección directa del MTB en muestras respiratorias positivas para BAAR: el Amplified® Mycobacterium tuberculosis Direct test (MTD) y el AMPLICOR®. Mycobacterium tuberculosis test. El MTD ha sido también aprobado para la detección de Mycobacterium tuberculosis en muestras respiratorias negativas o paucibacilares de pacientes con sospecha clínica de TB.13
Como era de esperar, la tasa de sensibilidad del MTD en pacientes con baciloscopia positiva (BK positivo) ha sido en general más alta que en quienes tienen baciloscopia negativa (90,9–98,4% frente a 83–85%), mientras que la de especificidad ha sido semejante independientemente del resultado de la baciloscopia (98–100% frente a 99%)10,13–15 El valor predictivo positivo (VPP) del MTD fue más alto que el de la baciloscopia en las muestras de pacientes con probabilidad baja o intermedia de tener TB, mientras que el valor predictivo negativo (VPN) lo fue en las muestras de pacientes cuya probabilidad era intermedia o alta.16 La sensibilidad del Amplicor® es menor y su VPP más bajo en comparación con los del MTD en pacientes con baciloscopia positiva (Tabla n.º 1).10,13,15,17
Debido a su alta especificidad y a su moderada sensibilidad, se recomienda no usar esta prueba en pacientes con baja sospecha clínica ni en muestras con alta probabilidad de ser positivas en la baciloscopia; el criterio clínico debe guiar el uso e interpretación de la prueba para lograr su máximo desempeño.18 Además, con el Amplicor® se puede detectar el MTB en muestras de pacientes en quienes se ha iniciado el tratamiento y cuyos cultivos son negativos, así como en las muestras con micobacterias de crecimiento rápido que impiden detectar el MTB en los cultivos.19 La sensibilidad del Amplicor® no varía entre poblaciones con endemicidad alta o baja para TB, y es independiente de la infección con VIH.20
Se han desarrollado otros métodos, entre ellos los siguientes: el BD Probe Tec ET system® que amplifica las secuencias de inserción IS6110 y 16S del ARN, y genera simultáneamente una reacción química que produce fluorescencia la cual permite detectar el MTB. Estudios preliminares13,21 han demostrado sensibilidad de 93,8%, especificidad de 99,8%, VPP de 93,8% y VPN de 99,8% (Tabla n.º 1)
El Automated Q–Beta Replicase Amplification Assay® amplifica la subunidad 23 del ARN ribosómico de las micobacterias del complejo MTB; la sensibilidad ha sido del 84% y la especificidad, del 97%; permite obtener resultados en un máximo de 6,5 horas. Sin embargo, su rendimiento es mucho más bajo en las muestras con pocos bacilos y se han informado falsos positivos con micobacterias no TB.22
En general, los métodos de amplificación de ácidos nucleicos tienen menor sensibilidad en las muestras provenientes de pacientes con procesos clínica y radiológicamente menos graves,23 así como en las muestras extrapulmonares24,25 y paucibacilares,26,27 aunque algunos estudios demuestran que la sensibilidad y especificidad son semejantes a las de las técnicas tradicionales.28
En la TB meníngea las pruebas de amplificación de ácidos nucleicos comerciales tienen alta la especificidad (98%) pero baja la sensibilidad (56%), explicable esta última por la escasez de bacilos en el sistema nervioso central, el uso de cantidades insuficientes de líquido cefalorraquídeo y la presencia de inhibidores; por lo anterior se las puede emplear para confirmar el diagnóstico pero no para descartarlo ni para decidir la suspensión del tratamiento.29 Al menos en un estudio el examen directo del LCR tuvo la misma sensibilidad que el MTD para detectar la TB meníngea antes de iniciar el tratamiento, pero el MTD fue mejor cuando la prueba se llevó a cabo 2 a 5 días después de iniciado.30
Estas pruebas presentan un comportamiento similar cuando se utiliza la PCR en muestras de líquido pleural, en las que se ha demostrado especificidad del 98% y sensibilidad del 62%,31 en comparación con la sensibilidad del examen directo (20%), el cultivo (33%), la biopsia pleural (64,2%), el interferón γ (INF–γ) (77,6%) y la adenosina deaminasa (ADA) (86,9%); la sensibilidad mejoró al emplear combinaciones de INF–γ, ADA y PCR.32,33
Por el contrario, el uso de PCR junto con la tinción de Ziehl Nielsen en muestras de esputo o lavado broncoalveolar (LBA) de pacientes con TB pulmonar permitió hacer un diagnóstico rápido en el 90% de los casos.34 Un metanálisis de los estudios de PCR en muestras del tracto respiratorio (esputo, LBA, aspirado de moco en jugo gástrico, aspirado traqueal), con examen directo negativo, encontró sensibilidad de 76%, especificidad de 97%, VPP de 96% y VPN de solo 80%; la seguridad diagnóstica fue mayor en las pruebas hechas en muestras de secreciones bronquiales y menor en las de jugo gástrico; sin embargo, se encontraron amplias variaciones entre los diferentes estudios analizados. Los autores de este estudio recomendaron no utilizar las pruebas de PCR como método único de diagnóstico en pacientes con TB pulmonar y baciloscopia negativa.35
En algunos casos ha sido baja la reproducibilidad de los resultados de las pruebas de amplificación desarrolladas por los laboratorios diagnósticos, al compararlas con las comercialmente disponibles; esto puede explicarse por las variaciones en los protocolos de laboratorio. Los valores de sensibilidad y especificidad han sido muy diferentes de uno a otro estudio.29,31,36
La certeza total de los métodos de amplificación de ácidos nucleicos [(Verdaderos positivos + Verdaderos negativos)/Total de muestras] es mucho más alta que la de la baciloscopia y se acerca a la de los cultivos. Si la pregunta es: ¿tiene mi paciente TB activa?, con la utilización de estos métodos la respuesta será correcta entre 92 y 95% de las veces.5
Las técnicas de amplificación de ácidos nucleicos pueden dar resultados falsos positivos para infección activa en pacientes con infección latente por MTB (prueba de tuberculina positiva), o por contaminación cruzada durante la manipulación de las muestras en el laboratorio. Los resultados falsos negativos se pueden deber a la presencia de inhibidores en la muestra, a la ausencia de las secuencias de ácidos nucleicos que se amplificarán (por ejemplo: IS6110) y a problemas en la obtención de la muestra o del ADN. El principal inconveniente para usar los métodos moleculares en el diagnóstico directo es su costo, que se calcula aproximadamente en US$25–50 por muestra para las pruebas comerciales y en US$15 para las desarrolladas por cada laboratorio. Aparentemente la principal ventaja de estos métodos es la aplicación en los pacientes muy sospechosos de tener TB pero cuyos exámenes de esputo son negativos, en quienes se puede iniciar más oportunamente el tratamiento y evitar exámenes invasivos.
Más recientemente se han desarrollado técnicas de PCR en tiempo real que permiten detectar el genoma del MTB en muestras clínicas utilizando amplificación con marcadores fluorescentes. Para esta técnica se deben utilizar equipos que detecten una señal específica generada a medida que se vaya dando la amplificación del segmento de ADN correspondiente. Esta técnica permite detectar y cuantificar temprana y rápidamente la amplificación. Algunos estudios con la técnica de PCR en tiempo real han demostrado que la sensibilidad es variable pero similar a la de los cultivos de muestras respiratorias con baciloscopias positivas. En muestras con baciloscopias negativas la sensibilidad ha sido menor.37 En un estudio, la PCR en tiempo real demostró sensibilidad mayor que la de las pruebas convencionales de amplificación y los cultivos.38 No existen suficientes estudios que hayan evaluado la PCR en tiempo real para definir su utilidad en el diagnóstico de la tuberculosis; sin embargo, por sus características, es un método que evita los inconvenientes de las técnicas usuales de amplificación y que puede llegar a reemplazarlas.
A diferencia de los métodos anteriores, basados en técnicas moleculares, el llamado FASTPlaqueTB® es un nuevo método, no molecular, que tarda entre 24 y 48 horas en detectar la presencia de micobacterias en muestras de pacientes; se basa en las técnicas de infección por fagos empleadas para detectar la resistencia a medicamentos antituberculosos. Su sensibilidad en muestras de esputo está entre 64 y 87,5% y la especificidad, entre 93 y 96,9%; el VPP es de 67,5% y el VPN, de 99,1%.39,40 Su costo es mucho menor que el de las pruebas de detección de ácidos nucleicos (US$13). Su interpretación puede ser difícil si el número de placas detectadas se aproxima al punto de corte; además, se pueden presentar falsos positivos por errores de procesamiento y falsos negativos por retardo en el mismo, por un bajo número de micobacterias vivas en la muestra, o por la presencia de sustancias inhibidoras que afecten la interacción de las micobacterias con los fagos.39
En un estudio sudafricano de costo–beneficio sobre el uso del FASTPlaqueTB® en cuatro algoritmos diferentes para el diagnóstico de TB se encontró una disminución en el costo y en el número de consultas necesarias para hacer el diagnóstico de TB en los algoritmos que utilizaron esta prueba; en menos de 48 horas se logró hacer el diagnóstico en más del 50% de los pacientes con baciloscopias negativas y se detectaron 28% más de casos al utilizarla simultáneamente con cultivos.41
MEDIOS DE CULTIVO RÁPIDO
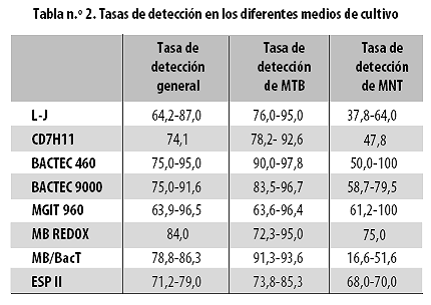
Tradicionalmente se han utilizado tres tipos distintos de medios de cultivo, a saber: los basados en huevo (Lowenstein–Jensen y Ogawa), los basados en agar (Middlebrook 7H10 y 7H11) y los líquidos (Middlebrook 7H9 y 7H12). El crecimiento de MTB en los medios basados en agar es más rápido que en los basados en huevo, pero estos últimos tienden a ser más sensibles. El crecimiento en los medios líquidos es más rápido que en los sólidos. El uso de una capa delgada del agar Middlebrook 7H11(CD7H11) permite la detección más temprana del crecimiento de las micobacterias.42 La especificidad y la sensibilidad informadas para el Lowenstein–Jensen (L–J) han sido de 98% y 80–86%, respectivamente. Un estudio local informó sensibilidad del 73,5% y especificidad del 99,2% para CD7H11.43
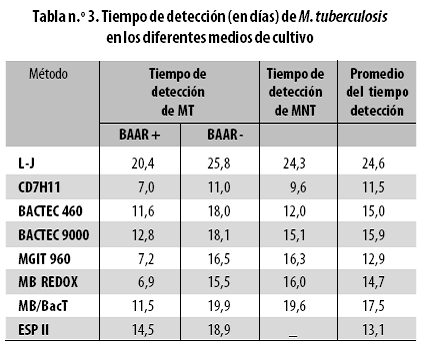
En un estudio multicéntrico latinoamericano se compararon los medio de cultivo de Lowenstein–Jensen y CD7H11; se encontró que la sensibilidad era más alta para este último (92,6%) que para el L–J (84,7%), mientras que la tasa de contaminación era mayor para el primero (5,1%) que para el segundo (3,0%). La mediana del tiempo necesario para detectar un cultivo positivo fue de 11,5 días para el CD7H11 y de 30,5 días para el L–J.44
En la década de los años 80 se dio un cambio radical en la micobacteriología con el desarrollo del BACTEC TB–460®, que permitió detectar el MTB en unos pocos días en lugar de semanas. Este método utilizaba un medio 7H12 con 14C–palmitato como única fuente de carbono, el cual, al ser metabolizado por el microorganismo, originaba 14CO2 detectable por el equipo. Sin embargo, el costo del aparato y la necesidad de usar radioisótopos restringieron su uso y llevaron a su descontinuación,13 a pesar de sus ventajas sobre los demás medios líquidos, como eran la rapidez, la sensibilidad para detectar micobacterias, especialmente micobacterias no tuberculosas, y la precisión con que identificaba el MTB.45,46 Más recientemente, el nuevo BACTEC 9000 MB® utiliza Middlebrook 7H9 y un sistema de fluorescencia que detecta el consumo de oxígeno debido a la actividad y crecimiento de los microorganismos.47
El Mycobacterial Growth Indicator Tube® (MGIT) es un método comercial totalmente automatizado que utiliza un tubo con Middlebrook 7H9 y un sensor de fluorescencia que detecta la reducción de oxígeno causada por las bacterias aerobias en crecimiento. Existe también una versión con capacidad para procesar gran cantidad de muestras clínicas, el MGIT 960, que combinado con un medio sólido da resultados comparables a los del BACTEC 460 para detectar el complejo MTB.48
Otro medio de detección rápida es el MB–Redox®, un sistema de cultivo no radiactivo que utiliza un tubo con medio de Kirchner y, como indicador, una sal incolora de tetrazolio que es reducida a formazán al crecer las micobacterias, dando gránulos de color rosa o violeta. Es fácil de manipular, detecta directamente el crecimiento sin necesidad de ningún instrumento, es rápido y sensible y no requiere revisiones diarias; sin embargo, aunque detecta el crecimiento de micobacterias, no permite distinguir el complejo MTB de otras micobacterias, por lo que se ha utilizado junto con un sistema de PCR para lograr la identificación de las especies patógenas.49 En un estudio se demostró que este método puede ser más rápido que el MGIT para detectar micobacterias en muestras con baciloscopia negativa (15,5 frente a 19,1 días).50
El MGIT y el MB Redox son sistemas rápidos, sensibles y de fácil manipulación y no requieren instrumentación costosa adicional. Sus tasas de contaminación son aceptables y no plantean mayores dificultades en la detección.50 En muestras con directos positivos el tiempo de detección del crecimiento es de 6,9 días con el MB Redox® y de 7,2 días con el MGIT; para muestras con directos negativos se han informado tiempos de crecimiento de 15,5 y 19,5 días, respectivamente.46
El sistema MB/BacT ®, basado en la detección colorimétrica continua de la producción de CO2 en medios líquidos, tiene varias ventajas: ser un sistema cerrado sin riesgo de contaminación, no generar radiactividad y permitir la obtención rápida de la mayoría de los aislamientos. Es una alternativa valiosa en los laboratorios donde no está permitido el manejo de desechos radiactivos.45
El ESP (Extra Sensing Power) culture system II® es otro sistema automatizado, con monitorización continua, que se basa en detectar los cambios de presión sobre el medio de cultivo en un recipiente sellado; este sistema permite el crecimiento y la identificación de microorganismos, incluyendo las micobacterias. 13,51
Se ha demostrado que la tasa de sensibilidad del ESP II es similar a la del BACTEC TB y superior a la del agar 7H11 para el aislamiento de MTB y micobacterias no tuberculosas (MNT); en cuanto al aislamiento de M. avium, es mayor la sensibilidad del ESP II. A pesar de sus ventajas, ni en el BACTEC TB ni en el ESP II se logra aislar todas las micobacterias, por lo que deben asociarse a un medio sólido. El ESP II tiene las ventajas de no requerir una gran carga laboral y no utilizar sustancias radiactivas.51,52
Heifets reportó el análisis de 243 aislamientos de MTB obtenidos entre los años 1994–1996 a partir de muestras inoculadas en tres medios de cultivo diferentes (agar 7H11, L–J y BACTEC 12B); solo la mitad de los aislamientos creció en los tres medios usados y el 14% lo hizo en el medio líquido BACTEC 12B, especialmente en las muestras provenientes de pacientes paucibacilares.46 En la actualidad es obligatorio en Estados Unidos asociar un medio líquido con uno sólido en los intentos de aislar micobacterias.12
En las tablas n.º 2 y 3 se presentan en forma comparativa los datos referentes al tiempo de crecimiento promedio y a las tasas de aislamiento en los medios de cultivo de uso actual.
La combinación de los métodos de detección rápida con los medios de cultivo sólidos permite aumentar las tasas de aislamiento, disminuir el tiempo necesario para que crezcan las micobacterias, hacer pruebas de identificación adecuadas e incubar prolongadamente.53 En la actualidad, la combinación de LJ y MGIT ha demostrado ser la mejor alternativa para el diagnóstico microbiológico de la TB.54
MÉTODOS DE IDENTIFICACIÓN
La identificación tradicional del MBT se logra mediante pruebas fenotípicas bioquímicas, cuyos resultados requieren varios días y pueden ser ambiguos en el caso de otras especies de micobacterias, por la variabilidad de las reacciones. Ello ha estimulado el desarrollo de nuevos métodos de identificación más rápidos y precisos que los tradicionales.13
La presencia de secuencias polimórficas en el ADN de las diferentes especies de micobacterias es una de las bases para identificarlas empleando métodos moleculares. Se ha demostrado que la hibridación de la secuencia de inserción IS6110 con técnicas de RFLP (Restriction fragment length polymorphism) es específica para identificar el complejo MTB.55 La amplificación de segmentos genéticos (16s del rARN y HSP de 65–kDa) con técnicas de PCR y su identificación con RFLP fluorescente, en gel de agarosa o con electroforesis capilar fluorescente, puede diferenciar hasta 22 especies de micobacterias. Estas técnicas son menos costosas que los métodos comerciales y tardan solo 1–2 días en identificar las especies.36,56,57
Uno de estos métodos de identificación es el PRA (PCR restriction enzyme analysis), que se basa en la amplificación por PCR de un fragmento de 441 pares de bases del gen hsp65 y la restricción posterior con dos enzimas BstEII y HaeIII; el número y el tamaño de las bandas generadas por la restricción y observadas en un gel de agarosa después de electroforesis permiten identificar las especies.58 Un estudio multicéntrico reciente, hecho por varios laboratorios latinoamericanos para evaluar el PRA en aislamientos codificados, demostró que, con este método, más de la mitad de los laboratorios podían identificar el 70% o más de las especies. La falta de precisión del método se asoció a falta de experiencia en la lectura y a errores en el procedimiento; el estudio concluyó que el PRA es un buen método para identificar especies de micobacterias cuando los laboratorios adquieren la suficiente experiencia y cuando los resultados se asocian con algunas características fenotípicas básicas.59
Se han desarrollado varios métodos comerciales para identificar micobacterias en cultivos; el primero de ellos fue el llamado Accuprobe®, basado en la detección del rRNA de la micobacteria utilizando una sonda de ADN, que puede identificar las especies del complejo MTB, M. intracellulare, el complejo de M. avium, M. gordonae y M. kansaii. Los resultados se obtienen en 1–2 horas con una certeza mayor del 90% para las especies descritas.53
LiPA (del inglés Line probe assay) es un método basado en técnicas de PCR que utiliza iniciadores específicos de género para amplificar el ADN e identificar las especies por comparación con las secuencias de referencia. Tiene especificidad del 99% y sensibilidad del 100%, pero su costo lo hace inasequible para el uso diario.13,53
Otro sistema comercial es TB PNA FISH® (del inglés Fluorescence in situ Hybridization Assay Using Peptide Nucleic Acid), basado en la hibridación de regiones del 16 rRNA con sondas de ácidos nucleicos y detectado por microscopía de fluorescencia.60 El sistema FISH es capaz de identificar correctamente el 100% de los aislamientos del complejo MTB y el 81,5% de las micobacterias no tuberculosas; sin embargo, no puede detectar M. marinum, M. xenopi, M. flavescens y M. fortuitum porque las sondas no son complementarias del rRNA de estas especies. El sistema no puede distinguir entre las diferentes especies de cada complejo. Otra limitación es que las sondas pueden reaccionar con el rRNA de Actinomyces spp. y Rickettsia spp. El problema más frecuente ha sido la debilidad de la fluorescencia, principalmente cuando se evalúan micobacterias no tuberculosas.61
Se ha empleado el sistema COBAS–Amplicor, que es la forma automatizada de Amplicor, para la identificación de MTB y M. avium en cultivos positivos en BACTEC 12B, lo que permite disminuir a 3–4,5 días el tiempo necesario para el aislamiento y la identificación. Sorprendentemente este sistema ha mostrado tasas más altas de inhibición de la amplificación cuando se lo utiliza en muestras provenientes de BACTEC 12B que en especímenes clínicos (19% frente a 4,7%).62
La cromatografía líquida de alto desempeño (High performance liquid chromatography, HPLC) identifica las especies de micobacterias por el patrón de ácidos micólicos de la pared; en pocas horas puede diferenciar más de 50 especies a partir de cultivos puros. El costo inicial del equipo limita su disponibilidad como método de identificación.12
El uso de estos métodos rápidos de identificación asociado a los medios de cultivo rápido permite la identificación precisa de una micobacteria en menos de 2 semanas en las muestras con directo positivo, y aproximadamente en 3 semanas en aquellas con directo negativo.5
CONCLUSIONES
La magnitud mundial del problema de la tuberculosis y su potencial de incremento futuro, han impulsado el mejoramiento de las técnicas de diagnóstico como una de las estrategias necesarias para el control.
Se ha calculado que para lograr una disminución del 22% en la mortalidad por TB se requieren pruebas diagnósticas rápidas con tasas de sensibilidad por encima del 85%, tanto para muestras positivas como para las negativas en la baciloscopia, asociadas a tasas de especificidad mayores del 97% y a una fácil asequibilidad;63 aunque las nuevas técnicas de diagnóstico microbiológico han representado grandes avances en la detección de la TB, están lejos de lograr estas metas, especialmente la de su disponibilidad en los países en vías de desarrollo, donde se presentan más del 80% de los casos mundiales de TB.
Aunque se han logrado avances en las técnicas de diagnóstico, todavía no existen pruebas con mejor sensibilidad que la baciloscopia y que tengan, además, su simplicidad y bajo costo. Para disminuir el impacto de la enfermedad en las poblaciones más afectadas se requiere una prueba rápida (resultados disponibles en una hora), de bajo costo, con altas sensibilidad y especificidad, incluso para pacientes con baciloscopia negativa, y que no requiera electricidad, refrigeración ni agua limpia.
Además de los métodos tradicionales para el diagnóstico de la tuberculosis (baciloscopia y cultivo en L–J) están disponibles en Colombia algunas de estas otras pruebas. Algunas instituciones hospitalarias emplean pruebas basadas en PCR, adecuadamente validadas, para amplificar el ADN de las micobacterias, especialmente para el diagnóstico de la tuberculosis meníngea. Nuestro laboratorio viene empleando de rutina el cultivo en capa delgada y en MGIT para el estudio de todas las muestras en las que se sospeche que pueda aislarse el MTB; también se intenta constantemente la difusión del método de capa delgada como una técnica de bajo costo, rápida y sensible que puede utilizarse de rutina, incluso en instituciones de menor complejidad.
REFERENCIAS BIBLIOGRÁFICAS
1. Global tuberculosis control: surveillance, planning, financing. WHO report 2005. Geneva, World Health Organization (WHO/HTM/TB/2005.349). Disponible en: http://www.who.int/tb/publications/global_report/2005/en/index.html Consultado en junio 16 de 2008 [ Links ]
2. World Health Organization. 1996. Groups at risk: WHO report on the tuberculosis epidemic. World Health Organization, Geneve, Switzerland. Disponible en: http://whqlibdoc.who.int/hq/1996/WHO_TB_96.198.pdf consultado en junio 16 de 2008 [ Links ]
3. Iseman MD. A clinician's guide to tuberculosis. Philadelphia: Lippincott, Williams and Wilkins; 2000. p. 460 [ Links ]
4. Frieden TR, Sterling TR, Munsiff SS, Watt CJ, Dye C. Tuberculosis. Lancet 2003; 362: 887–899. [ Links ]
5. Schluger NW. Changing approaches to the diagnosis of tuberculosis. Am J Resp Crit Care Med 2001; 164: 2020–2024. [ Links ]
6. Behr MA, Warren SA, Salamon H, Hopewell PC, Ponce de Leon A, Daley CL, et al. Transmission of Mycobacterium tuberculosis from patients smear–negative for acid fast bacilli. Lancet 1999; 353: 444–449. [ Links ]
7. Andersen P, Munk ME, Pollock JM, Doherty TM. Specific immune–based diagnosis of tuberculosis. Lancet 2000; 356: 1099–1104. [ Links ]
8. Greenaway C, Menzies D, Fanning A, Grewal R, Yuan L, FitzGerald JM, et al. Delay in diagnosis among hospitalized patients with active tuberculosis. Predictors and outcomes. Am J Respir Crit Care Med 2002; 165: 927–933. [ Links ]
9. Pablos–Mendez A, Sterling TR, Frieden TR. The relationship between delayed or incomplete treatment and all cause mortality in patients with tuberculosis. JAMA 1996; 276: 1223–1228. [ Links ]
10. Woods G. Molecular techniques in mycobacterial detection. Arch Patol Lab Med 2001; 125: 122–126. [ Links ]
11. Tenover FC, Crawford JT, Huebner RE, Geiter LJ, Horsburgh CR, Good RC. The resurgence of tuberculosis: is your laboratory ready? J Clin Microbiol 1993; 31: 767–770. [ Links ]
12. American Thoracic Society and the Centers for Disease Control and prevention. Diagnostic standards and classification of tuberculosis in adults and children. Am J Respir Crit Care Med 2000; 161: 1376–1395. [ Links ]
13. Palomino JC, Martin A, Portaels F. New methods for the diagnosis and drug resistance detection in mycobacteria. Recen Res Devel Microbiol 2002; 6: 297–318. [ Links ]
14. Vlaspolder F, Singer P, Roqqeveen C. Diagnostic value of an amplification method (Gen Probe) compared with that of culture for diagnosis of tuberculosis. J Clin Microbiol 1995; 33: 2699–2703. [ Links ]
15. Wang SX, Tay L. Evaluation of three nucleic acid amplification methods for direct detection of Mycobacterium tuberculosis complex in respiratory specimens. J Clin Microbiol 1999; 37: 1932–1934. [ Links ]
16. Catanzaro A, Perry S, Clarridge JE, Dunbar S, Goodnight– White S, LoBue PA, et al. The role of clinical suspicion in evaluating a new diagnostic test for active tuberculosis: results of a multicenter prospective trial. JAMA 2000; 283: 639–645. [ Links ]
17. Cartuyvels R, De Ridder C, Jonckheere S, Verbist L, Van Eldere J. Prospective clinical evaluation of Amplicor Mycobacterium tuberculosis PCR test as a screening method in a low prevalence population. J Clin Microbiol 1996; 34: 2001–2003. [ Links ]
18. Lim TK, Mukhopadhyay A, Gough A, Khoo KL, Khoo SM, Lee KH, et al. Role of clinical judgement in the application of a nucleic acid amplification test for the rapid diagnosis of pulmonary tuberculosis. Chest 2003; 124: 902–908. [ Links ]
19. Moore DF, Curry JI. Detection and identification of Mycobacterium tuberculosis directly from sputum sediments by Amplicor PCR. J Clin Microbiol 1995; 33: 2686–2691. [ Links ]
20. Kivihya–Ndugga L, Van Cleeff M, Juma E, Kimwomi J, Githui W, Oskam L, et al. Comparison of PCR with the routine procedure for diagnosis of tuberculosis in a population with high prevalences of tuberculosis and human immunodeficiency virus. J Clin Microbiol 2004; 42: 1012–1015. [ Links ]
21. Bergmann JS, Keating WE, Woods GL. Clinical evaluation of the BDProbeTec ET System for rapid detection of Mycobacterium tuberculosis. J Clin Microbiol 2000; 38: 863–865. [ Links ]
22. Smith JH, Radcliffe G, Rigby S, Mahan D, Lane DJ, Klinger JD. Performance of an automated Q–Beta Replicase Amplification Assay for Mycobacterium tuberculosis in a clinical trial. J Clin Microbiol 1997; 35: 1484–1491. [ Links ]
23. Al Zahrani K, Al Jahdali H, Poirier L, Rene P, Menzies D. Yield of smear, culture and amplification tests from repeated sputum induction for the diagnosis of pulmonary tuberculosis. Int J Tuberc Lung Dis 2001; 5: 855–860. [ Links ]
24. Mitarai S, Tanoue S, Sugita C, Sugihara E, Tamura A, Nagono Y, et al. Potencial use of amplicor PCR kit in diagnosing pulmonary tuberculosis from gastric aspirate. J Microbiol Methods 2001; 47: 339–344. [ Links ]
25. Rolfs A, Beige J, Finckh U, Köhler B, Schaberg T, Lokies J, et al. Amplification of Mycobacterium tuberculosis from peripheral blood. J Clin Microbiol 1995; 33: 3312–3314. [ Links ]
26. Garrino MG, Glupczynski Y, Degraux J, Nizet H, Delmee M. Evaluation of the Abbott LCx Mycobacterium tuberculosis assay for direct detection of Mycobacterium tuberculosis complex in human samples. J Clin Microbiol 1999; 37: 229–232. [ Links ]
27. Chen NH, Liu YC, Tsao TC, Wu TL, Hsieh MJ, Chuang ML, et al. Combined bronchoalveolar lavage and polymerase chain reaction in the diagnosis of pulmonary tuberculosis in smear negative patients. Int J Tuberc Lung Dis 2002; 6: 350–355. [ Links ]
28. Hasaneen NA, Zaki ME, Shalaby HM, El–Morsi AS. Polymerase chain reaction of pleural biopsy is a rapid and sensitive method for the diagnosis of tuberculous pleural effusion. Chest 2003; 124: 2105–2111. [ Links ]
29. Pai M, Flores LL, Pai N, Hubbard A, Riley LW, Colford JM. Diagnostic accuracy of nucleic amplification tests for tuberculosis meningitis: a systematic review and metaanalysis. Lancet Infect Dis 2003; 3: 633–643. [ Links ]
30. Thwaites GE, Caws M, Chau TT, Dung NT, Campbell JI, Phu NH, et al. Comparison of conventional bacteriology with nucleic acid amplification (amplified mycobacterium direct test) for diagnosis of tuberculous meningitis before and after inception of antituberculosis chemotherapy. J Clin Microbiol 2004; 42: 996–1002. [ Links ]
31. Pai M, Flores LL, Hubbard A, Riley LW, Colford JM. Nucleic acid amplification tests in the diagnosis of tuberculous pleuritis: a systematic review and meta–analysis. BMC Infect Dis 2004; 4: 6. [ Links ]
32. Nagesh BS, Sehgal S, Jindal SK, Arora SK. Evaluation of polymerase chain reaction for detection of Mycobacterium tuberculosis in pleural fluid. Chest 2001; 119: 1737–1741. [ Links ]
33. Villegas MV, Labrada LA, Saravia NG. Evaluation of polymerase chain reaction, adenosine deaminase, and interferon–gamma in pleural fluid for the differential diagnosis of pleural tuberculosis. Chest 2000; 118: 1355–1364. [ Links ]
34. Tueller C, Chhajed PN, Buitrago–Tellez C, Frei R, Frey M, Tamm M. Value of smear and PCR in bronchoalveolar lavage fluid in culture positive pulmonary tuberculosis. Eur Resp J 2005; 26: 767–772. [ Links ]
35. Sarmiento OL, Weigle KA, Alexander J, Weber DJ, Miller WC. Assesment by meta–analysis of PCR for diagnosis of smear–negative pulmonary tuberculosis. J Clin Microbiol 2003; 41: 3233–3240. [ Links ]
36. Suffys P, Palomino JC, Cardoso–Leao S, Espitia C, Cataldi A, Alito A, et al. Evaluation of the polymerase chain reaction for the detection of Mycobacterium tuberculosis. Int J Tuberc Lung Dis 2000; 4: 179–183. [ Links ]
37. Parashar D, Chauhan DS, Sharma VD, Katoch VM. Applications of real–time PCR technology to mycobacterial research. Indian J Med Res 2006; 124: 385–398. [ Links ]
38. Broccolo F, Scarpellini P, Locatelli G, Zingale A, Branbilla AM, Cichero P, et al. Rapid diagnosis of mycobacterial infections and quantification of M. tuberculosis load by two real–time calibrated PCR assays. J Clin Microbiol 2003; 41: 4565–4572. [ Links ]
39. Albay A, Kisa O, Baylan O, Dogaci L. The evaluation of FASTPlaqueTB test for the rapid diagnosis of tuberculosis. Diagn Microbiol Infect Dis 2003; 46: 211–215. [ Links ]
40. Marei AM, El–Behedy EM, Mohtady HA, Afify AF. Evaluation of a rapid bacteriophage–based method for the detection of Mycobacterium tuberculosis in clinical samples. J Med Microbiol 2003; 52: 331–335. [ Links ]
41. Albert H. Economic analysis of the diagnosis of smearnegative pulmonary tuberculosis in South Africa: incorporation of a new rapid test, FASTPlaqueTB, into the diagnostic algorithm. Int J Tuberc Lung Dis 2004; 8: 240–247. [ Links ]
42. Brodie D, Schluger NW. The diagnosis of tuberculosis. Clin Chest Med 2005; 26: 247–271. [ Links ]
43. Mejía GI, Guzmán A, Agudelo CA, Trujillo H, Robledo J. Cinco años de experiencia con el agar de capa delgada para el diagnóstico rápido de tuberculosis. Biomédica. 2004; 24 (Suppl.1): 52–59. [ Links ]
44. Robledo J, Mejia GI, Morcillo N, Chacon L, Camacho M, Luna J, et al. Evaluation of a rapid culture method for tuberculosis diagnosis: a Latin American multi–center study. Int J Tuberc Lung Dis 2006; 10: 613–619. [ Links ]
45. Piersimoni C, Scarparo C, Callegaro A, Tosi CP, Nista D, Bornigia S, et al. Comparison of MB/Bact alert 3D system with radiometric BACTEC system and Lowenstein–Jensen medium for recovery and identification of mycobacteria from clinical specimens: a multicenter study. J Clin Microbiol 2001; 39: 651–657. [ Links ]
46. Heifets L, Linder T, Sanchez T, Spencer D, Brennan J. Two liquid medium systems, Mycobacteria Growth Indicator Tube and MB Redox Tube, for Mycobacterium tuberculosis isolatiom from sputum specimens. J Clin Microbiol 2000; 38: 1227–1230. [ Links ]
47. Pfyffer GE, Cieslak C, Welscher HM, Kissling P, Rüsch–Gerdes S. Rapid detection of mycobacteria in clinical specimens by using the automated BACTEC 9000 MB system and comparison with radiometric and solid culture systems. J Clin Microbiol 1997; 35: 2229–2234. [ Links ]
48. Hanna BA, Ebraimzadeh A, Elliott LB, Morgan MA, Novak SM, Rush–Gerdes S, et al. Multicenter evaluation of the BACTEC MGIT 960 system for recovery of mycobacteria. J Clin Microbiol 1999; 37: 748–752. [ Links ]
49. Liu YC, Huang TS, Huang WK. Comparison of a nonradiometric liquid–medium method (MB REDOX) with the BACTEC system for growth and identification of mycobacteria in clinical specimens. J Clin Microbiol 1999; 37: 4048–4050. [ Links ]
50. Somoskovi A, Magyar P. Comparison of the mycobacteria growth indicator tube with MB redox, Lowenstein–Jensen, and Middlebrook 7H11 media for recovery of mycobacteria in clinical specimens. J Clin Microbiol 1999; 37: 1366–1339. [ Links ]
51. Woods GL, Fish G, Plaunt M, Murphy T. Clinical evaluation of Difco ESP culture system II for growth and detection of mycobacteria. J Clin Microbiol 1997; 35: 121–124. [ Links ]
52. Williams–Bouyer N, Yorke R, Lee HI, Woods GL. Comparison of the BACTEC MGIT 960 and ESP culture system II for growth and detection of mycobacteria. J Clin Microbiol 2000; 38: 4167–4170. [ Links ]
53. Drobniewsky FA, Caws M, Gibson A, Young D. Modern laboratory diagnosis of tuberculosis. Lancet Infect Dis 2003; 3: 141–147. [ Links ]
54. Somoskovi A, Kodmon C, Lantos A, Bártfai Z, Tamási L, Füzy J, et al. Comparison of recoveries of Mycobacterium tuberculosis using the automated BACTEC MGIT 960 system, the BACTEC 460 TB system, and Lowenstein–Jensen medium. J Clin Microbiol 2000; 38: 2395–2397. [ Links ]
55. Githui WA, Wilson SM, Drobniewski FA. Specificity of IS6110–based DNA fingerprinting and diagnostic techniques for Mycobacterium tuberculosis complex. J Clin Microbiol 1999; 37: 1224–1226. [ Links ]
56. Watterson SA, Drobniewsky FA. Modern laboratoy diagnosis of mycobacterial infections. J Clin Pathol 2000; 53: 727–732. [ Links ]
57. Hernandez SM, Morlock GP, Butler WR, Crawford JT, Cooksey RC. Identification of Mycobacterium species by PCR–restriction fragment length polymorphism analyses using fluorescence capillary electrophoresis. J Clin Microbiol 1999; 37: 3688–3692. [ Links ]
58. Telenti A, Marchesi F, Balz M, Bally F, Böttger EC, Bodmer T. Rapid identification of mycobacteria to the species level by polymerase chain reaction and restriction enzyme analysis. J Clin Microbiol 1993; 31: 175–178. [ Links ]
59. Leao SC, Bernardelli A, Cataldi A, Zumarraga M, Robledo J, Realpe T, et al. Multicenter evaluation of mycobacteria identification by PCR restriction enzyme analysis in laboratories from Latin America and the Caribbean. J Microbiol Methods 2005; 61: 193–199. [ Links ]
60. Stender H, Lund K, Petersen KH, Rasmussen OF, Hongmanee P, Miorner H, et al. Fluorescence in situ hybridization assay, using peptide nucleic acid probes for differentiation between tuberculous and nontuberculous mycobacterium species in smears of mycobacterium cultures. J Clin Microbiol 1999; 37: 2760–2765. [ Links ]
61. Drobniewsky FA, More PG, Harris S. Differentiation of Mycobacterium tuberculosis complex and nontuberculous mycobacterium liquid cultures by using peptide nucleic acid–fluorescence in situ hibridization probes. J Clin Microbiol 2000; 38: 444–447. [ Links ]
62. Ninet B, Rohner P, Metral C, Auckenthaler R. Assessment of use of the COBAS AMPLICOR system with BACTEC 12B cultures for rapid detection of frequently identified mycobacteria. J Clin Microbiol 1999; 37: 782–784. [ Links ]
63. Keeler E, Perkins MD, Small P, Hanson C, Reed S, Cunningham J, et al. Reducing the global burden of tuberculosis: the contribution of improved diagnostics. Nature 2006; 444 (Suppl. 1): 49–57. [ Links ]
Recibido: febrero 12 de 2008
Aceptado: marzo 31 de 2008