Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Iatreia
Print version ISSN 0121-0793
Iatreia vol.22 no.2 Medellín Apr./June 2009
ARTÍCULO DE REVISIÓN
Staphylococcus aureus resistente a meticilina: bases moleculares de la resistencia, epidemiología y tipificación
Methycillin–resistant Staphylococcus aureus: molecular bases of resistance; epidemiology, and typification
Judy Natalia Jiménez Quiceno1,2; Margarita María Correa Ochoa1
1. Docente Escuela de Microbiología, Grupo de Microbiología Molecular, Universidad de Antioquia.
2. Estudiante de Doctorado en Ciencias Básicas Biomédicas Universidad de Antioquia.
Dirección para la correspondencia: judynatalia@yahoo.com
Resumen
Staphylococcus aureus es responsable de un amplio espectro de cuadros clínicos que van desde infecciones en la piel y los tejidos blandos hasta enfermedades sistémicas muy graves que amenazan la vida; tiene gran importancia en la comunidad y está comúnmente implicado en infecciones nosocomiales. Además, un alto porcentaje de individuos está colonizado por S. aureus, lo cual constituye un factor de riesgo para su diseminación. S. aureus tiene gran capacidad de adquirir resistencia a los antimicrobianos y en la actualidad se encuentran cepas resistentes a la mayoría de los antibióticos disponibles; en particular, su resistencia a la meticilina, inicialmente en el ambiente hospitalario (HA–MRSA) y posteriormente en la comunidad (CA–MRSA), ha dificultado aún más el control mundial de este microorganismo. Los estudios de epidemiología molecular han permitido entender mejor las relaciones evolutivas de las cepas y definir el origen de los clones durante brotes epidémicos. En Colombia se sabe poco sobre la epidemiología de S. aureus y aún menos sobre su comportamiento en la comunidad. Por ello, estudios de vigilancia epidemiológica que involucren la tipificación molecular de las cepas permitirán comprender mejor su epidemiología y establecer estrategias más certeras de tratamiento y control.
Palabras clave: Epidemiología molecular, Resistencia a la meticilina, S. aureus meticilino–resistente adquirido en la comunidad (CA–MRSA), S. aureus meticilino–resistente asociado con los hospitales (HA–MRSA), Tipificación molecular
SUMMARY
Staphylococcus aureus is responsible for a wide variety of clinical manifestations ranging from skin and soft tissue infections to severe systemic and life threatening diseases; it is of relevance in the community and is commonly associated with nosocomial infections. Additionally, a high percentage of the population is colonized by S. aureus, which constitutes a risk factor for its dissemination. S. aureus has great capacity to acquire antimicrobial resistance, and currently there are resistant strains to the majority of available antibiotics; in particular, its resistance to methycillin, initially in the hospital environment (Hospital associated methycillin resistant S. aureus, HA–MRSA) and later in the community (Community acquired methycillin resistant S. aureus, CA–MRSA), has made even more difficult the worldwide control of this microorganism. Molecular epidemiological studies have provided a better understanding of the evolutionary relationships of the strains, and the definition of the clonal source during epidemic outbreaks. In Colombia, very little is known about S. aureus epidemiology and even less about its behavior in the community. Therefore, studies based on epidemiological surveillance, involving molecular typing, will lead to a better understanding of its epidemiology, making it possible the design of more assertive control strategies.
Key words: CA– MRSA, HA–MRSA, Methicillin resistance, Molecular epidemiology, Molecular typing, Staphylococcus aureus
INTRODUCCIÓN
Staphylococcus aureus ha sido reconocido como uno de los principales patógenos humanos, responsable de gran cantidad de infecciones en la comunidad y de un número importante de infecciones nosocomiales.1,2 Dada la diversidad de factores de virulencia que posee, tanto bioquímicos como estructurales, se lo considera como un patógeno perfecto, equipado para colonizar, invadir y diseminarse; dichos factores son, en parte, los
responsables del amplio espectro de manifestaciones clínicas que puede causar.3,4 Su gran capacidad de adquirir elementos exógenos por transferencia horizontal, tanto dentro de la especie como con otras especies, le permite ser un patógeno exitoso y adaptarse fácilmente al medio y a los agentes antimicrobianos, mediante la adquisición de factores de resistencia a antibióticos codificados por plásmidos, secuencias de inserción y transposones;5,6 dicha resistencia ha agudizado el problema mundial de las infecciones por esta bacteria.6 El conocimiento del primer mecanismo de resistencia de S. aureus data de los años 40 del siglo XX, cuando se introdujo la penicilina como antibiótico para tratar las infecciones por este patógeno. Desde entonces, emergieron rápidamente cepas resistentes productoras de penicilinasas, codificadas por un gen denominado blaZ; la alteración de los genes represores, blaR1 y blaI, provoca su producción.7 Luego, en los años 50, se dispuso de los primeros β–lactámicos estables a la acción de las penicilinasas, tales como las cefalosporinas de primera generación y la meticilina semisintética; sin embargo, poco tiempo después aparecieron cepas de S. aureus resistentes a meticilina, conocidas como MRSA (por la sigla en inglés de Methicillyn resistant S. aureus), observadas por primera vez en Inglaterra en 1961, en aislamientos nosocomiales. Desde entonces, las cepas de MRSA se han diseminado gradualmente a todo el mundo, y se las encuentra principalmente en las instituciones que brindan atención especializada.2,8 En las primeras décadas a partir de su aparición, estas cepas se asociaron al ambiente hospitalario por lo que se las conoció como HA–MRSA (por la sigla en inglés de Hospital acquired MRSA);2 en los años 90 aparecieron los primeros informes de MRSA adquiridos en la comunidad y se los denominó CA–MRSA (por la sigla en inglés de Community acquired MRSA);9,10 fueron reportados con mayor frecuencia en niños y adultos jóvenes, sin factores de riesgo relacionados con los HA–MRSA.2 Los mecanismos que originaron las cepas CA–MRSA siguen siendo motivo de gran controversia.11 En la actualidad, la oxacilina continúa siendo el medicamento de elección para combatir las infecciones por S. aureus; no obstante, el panorama parece ser bastante preocupante con el nivel de resistencia reportado para la meticilina; lo es aún más, si se considera que en los años 1996 y1997 surgieron las primeras cepas con sensibilidad disminuida a los glicopéptidos (vancomicina y teicoplanina), que son los antibióticos de elección para el control del MRSA.12 Estas cepas se denominaron de la siguiente manera por sus siglas en inglés: VISA (Vancomycin intermediate S. aureus), y GISA (Glycopeptide intermediate S. aureus).13
La capacidad de S. aureus para desarrollar mecanismos de resistencia hace necesario el conocimiento de su epidemiología como una contribución al establecimiento de medidas racionales de prevención y control. La información contenida en esta revisión proviene, en su mayor parte, de publicaciones encontradas en Medline– PubMed, seleccionadas con base en la calidad del trabajo realizado y en los aportes de sus resultados al avance del conocimiento sobre las bases moleculares de la resistencia de S. aureus a meticilina y sus implicaciones, la evolución genética de S. aureus a HA–MRSA y CAMRSA y la situación de este patógeno en Colombia; se revisaron, además, aspectos generales de los métodos fenotípicos y moleculares utilizados para su tipificación. Aumentar el conocimiento de la comunidad científica y del público en general sobre la situación del MRSA permitirá crear una conciencia participativa en el establecimiento de medidas certeras para el manejo de las infecciones producidas por esta bacteria.
Bases moleculares de la resistencia del S. aureus a la meticilina
La resistencia del S. aureus a meticilina se debe a la presencia de una proteína alterada de unión a la penicilina (PBP2a, por la sigla en inglés de Penicillin binding protein 2a), codificada por el gen mecA.14 Esta proteína, de 76 kDa, se caracteriza por su afinidad disminuida para los antibióticos β–lactámicos y por tener solo un dominio transpeptidasa, por lo que necesita apoyarse en la actividad de transglicosilación de la proteína normal (PBP2) para la formación del peptidoglicano de la pared celular.15
El gen mecA se encuentra en un elemento genético móvil conocido como el casete cromosómico (Staphylococcal chromosome cassette mec, SCCmec),15,16 que se inserta en un sitio específico del cromosoma bacteriano (attBSCC), cerca del origen de replicación de S. aureus. Esta característica es de gran relevancia porque le permite replicarse en forma temprana y transcribir los genes de resistencia importados.17 Dada la complejidad de este mecanismo de resistencia, se ha considerado poco probable que haya surgido por la presión selectiva ejercida por la introducción de los medicamentosβ – lactámicos.11 Aún no está claro el origen del gen mecA pero se ha propuesto que pudo haber evolucionado mucho tiempo atrás, en especies de estafilococo libres de penicilinasas que se encontraban bajo presión selectiva por la penicilina, cuando se inició su utilización intensiva como medida profiláctica en veterinaria, luego de su introducción para uso en humanos. En este contexto, se presenta el Staphylococcus sciuri, un colonizador frecuente de animales domésticos, como una de las especies implicadas;17 en él se identificó un gen mecA homólogo, con un alto porcentaje de similitud con el de S. aureus.18 La mayoría de los aislamientos de esta especie fueron susceptibles a antibióticos β–lactámicos, incluyendo la meticilina; sin embargo, se ha demostrado que las cepas susceptibles pueden ser convertidas a resistentes en el laboratorio, lo que revela su facilidad de adquirir resistencia.19
Estructura del casete cromosómico SCCmec
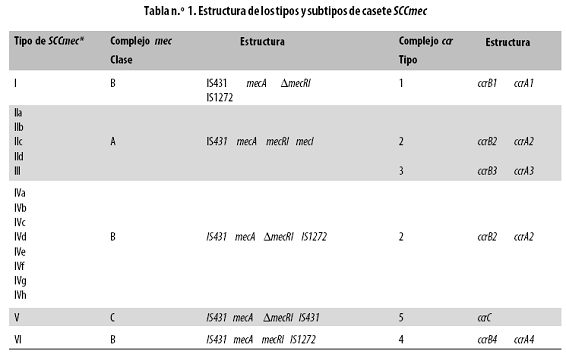
El SCCmec tiene tres componentes genéticos esenciales; son ellos: el complejo de genes mec, el complejo de genes ccr, que codifica para recombinasas, y una región conocida como J (junkyard), conformada por elementos cuya constitución puede variar, sin ser todos ellos fundamentales.11,20 El complejo mec está compuesto por el gen mecA y sus genes reguladores, mecR1 y mecI, que pueden aparecer intactos o truncados en diferentes aislamientos; cuando estos genes reguladores están intactos y son completamente funcionales parece que confieren una mayor represión en la expresión de la PBP2a. El complejo mec contiene además el elemento de inserción IS431mec, que ha sido frecuentemente asociado con genes que codifican resistencia a diversos antibióticos y al mercurio; en algunos aislamientos se encuentra también el IS1272.21 Con base en su estructura se han identificado cuatro clases de complejo mec: A, B, C, D (Tabla n.º 1).
Las clases A y B son más comunes en S. aureus y la C en Staphylococcus haemolyticus; sin embargo, se detectó un nuevo tipo de casete SCCmec en S. aureus que presentó la clase C.22 La clase D se ha detectado solo en Staphylococcus hominis.21
El complejo ccr está compuesto por genes que codifican para recombinasas responsables de la movilización del SCCmec; ellas median su integración y escisión del cromosoma.20 Hasta la fecha se han reportado cinco tipos de ccr; cuatro de ellos son alotipos denominados del 1 al 4, que comparten aproximadamente el 80% de la identidad y presentan los genes ccrA y ccrB; el quinto tipo, recientemente descrito, denominado ccr5 o ccrC, presenta el gen ccrC y no se encuentra relacionado con los genes ccrA y ccrB (Tabla n.º 1).16,22 El resto del casete cromosómico contiene secuencias de la región J, o Junkyard, que comprende tres fragmentos denominados J1, J2 y J3; puede contener plásmidos o transposones portadores de genes de resistencia a antibióticos noβ –lactámicos y a metales pesados.20El fragmento J1 se localiza entre el complejo ccr y el extremo cromosómico flanqueante derecho; el J2, entre los complejos ccr y mec, y el J3, entre el complejo mec y el orfX. Aunque los fragmentos J no son específicos de cada tipo de SCCmec, se observa que algunos de ellos se encuentran más comúnmente en ciertos tipos de SCCmec.23
Se ha reportado que el SCCmec no está restringido a la movilidad del gen mecA; él posee elementos adicionales, denominados no mec, que contribuyen a la supervivencia y al potencial patogénico de S. aureus. Entre los elementos no mec se encuentran secuencias que codifican para resistencia a metales pesados como el mercurio (SCCmer), o al ácido fusídico (SCC MSSA 476, por la sigla en inglés de Staphylococcal cassette chromosome methicillin–susceptible S. aureus) secuencias para biosíntesis capsular (SCCcap1), para la protección del ADN por sistemas de modificación–restricción (SCC CI) y para el catabolismo de la arginina (ACME, por la sigla en inglés de Arginine catabolic mobile element). La mayoría de estos elementos han sido reportados en estafilococos coagulasa negativa lo que sugiere, nuevamente, su transferencia desde estas especies a S. aureus.24
Tipos de casete cromosómico mec (SCCmec)
Diferentes combinaciones de las clases del complejo mec y de los alotipos del complejo ccr generan varios tipos de casete SCCmec (Tabla n.º 1). Además, variaciones en la región J, aun teniendo la misma combinación de los complejos mec y ccr, definen los subtipos o variantes.23En la actualidad se han descrito 6 tipos de SCCmec designados de I a VI; sus tamaños varían de 20 kb a 68 kb.20 Los tipos I, II y III se han asociado frecuentemente con infecciones nosocomiales; en contraste, los tipos IV y V se han encontrado en cepas de MRSA de la comunidad.2
Recientemente se describió una variante del SCCmec, similar al tipo IV, pero portadora de un nuevo alotipo, el ccrAB4, que fue denominado tipo VI.25
El SCCmec tipo IV es el más variable: hasta el momento se han descrito 8 subtipos que comprenden del IVa al IVh;26 el SCCmec tipo II contiene 5 subtipos, del IIa al IIe.17 Los tipos II y III se caracterizan por contener muchos determinantes de resistencia a antibióticos noβ –lactámicos y por su mayor tamaño, el II de 52 kb y el III de 66 kb. Los tipos I, IV y V solo contienen genes para recombinasas, genes estructurales y reguladores de la resistencia a meticilina, carecen de elementos transponibles y de genes que codifiquen para resistencia a antibióticos no betalactámicos;8 tienen un menor tamaño: el I es de 34 kb, el IV de 20 a 24 kb y el V de 28 kb, lo cual parece conferirles una ventaja evolutiva al facilitar su transferencia horizontal entre diferentes especies.27 El tipo V se diferencia de los otros por contener un grupo de genes foráneos que codifican para un sistema de restricción–modificación que parece estar involucrado en la estabilización del elemento en el cromosoma. En la década de los años 70, el elemento tipo IV se asoció con Staphylococcus epidermidis; sin embargo, solo a partir de los años 90 se reportó su asociación frecuente con S. aureus, lo que sugirió un evento de transferencia horizontal de mecA entre dos especies de estafilococo diferentes, una patógena y otra comensal.8
Origen del SCCmec
No se ha informado el elemento SCCmec en bacterias de otros géneros1,2 y su origen es aún desconocido;2,21,28 así mismo, se desconoce su mecanismo de transferencia pero está demostrado que ocurre en forma horizontal entre especies de Staphylococcus.19,28,29 Existe una teoría según la cual los genes ccr y mec fueron transferidos inicialmente a un estafilococo coagulasa negativa a partir de una fuente aún desconocida,25,28 y que en esa especie sufrieron una deleción de los genes reguladores mec antes de ser transferidos a S. aureus y dar origen al MRSA.17 La presencia de la secuencia de inserción IS1272, característica de Staphylococcus haemolyticus, en los tipos SCCmec I y IV, sugiere que esta secuencia fue transferida de esta especie a S. aureus.2 Existe evidencia de que la historia evolutiva del SCCmec es independiente de la del gen mecA. Esto lo demuestra el hecho de que el SCCmec puede existir en forma independiente del gen mecA, y que este último puede existir en ausencia de los elementos que componen el SCCmec, como ocurre en especies de estafilococos coagulasa negativa.21
Staphylococcus aureus sensible a meticilina (MSSA), S. aureus resistente a meticilina (MRSA) nosocomial y de comunidad
La presión de los antibióticos ha favorecido la evolución genética del S. aureus, con la consecuente aparición de cepas con diferentes determinantes de virulencia y patrones variables de susceptibilidad, que difieren no solo epidemiológicamente sino también en los tipos de infecciones que producen; así, se reportan cepas de S. aureus sensibles a meticilina (MSSA), de S. aureus resistentes a meticilina asociadas al ambiente hospitalario o nosocomiales (HA–MRSA) y de S. aureus resistente a meticilina asociado a la comunidad (CA–MRSA).1,2 MSSA se aísla más comúnmente en pacientes con infecciones adquiridas en la comunidad y causa principalmente infecciones de la piel, osteomielitis hematógena, artritis séptica y neumonía con derrame. Produce además varios síndromes asociados a toxinas, como el de choque tóxico, el de piel escaldada e intoxicaciones alimentarias.1 En años recientes se ha observado un incremento progresivo de las cepas de S. aureus resistentes a meticilina (MRSA), que se ha vuelto endémico en muchos hospitales, sobre todo en los que brindan atención compleja y manejan un alto número de camas, donde puede ser el agente causal de cerca del 50% de las infecciones nosocomiales.30 Se ha asociado el HA–MRSA a varios factores de riesgo, entre los que se destacan, hospitalización reciente y permanencia en unidades de cuidados intensivos (UCI);30 con el agravante de que los trabajadores de la salud y los pacientes colonizados por MRSA pueden convertirse en nuevos focos de infección.31 Además de deteriorar la salud de los individuos, las infecciones por HA–MRSA generan una gran carga para las instituciones de salud, por su fuerte asociación con altas tasas de morbilidad y mortalidad y por el incremento en los costos de la estancia hospitalaria. Se ha calculado que el costo promedio de las hospitalizaciones de pacientes con infecciones por MRSA es 30% más alto que el de los pacientes con otras dolencias.32 En la década de los años 90 adquirió gran importancia la emergencia de cepas de MRSA en la comunidad (CA–MRSA). Los primeros casos se reportaron en Australia9 y Estados Unidos,10 en 1993 y 1998, respectivamente; desde entonces se ha incrementado su aparición mundial. Las cepas de CA–MRSA poseen numerosos factores de virulencia, entre ellos la toxina Panton–Valentine (PVL), una leucocidina bicompuesta responsable de destruir leucocitos y producir necrosis tisular; la codifican dos genes cotranscritos (lukS–PV y lukF–PV), que residen en un profago.33 La presencia de la PVL se ha asociado con agravamiento de la enfermedad en adultos jóvenes y niños; las infecciones en la piel y los tejidos blandos evolucionan para dar lugar a la aparición de osteomielitis crónica y neumonía necrosante.8,33,34 CAMRSA se caracteriza por causar brotes en comunidades cerradas, que afectan a grupos familiares, militares, reclusos, niños en guarderías y deportistas.35 Las cepas de CA–MRSA difieren de las de HA–MRSA en su susceptibilidad a otros antibióticos: por lo general las cepas nosocomiales (HA–MRSA) son resistentes a todos los antibióticos β–lactámicos y a muchos no β –lactámicos.8 Ha habido controversia sobre cómo definir la infección por CA–MRSA; muchos autores han utilizado el término para referirse a la colonización o infección ocurrida en la comunidad, lo cual no implica necesariamente que las cepas se hayan adquirido en ella, pues pudo tratarse de una exposición previa.8 Se sabe que cepas de S. aureus, incluyendo MRSA, pueden colonizar al individuo por períodos largos sin ser detectadas y presentarse la infección un tiempo después; este hecho es el que dificulta determinar el origen real de las cepas que causan infección en la comunidad.8 Lo anterior condujo al establecimiento de pautas para lograr una definición precisa de lo que constituye una infección por CA–MRSA; para ello debe haber evidencias claras de su inicio en la comunidad y de que el individuo afectado no presenta ninguno de los siguientes factores de riesgo o contacto estrecho con personas que los presenten: hospitalización reciente, cirugía previa, diálisis, visitas a hospitales, exposición reciente a antibióticos, enfermedad crónica, inyecciones, residencia prolongada en instituciones de cuidado.10,36 La dificultad para definir el origen de estas cepas ha generado un auge en el desarrollo de estudios moleculares, que han contribuido, entre otras cosas, a distinguir entre las cepas realmente adquiridas en la comunidad y aquellas de origen nosocomial.35,37La evolución genética de las cepas de HAMRSA y CA–MRSA también ha mostrado grandes diferencias. Inicialmente las cepas de HA–MRSA exhibieron una expansión clonal; por más de un decenio un solo clon fue el responsable de las epidemias de HAMRSA en los diversos hospitales, pero en los años subsiguientes empezaron a surgir y a extenderse por todo el mundo nuevos clones. En la actualidad existe gran cantidad de clones epidémicos de HA–MRSA que muestran grandes diferencias genéticas; así se ha demostrado por métodos como la secuenciación de múltiples locus (Multilocus sequence typing– MLST), que portan diferentes tipos de SCCmec. Este comportamiento contrasta con la evolución de las cepas de CA–MRSA, que en sus orígenes mostraron una mayor diversidad genética pero en la actualidad solo unos pocos clones son responsables de las epidemias de CA–MRSA en el á mbito mundial.11
Recientemente se han descrito cepas de MSSA derivadas de MRSA que sufren una escisión del SCCmec pero conservan otros determinantes de resistencia a antibióticos no β–lactámicos,38,39 lo que hace muy importante su seguimiento porque pueden presentar un fenotipo inusual de resistencia. En contraste con la información mundial disponible sobre MRSA, existe un gran vacío en el conocimiento de la epidemiología de estas cepas de MSSA derivadas de MRSA, lo que enfatiza aún más la pertinencia de considerar su inclusión en estudios epidemiológicos que permitan dilucidar su papel en la diseminación de la resistencia.
Tipificación de MRSA
La tipificación de MRSA se puede hacer por métodos fenotípicos y moleculares. Los primeros han sido útiles pero algunos pueden presentar desventajas por ser dispendiosos y por las limitaciones de su poder discriminatorio; además, algunos de ellos no son aplicables a todas las cepas de S. aureus.40 Los métodos moleculares de tipificación se han empleado recientemente, con mayor frecuencia, en estudios de epidemiología molecular de MRSA, lo que ha permitido entender mejor las relaciones evolutivas de estos clones.41 Entre los métodos empleados para tipificar MRSA se encuentran los siguientes: la ribotipificación,42 los patrones de macrorrestricción de ADN cromosómico por electroforesis de campo pulsado (PFGE, por la sigla en inglés de Pulsed field gene electrophoresis) empleando la enzima SmaI 43,44 las técnicas basadas en PCR 20,23,45 y, más recientemente, las basadas en el secuenciamiento de ADN.46,47
La PFGE empleando la enzima SmaI es el método más comúnmente utilizado para estudios de la epidemiología molecular de S. aureus y ha sido de gran valor en estudios de brotes nosocomiales.48 Inicialmente esta técnica estuvo restringida a estudios locales y de corto plazo, debido a la falta de reproducibilidad entre laboratorios; sin embargo, gracias a la estandarización de la metodología para hacer la electroforesis y al esquema de interpretación de los patrones producidos siguiendo los criterios de Tenover y colaboradores,49 se pudieron adelantar estudios multicéntricos que han sido de gran utilidad para determinar las relaciones genéticas de las cepas de MRSA; en la actualidad se cuenta con un software que permite comparar los perfiles obtenidos en la PFGE e identificar a qué linaje de MRSA, entre los reportados, pertenecen las cepas en estudio.50
Se ha utilizado ampliamente la estrategia de tipificar el MRSA combinando la PFGE con otros métodos; en particular, se empleó la combinación con el polimorfismo del gen mec y de los patrones de inserción del transposón Tn455 para tipificar alrededor de 3.000 aislamientos de MRSA provenientes de varias regiones del mundo, lo que permitió identificar seis clones epidémicos: Ibérico, Brasilero, Pediátrico, Húngaro, NewYork /Japón y EMRSA– 16, que derivaron sus nombres de las regiones donde se detectaron primero o de alguna propiedad epidemiológica.2 recuentemente se usan estos clones como referentes de tipificación en estudios epidemiológicos.51,52
Recientemente, debido a la mayor disponibilidad y accesibilidad del secuenciamiento de ADN, los métodos de tipificación basados en este principio han ganado en popularidad entre los investigadores y se han convertido en los más utilizados para la tipificación de MRSA. Estos métodos han incrementado la reproducibilidad entre los laboratorios, así como la rapidez para obtener los resultados. Uno de ellos es la tipificación de secuencias multilocus (MLST, por la sigla en inglés de Mutilocus sequence typing), que está basada en la amplificación y secuenciación de fragmentos internos de siete genes esenciales, no ligados (house keeping). Su principal ventaja frente a la PFGE es que, al utilizar secuencias, no da lugar a las ambigüedades que se pueden presentar con la PFGE. Además, es una técnica muy estandarizada y cuenta con un sitio web que contiene información de más de 1.500 aislamientos de humanos y animales, provenientes de cerca de 40 países, lo que permite comparar las secuencias obtenidas con las reportadas en la base de datos para obtener el perfil alélico. Aunque la MLST brinda información acerca del linaje de las cepas, lo cual es muy importante para entender la epidemiología de los MRSA, esta técnica no es muy apropiada para investigar brotes en el ámbito clínico.48
Otro método basado en secuenciamiento, que ha sido ampliamente utilizado en estudios de epidemiología molecular de MRSA, es la tipificación del gen de la proteína A. Se lo denomina Staphyloccocus protein A (spa), involucra la amplificación y secuenciamiento de la región polimórfica X, una secuencia corta repetida del gen de la proteína A, un superantígeno de S. aureus.2,46 Esta región posee un alto grado de polimorfismo y, por consiguiente, es potencialmente adecuada para estudiar brotes por su alto poder de discriminación. La información empleada para la tipificación de spa se obtiene de un solo locus, en contraste con MLST que combina la información de siete locus, lo que hace que esta última sea mucho más costosa.53
Estudios de tipificación molecular de MRSA han permitido concluir que para una mejor caracterización de estas cepas se requiere definir no solo su perfil genético, sino también identificar los tipos estructurales del casete cromosómico SCCmec.54 Es así como recientemente se han descrito numerosos métodos de PCR que permiten detectar y tipificar MRSA con base en el SCCmec.20,23,45,55 La PCR ofrece ventajas como la rapidez, sensibilidad, especificidad y menor costo, al compararla con métodos basados en secuenciación; además, permite detectar MRSA directamente de muestras que contienen una mezcla de microorganismos.56 Estudios realizados por Ito y colaboradores,16 dirigidos a tipificar cepas de MRSA, permitieron dilucidar las diferencias en la estructura del SCCmec; estos autores diseñaron una PCR basada en las variaciones de tamaño y composición genética de los complejos mecA y ccr, la cual permitió identificar los 5 tipos de SCCmec. Francois y colaboradores55 desarrollaron una
PCR en tiempo real, con iniciadores (primers) dirigidos a los genes de las recombinasas (ccr); la desventaja de esta estrategia consiste en que solo identifica 4 tipos de casete y deja por fuera el tipo V.
Oliveira y colaboradores45 desarrollaron una PCR múltiple (multiplex PCR), basada solo en las variaciones de las secuencias del complejo mecA, sin tener en cuenta el complejo ccr. El ensayo determina los tipos de SSCmec al identificar 8 locus situados corriente arriba y corriente abajo del gen mecA, principalmente en la región J. Este método permitió la discriminación rápida de los tipos estructurales del elemento mec; sin embargo, tiene la limitación de que no detecta todos los tipos y subtipos descritos de casete SCCmec, como son el tipo V y los subtipos IVa, b, c y d. Posteriormente, Zhang y colaboradores20 describieron una PCR múltiple que detecta los tipos I a V y varios subtipos. Más recientemente, Kondo y colaboradores23 diseñaron una estrategia basada en seis PCR múltiples, que supera las limitaciones de las PCR anteriores, al permitir la discriminación y clasificación de la mayoría de los tipos y subtipos descritos hasta la fecha. Entre las ventajas de la prueba están sus altas sensibilidad y especificidad y su formato de PCR múltiple, que la hace muy práctica al combinar la rapidez con la relativa facilidad técnica.
La tipificación del SCCmec es primordial para la identificación adecuada de los clones de MRSA en estudios epidemiológicos; no obstante, se han presentado dificultades relacionadas con la unificación de criterios para determinar los tipos. Es así como Oliveira y colaboradores45 denominaron los elementos SCCmec basados principalmente en diferencias de la región J, mientras que Zhang y colaboradores20 y Chongtrakool y colaboradores57 asignaron los tipos con base en la determinación de clases del complejo mec y de tipos del complejo ccr. Por lo anterior, Chongtrakool y colaboradores57 han propuesto establecer una nueva nomenclatura que permita unificar criterios para la denominación de tipos de SCCmec, que sea aceptada y puesta en práctica por los investigadores en todo el mundo.
En vista de la amplia variedad de métodos disponibles para tipificar MRSA, la elección del más adecuado depende de las necesidades y del tipo de pregunta que se quiera responder. El método óptimo debe mostrar una alta capacidad de discriminación y de reproducibilidad y ser fácil de usar, rápido, asequible, de costo razonable y fácil interpretación.48,53
Recientemente se han puesto en práctica métodos que combinan la tipificación del cromosoma bacteriano, mediante PFGE o por estrategias de secuenciación, con la tipificación del SCCmec por PCR; estos métodos tienen gran especificidad y han resultado muy útiles en estudios epidemiológicos y, principalmente, en los de tipo evolutivo.53
La estrategia recomendada para optimizar el proceso de tipificación, lograr una mayor discriminación de las cepas y obtener información relevante que permita establecer medidas preventivas y de control, consiste en emplear inicialmente la PFGE y luego, en los aislamientos que muestran clonalidad, hacer el análisis de las cepas por SCCmec o MLST. Se podría emplear la tipificación spa en lugar de PFGE si no existieran limitaciones por los costos de la secuenciación. La tipificación spa provee datos concordantes con los obtenidos por MLST o PFGE; sin embargo, su uso no obvia la necesidad de efectuar la tipificación del SCCmec para determinar la clonalidad de los aislamientos.48
Situación en Colombia
En Colombia el primer estudio publicado de caracterización molecular de S. aureus resistente a meticilina se llevó a cabo entre 1996 y 1998, como parte de una estrategia global de vigilancia de la resistencia, con muestras de hospitales de Bogotá y Manizales. En ese trabajo la tipificación molecular mostró que todos los aislamientos compartían propiedades idénticas a las de un solo clon, el denominado Clon pediátrico, que había sido descrito en los años 90 en Europa, Nueva York y Sur América. Sin embargo, los aislamientos de Colombia diferían del Clon pediátrico en su amplia resistencia a muchos medicamentos y en que provenían de pacientes de todas las edades.52 En el año 2005, Cruz y colaboradores51 evaluaron 200 muestras de MRSA recolectadas entre 2001 y 2003, provenientes en su mayoría de hospitales de Bogotá y Cali (48% y 45%, respectivamente); estos autores no detectaron el Clon pediátrico, sino solamente el denominado Clon chileno.Este fue el primer reporte de dicho clon en Colombia, lo cual, según los autores, indicaba un cambio en la población genética de MRSA en el país.
Recientemente, Álvarez y colaboradores58 hicieron el primer reporte de CA–MRSA en Colombia; posteriormente Cortés y colaboradores,59 en un estudio realizado en Bogotá, a partir de una base de datos de aislamientos ambulatorios de los años 2001 a 2005, reportaron la presencia de S. aureus sugestivo de presentar un perfil de CA–MRSA, hecho que tiene implicaciones importantes en salud pública. Además, en 2008 se documentó la presencia de CA–MRSA en varias instituciones hospitalarias de Bogotá.60,61
En otras capitales, como Medellín, que tienen hospitales de alta complejidad, solo se conoce un estudio longitudinal, llevado a cabo por Vesga y colaboradores62 en el Hospital Universitario San Vicente de Paúl (HUSVP), entre los años 1987 y 1993. En ese trabajo se demostró, por métodos fenotípicos, resistencia a la penicilina en el 100% de las cepas de S. aureus analizadas y resistencia a la meticilina en el 10%; de estas últimas, el 60% fueron de MRSA adquirido en la comunidad. Recientemente, el Grupo de Microbiología Molecular de la Universidad de Antioquia inició un estudio dirigido a caracterizar clínica, molecular y epidemiológicamente las cepas de MRSA, tanto de origen hospitalario como de la comunidad; hasta la fecha, los resultados sugieren la aparición de cepas de CA–MRSA en el ambiente hospitalario, tal como se ha observado en Bogotá (datos no publicados). Otro estudio hecho recientemente por este grupo, documentó la presencia de MRSA colonizando las manos en población general.63 En la actualidad la resistencia de S. aureus a meticilina ha aumentado considerablemente en los hospitales de mediana y alta complejidad, y se encuentran porcentajes de resistencia superiores al 40%.64
Informes internacionales indican que hay una variación considerable en la prevalencia de MRSA entre países y hospitales, incluso dentro de una misma región.8 En Colombia, el uso de antibióticos no está regulado, es indiscriminado y muchas veces inadecuado, por lo que las condiciones son ideales para el desarrollo de la resistencia; por ello, la situación en este país es preocupante. La prevalencia de MRSA asociada al ambiente hospitalario es alta,51,52,62 es poco lo que se sabe sobre su epidemiología y aún menos sobre el comportamiento en la comunidad. Lo anterior pone de presente la necesidad de trabajar localmente en este campo para poder conocer la magnitud real de la prevalencia de S. aureus meticilino–resistente y la dinámica de su transmisión nosocomial y comunitaria. Una mayor comprensión de la epidemiología molecular de S. aureus posibilitará el establecimiento de medidas eficientes en el manejo racional de las infecciones que produce, lo cual redundará en el mejoramiento de las condiciones de salud de la población.
REFERENCIAS BIBLIOGRÁFICAS
1. Moreillon P, Que Y, Glauser M. Staphylococcus aureus. In: Mandell GL, Bennett JE, Dolin R, eds. Principles and Practice of Infectious Diseases, 6ª ed. Philadelphia: Churchill–Livingston; 2005. p. 2321–2351. [ Links ]
2. Aires de Sousa M, de Lencastre H. Bridges from hospitals to the laboratory: genetic portraits of methicillin–resistant Staphylococcus aureus clones. FEMS Immunol Med Microbiol 2004; 40: 101–111. [ Links ]
3. Lowy FD. Staphylococcus aureus infections. N Engl J Med 1998; 339: 520–532. [ Links ]
4. McCormick J, Yarwood J, Schlievert P. Toxic shock syndrome and bacterial superantigens: an update. Annu Rev Microbiol 2001; 55: 77–104. [ Links ]
5. Kuroda M, Ohta T, Uchiyama I, Baba T, Yuzawa H, Kobayashi I, et al. Whole genome sequencing of methicillin–resistant Staphylococcus aureus. Lancet 2001; 357: 1218–1219. [ Links ]
6. Baba T, Takeuchi F, Kuroda M, Yuzawa H, Aoki K, Oguchi A, et al. Genome and virulence determinants of high virulence community acquired MRSA. Lancet 2002; 359: 1819–1827. [ Links ]
7. Nimmo G, Bell J, Mitchell D, Gosbell I, Pearman J, Turnidge J, et al. Antimicrobial resistance in Staphylococcus aureus in Australian teaching hospitals, 1989–1999. Microb Drug Resist 2003; 9: 155–160. [ Links ]
8. Zetola N, Francis J, Nuermberger E, Bishai W. Communityacquired methicillin–resistant Staphylococcus aureus: an emerging threat. Lancet Infect Dis 2005; 5: 275–186. [ Links ]
9. Udo E, Pearman J, Grubb W. Genetic analysis of community isolates of methicillin–resistant Staphylococcus aureus in Western Australia. J Hosp Infect 1993; 25: 97–108. [ Links ]
10. CDC, Centers for Disease Control. Four pediatric deaths from community–acquired methicillin–resistant Staphylococcus aureus – Minnesota and North Dakota, 1997–1999. Morbid Mortal Wkly Rep 1999; 48: 707–110. [ Links ]
11. De Lencastre H, Oliveira D, Tomasz A. Antibiotic resistant Staphylococcus aureus: a paradigm of adaptive power. Curr Opin Microbiol 2007; 10: 1–8. [ Links ]
12. Sieradzki K, Roberts R, Haber S, Tomasz A. The development of vancomycin resistance in a patient with methicillin–resistant Staphylococcus aureus infection. N Engl J Med 1999; 340: 517–523. [ Links ]
13. Waldvogel FA. New resistance in Staphylococcus aureus. N Engl J Med 1999; 340: 556–557. [ Links ]
14. Lindsay J, Ruzin A, Ross H, Kurepina N, Novick R. The gene for toxic shock toxin is carried by a family of mobile pathogenicity islands in Staphylococcus aureus. Mol Microbiol 1998; 29: 527–543. [ Links ]
15. Chambers H. Methicillin resistance in staphylococci: molecular and biochemical basis and clinical implications. Clin Microbiol Rev 1997; 10: 781–791. [ Links ]
16. Ito T, Katayama Y, Asada K, Mori N, Tsutsumimoto K, Tiensasitorn C, et al. Structural comparison of three types of staphylococcal cassette chromosome mec integrated in the chromosome in methicillin–resistant Staphylococcus aureus. Antimicrob Agents Chemother 2001; 45: 1323–1336. Erratum in: Antimicrob Agents Chemother 2001; 45: 3677. [ Links ]
17. Grundmann H, Aires–de–Sousa M, Boyce J, Tiemersma E. Emergence and resurgence of methicillin–resistant Staphylococcus aureus as a public–health threat. Lancet 2006; 368: 874–885. [ Links ]
18. Couto I, de Lencastre H, Severina E, Kloos W, Webster J, Hubner R, et al. Ubiquitous presence of a mecA homologue in natural isolates of Staphylococcus sciuri. Microb Drug Resist 1996; 2: 377–391. [ Links ]
19. Wu S, Lencastre H, Tomasz A. Expression of high–level methicillin resistance in Staphylococcus aureus from the Staphylococcus sciuri mecA homologue: role of mutation(s) in the genetic background and in the coding region of mecA. Microb Drug Resist 2005; 11: 215–224. [ Links ]
20. Zhang K, McClure J, Elsayed S, Louie T, Conly J. Novel multiplex PCR assay for characterization and concomitant subtyping of staphylococcal cassette chromosome mec types I to V in methicillin–resistant Staphylococcus aureus. J Clin Microbiol 2005; 43: 5026– 5033. [ Links ]
21. Nour M, Mastouri M, Ben Nejma M. Methicillin resistance in Staphylococcus aureus: emergence and molecular basis. Pathol Biol (Paris) 2005; 53: 334–340. [ Links ]
22. Ito T, Ma X, Takeuchi F, Okuma K, Yuzawa H, Hiramatsu K. Novel type V staphylococcal cassette chromosome mec driven by a novel cassette chromosome recombinase, ccrC. Antimicrob Agents Chemother 2004; 48: 2637–2651. [ Links ]
23. Kondo Y, Ito T, Ma X, Watanabe S, Kreiswirth B, Etienne J, et al. Combination of multiplex PCRs for staphylococcal cassette chromosome mec type assignment: Rapid identification system for mec, ccr, and major differences in junkyard regions. Antimicrob Agents Chemother 2007; 51: 264–274. [ Links ]
24. Hanssen A, Ericson Sollid J. SCCmec in staphylococci: genes on the move. FEMS Immunol Med Microbiol 2006; 46: 8–20. [ Links ]
25. Oliveira D, Milheiriço C, de Lencastre H. Redefining a structural variant of staphylococcal cassette chromosome mec, SCCmec type VI. Antimicrob Agents Chemother 2006; 50: 3457–3459. [ Links ]
26. Milheirico C, Oliveira D, de Lencastre H. Multiplex PCR strategy for subtyping the staphylococcal cassette chromosome mec type IV in methicillin–resistant Staphylococcus aureus: 'SCCmec IV multiplex'. J Antimicrob Chemother 2007; 60: 42–48. [ Links ]
27. Hiramatsu K, Cuil L, Kuroda M, Ito T. The emergence and evolution of methicillin–resistant Staphylococcus aureus. Trends Microbiol 2001; 9: 486–493. [ Links ]
28. Hanssen A, Kjeldsen G, Ericson SJ. Local variants of staphylococcal cassette chromosome mec in sporadic methicillin–resistant Staphylococcus aureus and methicillin–resistant coagulase–negative staphylococci: evidence of horizontal gene transfer? Antimicrob Agents Chemother 2004; 48: 285–296. [ Links ]
29. Rahman M, Kobayashi N, Alam M, Ishino M. Genetic analysis of mecA homologues in Staphylococcus sciuri strains derived from mastitis in dairy cattle. Microb Drug Resist 2005; 11: 205–214. [ Links ]
30. Hidron A, Kourbatova E, Halvosa J, Terrell B, McDougal L, Tenover F, et al. Risk factors for colonization with methicillin–resistant Staphylococcus aureus (MRSA) in patients admitted to an urban hospital: emergence of community–associated MRSA nasal carriage. Clin Infect Dis 2005; 41: 159–166. [ Links ]
31. Lu P, Chin L, Peng C, Chiang Y, Chen T, Ma L, et al. Risk factors and molecular analysis of community methicillinresistant Staphylococcus aureus carriage. J Clin Microbiol 2005; 43: 132–139. [ Links ]
32. Gould I. Costs of hospital–acquired methicillin–resistant Staphylococcus aureus (MRSA) and its control. Int J Antimicrob Agents 2006; 28: 379–384. [ Links ]
33. McClure J, Conly J, Lau V, Elsayed S, Louie T, Hutchins W, et al. Novel multiplex PCR assay for detection of the Staphylococcal virulence marker Panton–Valentine leukocidin genes and simultaneous discrimination of methicillin–susceptible from–resistant staphylococci. J Clin Microbiol 2006; 44: 1141–1144. [ Links ]
34. Lina G, Piemont Y, Godail–Gamot F, Bes M, Peter M, Gauduchon V, et al. Involvement of Panton–Valentine leukocidin–producing Staphylococcus aureus in primary skin infections and pneumonia. Clin Infect Dis 1999; 29: 1128–1132. [ Links ]
35. Ho P, Cheung C, Mak G, Tse C, Ng T, Cheung C, et al. Molecular epidemiology and household transmission of community–associated methicillin–resistant Staphylococcus aureus in Hong Kong. Diagn Microbiol Infect Dis 2007; 57: 145–151. [ Links ]
36. Dietrich D, Auld D, Mermel L. Community–acquired methicillin–resistant Staphylococcus aureus in southern New England. Child Pediatr 2004; 113: 347–52. [ Links ]
37. Aires de Sousa M, de Lencastre H. Evolution of sporadic isolates of methicillin–resistant Staphylococcus aureus (MRSA) in hospitals and their similarities to isolates of community–acquired MRSA. J Clin Microbiol 2003; 41: 3806–3815. [ Links ]
38. Donnio P, Oliveira D, Faria N, Wilhelm N, Le Coustumier A, de Lencastre H. Partial excision of the chromosomal cassette containing the methicillin resistance determinant results in methicillin–susceptible Staphylococcus aureus. J Clin Microbiol 2005; 43: 4191– 4193. [ Links ]
39. Donnio P, Février F, Bifani P, Dehem M, Kervégant C, Wilhelm N, et al. Molecular and epidemiological evidence of spread of multiresistant methicillin–susceptible Staphylococcus aureus in hospitals. Antimicrob Agents Chemother 2007; 51: 4342–4350. [ Links ]
40. Maslow J, Mulligan M, Arbeit R. Molecular epidemiology: application of contemporary techniques to the typing of microorganisms. Clin Infect Dis 1993; 17: 153–162. [ Links ]
41. Shopsin B, Kreiswirth B. Molecular epidemiology of methicillin–resistant Staphylococcus aureus. Emerg Infect Dis 2001; 7: 323–326. [ Links ]
42. Preheim L, Pitcher D, Owen R, Cookson B. Typing of methicillin resistant and susceptible Staphylococcus aureus strains by ribosomal RNA gene restriction patterns using a biotinylated probe. Eur J Clin Microbiol Infect Dis 1991; 10: 428–436. [ Links ]
43. Stranden A, Frei R, Widmer A. Molecular typing of methicillin–resistant Staphylococcus aureus: can PCR replace pulsed–field gel electrophoresis. J Clin Microbiol 2003; 41: 3181–3186. [ Links ]
44. Quelle L, Corso A, Galas M, Sordelli D. STAR gene restriction profile analysis in epidemiological typing of methicillin–resistant Staphylococcus aureus: description of the new method and comparison with other polymerase chain reaction (PCR)–based methods. Diagn Microbiol Infect Dis 2003; 47: 455–464. [ Links ]
45. Oliveira D, de Lencastre H. Multiplex PCR strategy for rapid identification of structural types and variants of the mec element in methicillin–resistant Staphylococcus aureus. Antimicrob Agents Chemother 2002; 46: 2155–2161. [ Links ]
46. Shopsin B, Gomez M, Montgomery S, Smith D, Waddington M, Dodge D, et al. Evaluation of protein A gene polymorphic region DNA sequencing for typing of Staphylococcus aureus strains. J Clin Microbiol 1999; 37: 3556–3563. [ Links ]
47. Maiden M, Bygraves J, Feil E, Morelli G, Russell J, Urwin R, et al. Multilocus sequence typing: a portable approach to the identification of clones within populations of pathogenic microorganisms. Proc Natl Acad Sci 1998; 95: 3140–3145. [ Links ]
48. Cookson B, Robinson D, Monk A, Murchan S, Deplano A, de Ryck R, et al. Evaluation of molecular typing methods in characterizing a European collection of epidemic methicillin–resistant Staphylococcus aureus strains: the HARMONY Collection. J Clin Microbiol 2007; 45: 1830–1837. [ Links ]
49. Tenover F, Arbeit R, Goering R, Mickelsen P, Murray B, Persing D, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed–field gel electrophoresis: criteria for bacterial strain typing. J Clin Microbiol 1995; 33: 2233–2239. [ Links ]
50. McDougal L, Steward C, Killgore G, Chairtram S, McAllister S, Tenover F. Pulsed–field gel electrophoresis typing of oxacillin–resistant Staphylococcus aureus isolates from the United States: Establishing a national database. J Clin Microbiol 2003; 41: 5113–5120. [ Links ]
51. Cruz C, Moreno J, Renzoni A, Hidalgo M, Reyes J, Schrenzel J, et al. Tracking methicillin–resistant Staphylococcus aureus clones in Colombian hospitals over 7 years (1996–2003): emergence of a new dominant clone. Int J Antimicrob Agents 2005; 26: 457–462. [ Links ]
52. Gomes A, Sanches I, Aires de Sousa M, Castaneda E, de Lencastre H. Molecular epidemiology of methicillinresistant Staphylococcus aureus in Colombian hospitals: dominance of a single unique multidrug–resistant clone. Microb Drug Resist 2001; 7: 23–32. [ Links ]
53. Faria N, Carrico J, Oliveira D, Ramirez M, de Lencastre H. Analysis of typing methods for epidemiological surveillance of both methicillin–resistant and methicillinsusceptible Staphylococcus aureus strains. J Clin Microbiol 2008; 46: 136–144. [ Links ]
54. Oliveira D, Tomasz A, de Lencastre H. Secrets of success of a human pathogen: molecular evolution of pandemic clones of methicillin–resistant Staphylococcus aureus. Lancet Infect Dis 2002; 2: 180–189. Erratum in: Lancet Infect Dis 2002; 2: 315. [ Links ]
55. Francois P, Renzi G, Pittet D, Bento M, Lew D, Harbarth S, et al. A novel multiplex real–time PCR assay for rapid typing of major staphylococcal cassette chromosome mec elements. J Clin Microbiol 2004; 42: 3309–3312. [ Links ]
56. Huletsky A, Giroux R, Rossbach V, Gagnon M, Vaillancourt M, Bernier M, et al. New real–time PCR assay for rapid detection of methicillin–resistant Staphylococcus aureus directly from specimens containing a mixture of staphylococci. J Clin Microbiol 2004; 42: 1875–1884. [ Links ]
57. Chongtrakool P, Ito T, Ma X, Kondo Y, Trakulsomboon S, Tiensasitorn C, et al. Staphylococcal cassette chromosome mec (SCCmec) typing of methicillinresistant Staphylococcus aureus strains isolated in 11 Asian countries: a proposal for a new nomenclature for SCCmec elements. Antimicrob Agents Chemother 2006; 50: 1001–1012. [ Links ]
58. Alvarez C, Barrientos O, Leal A, Contreras G, Barrero L, Rincón S, et al. Community associated methicillin resistant Staphylococcus aureus, Colombia. Emerg Infect Dis 2006; 12: 2000–2001. [ Links ]
59. Cortés J, Gómez C, Cuervo S, Leal A, GREBO. Implicaciones en salud Pública de Staphylococcus aureus meticilino resistente adquirido en la comunidad en Bogotá, Colombia. Rev Salud Pública 2007; 9: 448–454. [ Links ]
60. Buitrago G, Cortés JA, Castillo JS, Leal AL, Sánchez R, Alvarez CA. Emergencia de Staphylococcus aureus resistente a meticilina con perfil comunitario en hospitales de Bogotá. Infectio 2008; 12: 64. [ Links ]
61. Escobar J, Moreno J, Díaz P, Castro B, Leal A, Vanegas N. Caracterización molecular de Staphylococcus aureus resistente a meticilina adquirido en la comunidad (SARM–AC) en Colombia. Infectio 2008; 12: 72. [ Links ]
62. Vesga O, Toro JM. Sepsis por Staphyloccoccus aureus: estudio descriptivo de 61 casos. Acta Med Colomb 1994; 19: 116–124. [ Links ]
63. Jiménez JN, Correa M, Rúa A, Zapata M, Riaño R, Báez P, et al. Detección molecular de Staphylococcus aureus resistente a meticilina (MRSA) colonizando manos de individuos de la población general. Infectio 2008; 12: 73. [ Links ]
64. Ospina S, comunicación personal, Hospital Universitario San Vicente de Paúl, 2008. [ Links ]
Recibido: 28 julio de 2008
Aceptado: 18 de agosto de 2008