Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Iatreia
Print version ISSN 0121-0793
Iatreia vol.23 no.2 Medellín Apr./June 2010
INVESTIGACIÓN ORIGINAL
Presencia de un polimorfismo de la CSP–12 relacionado con susceptibilidad a sepsis grave en una muestra de tres poblaciones colombianas
Presence of a polymorphism of the csp–12 related with susceptibility to severe sepsis in colombian individuals from three different populations
Susana Pamela Mejía de Los Rios1; Pablo Javier Patiño Grajales2; Fabián Alberto Jaimes Barragán3; Gabriel de Jesús Bedoya4; Juan Álvaro López Quintero5; Julián Camilo Arango6; Laura Yaneth Gámez Díaz7
1 Bióloga, Universidad del Valle, Joven Investigador de Colciencias, Grupo de Inmunodeficiencias Primarias, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia
2 Profesor Titular, Grupo de Inmunodeficiencias Primarias, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia
3 Profesor asociado y Jefe de departamento de Medicina Interna y Grupo académico de Epidemiología Clínica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia
4 Profesor Titular y Jefe del Grupo de Genética Molecular, Universidad de Antioquia, Medellín, Colombia
5 Profesor asociado, Escuela de Microbiología, Grupo de Microbiología Molecular, Universidad de Antioquia, Medellín, Colombia
6 Profesor de ocasional, Escuela de Microbiología, Grupo de Inmunodeficiencias Primarias, Universidad de Antioquia, Medellín, Colombia
7 Bacterióloga, Universidad Industrial de Santander, Estudiante de Maestría de Biología, Universidad de Antioquia, Medellín, Colombia Correspondencia: Susana Pamela Mejía susana_mejia@hotmail.com
RESUMEN
La sepsis, un síndrome de respuesta sistémica a la infección, es un problema de salud pública asociado a alta morbilidad y mortalidad alrededor del mundo. Entre los múltiples genes asociados a esta enfermedad se encuentra el gen que codifica para la caspasa–12 (csp–12), en el cual se ha identificado un polimorfismo de un sólo nucleótido (125T>C) en el exón 4 que predice una forma larga (L) de la proteína, que a su vez se ha asociado con riesgo de sepsis grave y alta mortalidad. Además, se ha demostrado que la frecuencia del alelo L es mucho mayor en poblaciones afroamericanas. Este estudio evalúa la presencia ó el polimorfismo 125T®C de la csp–12 en 128 individuos: 81 pacientes de Medellín con diagnóstico de sepsis, 23 individuos sanos de una población afroamericana del Chocó y 24 individuos sanos provenientes de Medellín. En las tres poblaciones se encontraron 121 individuos homocigotos S/S (csp–12 corta) y 7 heterocigotos S/L discriminados así: 3 pacientes con diagnóstico de sepsis, 3 individuos afroamericanos y 1 de la población sana de Medellín. Nuestros resultados muestran que, a pesar de ser una muestra pequeña, en nuestra población existe el alelo L, encontrándose en mayor frecuencia en individuos afroamericanos y en una menor proporción en los mestizos, tanto pacientes como en los individuos sanos. Esto indica que la población afroamericana de Colombia podría tener mayor susceptibilidad a sepsis grave que las poblaciones mestizas, las cuales, se ha demostrado, son producto de mezcla europea, amerindia y africana, ésta última en una baja proporción. Por lo tanto, se deben efectuar estudios más amplios para un mejor entendimiento de las bases genéticas de la respuesta inmune de pacientes con sepsis, con el fin de diseñar terapias más racionales y personalizadas para prevenir este síndrome.
Palabras clave
Caspase–12, Inflamación, Sepsis, SNP
SUMMARY
Sepsis, a syndrome of systemic response to infection is a major public health problem, because it is associated with high morbidity and mortality. Among the genes shown to be associated with this syndrome, there is one which encodes for caspase–12 (csp–12). Within this gene, the single nucleotide polymorphism 125T>C located in exon 4, which predicts a long form of the protein, has been associated with severe sepsis and increased related mortality. On the other hand, higher frequency of allele L has been reported in African American populations. The present study evaluated the csp–12 polymorphism 125T>C in 128 individuals: 81 patients with sepsis, 23 healthy African Colombian subjects and 24 healthy individuals from Medellin–Colombia. We found 121 individuals homozygous S/S (csp–12 short) in these three populations and 7 heterozygotes S/L, discriminated as follows: 3 septic patients, 3 African Colombians and 1 healthy subject from Medellin. This preliminary data suggest that the csp–12L allele is present in the Colombian population, both in African Colombians and Mestizo individuals (either septic patients or healthy individuals).Therefore, more comprehensive studies should be performed to better understand the genetic basis of the immune response of patients with sepsis in order to design more rational and personalized therapies to prevent this syndrome.
Key words
Caspase–12, Inflammation, Sepsis, SNP
INTRODUCCIÓN
La sepsis es el síndrome de respuesta inflamatoria sistémica que se desarrolla cuando la defensa inicial y apropiada del hospedero a una infección se amplifica y no es controlada por los inhibidores naturales; se define como grave cuando se presenta con disfunción orgánica, y como choque séptico cuando se asocia a hipotensión refractaria.1 A pesar de los avances que se han alcanzado en la comprensión molecular de la relación patógenohospedero y la respuesta inmunológica a la infección, la sepsis sigue siendo una de las causas de muerte más importantes en las unidades de cuidados intensivos (UCI).
Se estima una ocurrencia de 750.000 pacientes por año en Estados Unidos, con un aumento en su incidencia de 1,5° por año y una tasa de mortalidad entre 30–70° .2 Se considera que además del tipo de microorganismo y del tamaño del inóculo, hay otras características propias del paciente, como la edad, la constitución genética y el estado inmune y nutricional, que pueden ayudar a determinar el curso y pronóstico de esta condición.
Son múltiples los mecanismos activados durante la respuesta inflamatoria que están implicados en la patogénesis de la enfermedad, entre éstos se incluyen células efectoras (neutrófilos, monocitos y macrófagos), así como sus productos: especies reactivas de oxígeno (ROS), metabolitos de nitrógeno (RNS), citocinas, quimiocinas y aumento en la expresión de receptores de superficie que participan activamente en el proceso de diapédesis.3 Reportes recientes demuestran que individuos con alteraciones en los genes que regulan estos mecanismos de respuesta pueden presentar susceptibilidad ó resistencia a enfermedades infecciosas. Entre estas alteraciones se han descrito polimorfismos en los receptores Fc, en los receptores tipo toll (TLR) y en receptores de citoquinas, como TNF e IL–1.4,5Estos últimos determinan las concentraciones de moléculas pro o anti inflamatorias en la sangre y de esta manera pueden hacer que una persona desencadene una marcada respuesta hiper o hipoinflamatoria a la infección.6 Por tanto, el conocimiento de los polimorfismos de los genes involucrados en la respuesta inmune ayudará a comprender mejor la enorme variabilidad de respuesta de los individuos afectados por sepsis y permitirá la instauración de terapias más eficientes y personalizadas.
Entre los genes descritos como relacionados con la respuesta inmunológica en sepsis se encuentran los que codifican para las caspasas, enzimas claves en la mediación de los eventos proteolíticos en las cascadas de inflamación y de muerte celular por apoptosis.7 Entre este grupo se encuentra la caspasa 12 (csp–12), la cual se relaciona filogenéticamente con las caspasas de maduración de citocinas (caspasas 1, 4 y 5, conocidas como caspasas inflamatorias). Sin embargo, se ha reportado que en ratones actúa como un mediador de apoptosis inducida por estrés en el retículo endoplasmático (RE).8 En humanos, Saleh y colaboradores reportaron un polimorfismo de un solo nucleótido (T >C) en el codón 125 del exón 4 del gen de la csp–12, el cual conduce a un cambio de un codón de terminación por una arginina, originando la síntesis de una proteína truncada (Csp–12S) o una proteína larga (Csp–12L), respectivamente. Además, encontraron que aproximadamente el 20° de 776 individuos norteamericanos de descendencia surafricana presentaban dicho polimorfismo, mientras que en poblaciones caucásicas y asiáticas estaba ausente.9 Sin embargo, estudios recientes acerca de la frecuencia del alelo de la csp–12L en diferentes poblaciones alrededor del mundo mostraron que la csp–12L no se presenta exclusivamente en descendientes afroamericanos, pues también se encuentra en poblaciones del norte de África (Argelia) y del medio oriente (Israel), así como en algunas poblaciones del sur (Pakistán) y del este de Asia (Bougainville, Combadie, China, Japón, Nueva Guinea), aunque en una baja frecuencia.10
Desde el punto de vista funcional se sugiere que la csp–12L humana tiene un papel en la modulación de la respuesta a endotoxinas y citocinas (IL–1ß , TNF–α, IFN–y), actuando como un regulador dominante–negativo de la activación de caspasas inflamatorias, potencialmente por antagonismo en los complejos de inflamación y asociaciones en las vías pro–inflamatorias, constituyendo un factor de riesgo para el desarrollo de la sepsis.9,11,12 Estos datos son soportados por un estudio hecho en ratones con delección en el exón 2 del gen de la csp–12 (csp–12–/–), lo cuales mostraron resistencia a peritonitis y a choque séptico; se observó sin embargo que en respuesta a ligandos de receptores tipo Toll y NOD los ratones silvestres mostraban una atenuación en la producción de citocinas pro–inflamatorias, como IL–1ß , IL–18, IFN–y, pero no de IFN–α y IL–6. Los autores propusieron por tanto una asociación de la csp–12 con la caspasa–1 que produce inhibición de su actividad proteasa impidiendo la maduración de las citocinas proinflamatorias. 13
Con base en el anterior conocimiento se diseñó el presente estudio con el fin de determinar la presencia de los alelos que codifican para las formas larga y corta de la csp–12 en individuos con diagnóstico de sepsis, en individuos sanos de una población afroamericana del Chocó y en individuos sanos de la ciudad de Medellín.
MÉTODOS
Población de estudio
Se emplearon 128 muestras de ADN genómico de individuos Colombianos discriminados en tres grupos: 81 pacientes con diagnóstico de sepsis (Ensayo clínico HETRASE14), 23 descendientes afroamericanos del Chocó y 24 individuos sanos de Medellín. Las muestras de la población del Chocó pertenecen al banco de ADN del grupo de Genética Molecular (GENMOL) de la Universidad de Antioquia y han sido plenamente definidas como provenientes de descendientes afroamericanos mediante marcadores moleculares específicos. Las muestras de sangre periférica de los pacientes con sepsis fueron obtenidas de individuos que participaron en el proyecto Heparina no Fraccionada Para el Tratamiento de la Sepsis: Un Experimento Clínico Controlado (Estudio HETRASE), en el Hospital Universitario San Vicente de Paúl de la ciudad de Medellín. Todos los sujetos, o sus representantes legales, firmaron un consentimiento informado aprobado por el Comité de Ética de la institución para la participación del proyecto HETRASE y en estudios ligados a este.
Recolección de muestras y extracción de DNA
Personal de enfermería entrenado especialmente para el proyecto de HETRASE recolectó 3 ml de sangre venosa periférica en tubos con EDTA (ácido etilen–diamino–tetraacético) en cada uno de los pacientes con diagnóstico de sepsis; en los individuos sanos se hizo la misma recolección, pero por bacteriólogos del grupo de Inmunodeficiencias Primarias. La extracción de ADN genómico y su purificación fueron efectuadas mediante un estuche comercial Gentra Systems® (Lot No. 8241468 – 8241543) siguiendo las instrucciones del fabricante.
Detección del polimorfismo 125 T C en el exón 4 del gen de la csp–12
C en el exón 4 del gen de la csp–12
La detección del polimorfismo de 125T  C se realizó mediante las técnicas de polimorfismos conformacionales de DNA de cadena sencilla (PCR–SSCP, por sus siglas en inglés) y de polimorfismos en la longitud de los fragmentos de restricción (PCR–RFLP, por sus siglas en inglés). En ambos casos se realizó la amplificación de un fragmento de 314 pb del exón 4 del gen de la csp–12 por PCR (Reacción en cadena de la polimerasa), utilizando los oligonucleótidos sentido, 5'–GTCATTCTGTGTGTAT TAATTGC–3' y antisentido, 5' –CCTATAATATCATACATCTT GCTC–3')9, bajo las siguientes condiciones: cinco min a 94 ° C – 30 ciclos de 94 ° C – 30'', 55 ° C – 30'', 72 ° C – 30'', diez min a 72 ° C y se conserva a 4 ° C. La amplificación de los fragmentos se confirmó en un gel de agarosa al 1,5° teñido con 1ml de bromuro de etidio (BrEt) con una concentración de 10mg/ml.
C se realizó mediante las técnicas de polimorfismos conformacionales de DNA de cadena sencilla (PCR–SSCP, por sus siglas en inglés) y de polimorfismos en la longitud de los fragmentos de restricción (PCR–RFLP, por sus siglas en inglés). En ambos casos se realizó la amplificación de un fragmento de 314 pb del exón 4 del gen de la csp–12 por PCR (Reacción en cadena de la polimerasa), utilizando los oligonucleótidos sentido, 5'–GTCATTCTGTGTGTAT TAATTGC–3' y antisentido, 5' –CCTATAATATCATACATCTT GCTC–3')9, bajo las siguientes condiciones: cinco min a 94 ° C – 30 ciclos de 94 ° C – 30'', 55 ° C – 30'', 72 ° C – 30'', diez min a 72 ° C y se conserva a 4 ° C. La amplificación de los fragmentos se confirmó en un gel de agarosa al 1,5° teñido con 1ml de bromuro de etidio (BrEt) con una concentración de 10mg/ml.
PCR–SSCP
Se tomaron 5 µ l del producto de la PCR y se realizó una electroforesis en un gel vertical de poliacrilamida al 4° en condiciones no desnaturalizantes. Los productos de la PCR se mezclaron con 1 µ l de tampón de carga (Fermentas Lot No. 00029840) y se calentaron a 90 °C por dos minutos para desnaturalizar el ADN. Los pozos se cargaron con 4 a 6 µl de la muestra, con los respectivos controles positivos y negativos. La electroforesis se corrió a 180 V durante cinco horas. Una vez finalizada, se procedió a revelar las bandas de ADN por tinción con plata (Marca Analyticals Carlo Erba).
PCR–RFLP
Se realizó de acuerdo con los reportes en la literatura:9 se tomó la secuencia completa del gen de csp–12 (GenBank No.AP002004 172081 bp DNA linear cromosoma 11q) para establecer la región de interés e identificar una enzima de restricción que reconociera la variante génica de csp–12 mediante el programa Restriction Mapper (RestrictionMapper – on line restriction mapping the easy way®). La enzima seleccionada fue LweI o su isoesquizomero SfaNI, que reconocen la secuencia del polimorfismo para csp–12L (5'...GCAT C(N)5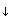 ..3'). Después de obtener los fragmentos amplificados de la región del exón 4 de la casp–12, se realizó la digestión con la enzima de restricción LweI (Fermentas No.ER1621) bajo las siguientes condiciones: 24 h a 37 °...C, 20 min a 65 ° C y luego se llevó a 4 ° C. Los productos se analizaron en geles de agarosa al 2,5° , teñidos con BrEt y en geles de poliacrilamida al 8° teñidos con plata con el fin de evidenciar los diferentes patrones de corte del ADN.
..3'). Después de obtener los fragmentos amplificados de la región del exón 4 de la casp–12, se realizó la digestión con la enzima de restricción LweI (Fermentas No.ER1621) bajo las siguientes condiciones: 24 h a 37 °...C, 20 min a 65 ° C y luego se llevó a 4 ° C. Los productos se analizaron en geles de agarosa al 2,5° , teñidos con BrEt y en geles de poliacrilamida al 8° teñidos con plata con el fin de evidenciar los diferentes patrones de corte del ADN.
Secuenciación del ADN
La confirmación de los resultados obtenidos en la genotipificación por PCR–RFLP de individuos que presentaron el polimorfismo, T125C, se analizaron mediante secuenciamiento de la región amplificada por PCR del exón 4. El secuenciemiento fue realizado por el laboratorio Macrogen Advancing through Genomics (Seúl–Corea). Para el análisis de las secuencias se utilizó el programa chromas version 1.45.
Análisis estadístico
En Colombia no existen estudios previos que permitan determinar el tamaño de muestra requerido para un análisis estadístico significativo de los polimorfismos en el gen de la csp–12, por tanto, en este estudio se realizó un muestreo por conveniencia. Las frecuencias alélicas y genotípicas se calcularon utilizando el programa Genepop versión 3.1. Las pruebas de significancia (chicuadrado (X2) o prueba exacta de Fisher) para las diferencias alélicas y genotípicas se hicieron mediante el programa Epi–Info versión 6.
RESULTADOS Y DISCUSIÓN
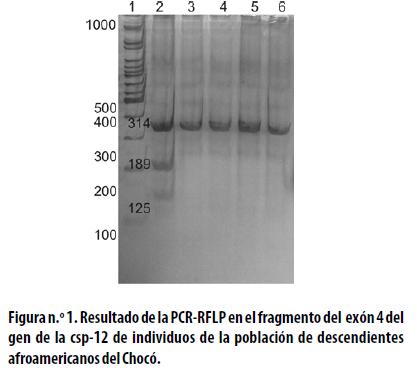
Con el propósito de identificar el SNP de interés se utilizó inicialmente SSCP, pero no fue posible evidenciar sin embargo diferencias en la electroforesis de los patrones de bandas analizados que permitiera evidenciar la presencia de los genotipos C/T y T/T (resultados no mostrados). Para tratar de suplir esta limitante en la identificación del SNP se procedió a utilizar el método de PCR–RFLP. Para esto, los productos de PCR obtenidos se digirieron con la enzima de restricción LWeI que permite identificar el cambio de T 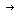 C en el codón 125 del exón 4 del gen csp–12, el cual se asocia con la forma larga de la proteína codificada por este gen. La digestión con esta enzima permitió distinguir los diferentes genotipos: T/T y C/T. En la figura n.° 1 se presenta un gel de poliacrilamida en el que se observan las bandas que se encontraron para cada genotipo.
C en el codón 125 del exón 4 del gen csp–12, el cual se asocia con la forma larga de la proteína codificada por este gen. La digestión con esta enzima permitió distinguir los diferentes genotipos: T/T y C/T. En la figura n.° 1 se presenta un gel de poliacrilamida en el que se observan las bandas que se encontraron para cada genotipo.
En el carril 1 marcador de peso molecular Fermentas, en el carril 2 se encuentra un individuo heterocigoto C/T con un patrón de bandas de 314 pb, 189 pb y 125 pb, en el carril 3, 4, 5 y 6 se muestran individuos homocigotos T/T con un patrón de bandas de 314 pb.
El genotipo homocigoto T/T se observó con mayor frecuencia en las poblaciones analizadas (tabla n.° 1). El genotipo heterocigótico C/T se encontró solo en siete individuos, discriminados así: tres individuos del grupo de pacientes con sepsis, tres individuos de la población afro descendiente del Chocó, y un sujeto de la población sana de Medellín; el genotipo C/C no se encontró en ninguno de los individuos estudiados (tabla n.° 1). Se puede observar por tanto que en las tres poblaciones existe una presencia elevada del alelo T y una baja del alelo C. Desde el punto de vista de la distribución de frecuencias genotípicas solo se encontraron diferencias significativas entre el grupo de pacientes y el grupo de afrodescendientes sanos y no se encontraron diferencias entre las frecuencias alélicas de todos los grupos de estudio (tabla n.° 2). Es de notar que debido a la muestra tan pequeña de las poblaciones de afroamericanas sanos del chocó y de los individuos sanos de Medellín los resultados no permitieron determinar acertadamente las frecuencias de los alelos para la csp–12 en la población colombiana, pero si se observó un comportamiento muy similar al reportado en estudios anteriores9 en los cuales el análisis de las secuencias de más de 1100 genomas de DNA de individuos de distintas etnias mostró predominantemente el genotipo T/T, el cual codifica para la forma truncada de la csp–12. En el estudio mencionado, la frecuencia del polimorfismo que resulta en la csp–12L (C/C) fue baja, encontrándose sólo en la población de descendientes afroamericanos (T/C= 20,3° , T/T= 1,3° ) y estuvo ausente en las otras poblaciones del estudio (caucásicos, asiáticos).9
Para confirmar los resultados obtenidos por el análisis de PCR–RFLP se realizó la secuenciacióndel ADN de dos pacientes con sepsis y en dos individuossanos de la ciudad de Medellín que presentaron el genotipo homocigoto T/T y heterocigoto T/C. Se confirmó la presencia del alelo C en el codón 125 del exón 4 (figura n.° 2), que predice el cambio de un codón de parada por uno que codifica para arginina (figura n.° 3). Estos resultados confirman que la técnica de PCR–RFLP reportada por primera vez en nuestro estudio puede ser utilizada como herramienta de identificación del polimorfismo que permite la posible expresión de la csp–12L.
A pesar de tratarse de una muestra pequeña, en los datos obtenidos en este estudio fue posible identificar el alelo C125 en nuestra población, el cual corresponde al polimorfismo que se asocia a la expresión de la csp–12L. La forma larga de esta caspasa se considera el genotipo ancestral de acuerdo con el estudio evolutivo realizado por Xue Y, et al, en el 2006, en el que mostró que desde hace unos 100.000 años, cuando las primeras poblaciones de humanos migraron fuera del África, se empezó a presentar una fuerza de selección positiva sobre dicho polimorfismo, de manera que permitió el aumento de la frecuencia del alelo T que codifica para la forma corta de la csp–12, dirigiéndolo cerca de la fijación. De acuerdo con esto, este estudio propone que la forma larga de la caspasa tiende a desaparecer. Dicha selección ocurrió cuando hubo un incremento del tamaño y densidad de la población en Europa y Asia expuesta a enfermedades infecciosas, lo que favoreció la reproducción de individuos que tenían la forma truncada de la caspasa.15 En Afrodescendientes, por su parte, el cruce entre distintos grupos raciales introdujo la csp–12S en su perfil genético, lo que favoreció la perpetuación de su raza. De acuerdo con lo anterior, y con base en los resultados del presente estudio, podemos observar que en nuestra población el alelo T de la csp–12 aún no se ha fijado completamente puesto que aún existen individuos heterocigotos T/C, pero se evidencia el proceso de selección positiva que existe sobre el alelo T por su alta frecuencia en la población estudiada. La presencia del polimorfismo csp–12L en un individuo se ha asociado con una mayor susceptibilidad a desarrollar sepsis grave,9,13 por lo tanto, y de acuerdo con nuestros resultados se podría sugerir que la población afrocolombiana tendría mayor riesgo de sufrir las complicaciones de la sepsis, en comparación con las poblaciones mestizas, predisposición debida a que se ha demostrado que en nuestro medio el mestizaje es producto de una mezcla europea, amerindia y africana , esta última en una baja proporción, situación especialmente válida para la población de Medellín, la cual presenta un 65° de europeo, 25° amerindio y solo un 10° africano.16
Como ya se había mencionado, se ha sugerido que la csp–12L actúa como un regulador dominante negativo inhibiendo el efecto de las caspasas proinflamatorias, y por lo tanto, induciendo una disminución en la producción de citoquinas como IL–1ß , IL–18 e INF–y Se esperaría entonces una menor severidad en el desarrollo de sepsis en los individuos con atenuación de producción de estas citoquinas, y no un estado de gravedad mayor, como lo han mostrado los estudios poblacionales.9
Esto podría explicarse por la naturaleza dual de las citoquinas proinflamatorias. Aunque ellas pueden causar daños inflamatorios perjudiciales contra las organismo, la inmunidad innata – la primera respuesta del cuerpo a los agentes infecciosos – induce una producción temprana de citoquinas para detectar y limitar la infección. Por ejemplo, ratones que son deficientes para el receptor tipo Toll 4 (TLR4), el cual detecta lipopolisacáridos de los microorganismos y activa una cascada celular que lleva a la producción de citoquinas, son altamente resistentes a los efectos sistémicos inducidos por estos productos bacterianos, pero más susceptible a infecciones causadas por los patógenos que expresan estas moléculas.12,17 Igualmente, pacientes que reciben terapia anti–TNF para reducir la inflamación, así como los individuos que tienen un defecto en la vía de señalización de los TLR, presentan un mayor riesgo de infección. Como consecuencia, se predice que muchos tratamientos desarrollados para la sepsis grave pueden aumentar el riesgo de inmunosupresión e infección secundaria. Las estrategias terapéuticas futuras contra la sepsis podrían necesitar equilibrar este riesgo contra el potencial benéfico de tratar a corto plazo un exceso de un mediador inflamatorio. Hasta ahora el desarrollo de nuevas estrategias terapéuticas para tratar la sepsis se ha basado en gran parte en la premisa de neutralizar toxinas y mediadores circulantes potencialmente tóxicos con el fin de detener o disminuir los efectos de este síndrome. Sin embargo, hasta el momento esta estrategia no ha tenido resultados alentadores puesto que ni los corticosteroides, ni los bloqueadores del lipopolisacarido, ni los bloqueadores del TNF–α , ni los antagonistas del receptor de la IL–1 han tenido éxito en los ensayos clínicos, lo que ha llevado al cuestionamiento de si la muerte en los pacientes con sepsis origina una inflamación incontrolada. De acuerdo con lo anterior, se ha propuesto como posibles causas de la falla terapéutica la heterogeneidad de los pacientes con sepsis, y se postula que los blancos de la terapia, su tiempo de administración, las dosis utilizadas y la combinación de terapias sean erradas.18
Poder diferenciar adecuadamente entre los desórdenes inmunes y las sub–poblaciones de pacientes que presentan la forma corta o larga de la caspasa–12 adquiriría entonces importancia para instaurar el tratamiento adecuado. De esta manera, se podría proponer que la identificación del alelo C de la csp–12 en pacientes con sepsis se asocia con un perfil de citoquinas determinado, y en éstos pacientes no se esperaría que se derive un beneficio significativo de la estrategia antiinflamatoria que se ha propuesto tradicionalmente. Nuestro estudio reporta entonces por primera vez la presencia del alelo C en la población colombiana y describe una técnica molecular para su caracterización. Estos datos pueden ser la base para realizar estudios de asociación entre la presencia de este alelo y un patrón de citoquinas característico, lo que podría identificar previamente un desorden inmunológico aun no reconocido que confiera susceptibilidad al desarrollo de entidades inflamatorias como la sepsis grave. Se necesitan estudios posteriores con muestreos más amplios que nos permitan definir la frecuencia del alelo C de la csp–12 en nuestra población y que nos ayuden a comprender si existe una relación entre los polimorfismos asociados a la csp–12 con algunas características clínicas de la sepsis, lo que a su vez permitiría diseñar nuevas alternativas terapéuticas dirigidas a pacientes que sean portadores de este polimorfismo.
REFERENCIAS BIBLIOGRÁFICAS
1. Marik PE, Varon J. Sepsis: state of the art. [Internet]. Disease–a–month: DM. 2001; 47(10): 465–532.Available from: http://www.ncbi.nlm.nih.gov/pubmed/11641650 [ Links ]
2. Angus DC, Wax RS. Epidemiology of sepsis: an update. [Internet]. Critical care medicine. 2001 ;29 (7 Suppl): S109–16.Available from: http://www.ncbi.nlm.nih.gov/pubmed/11445744 [ Links ]
3. Kellum JA, Angus DC. Genetic variation and risk of sepsis. [Internet]. Minerva anestesiologica. 2003; 69(4): 245–53. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12766715 [ Links ]
4. Barber RC, Aragaki CC, Rivera–Chavez FA, Purdue GF, Hunt JL, Horton JW. TLR4 and TNF–alpha polymorphisms are associated with an increased risk for severe sepsis following burn injury. [Internet]. Journal of medical genetics. 2004; 41(11): 808–13.Available from: http://www.ncbi.nlm.nih.gov/pubmed/15520404 [ Links ]
5. Holmes CL, Russell JA, Walley KR. Genetic polymorphisms in sepsis and septic shock: role in prognosis and potential for therapy. [Internet]. Chest. 2003; 124(3): 1103–15. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12970043 [ Links ]
6. Briceño I. Sepsis: definiciones y Aspectos Fisiopatológicos [Internet]. MEDICRIT. 2005; 2(8): 164–178. Available from: www.medicrit.com [ Links ]
7. Lamkanfi M, Kalai M, Vandenabeele P. Caspase–12: an overview. [Internet]. Cell death and differentiation. 2004; 11(4): 365–368. Available from: http://www.ncbi.nlm.nih.gov/pubmed/14685161 [ Links ]
8. Nakagawa T, Zhu H, Morishima N, Li E, Xu J, Yankner BA, et al. Caspase–12 mediates endoplasmicreticulum– specific apoptosis and cytotoxicity by amyloid–beta. [Internet]. Nature. 2000; 403(6765): 98–103. Available from: http://www.ncbi.nlm.nih.gov/pubmed/10638761 [ Links ]
9. Saleh M, Vaillancourt JP, Graham RK, Huyck M, Srinivasula SM, Alnemri ES, et al. Differential modulation of endotoxin responsiveness by human caspase–12 polymorphisms. [Internet]. Nature. 2004; 429(6987): 75–79. Available from: http://www.ncbi.nlm.nih.gov/pubmed/15129283 [ Links ]
10. Kachapati K, O'Brien TR, Bergeron J, Zhang M, Dean M. Population distribution of the functional caspase– 12 allele. [Internet]. Human mutation. 2006; 27(9): 975. Available from: http://www.ncbi.nlm.nih.gov/pubmed/16917906 [ Links ]
11. Stehlik C, Krajewska M, Welsh K, Krajewski S, Godzik A, Reed JC. The PAAD/PYRIN–only protein POP1/ASC2 is a modulator of ASC–mediated nuclear–factor–kappa B and pro–caspase–1 regulation. [Internet]. The Biochemical journal. 2003; 373(Pt 1): 101–113. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12656673 [ Links ]
12. Martinon F, Burns K, Tschopp J. The inflammasome: a molecular platform triggering activation of inflammatory caspases and processing of proIL–beta. [Internet]. Molecular cell. 2002; 10(2): 417–426. Available from: http://www.ncbi.nlm.nih.gov/pubmed/12191486 [ Links ]
13. Saleh M, Mathison JC, Wolinski MK, Bensinger SJ, Fitzgerald P, Droin N, et al. Enhanced bacterial clearance and sepsis resistance in caspase–12– deficient mice. [Internet]. Nature 2006 ;440(7087): 1064–1068. Available from: http://dx.doi.org/10.1038/nature04656 [ Links ]
14. Jaimes F, De La Rosa G, Morales C, Fortich F, Arango C, Aguirre D, et al. Unfractioned heparin for treatment of sepsis: A randomized clinical trial (The HETRASE Study). [Internet]. Critical Care Medicine. 2009;37(4): 1185–1196. Available from: http://www.ncbi.nlm.nih.gov/pubmed/19242322 [ Links ]
15. Xue Y, Daly A, Yngvadottir B, Liu M, Coop G, Kim Y, et al. Spread of an inactive form of caspase–12 in humans is due to recent positive selection. [Internet]. American journal of human genetics. 2006 ;78(4):659– 70.Available from: http://www.ncbi.nlm.nih.gov/pubmed/16532395 [ Links ]
16. Wang S, Ray N, Rojas W, Parra MV, Bedoya G, Gallo C, et al. Geographic patterns of genome admixture in Latin American Mestizos. [Internet]. PLoS genetics. 2008;4(3): e1000037. Available from: http://www.ncbi.nlm.nih.gov/pubmed/18369456 [ Links ]
17. Creagh EM, Conroy H, Martin SJ. Caspase–activation pathways in apoptosis and immunity. [Internet]. Immunological reviews. 2003; 19310–1921.Available from: http://www.ncbi.nlm.nih.gov/pubmed/12752666 [ Links ]
18. Hotchkiss RS, Tinsley KW, Swanson PE, Schmieg RE, Hui JJ, Chang KC, et al. Sepsis–induced apoptosis causes progressive profound depletion of B and CD4+ T lymphocytes in humans. [Internet]. Journal of immunology (Baltimore, Md. : 1950). 2001; 166(11): 6952–6963. Available from: http://www.ncbi.nlm.nih.gov/pubmed/11359857 [ Links ]
Recibido: julio 23 de 2009
Aceptado: marzo 16 de 2010