Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Iatreia
Print version ISSN 0121-0793
Iatreia vol.27 no.3 Medellín July/Sept. 2014
INVESTIGACIÓN ORIGINAL
Descripción de un modelo de infección in vitro con virus dengue empleando células mononucleares humanas de sangre periférica
Human peripheral blood mononuclear cells as an in vitro model for dengue virus infection
Félix Giovanni Delgado Tiria1; Adriana Marisol Pérez Acosta1; Jaime E Castellanos1
1 Grupo de Virología, Universidad del Bosque, Bogotá, Colombia. delgadofelix@unbosque.edu.co
Recibido: agosto 09 de 2013
Aceptado: octubre 11 de 2013
RESUMEN
No existen modelos animales apropiados para el estudio de la fisiopatología y las manifestaciones clínicas de la enfermedad causada por la infección con virus dengue, por lo que para ello se requiere desarrollar modelos experimentales. El propósito del presente trabajo fue establecer un modelo de infección in vitro con virus dengue serotipo-2 (DENV-2). Para esto se obtuvieron células mononucleares de sangre periférica (CMSP) usando un gradiente de Ficoll, y se las cultivó e infectó con DENV-2 a una baja multiplicidad de infección. La subpoblación celular que se infectó y produjo citocinas se identificó empleando un análisis multiparamétrico por citometría de flujo. Como resultado, las CMSP fueron permisivas a la infección, que se detectó a las 24 horas de inoculado el virus. Además, en este mismo tiempo, los monocitos CD14+, pero no los linfocitos CD3+ o CD19+, fueron la subpoblación celular preferencialmente infectada y responsable de la producción de TNF-α e IL-6. En conclusión, se estableció un modelo de infección in vitro usando CMSP no fraccionadas, en el que se identificó a los monocitos CD14+ como la principal célula blanco de la infección con DENV-2 y productora de citocinas proinflamatorias.
PALABRAS CLAVE
Células Mononucleares, Citometría de Flujo, IL-6; TNF-α, Virus Dengue
SUMMARY
To date, there are no appropriate animal models for the study of the pathophysiology and clinical manifestations of the disease caused by dengue virus infection; therefore, experimental models are required for that purpose. The objective of the present work was to establish a model of in vitro infection with DENV-2. To this end, human peripheral blood mononuclear cells (PBMC) were obtained using a Ficoll gradient, and infected with DENV-2 using a low multiplicity of infection. The cell populations infected and responsible for the production of cytokines were identified using a multiparametric analysis by flow cytometry. As a result, PBMC were permissive to infection that was detected 24 hours after virus inoculation. Additionally, at this same time, CD14+ cells, but not CD3+ or CD19+ cells, were preferentially infected and responsible for the production of TNF-α and IL-6. In conclusion, we established a model of in vitro infection using unfractionated PBMC, in which CD14+ cells were identified as the primary target cells for infection with DENV-2, and the production of proinflammatory cytokines.
KEY WORDS
Dengue Virus, Flow Cytometry, IL-6, Mononuclear Cells,TNF-α
INTRODUCCIÓN
El virus dengue (DENV) es un miembro del género Flavivirus de la familia Flaviviridae. Comprende cuatro diferentes serotipos (DENV-1 al 4) transmitidos por la hembra del género Aedes aegypti (vector principal) mediante el ciclo de transmisión hombre-mosquitohombre (1). Su genoma es un RNA de cadena única y polaridad positiva con aproximadamente 11 kb de longitud y presenta un solo marco de lectura abierto que codifica para una poliproteína precursora procesada antes y después de la traducción por proteasas celulares y virales en tres proteínas estructurales: C (cápside), M (membrana no glicosilada, que se forma a partir de un precursor denominado prM durante la maduración), E (envoltura glicosilada), y siete no estructurales: NS1, NS2a, NS2b, NS3, NS4a, NS4b, NS5 (2).
No se ha determinado claramente la afinidad por un grupo celular específico para la infección con DENV, debido a que no se tienen datos suficientes que definan el tejido de mayor susceptibilidad y replicación del DENV in vivo. Existen datos limitados respecto a la detección del DENV en tejidos humanos premortem y posmortem y lo que se ha sugerido es que la infección con DENV se inicia en el sitio de la inoculación, posiblemente mediante la infección de las células de Langerhans en la piel (3,4) para luego diseminarse hacia los nódulos linfoides regionales, el bazo, el hígado y otros órganos por la vía linfática y el sistema circulatorio (5,6). Hallazgos patológicos adicionales en monos rhesus infectados experimentalmente y en casos fatales de infección en seres humanos determinaron que el tipo celular que soporta la replicación del DENV in vivo son las células del linaje fagocítico-mononuclear (7-10). En este sentido, varios estudios han intentado detectar el DENV en células mononucleares de sangre periférica (CMSP), como una forma sencilla de examinar la replicación del DENV in vivo e in vitro.
Estudios in vivo han encontrado que el DENV se detecta con mayor frecuencia en las CMSP de pacientes durante una infección aguda (11,12) y que se observa además una relación directa entre la gravedad de la enfermedad y mayor detección de antígeno viral en estas células (11). Además, se ha logrado detectar hebras positivas y negativas de RNA viral en CMSP de pacientes infectados, lo que indica una replicación activa del DENV en estas células (13,14). Por otra parte, en trabajos in vitro de infección de CMSP con DENV-2 se detectó un pico en la replicación viral al segundo día postinfección al cuantificar el DENV por la técnica de plaqueo (15).
Por otro lado, no existen modelos animales apropiados para el estudio de la fisiopatogenia y de las manifestaciones clínicas de la enfermedad causada por el DENV, ni como referentes para la evaluación de tratamientos farmacológicos específicos. La enfermedad (leve o grave) solamente la padecen los humanos y ningún animal sufre manifestaciones clínicas similares, aunque sean infectados por mosquitos o experimentalmente. Los monos de algunas especies de la familia Cercopithecus al ser infectados muestran susceptibilidad a la infección, pero en ellos se detectan bajos títulos de virus producidos sin que aparezcan signos o síntomas de la enfermedad (16). Teniendo en cuenta lo anterior, con el presente trabajo nuestro grupo de investigación busca establecer en el laboratorio un modelo de infección in vitro con virus dengue que permita fundamentar estudios posteriores encaminados a la evaluación de moléculas para ejercer un mejor control farmacológico de la enfermedad, ya sea disminuyendo la replicación viral o regulando la respuesta inmune innata inducida durante la infección.
METODOLOGÍA
Líneas celulares: se emplearon en este estudio células LLC-MK2 (línea de epitelio de riñón de mono rhesus) y U937 (línea promonocítica humana obtenida de un linfoma histiocítico difuso) mantenidas en el medio Dulbecco's Modified Eagle Medium (DMEM, Sigma) suplementado con 10% de suero fetal bovino (SFB), antibióticos (penicilina 100 U/mL y estreptomicina 100 mg/mL, Gibco), e incubadas a 37 °C en una atmósfera con CO2 al 5%. La línea celular C6/36 (proveniente de Aedes albopictus) fue mantenida en las mismas condiciones excepto que la temperatura de incubación fue de 33 °C.
Stock viral: el DENV serotipo 2 (DENV-2) fue suministrado por el Instituto Nacional de Salud de Colombia y cosechado en células C6/36. La recolección de la cosecha viral se hizo entre el quinto y el octavo días postinfección, con base en la observación del efecto citopático inducido por el virus sobre la monocapa celular. El sobrenadante y las células (lisadas por cambios bruscos de temperatura entre 28 °C y -80 °C) se recolectaron y centrifugaron a 720 xg por 10 minutos a 4 °C y el sobrenadante se distribuyó en alícuotas y se almacenó a -80 °C. Adicionalmente, se preparó un control antigénico celular (Mock) en iguales condiciones sin inoculación del virus. La titulación viral se efectuó por inmunodetección enzimática de focos de células infectadas sobre monocapas de LLC-MK2.
Obtención de CMSP: se utilizó sangre venosa periférica de donantes adultos sanos quienes autorizaron, por medio de un consentimiento informado, el uso de sus componentes sanguíneos para este trabajo de investigación, aprobado por el Comité de Ética de la Universidad el Bosque. Se hizo una prueba rápida de ELISA (Dengue Duo Cassette, Panbio Diagnostics) usando el plasma obtenido, para detectar anticuerpos IgM o IgG específicos contra DENV. Se seleccionaron los donantes seronegativos para IgM e IgG para la obtención de las CMSP empleando un gradiente de ficoll (Histopaque-1077, Sigma). Las CMSP obtenidas se resuspendieron en medio de mantenimiento RPMI-1640 (Gibco) suplementado con SFB al 10% (Gibco), penicilina 100 U/mL y estreptomicina 100 mg/mL (Gibco), se sembraron 1 x 106 células/mL en tubos de polipropileno de 15 mL (Becton Dickinson) para evitar la adherencia, y se incubaron a 37 °C en atmósfera con 5% de CO2 y 70% de humedad. La obtención y procesamiento de las CMSP se llevaron a cabo en condiciones de apirogenicidad, usando para esto reactivos con niveles de endotoxina por debajo de 0,3 EU/mL.
Inmunocompetencia de las CMSP en cultivo: se evaluó la inmunocompetencia de estas células usando 5 ng/mL de PMA (phorbol 12-myristate 13-acetate, Sigma) y 500 ng/mL de ionomicina (Ionomycin calcium salt de Streptomyces, Sigma) como estímulo para la activación de linfocitos, y 50 ng/mL de LPS (lipopolisacárido de Escherichia coli, Sigma) como estímulo para la activación de los monocitos. Para ello, se sembraron 1 x 106 células/mL en tubos de 15 mL, se adicionaron el respectivo estímulo y 3 µg/mL de brefeldina (eBioscience) durante 12 horas. Posteriormente, se recuperaron las CMSP (5 x 105 células) y se analizaron por citometría de flujo para detectar la presencia de citocinas intracelulares (IL-6 y TNF-α).
Infección de las células con DENV-2: las infecciones se hicieron con DENV-2 a una multiplicidad de infección (MOI) de 0,1. Como control negativo se utilizaron células tratadas con el sobrenadante del lisado de células C6/36 no infectadas (Mock). Dependiendo del ensayo, las CMSP se infectaron por 12 y 24 horas. En el caso de la detección de citocinas intracelulares se adicionó brefeldina (3 µg/mL) 12 horas antes de recoger las células. Las células se usaron para detectar la presencia de marcadores de fenotipo (CD14, CD3 y CD19), antígeno viral o citocinas intracelulares (IL-6 y TNF-α). Además, los sobrenadantes de las CMSP de 24 horas postinfección (HPI) se usaron para infectar una monocapa de células LLC-MK2 sobre las que posteriormente se analizó, por inmunodetección enzimática, la presencia de células infectadas. Como control positivo de infección, las líneas celulares U937 y C6/36 fueron infectadas por dos y cinco días respectivamente, y luego analizadas por citometría de flujo para detectar la presencia intracelular de antígeno viral.
PCR: se extrajo el RNA de las CMSP adicionando 1 mL de Trizol® (Invitrogen) siguiendo las instrucciones del fabricante. Se resuspendió el RNA obtenido en agua tratada con dietil-pirocarbonato (DEPC) al 0,1% y se cuantificó a 260 nm. A partir de 0,5-1 µg de RNA total se efectuó una transcripción reversa utilizando la retrotranscriptasa M-MLV (Promega) y random primer (Promega) a 37 °C por 1 hora. Para detectar el genoma viral, se hizo una PCR convencional utilizando la enzima GoTaq polimerasa (Promega) y los primers sentido Md1 5'-TCA ATA TGC TGA AAC GCG AGA GAA ACC G-3', y antisentido D2 5'-TTG CAC CAA CAG TCA ATG TCT TCA GGT TC-3, que amplifican un producto de 511 pb correspondiente a un segmento del gen CprM del DENV, según lo reportado por Chien y colaboradores (17).
Anticuerpos para citometría de flujo: para la detección intracelular del DENV-2 se empleó una marcación indirecta utilizando un anticuerpo monoclonal antiflavivirus (10 mg/mL) (Chemicon, MAB8744. Isotipo IgG2a de ratón) específico para la proteína E viral y un anticuerpo anti-IgG2a de ratón acoplado a isotiocianato de fluoresceína (FITC, por su sigla en inglés) (8 mg/mL, Becton Dickinson). Todos los demás anticuerpos empleados poseen un isotipo IgG1. Los anticuerpos usados para detectar los marcadores de superficie fueron CD3-Pacific Blue (8 mg/mL, Becton Dickinson), CD19-APC-Cy7 (5 mg/mL, Becton Dickinson) y CD14-PerCP Cy5.5 (10 mg/mL, eBioscience). Los anticuerpos utilizados para detectar citocinas intracelulares fueron TNF-α-APC (5 mg/mL, Becton Dickinson) e IL-6-PE (5 mg/mL, eBioscience). Todos los anticuerpos se titularon previamente.
Citometría de flujo: en total, se centrifugaron 5x105 células a 250 xg por 5 minutos y se resuspendieron en 100 µL de FACSFlow™ (BD Biosciences). Posteriormente, se hizo una incubación con el colorante de viabilidad (LIVE⁄DEAD® Fixable Aqua Dead Cell Stain, INVITROGEN) y los anticuerpos para detección de fenotipo por 30 minutos a 4 °C. Luego de un lavado con FACSFlow™, las CMSP se fijaron y permeabilizaron con el reactivo Cytofix/Citoperm™ (Becton-Dickinson) por 20 minutos a 4 °C y se hizo un lavado con PermWash ™ (Becton-Dickinson). Después, dependiendo del ensayo, se hizo una incubación con el anticuerpo antiflavivirus por 1 hora a 37 °C y con el anticuerpo antirratón por 30 minutos a 4 °C, o se incubó con los anticuerpos para TNF-α e IL-6 por 30 minutos a 4 °C. Finalmente, se hizo un lavado con PermWash™ y las células se analizaron en un citómetro de flujo FACSCanto ™ II (Becton-Dickinson) empleando el software FACSDiva™.
RESULTADOS
Los monocitos CD14+ en las CMSP son permisivos a la infección con DENV-2
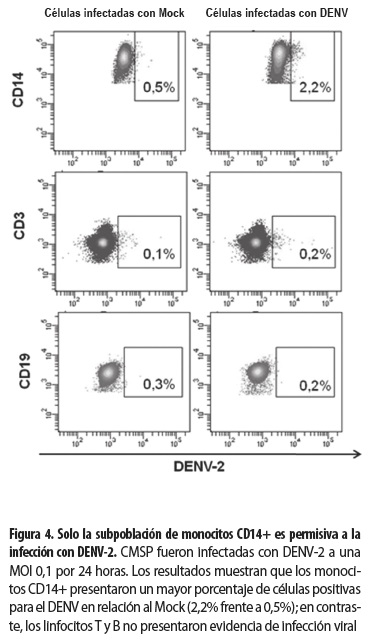
Para estudiar la infección desde un enfoque más parecido a lo que ocurre naturalmente, CMSP no fraccionadas fueron infectadas in vitro con DENV-2 a una MOI baja (0,1) por 24 horas. La infección de las CMSP se verificó empleando dos técnicas: PCR e inmunodetección enzimática del DENV en células LLC-MK2. Como resultado, en la figura 1A (línea 2) se observa una banda de 511pb correspondiente a un segmento del gen C-prM del DENV únicamente en las CMSP que fueron infectadas, y en la figura 1C se observa la presencia de antígeno viral en las células LLC-MK2 inoculadas con los sobrenadantes de CMSP infectadas. Estos dos hallazgos no se observaron en los controles negativos (células inoculadas con Mock; figuras 1A (línea 1) y 1B).
Posteriormente, se quiso identificar la subpoblación celular de las CMSP permisiva a la infección con DENV-2 y para ello se empleó citometría de flujo. Sin embargo, debido a que la detección del DENV se efectuó utilizando una marcación indirecta, se verificó previamente la eficacia de la misma. Para ello, se emplearon líneas celulares de diferente permisividad a la infección con DENV. En la figura 2 se muestra que el 74,8% de las células C6/36 y el 7,3% de las células U937 fueron positivas para DENV y se observó menos del 0,2% de inespecificidad en las células no infectadas.
Una vez definidas las condiciones de detección indirecta del DENV por citometría de flujo, se procedió a los ensayos con las CMSP. Para esto, como se ilustra en la figura 3, se aplicó la siguiente estrategia de análisis: 1. Se seleccionaron los singlets (células que pasan una a la vez frente al láser) definidos por tener un área (FSC-A) y un ancho de señal (FSC-W) uniformes, medidas en el forward scatter; de esta manera se descartaron del análisis los agregados celulares (dos o más células que pasan juntas frente al láser). 2. Sobre esta población de singlets, se identificaron las poblaciones de linfocitos y monocitos de acuerdo con su tamaño (forward scattered) y complejidad interna (side scattered). 3. Sobre cada una de estas dos poblaciones se identificaron las células no viables mediante el uso de un colorante de viabilidad que penetra en las células cuya membrana celular está dañada o alterada, reaccionando con los grupos amino presentes en el citoplasma. De esta manera fue posible seleccionar, para los análisis respectivos, los monocitos y linfocitos viables que emiten niveles bajos de fluorescencia. 4. Sobre la región de linfocitos viables, se evaluó la expresión de CD3, considerando la población CD3+ como linfocitos T, y sobre la región CD3- se identificó la población CD19+ como linfocitos B. Por otro lado, en los monocitos viables se evaluó la expresión de CD14, considerando la población CD14+ como monocitos. Esta estrategia de análisis nos permitió identificar plenamente nuestras poblaciones de interés (CD3+, CD19+, CD14+) para los experimentos de detección de antígeno viral o citocinas intracelulares.
Aplicando la estrategia de análisis ya expuesta, se examinó la permisibilidad de los diferentes subtipos celulares de las CMSP al DENV. Los resultados mostraron que, de las subpoblaciones analizadas, los monocitos CD14+ presentaron un porcentaje significativamente mayor de células positivas para el DENV-2 en relación al Mock (2,2% frente a 0,5%); en contraste, los linfocitos T (CD3+) y B (CD19+) no presentaron evidencia de infección viral (figura 4).
La infección de las CMSP con DENV-2 induce la producción de TNF-α e IL-6 en monocitos CD14+
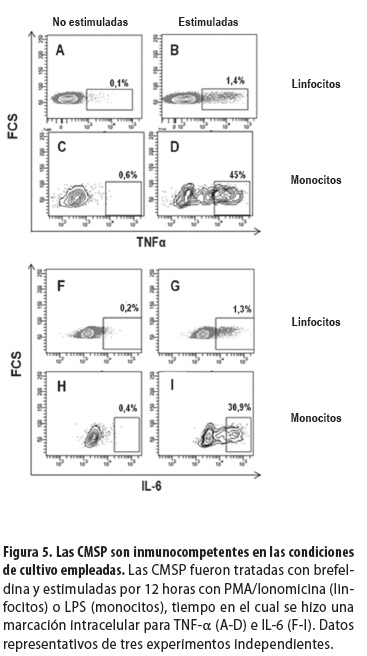
Antes de analizar la producción de TNF-α e IL-6 en las diferentes subpoblaciones celulares durante la infección, se verificó la inmunocompetencia de las CMSP en las condiciones de cultivo empleadas. Las células se mantuvieron por 12 horas en presencia o ausencia de PMA/Ionomicina (estímulos policlonales para la activación de linfocitos T y B) o LPS (estímulo para la activación de monocitos) y posteriormente se analizaron por citometría de flujo, en la que se evaluó la presencia intracelular de las citocinas IL-6 y TNF-α. En la figura 5 se observa que el tratamiento con PMA/ Ionomicina indujo a 1,4% y 1,3% de linfocitos a la producción de TNF-α e IL-6, respectivamente (figura 5B y G). Cuando se usó el estímulo con LPS, se indujo la producción de TNF-α e IL-6 en 45% y 30,9% de los monocitos, respectivamente (figura 5D e I). De esta manera, los resultados confirman que bajo las condiciones de cultivo empleadas, las CMSP son susceptibles de ser activadas.
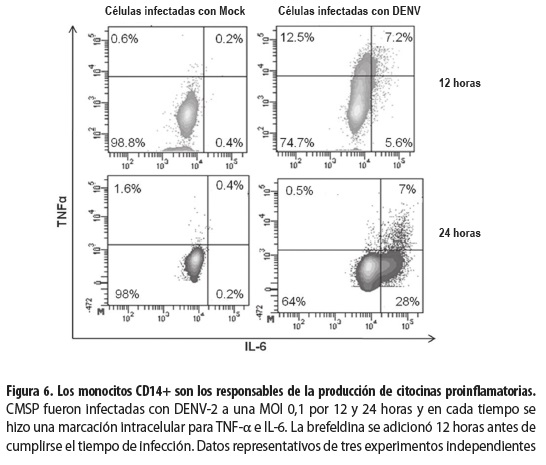
Posteriormente se llevaron a cabo los ensayos con las CMSP inoculadas con Mock o DENV-2 por 12 y 24 horas, tiempos en los que se recogieron las células para detectar IL-6 y TNF-α intracelularmente empleando citometría de flujo. De esta manera, se identificó la subpoblación celular de las CMSP responsable de la producción de las citocinas proinflamatorias durante la infección. De las subpoblaciones analizadas, los monocitos CD14+ produjeron TNF-α e IL-6 a las 12 y 24 horas postinfección (figura 6); sin embargo, en los linfocitos T y B no se detectó ninguna de las dos citocinas evaluadas (datos no mostrados).
DISCUSIÓN
En el presente trabajo se estableció un modelo de infección in vitro con DENV en el que se identificaron la población celular permisiva a la infección y la población celular responsable de la producción de citocinas proinflamatorias como TNF-α e IL-6. Una de las ventajas de este modelo fue la utilización de CMSP no fraccionadas y una MOI baja de infección (0,1), ya que de una manera aproximada permitió semejar el ambiente natural en que ocurre la infección in vivo. Además, el cultivo de las CMSP se hizo en tubos de polipropileno porque este material no permite la adherencia de los monocitos a la superficie del tubo, sino que los mantiene en suspensión durante todo el tiempo de cultivo; en estas condiciones se alcanzan porcentajes de recuperación más altos para los análisis por citometría (18). Este enfoque metodológico se ha empleado en varios trabajos de investigación con los monocitos como objeto de estudio (18-20) lo que sugiere que es una forma aceptada de cultivo para estas células. Por otro lado, se tuvo la precaución de seleccionar donantes seronegativos para anticuerpos contra DENV, pues se sabe que tanto la neutralización como la amplificación virales dependiente de anticuerpos (ADE) son fenómenos que modifican la infección viral (21) y los niveles de citocinas durante la infección por dengue (22).
Las CMSP fueron infectadas por 24 horas, debido a que se ha reportado que para este tiempo ocurre la primera ronda de infección (23). Además, Torrentes- Carvalho y colaboradores (24) reportaron que monocitos primarios infectados con DENV-2 por 48 horas expresaron marcadores apoptóticos como fosfatidilserina y el receptor de muerte Fas, a diferencia de los monocitos infectados por 24 horas que presentaron niveles de expresión más bajos de estos marcadores de apoptosis. En el presente trabajo no se consideró la evaluación de estos marcadores; sin embargo, durante los análisis multiparamétricos por citometría de flujo, en los que se utilizó un colorante de viabilidad, se pudo observar que las células inoculadas por 24 horas con Mock o con DENV-2 presentaron porcentajes bajos de células no viables (en promedio 4,1% y 3,4%, respectivamente), que fueron muy similares.
Una vez establecido que las CMSP eran susceptibles de ser infectadas in vitro con DENV-2 (figura 1), y antes de proceder a identificar el fenotipo de la subpoblación celular blanco de la infección, se verificó la marcación indirecta empleada para la detección del virus por citometría de flujo. Para esto se emplearon líneas celulares de diferente permisividad a la infección con DENV. Se usaron células altamente permisivas a la infección (C6/36) (23) y otras que se consideran de baja permisividad (U937) (25). Los resultados obtenidos en este estudio están de acuerdo con lo esperado, pues, como se muestra en la figura 2, el porcentaje de células infectadas con DENV-2 fue del 74,8% para las C6/36 y de 7,3% para las U937. Es importante resaltar que las células inoculadas con el virus o con solo el medio se marcaron de igual manera para la detección de la proteína E viral. El background observado en las células no infectadas con esta marcación indirecta fue bajo (0,1%), lo que sugiere que esta estrategia de marcación permite identificar de manera específica las células infectadas con el virus.
Una vez obtenidos estos resultados, se hicieron los ensayos empleando CMSP y se continuaron los análisis por citometría de flujo. Esta técnica permite análisis detallados de subpoblaciones celulares sin necesidad de hacer separaciones físicas de las mismas, mediante análisis multiparamétricos como el que se muestra en la figura 3. Esta estrategia de análisis, que se aplicó en todos los ensayos posteriores, permitió además identificar agregados celulares (mediante FSC-A y FSC-W) y células muertas (mediante la marcación con un colorante de viabilidad), que fueron excluidas porque presentan características diferentes de autofluorescencia y unen de manera inespecífica los anticuerpos; con ello se mejoraron en gran medida la sensibilidad y la precisión en los análisis de los datos (26). Teniendo en cuenta lo anterior, se analizaron las CMSP infectadas o no con DENV-2 y los resultados indicaron que de las tres subpoblaciones identificadas (CD14+, CD19+, CD3+) solamente los monocitos CD14+ fueron infectados con DENV-2 (figura 4). Este resultado es similar al obtenido en dos trabajos en los que infectaron in vitro CMSP totales no fraccionadas, provenientes de donantes seronegativos para DENV, y lograron determinar por citometría de flujo que los monocitos humanos CD14+ son las células preferencialmente infectadas (6,27).
Por otro lado, el porcentaje de infección de los monocitos CD14+ obtenido en este trabajo se encuentra dentro de los valores esperados debido a que estudios previos como el de Wu y colaboradores (3) hallaron entre 1% y 5% de monocitos/macrófagos infectados con una MOI de 2. De forma similar, Kou y colaboradores (6) reportaron en promedio un 3,5% de monocitos CD14+ infectados cuando se usó una MOI de 5. Una consideración muy importante para tener en cuenta es que las células primarias humanas son más difíciles de infectar in vitro que las líneas celulares; además, la cinética de detección del DENV en fagocitos mononucleares varía de acuerdo con la cepa del virus y la MOI empleadas (28). Por otro lado, estudios llevados a cabo empleando CMSP provenientes de pacientes con dengue identificaron por citometría de flujo a los monocitos CD14+ como las células blanco para el DENV (29,30), lo que corrobora nuestro hallazgo in vitro.
En contraste con lo anterior, otros estudios sugieren que el DENV tiene la capacidad de replicarse activamente tanto en linfocitos B como en linfocitos T y que estos grupos celulares podrían también jugar un papel importante en la patogenia del dengue como hospederos del DENV (31-33). Sin embargo, en nuestros ensayos no observamos infección de linfocitos T (CD3+) o B (CD19+), como se muestra en la figura 4. Estos resultados contradictorios pudieron deberse a que en los estudios en que se detectó infección en linfocitos usaron principalmente líneas celulares o poblaciones celulares purificadas.
Adicionalmente, se ha sugerido que una de las características más importantes que se presentan en los pacientes con dengue grave es la pérdida de la integridad endotelial, que se explica como consecuencia de una respuesta inmune anormal contra el DENV (34). Estudios clínicos muestran que los niveles de citocinas como TNF-α e IL-6 se encuentran significativamente aumentados en pacientes con dengue grave (35-38). Estas citocinas proinflamatorias se asocian in vivo con una respuesta de fase aguda que puede resultar en la liberación de péptidos quimiotácticos, fiebre y la activación de células endoteliales que conduce a la permeabilidad vascular, rasgo clínico dominante en el dengue grave (39).
Se han descrito como fuente de TNF-α e IL-6 a monocitos y linfocitos B purificados al infectarlos con DENV-2, y a linfocitos T CD4+ al exponerse a antígenos del DENV-2 in vitro (40). En el presente estudio, se detectó la presencia de TNF-α e IL-6 en los monocitos de las CMSP infectadas. Los resultados indican que solo los monocitos CD14+ son los responsables de la producción de TNF-α e IL-6 durante la infección (figura 6). Por otro lado, estudios in vivo han mostrado que los monocitos activados podrían jugar un papel importante en la inmunopatogénesis de la enfermedad. Durbin y colaboradores (30) informaron la presencia de un alto porcentaje de monocitos activados en pacientes con dengue grave.
En conclusión, con el presente trabajo se logró establecer en nuestro laboratorio un modelo de infección in vitro con DENV-2 empleando CMSP que se asemeja de cerca a lo que ocurre in vivo, ya que a una baja MOI se detectó la infección de monocitos y la producción de citocinas proinflamatorias que median procesos inmunopatológicos característicos de la enfermedad.
REFERENCIAS BIBLIOGRÁFICAS
1. Velandia M, Castellanos J. Virus del dengue: estructura y ciclo viral. Infectio. 2011;15(1):33–43. [ Links ]
2. Henchal EA, Putnak JR. The dengue viruses. Clin Microbiol Rev. 1990;3(4):376–96. [ Links ]
3. Wu SJ, Grouard-Vogel G, Sun W, Mascola JR, Brachtel E, Putvatana R, et al. Human skin Langerhans cells are targets of dengue virus infection. Nat Med. 2000;6(7):816–20. [ Links ]
4. Tassaneetrithep B, Burgess TH, Granelli-Piperno A, Trumpfheller C, Finke J, Sun W, et al. DC-SIGN (CD209) mediates dengue virus infection of human dendritic cells. J Exp Med. 2003;197(7):823–9. [ Links ]
5. Jessie K, Fong MY, Devi S, Lam SK, Wong KT. Localization of dengue virus in naturally infected human tissues, by immunohistochemistry and in situ hybridization. J Infect Dis. 2004;189(8):1411–8. [ Links ]
6. Kou Z, Quinn M, Chen H, Rodrigo WWSI, Rose RC, Schlesinger JJ, et al. Monocytes, but not T or B cells, are the principal target cells for dengue virus (DV) infection among human peripheral blood mononuclear cells. J Med Virol. 2008;80(1):134–46. [ Links ]
7. Halstead SB, O'Rourke EJ. Dengue viruses and mononuclear phagocytes. I. Infection enhancement by non-neutralizing antibody. J Exp Med. 1977;146(1):201–17. [ Links ]
8. Bhamarapravati N, Tuchinda P, Boonyapaknavik V. Pathology of Thailand haemorrhagic fever: a study of 100 autopsy cases. Ann Trop Med Parasitol. 1967;61(4):500–10. [ Links ]
9. Marchette NJ, Halstead SB, Falkler WA, Stenhouse A, Nash D. Studies on the pathogenesis of dengue infection in monkeys. 3. Sequential distribution of virus in primary and heterologous infections. J Infect Dis. 1973;128(1):23–30. [ Links ]
10. Morens DM, Marchette NJ, Chu MC, Halstead SB. Growth of dengue type 2 virus isolates in human peripheral blood leukocytes correlates with severe and mild dengue disease. Am J Trop Med Hyg. 1991;45(5):644–51. [ Links ]
11. Kittigul L, Meethien N, Sujirarat D, Kittigul C, Vasanavat S. Comparison of dengue virus antigens in sera and peripheral blood mononuclear cells from dengue infected patients. Asian Pac J Allergy Immunol. 1997;15(4):187–91. [ Links ]
12. Fung CP, Lee YM, Kuo BI, Yang SP, Chan YJ, Liu CY, et al. Using buffy coat for reverse transcriptase-polymerase chain reaction in the diagnosis of dengue virus infection: preliminary study. J Microbiol Immunol Infect. 2000;33(4):217–22. [ Links ]
13. Wang WK, Lee CN, Kao CL, Lin YL, King CC. Quantitative competitive reverse transcription-PCR for quantification of dengue virus RNA. J Clin Microbiol. 2000;38(9):3306–10. [ Links ]
14. Wang W-K, Sung T-L, Tsai Y-C, Kao C-L, Chang S-M, King C-C. Detection of dengue virus replication in peripheral blood mononuclear cells from dengue virus type 2-infected patients by a reverse transcription-realtime PCR assay. J Clin Microbiol. 2002;40(12):4472–8. [ Links ]
15. Rabablert J, Wasi C, Kinney R, Kasisith J, Pitidhammabhorn D, Ubol S. Attenuating characteristics of DEN-2 PDK53 in flavivirus-naïve peripheral blood mononuclear cells. Vaccine. 2007;25(19):3896–905. [ Links ]
16. Bente DA, Rico-Hesse R. Models of dengue virus infection. Drug Discov Today Dis Models. 2006 ;3(1):97–103. [ Links ]
17. Chien L-J, Liao T-L, Shu P-Y, Huang J-H, Gubler DJ, Chang G-JJ. Development of real-time reverse transcriptase PCR assays to detect and serotype dengue viruses. J Clin Microbiol. 2006;44(4):1295–304. [ Links ]
18. Baran J, Kowalczyk D, Ozóg M, Zembala M. Three-color flow cytometry detection of intracellular cytokines in peripheral blood mononuclear cells: comparative analysis of phorbol myristate acetate-ionomycin and phytohemagglutinin stimulation. Clin Diagn Lab Immunol. 2001;8(2):303–13. [ Links ]
19. Van der Bruggen T, Nijenhuis S, van Raaij E, Verhoef J, van Asbeck BS. Lipopolysaccharide-induced tumor necrosis factor alpha production by human monocytes involves the raf-1/MEK1-MEK2/ERK1-ERK2 pathway. Infect Immun. 1999;67(8):3824–9. [ Links ]
20. Soga F, Katoh N, Inoue T, Kishimoto S. Serotonin activates human monocytes and prevents apoptosis. J Invest Dermatol. 2007;127(8):1947–55. [ Links ]
21. Van der Schaar HM, Wilschut JC, Smit JM. Role of antibodies in controlling dengue virus infection. Immunobiology. 2009;214(7):613–29. [ Links ]
22. Chareonsirisuthigul T, Kalayanarooj S, Ubol S. Dengue virus (DENV) antibody-dependent enhancement of infection upregulates the production of anti-inflammatory cytokines, but suppresses anti- DENV free radical and pro-inflammatory cytokine production, in THP-1 cells. J Gen Virol. 2007;88(Pt 2):365–75. [ Links ]
23. Lambeth CR, White LJ, Johnston RE, de Silva AM. Flow cytometry-based assay for titrating dengue virus. J Clin Microbiol. 2005;43(7):3267–72. [ Links ]
24. Torrentes-Carvalho A, Azeredo EL, Reis SRI, Miranda AS, Gandini M, Barbosa LS, et al. Dengue-2 infection and the induction of apoptosis in human primary monocytes. Mem Inst Oswaldo Cruz. 2009;104(8):1091–9. [ Links ]
25. Diamond MS, Edgil D, Roberts TG, Lu B, Harris E. Infection of human cells by dengue virus is modulated by different cell types and viral strains. J Virol. 2000;74(17):7814–23. [ Links ]
26. Mahnke YD, Roederer M. Optimizing a multicolor immunophenotyping assay. Clin Lab Med. 2007;27(3):469–85, v. [ Links ]
27. Sydow FF, Santiago MA, Neves-Souza PC, Cerqueira DI, Gouvea AS, Lavatori MF, et al. Comparison of dengue infection in human mononuclear leukocytes with mosquito C6/36 and mammalian Vero cells using flow cytometry to detect virus antigen. Mem Inst Oswaldo Cruz. 95(4):483–9. [ Links ]
28. Cologna R, Rico-Hesse R. American genotype structures decrease dengue virus output from human monocytes and dendritic cells. J Virol. 2003;77(7):3929–38. [ Links ]
29. Neves-Souza PCF, Azeredo EL, Zagne SMO, Valls-de- Souza R, Reis SRNI, Cerqueira DIS, et al. Inducible nitric oxide synthase (iNOS) expression in monocytes during acute Dengue Fever in patients and during in vitro infection. BMC Infect Dis. 2005;5:64. [ Links ]
30. Durbin AP, Vargas MJ, Wanionek K, Hammond SN, Gordon A, Rocha C, et al. Phenotyping of peripheral blood mononuclear cells during acute dengue illness demonstrates infection and increased activation of monocytes in severe cases compared to classic dengue fever. Virology. 2008;376(2):429–35. [ Links ]
31. King AD, Nisalak A, Kalayanrooj S, Myint KS, Pattanapanyasat K, Nimmannitya S, et al. B cells are the principal circulating mononuclear cells infected by dengue virus. Southeast Asian J Trop Med Public Health. 1999;30(4):718–28. [ Links ]
32. Lin Y-W, Wang K-J, Lei H-Y, Lin Y-S, Yeh T-M, Liu H-S, et al. Virus replication and cytokine production in dengue virus-infected human B lymphocytes. J Virol. 2002;76(23):12242–9. [ Links ]
33. Mentor NA, Kurane I. Dengue virus infection of human T lymphocytes. Acta Virol. 1997;41(3):175–6. [ Links ]
34. Rodenhuis-Zybert IA, Wilschut J, Smit JM. Dengue virus life cycle: viral and host factors modulating infectivity. Cell Mol Life Sci. 2010;67(16):2773–86. [ Links ]
35. Chaturvedi U, Raghupathy R, Pacsa A, Elbishbishi E, Agarwal R, Nagar R, et al. Shift from a Th1-type response to Th2-Type in dengue haemorrhagic fever. Curr Sci. 1999;76(1): 63-9. [ Links ]
36. Pinto LM, Oliveira SA, Braga EL, Nogueira RM, Kubelka CF. Increased pro-inflammatory cytokines (TNFalpha and IL-6) and anti-inflammatory compounds (sTNFRp55 and sTNFRp75) in Brazilian patients during exanthematic dengue fever. Mem Inst Oswaldo Cruz. 1999;94(3):387–94. [ Links ]
37. Bethell DB, Flobbe K, Cao XT, Day NP, Pham TP, Buurman WA, et al. Pathophysiologic and prognostic role of cytokines in dengue hemorrhagic fever. J Infect Dis. 1998;177(3):778–82. [ Links ]
38. Houghton-Triviño N, Martín K, Giaya K, Rodríguez JA, Bosch I, Castellanos JE. Comparison of the transcriptional profiles of patients with dengue fever and dengue hemorrhagic fever reveals differences in the immune response and clues in immunopathogenesis. Biomedica. 2010;30(4):587–97. [ Links ]
39. Anderson R, Wang S, Osiowy C, Issekutz AC. Activation of endothelial cells via antibody-enhanced dengue virus infection of peripheral blood monocytes. J Virol. 1997;71(6):4226–32. [ Links ]
40. Gagnon SJ, Ennis FA, Rothman AL. Bystander target cell lysis and cytokine production by dengue virusspecific human CD4(+) cytotoxic T-lymphocyte clones. J Virol. 1999;73(5):3623–9. [ Links ]