Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Iatreia
Print version ISSN 0121-0793
Iatreia vol.27 no.4 Medellín Oct./Dec. 2014
INVESTIGACIÓN ORIGINAL
Frecuencia de los transcriptos p190BCR-ABL y p210BCR-ABL en una población colombiana con leucemia mieloide crónica (LMC) usando RT-PCR cualitativa
Detection of p190BCR-ABL AND p210BCR-ABL fusion transcripts in patients with chronic myeloid leukemia (CML) using qualitative RT-PCR
Frequência dos transcritos p190BCR-ABL e p210BCR- ABL numa população colombiana com leucemia mielóide crônica (LMC) usando RT-PCR qualitativa
Carlos Alberto Aya Bonilla1; José Domingo Torres2; Carlos Enrique Muskus3; Gloria Ramírez Gaviria4; Jorge Cuervo Sierra5; Margarita Sierra Sánchez6; Francisco Cuéllar-Ambrosi7; Jorge Humberto Botero Garcés8; Carmen Gloria Artigas A.9; Carlos Mario Muñetón10; Gonzalo Vásquez Palacio11
1 Licenciado en Biología y Química, Universidad del Tolima. Magíster en Biología, Unidad de Genética Médica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
2 Médico cirujano y Especialista en Medicina Interna, Universidad de Antioquia. Especialista en Hematología, Universidad Nacional de Colombia. Profesor Titular, Departamento de Medicina Interna, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
3 Doctor en Ciencias Básicas Biomédicas, Universidad de Antioquia. Profesor, Programa de Estudio y Control de Enfermedades Tropicales, PECET, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
4 Bacterióloga y Laboratorista Clínica, Universidad de Antioquia. Magíster en Ciencias Básicas Biomédicas, área Citogenética, Unidad de Genética Médica, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
5 Médico cirujano, Universidad de Antioquia. Especialista en Medicina Interna, Universidad de Antioquia. Especialista en Hematología Clínica, Hospital Universitario UANL de Monterrey, México.
6 Médico cirujano, Universidad Pontificia Bolivariana, Medellín, Colombia. Especialista en Pediatría, Universidad de Antioquia, Medellín, Colombia. Especialista en Oncología Pediátrica, Pontificia Universidad Javeriana, Bogotá, Colombia. Oncólogo pediátrico, Hospital Universitario San Vicente Fundación, Medellín, Colombia.
7 Médico cirujano, Universidad del Valle, Cali, Colombia. Especialista en Medicina Interna, Universidad de Antioquia. Sección de Hematología de Adultos, IPS Universitaria, Universidad de Antioquia, Medellín, Colombia.
8 Médico cirujano, Universidad de Antioquia. Magíster en Inmunología. Especialista en Estadística, Universidad Nacional de Colombia. Profesor, Departamento de Microbiología y Parasitología, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
9 Licenciado en Tecnología Médica, Universidad Austral de Chile. Profesor de Hematología, Universidad San Sebastián, Santiago de Chile.
10 Biólogo y Magíster en Genética. Profesor Asociado, Departamento de Pediatría y Puericultura, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
11 Biólogo y Magíster en Ciencias Básicas Biomédicas. Profesor, Departamento de Pediatría y Puericultura, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia. gvasquezp@gmail.com
Recibido: junio 19 de 2013
Aceptado: marzo 03 de 2014
RESUMEN
Introducción: la leucemia mieloide crónica (LMC) se caracteriza por la presencia del cromosoma Filadelfia (Ph) que resulta de la translocación recíproca balanceada t(9;22)(q34;q11); este marcador cromosómico se encuentra con menor frecuencia en pacientes con leucemia linfoide aguda (LLA).
Objetivo: determinar la frecuencia de las fusiones génicas BCR-ABL, que codifican para los transcriptos p210BCR-ABL y p190 BCR-ABL en pacientes colombianos con diagnóstico de LMC, en diferentes fases de la enfermedad o de su tratamiento.
Materiales y métodos: estudio descriptivo de corte transversal de 31 pacientes con LMC (15-78 años). El análisis se hizo a partir de muestras de sangre periférica con la técnica PCR anidada cualitativa para las isoformas P210 BCR-ABL (b3a2 e b2a2) y P190 BCR-ABL (e1a2).
Resultados: se detectó el transcripto p210BCR-ABL en 29 de los 31 casos (93,6%). En ellos se identificaron las fusiones génicas b2a2 (16/29; 55,2%), b3a2 (10/29; 34,5%) y la coexpresión b3a2 y b2a2 (3/29; 10,3%).
Conclusión: la fusión génica b2a2 fue la más frecuente en esta población con LMC.
PALABRAS CLAVE
Leucemia Mieloide Crónica, PCR Anidada, Proteínas de Fusión bcr-abl
SUMMARY
Introduction: Chronic myelogenous leukemia (CML) is characterized by the presence of the Philadelphia chromosome (Ph), resulting from the balanced reciprocal translocation t(9;22)(q34;q11). This marker chromosome is found less frequently in patients suffering from acute lymphoblastic leukemia.
Objective: To determine the frequency of BCR-ABL gene fusions encoding the p210BCR-ABL y p190 BCR-ABL transcripts in Colombian patients diagnosed with CML in different stages of the disease and/or its treatment.
Materials and methods: Cross sectional, descriptive study of thirty one CML patients (aged 15-78). Analysis was carried out through qualitative nested PCR for the isoforms P210 BCR-ABL (b3a2 e b2a2) and P190 BCR-ABL (e1a2), and based on peripheral blood samples.
Results: In 29 of the 31 patients (93.6%) transcript p210BCR-ABL was detected; b2a2 and b3a2 gene fusions and the coexpression b3a2 y b2a2 were identified in 55.2% (16/29), 34.5% (10/29) and 10.3% (3/29) of the cases, respectively. Conclusion: b2a2 gene fusion was the most frequent in this CML population.
KEY WORDS
Leukemia, Myelogenous, Chronic; Nested PCR, Fusion Proteins, bcr-abl
RESUMO
Introdução: a leucemia mielóide crônica (LMC) caracteriza- se pela presença do cromossomo Filadélfia (Ph) que resulta da translocação recíproca balanceada t(9;22)(q34;q11); este marcador cromossômico se encontra com menor frequência em pacientes com leucemia linfoide aguda (LLA).
Objetivo: determinar a frequência das fusões genéticas BCR-ABL, que codificam para os transcritos p210BCR-ABL e p190 BCR-ABL em pacientes colombianos com diagnóstico de LMC, em diferentes fases da doença ou de seu tratamento.
Materiais e métodos: estudo descritivo de corte transversal de 31 pacientes com LMC (15-78 anos). A análise se fez a partir de mostras de sangue periférico com a técnica PCR aninhada qualitativa para as isoformas P210 BCR-ABL (b3a2 e b2a2) e P190 BCR-ABL (e1a2).
Resultados: detectou-se o transcrito p210BCR-ABL em 29 dos 31 casos (93,6%). Neles se identificaram as fusões genéticas b2a2 (16/29; 55,2%), b3a2 (10/29; 34,5%) e a co-expressão b3a2 e b2a2 (3/29; 10,3%).
Conclusão: a fusão genética b2a2 foi a mais frequente nesta população com LMC.
PALAVRAS IMPORTANTES
Leucemia Mielóide Crônica, PCR Aninhada, Proteínas de Fusão bcr-abl
INTRODUCCIÓN
La leucemia mieloide crónica (LMC) se caracteriza por la proliferación sanguínea de las células granulocíticas como resultado de la transformación maligna de las células madre hematopoyéticas pluripotentes. Citogenéticamente, se define por la presencia del cromosoma Filadelfia (Ph), que se origina de la translocación recíproca balanceada t(9;22)(q34; q11) en la que se unen las secuencias del gen BCR del cromosoma 22 con las del gen ABL1 en la región q34 del cromosoma 9 generando el oncogén BCR-ABL1; el producto de la fusión codifica para la proteína quimérica p210, que presenta actividad descontrolada de tirosina-cinasa y capacidad de transformación celular. La gran mayoría de los pacientes con LMC exhiben el transcripto BCR-ABL b3a2 y/o la unión b2a2 y con baja frecuencia se detectan los transcriptos b2a3, b3a3, e1a2 o e6a2 (1).
El cromosoma Ph no solo se presenta en la LMC, sino también en 5% de los niños con leucemia linfoide aguda (LLA), en 15% a 30% de los adultos con LLA y en 2% de los casos de leucemia mieloide aguda (LMA) (2).
La biología molecular ha permitido hacer estudios en pacientes con LMC y demostrar que el transcripto p210BCR-ABL está presente en 99% de dichos pacientes, que corresponde a las fusiones génicas b3a2 (55%) y b2a2 (40%) y, en 5% de los casos, a la coexpresión de los reordenamientos génicos b3a2 y b2a2 (3). Por otra parte, la fusión génica e1a2 se expresa en 95% de los pacientes con el transcripto p190BCR-ABL y en una baja proporción e1a3 (4,5). El seguimiento de la terapia con inhibidores de la tirosina-cinasa (ITK, por su sigla en inglés) es una estrategia fundamental porque permite cuantificar las respuestas hematológica, citogenética y molecular de los pacientes con LMC. La detección de los transcriptos de fusión BCR-ABL p210/ p190 en el momento del diagnóstico o en pacientes en tratamiento, sea con ITK o con trasplante alogénico (Alo-TMO), tiene implicaciones pronósticas, porque la presencia del transcripto p210 es evidencia suficiente para iniciar o cambiar el tratamiento en estos pacientes (6).
El término enfermedad mínima residual (EMR) se refiere a la persistencia de pocas células malignas tras el tratamiento con intención curativa. Para identificar a los pacientes con LMC y EMR se pueden emplear diversas técnicas: citogenética convencional, técnicas moleculares como la reacción en cadena de la polimerasa acoplada a la transcriptasa reversa (RT-PCR) y PCR en tiempo real (qPCR); estas dos últimas tienen distintos grados de especificidad entre 0 y 1 y sensibilidad para detectar una célula con la fusión BCRABL entre 1 x 105 y 1 x 106 (7) células normales. Su aplicación permite evaluar la efectividad de la terapia antileucémica, según la variación de la carga tumoral (células Ph o cantidad del transcripto BCR-ABL) y la velocidad de disminución de la misma; por estas razones, en la actualidad las pruebas moleculares son las de mayor aceptación (8).
Si bien existen en Latinoamérica otros estudios en los que se determinan las frecuencias de los transcriptos p210 y p190, en Colombia no aparecen en las bases de datos PubMed y MEDLINE. El objetivo de este trabajo fue determinar la frecuencia de las fusiones génicas BCR-ABL, que codifican para los transcriptos p190BCR-ABL y p210BCR-ABL en una cohorte de pacientes antioqueños con LMC, en diferentes fases de la enfermedad y del tratamiento.
MATERIALES Y MÉTODOS
Casos
Se estudiaron 31 muestras de sangre periférica de pacientes remitidos del Hospital Universitario San Vicente Fundación (HUSVF) y de la Clínica León XIII, ambos de Medellín. Todos los pacientes firmaron voluntariamente el consentimiento informado, avalado por los comités de bioética para la experimentación en humanos de la Universidad de Antioquia (Sede de Investigaciones Universitarias) y de los respectivos centros hospitalarios. Se recibieron las muestras en la Unidad de Genética Médica de la Universidad de Antioquia entre junio de 2005 y junio de 2007.
Controles
Como controles positivos se utilizaron, en la detección del transcripto p210, la línea celular k562 para el gen de fusión b3a2 y una muestra de un paciente diagnosticado con LMC para b2a2. Los controles negativos fueron muestras de sangre periférica de cinco personas sanas, que también firmaron voluntariamente el consentimiento informado, con diagnóstico confirmado de t(9;22)(q34;q11) por citogenética convencional e hibridación in situ con fluorescencia (FISH).
Recolección de muestras y PCR anidada
Se procesaron muestras de sangre periférica recolectadas en tubos con EDTA; el ARN total se extrajo por el método de Trizol® (Invitrogen™) y fenol cloroformo de Chomczynnky (9). A partir de ARN mensajero se obtuvo el ADN copia mediante el uso de la transcriptasa reversa SuperScript™ lll First Strand Synthesis System (Invitrogen™).
Para el análisis molecular de los transcriptos p190BCR-ABL y p210BCR-ABL, a todas las muestras de los pacientes con LMC, así como a los controles negativos y positivos, se les hicieron, como control interno, una PCR convencional para amplificar el gen BCR normal, y una PCR anidada para detectar por separado los transcriptos de fusión p210BCR-ABL y p190BCR-ABL. Los productos amplificados se observaron por electroforesis en geles de agarosa al 1,5% teñidos con bromuro de etidio. Como controles internos del gen BCR se utilizaron un par de iniciadores C5e- (Reverse R) y BCR-b2-C (Forward F) que amplificaron un fragmento de 848 pb (10-12). Luego se hizo una PCR convencional en las siguientes condiciones: desnaturalización a 95°C por 30 segundos, seguida de 35 ciclos a 94°C por 30 segundos, 60°C por 1 minuto y 72°C por 1 minuto. Para el análisis molecular del transcripto p190BCR-ABL por la PCR anidada en la primera corrida se emplearon los oligonucleótidos BCR-e1-A (5'GACTGCAGCTCCAATGAGAAC3') y ABL-a3-B (5'GTTTGGGCTTCACACCATTCC 3'), según lo reportado por Van Dongen y colaboradores (10), y para la segunda, BCR-e1-C y ABL-a3-D, que amplificaron los fragmentos de 521pb y 381 pb, respectivamente (figura 1).
Para el análisis del reordenamiento p210 se utilizaron en la primera ronda los siguientes cebadores internos: BCR-b1-A y ABL-a3-B, para amplificar fragmentos de 417pb, 342pb, 243pb y 168pb, que corresponden a las fusiones génicas b3a2, b2a2, b3a3 y b2a3, respectivamente. En la segunda ronda se emplearon BCR-b2-C y ABL– a3 -D, que generaron productos de amplificación de 360pb, 285pb, 186pb y 111pb propios de las fusiones génicas b3a2, b2a2, b3a3 y b2a3, respectivamente (tabla 1, figuras 1 y 2).
Análisis estadístico
Se utilizaron frecuencias absolutas y relativas para describir la población del estudio. Cuando el análisis de diferencias de proporciones entre el grupo control y el de referencia dio un valor de N superior a 30 y los datos mostraron una distribución normal, se hizo una comparación de frecuencias de las fusiones génicas de los transcriptos BCR-ABL halladas en nuestro estudio con las informadas en la literatura. Para este fin se utilizó la prueba de comparación de proporciones mediante el software EPIDAT 3.1. Cuando el valor de N era de 25 o menos o el valor esperado era menor de 5 en una o más casillas en una tabla 2 x 2, se hizo la prueba exacta de Fischer. Se utilizó un α de 0,05 y p < 0,05 como valor significativo.
RESULTADOS
Se incluyeron 31 pacientes, de los cuales 16 (51,6%) eran mujeres (edad promedio de 41,4 años; rango: 15-73 años) y 15 (48,4%), hombres (edad promedio de 50,4 años; rango: 25-78 años). Todos se confirmaron con diagnóstico clínico, morfológico y citogenético.
Frecuencias de los transcriptos de fusión BCRABL y sus fusiones génicas
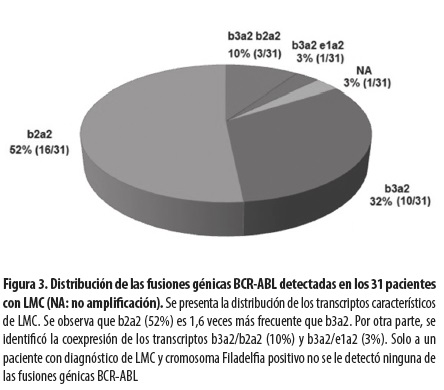
Se detectó el transcripto p210BCR-ABL en 30 de los 31 casos (96,8%). La distribución de las fusiones génicas BCR-ABL detectadas se presenta en la figura 3. Cabe anotar que 13 de los 31 (41,9%) se encontraban en la etapa diagnóstica y los restantes, en diferentes esquemas de tratamiento. Otros hallazgos importantes fueron: 29 con p210BCR-ABL (de los cuales 16 tenían la fusión génica b2a2, 10 expresaban b3a2 y 3, la coexpresión b3a2/b2a2); 1 presentó p210BCR-ABL /p190BCR-ABL (b3a2/e1a2) y otro fue negativo para estos transcriptos. Se hicieron comparaciones de las frecuencias de las fusiones génicas que codifican para el transcripto p210 (b3a2, b2a2 y la coexpresión b3a2/b2a2) con las informadas en la literatura en trabajos similares. En el análisis de los resultados, de las ocho poblaciones informadas solo tres fueron estadísticamente significativas (13-17). Para el transcripto b3a2 fueron significativas las diferencias con las poblaciones europea, paquistaní y cubana (10,12,15) en relación a la estudiada en este trabajo, y para el transcripto b2a2 no se hallaron diferencias significativas (tabla 2).
DISCUSIÓN
En Latinoamérica existen otros estudios en los que se determinan las frecuencias de los transcriptos p210 y p190 en pacientes con LMC, pero no aparecen en PUBMED trabajos colombianos similares. Por lo tanto, se desconocen estos aspectos en nuestra población y este trabajo sería el primero de su género en el país.
La frecuencia del transcripto p210 en pacientes con LMC varía entre 90% y 100%, como se observa en la tabla 2. En este trabajo fue de 96,7% y al compararla con las informadas en la literatura se hallaron diferencias estadísticamente significativas con las poblaciones mexicana (91%; p = 0,038) (13), cubana (100%; p = 0,036) (15) y brasileña (72%; p = 0,0298) (18). Los valores publicados para los transcriptos p190 y/o las coexpresiones p210/p190/p230 varían entre 0% y 10%. En este estudio se obtuvo un valor del 3,2% para dichos transcriptos, sin diferencias significativas en comparación con las poblaciones mencionadas.
Por otra parte, encontramos una frecuencia del 34,5% para la fusión génica b3a2, estadísticamente diferente de las informadas en las poblaciones europea (55%; p = 0,043) (10), paquistaní (63,3%; p = 0,029) (12), brasileña (55,3%; p = 0,040) (14) y cubana (54,8%; p = 0,0295) (15). Para la fusión génica b2a2 encontramos una frecuencia de 55,2%, que es 1,6 veces mayor que la de b3a2; al comparar con los hallazgos informados en la literatura, no se encontraron diferencias significativas. Para la coexpresión b3a2/b2a2, solo se encontraron diferencias significativas con la población brasileña (8,5%; p = 0,025) (14).
Las diferencias encontradas en las frecuencias de los transcriptos p210 (b3a2 y b2a2) y p190 se pueden atribuir a distintos factores: (1) al número de individuos de las poblaciones en estudio; (2) a la fase de la enfermedad en el momento del estudio; (3) a variaciones en el proceso cotranscripcional de corte y empalme del ARN (splicing); (4) a diferencias étnicas, raciales y relacionadas con la situación geográfica.
Con respecto a las implicaciones clínicas de los transcriptos b2a2 y b3a2, varios autores han informado que los pacientes con LMC con el transcripto b2a2 tienen mejor pronóstico y responden más tempranamente al imatinib. De Lemos y colaboradores (19) encontraron diferencias estadísticamente significativas en la cuantificación de los transcriptos b2a2 y b3a2 después de seis meses de tratamiento con imatinib, indicativas de que aquellos con el transcripto b2a2 tienen mejor respuesta molecular. Posteriormente, Verma y colaboradores (20) informaron que los pacientes con las variantes raras como e1a2 presentan menor respuesta al imatinib. Además, Sharma y colaboradores (21) encontraron que 59% de los pacientes con el transcripto b2a2 lograban la respuesta citogenética completa (RCyC), en contraste con 28% de aquellos con b3a2 (p = 0,04). Por otro lado, se encontró que el 25% de quienes no tuvieron RCyC tenían el transcripto b2a2 y 75%, el b3a2 (p = 0,04). Cabe anotar que el número de pacientes analizados en el presente trabajo fue de solo 31 en comparación con los 252 del estudio cubano (15) y los 250 del mexicano (13); estas poblaciones más numerosas permiten encontrar con mayor probabilidad los transcriptos de baja frecuencia. Además, todos los pacientes de este trabajo se encontraban en distintas fases de la enfermedad, situación similar a la de los trabajos de referencia. Hoy se sabe que al avanzar la enfermedad pueden aparecer alteraciones secundarias en el cromosoma Ph; las más comunes son: la trisomía del cromosoma 8, el isocromosoma 17q (i17q) y la presencia de doble Ph. Con menor frecuencia pueden aparecer trisomías de los cromosomas 19, 21 y 17, monosomía del 7 y alteraciones del 3q (22), y a nivel molecular la coexpresión de los transcriptos p210 y p190. En este proceso de transformación se pueden seleccionar clones que expresan selectivamente uno de los transcriptos b3a2 o b2a2, lo que podría explicar la aparición con mayor frecuencia del b2a2 como ocurrió en este estudio (23,24).
En la última década se les ha dado gran importancia a las diferencias étnicas, al estado socioeconómico y al sexo en relación con la incidencia, el diagnóstico y el pronóstico de la enfermedad. Lee y colaboradores (25) examinaron las características demográficas y clínicas de los pacientes con LMC en diferentes poblaciones y encontraron que los hispanos presentaban un perfil de bajo riesgo y lograban mejor respuesta al tratamiento comparados con los no hispanos. Además, propusieron que los factores biológicos/genéticos pueden contribuir a las diferencias étnicas observadas en la presentación y el comportamiento de la enfermedad; los grupos étnicos hispanos tienen menor diversidad étnica que los descendientes de africanos.
Con respecto a la estructura étnica de la población colombiana y específicamente del departamento de Antioquia, Rojas y colaboradores (26), Reich y colaboradores (27) y Duque y colaboradores (28) hicieron varios estudios y determinaron la composición genética ancestral de la población antioqueña en la que hallaron componentes europeo (60%), amerindio (28%) y africano (12%). Lo anterior indica claramente que esta población tiene una estructura genética distinta a la de otras poblaciones latinoamericanas e incluso a las del resto del país.
Se consideró que los pacientes LMC20, LMC45 y LMC54 tenían resistencia citogenética a la terapia con mesilato de imatinib, y que los pacientes LMC12, LMC53 y LMC67 cursaban con crisis blástica en el momento de tomar la muestra. Aunque las frecuencias de las fusiones génicas BCR-ABL (b3a2 y/o b2a2) cambian de una población a otra, el proceso leucemogénico inducido por la proteína p210BCR-ABL es el mismo aunque exista diferencia pronóstica entre estas fusiones génicas; se podría sugerir que estas variaciones se deben al origen étnico de la población con LMC. Lo anterior sustenta que la respuesta clínica podría depender del tipo de proteína quimérica BCR-ABL y de la fusión génica que la codifique y explicaría diferencias en la respuesta al tratamiento con ITK de los pacientes según tengan las fusiones génicas b3a2 y/o b2a2 (29,30).
Otra explicación para las diferencias encontradas en los transcriptos puede ser que las variaciones en el proceso cotranscripcional se manifiestan como la coexpresión de los transcriptos p190 BCR-ABL y p210BCR-ABL debido a splicing alternativo en la porción génica BCR del transcripto primario BCR-ABL; esta expresión conjunta de transcriptos de fusión se puede detectar incluso en concentraciones bajas en la mayoría de los pacientes con LMC en el momento del diagnóstico con distinto valor pronóstico (31,32).
Por el contrario, en el presente estudio a ninguno de los pacientes con LMC en etapa diagnóstica se le detectó coexpresión de los transcriptos p190BCR-ABL y p210BCR-ABL, y el único que la presentó fue un individuo con 35 meses de tratamiento con mesilato de imatinib, al que era resistente. Otros autores consideran que la coexpresión de estos transcriptos en un paciente con LMC es un marcador de mal pronóstico que indica progresión de la enfermedad a la fase blástica debido a que la proteína p190BCR-ABL tiene un potencial oncogénico mayor que p210BCR-ABL y p230BCR-ABL situación que ocurrió en este individuo (33,34). Este paciente finalmente fue sometido a un Alo-TMO y 172 días después su análisis molecular fue positivo para la fusión génica b3a2. Lo anterior determinó el cambio de la conducta terapéutica: se llevó a cabo la infusión de linfocitos del donante con la finalidad de reducir las células leucémicas Ph+ mediante el efecto de injerto contra leucemia.
En esta investigación se diagnosticó por citogenética un caso de LMC con Ph+. Debido a esto, recibió terapia inicial con hidroxiúrea durante 42 meses y después mesilato de imatinib por 18 meses, momento en el que se efectuó la prueba molecular con RT-PCR cualitativa en la que resultó positivo para la amplificación del gen BCR normal (control interno), pero negativo para BCR-ABL. Esto indica una respuesta molecular completa (RMC) luego de la terapia en mención.
Otro caso interesante fue el de un paciente que presentó respuesta subóptima a los seis meses de terapia con mesilato de imatinib; en el análisis citogenético y el FISH se observaron células leucémicas con doble cromosoma Filadelfia, que es uno de los marcadores cromosómicos de progresión a crisis blástica; con frecuencia estos pacientes desarrollan alteraciones adicionales que afectan los cromosomas 8, 17 y 22 con duplicaciones del cromosoma Ph o trisomía 8 (35,36). No obstante, en este paciente la RT-PCR cualitativa detectó solo la presencia de la fusión génica b2a2, lo que significa que ambos cromosomas Filadelfia tenían el mismo transcripto de fusión y se originaron por ruptura en el mismo sitio; esto indica que posiblemente el agente leucemogénico se originó al mismo tiempo, con una exposición intensa y/o prolongada al ITK, generando dos cromosomas Filadelfia en la misma célula, factores oncogénicos cuya génesis está aún por esclarecer (37).
Se destaca además la importancia de hacerles análisis citogenético (cariotipo) a todos los pacientes, debido a que el análisis molecular, por sí solo, no detecta la presencia de un doble cromosoma Filadelfia ni la evolución clonal (38). Este paciente presentó resistencia al imatinib por lo que recibió un Alo-TMO; 237 días después continuaba positivo para la fusión génica, pero Filadelfia negativo por citogenética, lo cual es de mal pronóstico e indujo a recurrir a la infusión de linfocitos del donante. Esta situación particular permitió corroborar aún más la sensibilidad que tiene la RT-PCR y su utilidad en el tratamiento y pronóstico de los pacientes con LMC trasplantados (39-41).
En este trabajo se empleó una RT-PCR cualitativa, que solo permitió determinar la ausencia o la presencia del transcripto BCR-ABL y, por tanto, no se pudo establecer la correlación entre los grados de respuesta citogenética y/o molecular y la reducción de los niveles del transcripto ni conocer el grado de respuesta de estos pacientes al mesilato de imatinib. Tampoco se contó con los datos del seguimiento citogenético, y se desconoce si se suministró el fármaco de manera continua a todos los pacientes.
Con respecto a esta última situación, se encontraron dos pacientes que recibían de manera irregular el mesilato de imatinib, debido a retraso en la entrega del medicamento. Es importante resaltar que el suministro de este a algunos pacientes con diagnóstico de LMC no se hace de manera eficiente y oportuna, lo que favorece que los clones leucémicos Ph+ continúen proliferando y se pierdan las respuestas citogenética y/o molecular durante la terapia, además de la aparición de mutaciones en el dominio tirosina-cinasa de BCR-ABL.
Estas mutaciones se originan por la inestabilidad genómica de la célula madre leucémica inducida por la proteína BCR-ABL, que puede conferir resistencia a este fármaco y aumentar el riesgo de progresión de la fase crónica a la fase acelerada o a una crisis blástica (42,43). Lo anterior podría explicar los eventos de resistencia a este medicamento y sugerir que los individuos con LMC incluidos en el presente estudio, y los que están en tratamiento con mesilato de imatinib en Colombia, podrían presentar tasas de respuestas citogenéticas y/o moleculares distintas a las de pacientes con LMC tratados de manera adecuada y continua con este inhibidor de la tirosinacinasa (44-46). Con base en estudios epidemiológicos, GLOBOCAN informó, en el año 2008, para Colombia una incidencia anual de leucemias de 2.073 con 560 muertes, que podrían disminuir con un diagnóstico molecular oportuno y un tratamiento adecuado (47).
En este estudio, además, se demostró que la RT-PCR es de gran utilidad en la detección de los genes de fusión BCR-ABL: (1) para el diagnóstico de la LMC y de síndromes mieloproliferativos crónicos con sospecha de cromosoma Filadelfia; (2) para la detección de la enfermedad mínima residual en pacientes con LMC tratados con mesilato de imatinib, al confirmar la respuesta molecular completa a este fármaco; (3) en el seguimiento molecular cualitativo de pacientes con LMC tratados con Alo-TMO, con implicación pronóstica a los seis meses postrasplante al predecir tempranamente la pérdida del trasplante por la presencia de clones leucémicos cromosoma Ph+; (4) para permitir al médico tratante cambiar el esquema terapéutico, por ejemplo, hacer el refuerzo con linfocitos del donante; (5) para detectar la coexpresión de las fusiones génicas o transcriptos BCR-ABL, para determinar, durante el seguimiento, el clon leucémico resistente a la terapia con ITK.
En contraste, esta técnica mostró las siguientes desventajas: (1) solo detecta de manera cualitativa la presencia de las fusiones génicas y los transcriptos BCR-ABL y no permite la cuantificación de estos marcadores moleculares durante el tratamiento. Otros métodos, como la qPCR, dan solución a este problema midiendo los niveles basales de los transcriptos en las distintas etapas del seguimiento, lo que permite orientar el esquema terapéutico en estos pacientes. (2) no pueden determinar los grados de respuesta citogenética y molecular del paciente, hasta que alcance la remisión molecular completa; (3) no permite establecer el clon leucémico resistente en pacientes con doble cromosoma Filadelfia, que expresan solo una fusión génica.
En la actualidad, el uso de la qPCR para el diagnóstico de la enfermedad y durante su seguimiento permite identificar y cuantificar los transcriptos p210 y p190 estableciendo, principalmente, el grado de respuesta molecular. Sin embargo, como la RT-PCR es un poco más sensible que la qPCR, la primera es aún de gran utilidad para corroborar la remisión molecular completa o la negatividad del transcripto BCR-ABL por RTPCR cualitativa (48,49) y, además, diferenciar entre los tipos de transcripto (b3a2, b2a2, e1a2). Por último, su implementación, equipos e infraestructura no implican altos costos.
En conclusión, la prueba de RT-PCR cualitativa permitió conocer, en los pacientes estudiados, la frecuencia de los transcriptos p210BCR-ABL y p190BCR-ABL, así como los genes de fusión BCR-ABL que codifican para cada uno de ellos.
AGRADECIMIENTOS
Agradecemos a la Universidad de Antioquia, CODI CPT- 0329, la financiación de este proyecto. A los hematólogos, por el acompañamiento durante el desarrollo y ejecución de este trabajo. A los pacientes, por su colaboración y participación. A los hospitales y clínicas de la ciudad donde se llevaron a cabo la selección de los pacientes y la toma de muestras. Al personal de la Unidad de Genética Médica, en especial al profesor José Luis Ramírez, por su apoyo y colaboración. Al Programa para el Estudio y Control de Enfermedades Tropicales (PECET), por permitir desarrollar una parte del trabajo experimental. Al Joven Investigador, Juan David Vélez Aguirre, por su colaboración. A Susana Osorio Cardona, por su ayuda con la traducción de los textos.
REFERENCIAS BIBLIOGRÁFICAS
1. Goldman JM, Melo J V. Chronic myeloid leukemia-- advances in biology and new approaches to treatment. N Engl J Med. 2003 Oct 9;349(15):1451–64. [ Links ]
2. Melo J V. The molecular biology of chronic myeloid leukaemia. Leukemia. 1996 May;10(5):751–6. [ Links ]
3. Quintás-Cardama A, Cortes J. Molecular biology of BCR-ABL1-positive chronic myeloid leukemia. Blood. 2009 Feb 19;113(8):1619–30. [ Links ]
4. Laurent E, Talpaz M, Kantarjian H, Kurzrock R. The BCR gene and philadelphia chromosome-positive leukemogenesis. Cancer Res. 2001 Mar 15;61(6):2343–55. [ Links ]
5. Fujisawa S, Nakamura S, Naito K, Kobayashi M, Ohnishi K. A variant transcript, e1a3, of the minor BCRABL fusion gene in acute lymphoblastic leukemia: case report and review of the literature. Int J Hematol. 2008 Mar;87(2):184–8. [ Links ]
6. Swerdlow SH, Campo E, Harris NL, Jaffe ES, Pileri SA, Stein H, et al. WHO Classification of Tumours of Haematopoietic and Lymphoid Tissues. 4th ed. Geneva: WHO; 2008. [ Links ]
7. Faderl S, Hochhaus A, Hughes T. Monitoring of minimal residual disease in chronic myeloid leukemia. Hematol Oncol Clin North Am. 2004 Jun;18(3):657–70, ix–x. [ Links ]
8. Schrappe M. Minimal residual disease: optimal methods, timing, and clinical relevance for an individual patient. Hematology Am Soc Hematol Educ Program. 2012 Jan;2012:137–42. [ Links ]
9. Chomczynski P, Sacchi N. Single-step method of RNA isolation by acid guanidinium thiocyanatephenol- chloroform extraction. Anal Biochem. 1987 Apr;162(1):156–9. [ Links ]
10. van Dongen JJ, Macintyre EA, Gabert JA, Delabesse E, Rossi V, Saglio G, et al. Standardized RT-PCR analysis of fusion gene transcripts from chromosome aberrations in acute leukemia for detection of minimal residual disease. Report of the BIOMED-1 Concerted Action: investigation of minimal residual disease in acute leukemia. Leukemia. 1999 Dec;13(12):1901–28. [ Links ]
11. Cross NC. Detection of BCR-ABL in Hematological Malignancies by RT-PCR. Methods in molecular medicine. 1996 Jan;6:25–36. [ Links ]
12. Iqbal M, Ali S, Sheikh N, Khan M, et al. Frequency of BCR-Abl fusion oncogene splice variants associated with chronic myeloid leukemia (CML). JCT. 2011;02(02):176–80. [ Links ]
13. Arana-Trejo RM, Ruíz Sánchez E, Ignacio-Ibarra G, Báez de la Fuente E, Garces O, Gómez Morales E, et al. BCR/ABL p210, p190 and p230 fusion genes in 250 Mexican patients with chronic myeloid leukaemia (CML). Clin Lab Haematol. 2002 Jun;24(3):145–50. [ Links ]
14. de Carvalho DL, Barbosa CD, de Carvalho AL, Beck ST. Association of HLA antigens and BCR-ABL transcripts in leukemia patients with the Philadelphia chromosome. Rev Bras Hematol Hemoter. 2012 Jan;34(4):280–4. [ Links ]
15. Casanueva-Calero K, Pantaleón-Florido G, Ruiz- Noa Y, Mato-Luis J, Quesada-Dorta M, Carnot-Uria J. Estudio del reordenamiento molecular BCR-ABL en pacientes cubanos con diagnóstico presuntivo de leucemia mieloide crónica. Rev Hematol Mex. 2011;12(4):243–8. [ Links ]
16. Sastre DA, Argaraña CE, Heller VB, Gallo M, Fernández EN, Rodríguez CM. An analysis of multiplex-PCR in the detection of BCR-ABL transcripts in hematological disorders. Genet Mol Biol. 2007;30(3):520–3. [ Links ]
17. Artigas CG, Melo A, Roa JC, Roa I, Quijada I, Vittini C, et al. Transcriptos de fusión del gen BCR/ABL en pacientes con leucemia mieloide crónica. Int J Morphol. 2003;21(3) [ Links ].
18. Andrade G V. Papel da P190 BCR-ABL como parâmetro de recaída na leucemia mielóide crônica. Rev Bras Hematol. 2008 Aug;30(4) [ Links ].
19. de Lemos JAR, de Oliveira CM, Scerni ACC, Bentes AQ, Beltrão AC, Bentes IRG, et al. Differential molecular response of the transcripts B2A2 and B3A2 to imatinib mesylate in chronic myeloid leukemia. Genet Mol Res. 2005 Jan;4(4):803–11. [ Links ]
20. Verma D, Kantarjian HM, Jones D, Luthra R, Borthakur G, Verstovsek S, et al. Chronic myeloid leukemia (CML) with P190 BCR-ABL: analysis of characteristics, outcomes, and prognostic significance. Blood. 2009 Sep 10;114(11):2232–5. [ Links ]
21. Sharma P, Kumar L, Mohanty S, Kochupillai V. Response to Imatinib mesylate in chronic myeloid leukemia patients with variant BCR-ABL fusion transcripts. Ann Hematol. 2010 Mar;89(3):241–7. [ Links ]
22. Ossard-Receveur A, Bernheim A, Clausse B, Danglot G, Fauvet D, Leon B, et al. Duplication of the Phchromosome as a possible mechanism of resistance to imatinib mesylate in patients with chronic myelogenous leukemia. Cancer Genet Cytogenet. 2005 Dec;163(2):189–90. [ Links ]
23. Otero L, Ornellas MH, Dobbin J, de Souza Fernandez T. Double Philadelphia-chromosome: a resistance factor on the imatinib mesylate therapy for chronic myeloid leukemia. Int J Lab Hematol. 2008 Aug;30(4):346–8. [ Links ]
24. Kaur A, Kaur SP, Singh A, Singh JR. Karyotypic findings in chronic myeloid leukemia cases undergoing treatment. Indian J Hum Genet. 2012 Jan;18(1):66–70. [ Links ]
25. Lee JP, Birnstein E, Masiello D, Yang D, Yang AS. Gender and ethnic differences in chronic myelogenous leukemia prognosis and treatment response: a singleinstitution retrospective study. J Hematol Oncol. 2009 Jan;2:30. [ Links ]
26. Rojas W, Parra MV, Campo O, Caro MA, Lopera JG, Arias W, et al. Genetic make up and structure of Colombian populations by means of uniparental and biparental DNA markers. Am J Phys Anthropol. 2010 Sep;143(1):13–20. [ Links ]
27. Reich D, Patterson N, Campbell D, Tandon A, Mazieres S, Ray N, et al. Reconstructing Native American population history. Nature. 2012 Aug 16;488(7411):370–4. [ Links ]
28. Duque C, Parra MV, Valencia AV, Bedoya G, Ruiz-Linares A. Comparación de cuatro programas utilizados en la determinación de la composición genética ancestral de la población antioqueña. Revista Colombiana de Antropología. 2012;48(1):233–57. [ Links ]
29. Radich J, Gehly G, Lee A, Avery R, Bryant E, Edmands S, et al. Detection of BCR-abl transcripts in Philadelphia chromosome-positive acute lymphoblastic leukemia after marrow transplantation. Blood. 1997 Apr 1;89(7):2602–9. [ Links ]
30. Dombret H, Gabert J, Boiron J-M, Rigal-Huguet F, Blaise D, Thomas X, et al. Outcome of treatment in adults with Philadelphia chromosome-positive acute lymphoblastic leukemia--results of the prospective multicenter LALA-94 trial. Blood. 2002 Oct 1;100(7):2357–66. [ Links ]
31. van Dongen JJ, Macintyre EA, Gabert JA, Delabesse E, Rossi V, Saglio G, et al. Standardized RT-PCR analysis of fusion gene transcripts from chromosome aberrations in acute leukemia for detection of minimal residual disease. Report of the BIOMED-1 Concerted Action: investigation of minimal residual disease in acute leukemia. Leukemia. 1999 Dec;13(12):1901–28. [ Links ]
32. van Rhee F, Hochhaus A, Lin F, Melo J V, Goldman JM, Cross NC. p190 BCR-ABL mRNA is expressed at low levels in p210-positive chronic myeloid and acute lymphoblastic leukemias. Blood. 1996 Jun 15;87(12):5213–7. [ Links ]
33. Advani AS, Pendergast AM. BCR-Abl variants: biological and clinical aspects. Leuk Res. 2002 Aug;26(8):713– 20. [ Links ]
34. Agirre X, Román-Gómez J, Vázquez I, Jiménez-Velasco A, Larráyoz MJ, Lahortiga I, et al. Coexistence of different clonal populations harboring the b3a2 (p210) and e1a2 (p190) BCR-ABL1 fusion transcripts in chronic myelogenous leukemia resistant to imatinib. Cancer Genet Cytogenet. 2005 Jul 1;160(1):22–6. [ Links ]
35. Tauchi T, Ohyashiki K. Molecular mechanisms of resistance of leukemia to imatinib mesylate. Leuk Res. 2004 May;28 Suppl 1:S39–45. [ Links ]
36. Calabretta B, Perrotti D. The biology of CML blast crisis. Blood. 2004 Jun 1;103(11):4010–22. [ Links ]
37. Agarwal S, Tafel AA, Kanaar R. DNA double-strand break repair and chromosome translocations. DNA Repair (Amst). 2006 Sep 8;5(9-10):1075–81. [ Links ]
38. Jabbour E, Kantarjian H, Ghanem H, O'Brien S, Quintas- Cardama A, Garcia-Manero G, et al. The achievement of a 3-month complete cytogenetic response to second-generation tyrosine kinase inhibitors predicts survival in patients with chronic phase chronic myeloid leukemia after imatinib failure. Clin Lymphoma Myeloma Leuk. 2013 Jun;13(3):302–6. [ Links ]
39. Kantarjian H, Schiffer C, Jones D, Cortes J. Monitoring the response and course of chronic myeloid leukemia in the modern era of BCR-ABL tyrosine kinase inhibitors: practical advice on the use and interpretation of monitoring methods. Blood. 2008 Feb 15;111(4):1774–80. [ Links ]
40. Branford S. Monitoring after successful therapy for chronic myeloid leukemia. Hematology Am Soc Hematol Educ Program. 2012 Jan;2012:105–10. [ Links ]
41. O'Brien S, Radich JP, Abboud CN, Akhtari M, Altman JK, Berman E, et al. Chronic Myelogenous Leukemia, Version 1.2014. J Natl Compr Canc Netw. 2013 Nov;11(11):1327–40. [ Links ]
42. Weisberg E, Manley PW, Cowan-Jacob SW, Hochhaus A, Griffin JD. Second generation inhibitors of BCR-ABL for the treatment of imatinib-resistant chronic myeloid leukaemia. Nat Rev Cancer. 2007 May;7(5):345–56. [ Links ]
43. Koptyra M, Falinski R, Nowicki MO, Stoklosa T, Majsterek I, Nieborowska-Skorska M, et al. BCR/ABL kinase induces self-mutagenesis via reactive oxygen species to encode imatinib resistance. Blood. 2006 Jul 1;108(1):319–27. [ Links ]
44. Cohen MH, Williams G, Johnson JR, Duan J, Gobburu J, Rahman A, et al. Approval summary for imatinib mesylate capsules in the treatment of chronic myelogenous leukemia. Clin Cancer Res. 2002 May;8(5):935–42. [ Links ]
45. O'Brien SG, Guilhot F, Larson RA, Gathmann I, Baccarani M, Cervantes F, et al. Imatinib compared with interferon and low-dose cytarabine for newly diagnosed chronic-phase chronic myeloid leukemia. N Engl J Med. 2003 Mar 13;348(11):994–1004. [ Links ]
46. Krause DS, Van Etten RA. Tyrosine kinases as targets for cancer therapy. N Engl J Med. 2005 Jul 14;353(2):172–87. [ Links ]
47. World Health Organization. GLOBOCAN 2012: estimated cancer incidence, mortality, prevalence and disability-adjusted life years (DALYs) Worldwide in 2008 [Internet]. http://globocan.iarc.fr. 2012. Available from: http://globocan.iarc.fr/Default.aspx [ Links ]
48. White HE, Hedges J, Bendit I, Branford S, Colomer D, Hochhaus A, et al. Establishment and validation of analytical reference panels for the standardization of quantitative BCR-ABL1 measurements on the international scale. Clin Chem. 2013 Jun;59(6):938–48. [ Links ]
49. Moore FR, Rempfer CB, Press RD. Quantitative BCRABL1 RQ-PCR fusion transcript monitoring in chronic myelogenous leukemia. Methods Mol Biol. 2013 Jan;999:1–23. [ Links ]