Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Iatreia
Print version ISSN 0121-0793
Iatreia vol.28 no.3 Medellín July/Aug. 2015
https://doi.org/10.17533/udea.iatreia.v28n3a05
ARTÍCULO DE REVISIÓN
DOI 10.17533/udea.iatreia.v28n3a05
Determinación de la clonalidad en tejidos humanos
Clonality evaluation in human tissues
Determinação da clonagem em tecidos humanos
Nicolás Villamizar-Rivera1; Natalia Olaya2
1 Licenciado en Biología, Coordinador del proyecto de investigación Estudio de la clonalidad linfoide por medio del análisis de reordenamientos del receptor del antígeno. Grupo Patología Oncológica, Instituto Nacional de Cancerología, E.S.E. Bogotá Colombia
2 Doctora en Ciencias. Patóloga, Médica Especialista. Instituto Nacional de Cancerología. E.S.E. Bogotá Colombia. nolaya@cancer.gov.co
Recibido: marzo 27 de 2014
Aceptado: septiembre 29 de 2014
RESUMEN
Las proliferaciones malignas suelen ser clonales. La mayoría de las veces el potencial de una lesión se establece por medio del análisis clínico y el estudio anatomopatológico, pero algunos casos son de difícil diagnóstico. Por otra parte, existen situaciones en las que se producen clonas dominantes cuyo análisis es importante, tal como ocurre en enfermedades autoinmunes e inmunodeficiencias. Este artículo presenta de manera comprensible las técnicas principales para el estudio de la clonalidad, a saber: la evaluación de los reordenamientos génicos del receptor de antígeno y la evaluación del gen del receptor de antígeno humano.
PALABRAS CLAVE
Cáncer, Inactivación del Cromosoma X, Leucemia, Linfoma, Reordenamiento Génico
SUMMARY
Malignant proliferations are usually clonal. While most times the biological potential can be established through routine pathologic and clinical examinations, some cases are difficult to classify. Moreover, in some situations there are dominant clones whose analysis is important, such as in autoimmune diseases and immunodeficiency. This paper presents in an understandable way the main techniques for the study of clonality, namely: evaluation of gene rearrangements of antigen receptor, and evaluation of human antigen receptor gene.
KEY WORDS
Cancer, DNA Rearrangements, Leukemia, Lymphoma, X Chromosome Inactivation
RESUMO
As proliferações malignas costumam ser clonadas. A maioria das vezes o potencial biológico de uma lesão se estabelece por meio da análise clínica e o estudo anatomopatológico, mas alguns casos são de difícil diagnóstico. Por outra parte, existem situações nas que se produzem clones dominantes cujo análise é importante, tal como ocorre em doenças autoimunes e imunodeficiências. Este artigo apresenta de maneira compreensível as técnicas principais para o estudo da clonagem, a saber: a avaliação dos reordenamentos genéticos do receptor de antígeno e a avaliação do gene do receptor de antígeno humano.
PALAVRAS CHAVE
Câncer, Inativação do Cromossomo X, Leucemia, Linfoma, Reordenamento Genético
Cómo citar: Villamizar-Rivera N, Olaya N. Determinación de la clonalidad en tejidos humanos. Iatreia. 2015 Jul-Sep;28(3): 269-282. DOI 10.17533/udea.iatreia.v28n3a05.
INTRODUCCIÓN
La teoría en boga para explicar el desarrollo del cáncer es la evolución. Se supone que las células somáticas sufren un proceso evolutivo gradual en el que se produce la selección y triunfo de células capaces de escapar a las limitaciones impuestas por el ambiente, es decir, la lucha por el espacio y los nutrientes, y a los mecanismos celulares de control de la división celular y del envejecimiento (1). La presión selectiva es tal que la mayoría de los procesos neoplásicos progresivos ocurren solo a edades avanzadas. Según la teoría, una célula transformada da origen a una población de células hijas o clon, la cual conforma la lesión tumoral. En general, pero no en todos los casos, clonal equivale a neoplásico (1).
Las leucemias y los linfomas son cánceres originados en los linfocitos o sus progenitores; afectan cualquier topografía corporal y a personas de todas las edades. Para diagnosticarlos y clasificarlos, se evalúan la morfología, el inmunofenotipo y las alteraciones genéticas (2,3). En las proliferaciones linfoides maduras se determina la relación de expresión de las cadenas λ y κ de las inmunoglobulinas (4-6).
Estos métodos permiten clasificar la mayoría de las lesiones linfoides, pero un porcentaje de los casos son difíciles y los reportes diagnósticos sufren retraso o se encarecen por la solicitud de grandes paneles de inmnohistoquímica, no siempre concluyentes. Esto necesariamente afecta la oportunidad o calidad del tratamiento. Este es un problema en general invisible en la literatura pues a la larga el patólogo o los médicos tratantes se ven obligados a tomar una decisión. Además, a veces es necesario determinar si dos lesiones presentes en el mismo paciente tienen el mismo origen o son diferentes (7,8).
El rearreglo de los genes del receptor de antígeno es un proceso fisiológico a partir de cuyo estudio se puede determinar la clonalidad linfoide. Son candidatos a este análisis las proliferaciones de células B de clasificación difícil, todas las lesiones de células T y aquellas que ocurren en pacientes trasplantados, inmunodeficientes, con enfermedades autoinmunes o asociadas con linfoproliferación, tales como la enfermedad inflamatoria intestinal y la enfermedad celíaca (9,10).
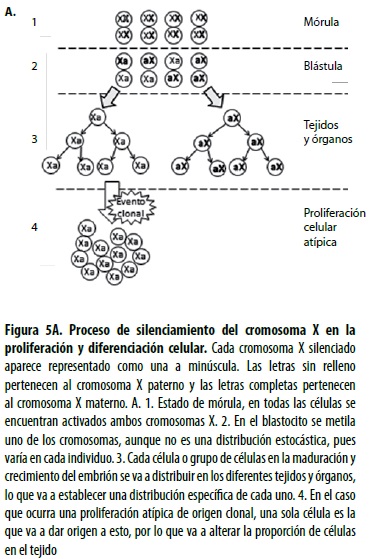
Entre tanto, para evaluar la clonalidad de lesiones no linfoides se pueden utilizar las técnicas basadas en el proceso de lionización o inactivación del cromosoma X. Durante la morulación, las hembras inactivan los alelos paternos o maternos de una parte de los genes del cromosoma X (11). La expresión de uno u otro se altera cuando se produce una proliferación clonal. No se han generalizado para uso diagnóstico las pruebas basadas en la inactivación de genes del cromosoma X, pues solo es posible utilizarlas en mujeres y su interpretación es controversial, pero son las únicas que hasta el momento permiten hacer un diagnóstico de clonalidad en lesiones no linfoides.
Nos proponemos introducir a los lectores en los principios científicos y de aplicación de las principales técnicas de evaluación de la clonalidad.
EVALUACIÓN DE LOS REORDENAMIENTOS DE GENES DEL RECEPTOR DEL ANTÍGENO
Los genes de las inmunoglobulinas (Ig) y del receptor de los linfocitos T (TCR) están presentes en todas las células del organismo, pero a la manera de un rompecabezas sin armar. Por esta razón, los linfocitos requieren convertir los fragmentos génicos apartados en genes funcionales para los receptores de antígeno y generar diversidad antigénica (12). Este proceso se conoce como rearreglo o reordenamiento, términos que se utilizarán indistintamente en este texto. Cuando el reordenamiento de los genes del receptor de antígeno no es perfecto, bien sea por falta de alguna enzima o por otro defecto, se presentan graves inmunodeficiencias (13). Por otra parte, cuando una célula linfoide se transforma en cancerosa y se divide sin cesar, sus células hijas poseen el mismo reordenamiento de los genes de receptor de antígeno que la célula madre que las originó.
Los loci que codifican para las cadenas pesadas de las Ig, las cadenas ligeras κ y λ de Ig, así como las cadenas α, β, γ y δ del TCR se encuentran en cromosomas diferentes. Además, existen varias secuencias posibles para cada uno de los segmentos que constituyen la región variable del gen. La organización germinal de los genes de las Ig y del TCR (figura 1), no puede transcribirse en ARNm (12).
En cada linfocito, la recombinación somática elabora un exón que codificará para la región variable a partir de la selección de un segmento variable (V), un segmento de unión (J) y un segmento de diversidad (D) y la adición de nucleótidos N y P, que posteriormente serán transcritos en ARNm y finalmente traducidos y madurados en una proteína funcional (figura 2). El segmento D solo está presente en los loci de las cadenas pesadas de Ig y los loci β y δ son exclusivos de las del TCR (figura 2). La recombinación alélica ocurre cuando se unen los fragmentos variables, de diversidad y unión, llamada recombinación V(D)J que se regula por medio de exclusión alélica; una vez que un alelo se ha reordenado, se envía una señal al otro para interrumpir el proceso (12,13).
Los reordenamientos se efectúan por medio de pasos secuenciales e invariables:
1. Sinapsis: el mecanismo enzimático detecta las secuencias señal de la recombinación o RSS. Estas constan de tres porciones: un heptámero de nucleótidos conservados, un espaciador de 12 o 23 nucleótidos variables y un nonámero de nucleótidos rico en AT. El espaciador corresponde a giros de la hélice de ADN, de manera que los heptámeros se encuentran en posiciones vulnerables al ataque enzimático por la V(D)J recombinasa. Estas enzimas son propias de los linfocitos en desarrollo y se encuentran inactivas durante la proliferación celular.
2. Escisión: roturas en la doble cadena de ADN en las uniones entre la RSS y la secuencia codificadora. La unión entre los heptámeros se lleva a cabo eliminando el ADN entre ellos, o formando un bucle por inversión de las cadenas. Los extremos codificadores rotos finalizan por una horquilla cerrada (14).
3. Apertura de las horquillas por medio de la enzima Artemisa, una endonucleasa, con el fin de que la enzima TDT (por la sigla en inglés de Terminal deoxynucleotydil transferase) añada nuevas bases en los 2. Proceso de recombinación del receptor del antígeno. En la se muestra el procesamiento de la cadena pesada de las inmunoglobulinas, pero los procesos son similares para las cadenas ligeras de Ig y el TCR. Este proceso permite obtener entre 1x1010 -1x1018 combinaciones. Momentos antes de la transcripción del ADN se eliminan todos los segmentos constantes de la línea germinal excepto uno, y este es el que se expresa. El segmento líder no se traduce a proteína extremos expuestos, aumentando la diversidad de las secuencias.
4. Unión y finalización por medio del sistema de unión de extremos no homólogo, un mecanismo de reparación de ADN presente en todas las células. Más tarde, los linfocitos B maduros aumentan su repertorio de combinación por medio de hipermutación somática en el centro germinal. Se caracteriza por la adición o sustracción de nucleótidos (14,15).
Todavía se desconoce cómo un locus determinado se selecciona en un caso específico o por qué algunos fragmentos son preferidos con mayor reiteración: por ejemplo, el segmento VH81X, el más proximal de los variables de la cadena pesada, se reordena con mayor frecuencia que los segmentos distales (12). En las neoplasias linfoides ocurren reordenamientos de uno u otro receptor independientemente del linaje de la lesión, pero durante el proceso fisiológico las regiones variables de las Ig son ensambladas en los linfocitos B, mientras que las regiones variables del TCR son acopladas solo en los linfocitos T.
Cuando en un tejido linfoide hay una población neoplásica, predomina un grupo de reordenamientos génicos. Cuando se trata de una población policlonal, hay enorme variación de reordenamientos. Este hecho sirve para diferenciar una población de células linfoides neoplásicas de una reactiva. Durante largo tiempo se utilizó con este fin el Southern Blot. La técnica es muy específica, pero sus exigencias metodológicas limitan la aplicación (13). Desde hace diez años existe un grupo de protocolos basados en PCR (reacción en cadena de la polimerasa, por la sigla en inglés de polymerase chain reaction) (7). La estrategia, denominada BIOMED-2, fue desarrollada por un conjunto de laboratorios europeos, que más tarde conformó el consorcio EUROCLONALITY (16). Desde entonces han aparecido numerosas publicaciones acerca de su desempeño y otras que ofrecen mejoras de la técnica original (17,18).
Se puede evaluar rápidamente el repertorio de combinaciones TCRVβ por medio de citometría de flujo (19,20). El consorcio Euroflow, conformado en parte por los mismos laboratorios del consorcio BIOMED-2, propone un panel de 24 anticuerpos, que permiten determinar el 70% de los dominios del TCRβ (21). Según Lima y colaboradores (22), este análisis citométrico es una buena herramienta de tamización para la clonalidad de lesiones linfoides de linaje T en especial en el caso de la leucemia de células grandes granulares. No obstante, su uso no se ha generalizado para todas las proliferaciones linfoides, para las cuales se considera que el estándar de oro es la evaluación por PCR.
El estudio de los reordenamientos génicos del receptor de antígeno exige un ejercicio multidisciplinario entre la patología, la citometría de flujo, la genética y la biología molecular (23). A diferencia de las demás pruebas que evalúan cambios patológicos del ADN, el principio de las pruebas para la evaluación de la clonalidad linfoide es un proceso fisiológico, de manera que el significado de los clones, en caso de encontrarse, se debe determinar en un contexto adecuado (24). Además de utilizarse en oncología, el estudio de los rearreglos es útil en casos de trasplante y enfermedades autoinmunes (17,25).
PROCEDIMIENTO TÉCNICO
Indicaciones de la técnica y muestras
Los protocolos de PCR multiplex propuestos por BIOMED- 2 están diseñados para funcionar en muestras de tejido fresco o congelado. Sin embargo, se han realizado con éxito en tejidos parafinados (26,27). Para cualquier tejido la condición es que contenga una cantidad representativa de células problema (7,20,28).
Las pruebas están indicadas en pacientes que presenten proliferaciones linfoides de difícil clasificación, en las cuales el diagnóstico diferencial sea entre lesiones reactivas y neoplásicas. Son de particular utilidad en el caso de lesiones linfoides en la piel y de linfomas asociados con las mucosas (MALT), entre otros.
El patólogo escoge el área de tejido que se va a examinar. Si es del caso, hace macro o microdisección de la muestra con el fin de enriquecerla en células problema, pero es importante que persistan poblaciones policlonales. Se debe evitar seleccionar regiones naturalmente oligoclonales, como los centros germinales de los folículos linfoides.
Los controles negativos son poblaciones policlonales de linfocitos, obtenidos a partir de sangre periférica o de tejidos linfoides normales. Se debe verificar que en ellos no existan clones dominantes (29,30). Como controles positivos se usan líneas celulares comerciales con reordenamientos conocidos o lesiones clonales caracterizadas (7,23).
Para facilitar la interpretación, es necesario tener información sobre el caso en estudio: datos demográficos y clínicos, informe de patología, inmunofenotipo, presencia y cantidad de linfocitos reactivos, etc. Los tratamientos recibidos afectan los resultados; por ejemplo, la administración de anti-CD20 dificulta la determinación de la clonalidad de células B, y algunas traslocaciones, tales como t(11; 14) y t(14; 18) pueden dar resultados falsos negativos, porque involucran rearreglos aberrantes de las cadenas pesadas de las Ig (31).
Aislamiento de ácidos nucleicos y evaluación de la calidad
El tiempo de isquemia, los procedimientos de congelación y almacenamiento y la inclusión en parafina afectan la calidad del ADN extraído de los tejidos. Son importantes el tipo y tiempo de fijación, el espesor del tejido, los procedimientos de extracción del ADN y la presencia de inhibidores de la PCR (28,32).
Las pruebas requieren ADN de alta pureza y baja fragmentación. Si se utilizan muestras frescas o congeladas, cualquier método de extracción, tanto comercial como tradicional, funciona bien, incluyendo el uso de extractores automáticos. En el caso del tejido en parafina, se recomienda el uso de formalina tamponada al 10% como fijador y un cuidadoso control de los tiempos de fijación. Aunque los protocolos BIOMED-2 recomiendan el kit QIAMP DNA Mini kit (QIAGEN®), otras publicaciones sugieren extracción orgánica y purificación (27,32). Una de las ventajas de la extracción orgánica es su eficiencia; la concentración de ADN obtenida es mayor y puede considerarse la dilución de la muestra para disminuir el efecto de los inhibidores de la PCR que puedan estar presentes. En cualquier caso, si se utiliza tejido en parafina se recomienda verificar que sea amplificable al menos hasta 300 nucleótidos. BIOMED-2 incluye un protocolo de amplificación que permite evaluar productos de varios tamaños, pero existen otras opciones (7,27).
Selección de blancos
La selección de los fragmentos que se desea amplificar depende de la pregunta clínico-patológica y de la cantidad y la calidad del ADN extraído. En el caso de los tejidos parafinados, casi siempre se deberán evaluar los productos de PCR o amplicones de menor tamaño. En algunos casos es posible amplificar fragmentos mayores de lo predicho por medio del control de calidad. Además, la pregunta clínica ayuda, ya que la frecuencia y distribución de los reordenamientos varían según la taxonomía tumoral (tablas 1a y 1b).
Si la cantidad de células linfoides en la muestra es baja, como en las infiltraciones de piel o intestino, es necesario ajustar las cantidades de ADN y efectuar las pruebas por duplicado. Se propone un algoritmo para la selección de los blancos moleculares (32) (figura 3).
Amplificación y análisis de los productos de PCR
No se pueden detectar por medio de PCR todos los segmentos génicos posibles, ya que se requerirían demasiados primers y tubos. Sin embargo, los primers consensus que propone el consorcio reconocen secuencias conservadas entre los fragmentos y permiten detectar la mayor parte de combinaciones posibles. El protocolo de amplificación permite la utilización de cualquier termociclador (7,21).
El análisis de los productos de las PCR multiplex se hace por medio de electroforesis en gel de acrilamida o capilar. También se ha usado electroforesis microcapilar de alta resolución por medio del equipo Agilent 2100 Bioanalyzer (33). Se puede hacer el análisis de fragmentos de doble cadena (formación de heterodupletas) y el de productos de una sola cadena (34,35). Con el fin de inducir la formación de heterodupletas, los productos de la PCR multiplex se someten a calentamiento y enfriamiento rápido (36). La de un pico o banda dominante sugiere la presencia de un clon (7) (figura 4).
INTERPRETACIÓN Y DIFICULTADES
Tanto para la ejecución como para la interpretación es necesario conocer la estructura de los genes que se van a evaluar, sus patrones de reordenamiento y las variaciones de estos; además, se deben verificar los patrones de expresión de los controles. Los resultados experimentales deben ser reproducibles y analizarse de manera multidisciplinaria (37).
Ocasionalmente ocurren falsos positivos o negativos en la aplicación de estos ensayos. Por ejemplo, el evaluador puede enfrentarse con productos de PCR que no son del tamaño esperado o que son múltiples. En el caso de muestras que contienen escasos linfocitos, se pueden obtener picos únicos no específicos, lo cual se denomina pseudoclonalidad. Además los tejidos, incluso los tumorales, suelen tener una población de fondo policlonal normal, que, si es muy prominente, puede ser de difícil interpretación.
Los falsos negativos ocurren por varias razones: 1. Se utilizan primers o iniciadores consenso; entonces se amplifican alrededor del 80% de los posibles segmentos, pero no todos. 2. En las neoplasias de células B maduras del centro germinal, cuyas células pueden haber sufrido hipermutación somática, no habrá amplificación. 3. La lesión está insuficientemente representada en el tejido examinado (7). Creemos que con el tiempo se desarrollarán primers que permitan la amplificación de todos los segmentos posibles y que sean menos sensibles a la hipermutación somática.
La detección de uno o varios clones no implica la presencia de una neoplasia, razón por la cual insistimos en que los resultados se deben interpretar en colaboración con todas las personas a cargo del diagnóstico y tratamiento del paciente (23). Es posible recurrir a los servicios de EUROCLONALITY para la interpretación de resultados confusos (15). Aunque en América Latina algunos grupos tienen experiencia con la utilización de las técnicas, esta sigue siendo relativamente marginal, al menos en lo que concierne a las publicaciones que pueden encontrarse sobre el tema en los buscadores de literatura más comunes (38-40).
ENFERMEDAD MÍNIMA RESIDUAL (EMR)
Cuando se diagnostica una leucemia aguda el paciente tiene alrededor de 1011-1012 células. Lo que se denomina remisión completa después de la inducción es la reducción de 10 veces esta cantidad, a 1010 (41). Con el fin de predecir y evitar una recaída clínica se requiere detectar los blastos en las fases subclínicas, lo cual se conoce como enfermedad mínima residual (EMR) (42).
En general, la EMR es un factor pronóstico importante en leucemias y linfomas (43,44). La finalidad de las pruebas para determinación de EMR es detectar hasta el último blasto. Cuando la enfermedad se asocia con un daño genético específico, como la t (9; 22), se puede utilizar este como marcador de EMR. En el caso de las leucemias agudas, la técnica más utilizada es la inmunofenotipificación por medio de citometría de flujo; a veces se usa la determinación de proteínas quiméricas (45) (tabla 2).
Las técnicas de biología molecular para la determinación de reordenamientos de los genes del receptor de antígeno tienen una sensibilidad comparable o mayor que la de la citometría de flujo (30). Se han estudiado incluso para el seguimiento en linfoma de Hodgkin, que se caracteriza por la escasez de células tumorales y la carencia de traslocación específica (33).
Para determinar la EMR por medio de las pruebas de reordenamientos de Ig y TCR, se aplican los protocolos sobre una muestra diagnóstica rica en células tumorales, y se secuencian los reordenamientos más representativos; a partir de los resultados se diseñan primers específicos y se ponen en marcha ensayos cuantitativos por PCR (44). Un 30% de los casos presentan evolución clonal y requieren seguimiento por varios rearreglos (45); sin embargo, puede decirse que el rastreo por medio de esta técnica no solo es muy preciso, sino personalizado, además de considerarse como el estándar de oro (46).
EVALUACIÓN DE LA INACTIVACIÓN DEL GEN DEL RECEPTOR DE ANDRÓGENO HUMANO
Antes de que ocurra la diferenciación, las células del embrión de sexo femenino inactivan, al azar, la mayor parte de los genes de uno de los dos cromosomas X que poseen (47). El cromosoma X codifica para numerosos genes, de los cuales el 15% nunca se inactivan y el 10% se inactivan de forma incompleta (47). Como resultado, se obtienen tejidos en los cuales se expresa más o menos el cromosoma X paterno o el X materno (48). El mecanismo principal mediante el cual se inactivan grandes porciones de uno u otro cromosoma es la metilación de las islas CpG en los extremos 5´de los genes. Luego, estos patrones de metilación son heredados por las células somáticas (10) (figura 5A).
Se pueden determinar los linajes originales de las poblaciones celulares en hembras evaluando la inactivación de los genes codificados por el cromosoma X, con la condición de que dichos genes sean heterocigotos; puede realizarse evaluando la metilación de la secuencia, por análisis de polimorfismos o por medio de la determinación de las isoformas proteicas que se expresan en cada línea. Se han descrito varios genes informativos que podrían utilizarse para el análisis, entre los cuales están G6PD, P55, BTK, FHL-1 y fosfoglicerato quinasa (49). Además del receptor de andrógeno humano AR se han usado otros genes, tales como FMR1, M27 beta e hipoxantina fosforribosiltransferasa (HPRT) (10).
PROCEDIMIENTO TÉCNICO
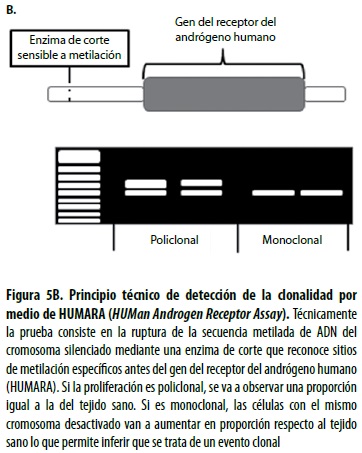
Para determinar el patrón de metilación del cromosoma X se efectúa el ensayo del receptor de andrógenos humanos (HUMARA, por la sigla en inglés de HUMan Androgen Receptor Assay), que es la técnica más conocida y permite establecer la clonalidad en lesiones no linfoides de mujeres. Utiliza las enzimas de restricción sensibles a la metilación HPAII y HhaI. Las enzimas digieren las citocinas no metiladas. Los sitios de clivaje de las enzimas están cercanos a un SRT (short tandem repeat) polimórfico del gen. Posteriormente se hace una PCR con el fin de amplificar un fragmento que incluya las regiones de flanqueo HPAII y HhaI y el SRT. En caso de que haya metilación de la secuencia, se producirá amplificación de esta y habrá un producto de PCR evaluable. Se ha encontrado que la metilación se correlaciona con la inactivación del cromosoma X. En tal caso, se considera que el alelo inactivado es el que amplifica de manera preferencial (figura 5B).
INTERPRETACIÓN Y DIFICULTADES
HUMARA ha permitido diferenciar entre lesiones malignas y reactivas, tanto linfoides como epiteliales (48,49). Sin embargo, la prueba solo puede utilizarse en mujeres. Además, en edades avanzadas se pueden producir alteraciones del patrón de expresión que pueden crear confusión (50). Por otra parte, no hay versiones comerciales de la técnica, y su uso en un contexto clínico requiere un cuidadoso proceso de estandarización y validación.
CONCLUSIÓN
El diagnóstico correcto es el primer requisito para un tratamiento exitoso, en particular en el caso de las neoplasias. Los dos procedimientos mencionados para confirmar la clonalidad, es decir, el análisis de rearreglos de receptor de antígeno y el HUMARA se han conocido por decenios. El HUMARA no es, por ahora, una técnica que se utilice de rutina en casos clínicos, pero los reordenamientos de receptor de antígeno son la regla de oro para la confirmación de clonalidad y para la determinación de recaída molecular en muchos casos de proliferación linfoide. Es importante que los médicos tratantes conozcan los principios técnicos mediante los cuales se llevan a cabo ambos procedimientos, sus indicaciones y su interpretación.
CONFLICTOS DE INTERESES
Ninguno para declarar.
REFERENCIAS BIBLIOGRÁFICAS
1. Greaves M, Maley CC. Clonal evolution in cancer. Nature. 2012 Jan;481(7381):306-13. [ Links ]
2. Swerdlow S, Campo E, Harris NL, Jaffe E., Pileri S., Stein H, et al. WHO Classification of Tumours of Haematopoietic and Lymphoid Tissues. 4th ed. Lyon: International Agency for Research on Cancer; 2008. [ Links ]
3. Rezuke WN, Abernathy EC, Tsongalis GJ. Molecular diagnosis of B- and T-cell lymphomas: fundamental principles and clinical applications. Clin Chem. 1997 Oct;43(10):1814-23. [ Links ]
4. Jaffe ES, Harris NL, Vardiman J, Elias Campo E, Arber DM. Hematopathology. St. Louis: Elsevier; 2010. [ Links ]
5. Goud KI, Dayakar S, Prasad SVSS, Rao KN, Shaik A, Vanjakshi S. Molecular diagnosis of lymphoblastic leukemia. J Cancer Res Ther. 2013 Jul;9(3):493-6. [ Links ]
6. Ståhlberg A, Aman P, Ridell B, Mostad P, Kubista M. Quantitative real-time PCR method for detection of B-lymphocyte monoclonality by comparison of kappa and lambda immunoglobulin light chain expression. Clin Chem. 2003 Jan;49(1):51-9. [ Links ]
7. van Dongen JJM, Langerak AW, Brüggemann M, Evans PAS, Hummel M, Lavender FL, et al. Design and standardization of PCR primers and protocols for detection of clonal immunoglobulin and T-cell receptor gene recombinations in suspect lymphoproliferations: report of the BIOMED-2 Concerted Action BMH4-CT98-3936. Leukemia. 2003 Dec;17(12):2257-317. [ Links ]
8. Gazzola A, Mannu C, Rossi M, Laginestra MA, Sapienza MR, Fuligni F, et al. The evolution of clonality testing in the diagnosis and monitoring of hematological malignancies. Ther Adv Hematol. 2014 Apr;5(2):35-47. [ Links ]
9. Perfetti V, Brunetti L, Biagi F, Ciccocioppo R, Bianchi PI, Corazza GR. TCRβ clonality improves diagnostic yield of TCRγ clonality in refractory celiac disease. J Clin Gastroenterol. 2012 Sep;46(8):675-9. [ Links ]
10. Sirsath NT, Channaviriappa LK, Nagendrappa LK, Dasappa L, Sathyanarayanan V, Setty GBK. Human immunodeficiency virus - associated lymphomas: a neglected domain. N Am J Med Sci. 2013 Jul;5(7):432-7. [ Links ]
11. Chen GL, Prchal JT. X-linked clonality testing: interpretation and limitations. Blood. 2007 Sep;110(5):1411-9. [ Links ]
12. Abbas AK, Lichtman AH, Pillai S, Igea Aznar JM. Inmunología celular y molecular. 6a ed. Barcelona: Elsevier; 2009. [ Links ]
13. Giallourakis CC, Franklin A, Guo C, Cheng H-L, Yoon HS, Gallagher M, et al. Elements between the IgH variable (V) and diversity (D) clusters influence antisense transcription and lineage-specific V(D) J recombination. Proc Natl Acad Sci U S A. 2010 Dec;107(51):22207-12. [ Links ]
14. Deriano L, Chaumeil J, Coussens M, Multani A, Chou Y, Alekseyenko AV, et al. The RAG2 C terminus suppresses genomic instability and lymphomagenesis. Nature. 2011 Mar;471(7336):119-23. [ Links ]
15. Knowles DM, Neri A, Pelicci PG, Burke JS, Wu A, Winberg CD, et al. Immunoglobulin and T-cell receptor beta-chain gene rearrangement analysis of Hodgkin's disease: implications for lineage determination and differential diagnosis. Proc Natl Acad Sci U S A. 1986 Oct;83(20):7942-6. [ Links ]
16. Rombout PDM, Diss TC, Hodges E; Hummel M, van Dongen JJM, Langerak AW, Groenen PJTA. The Euro- Clonality website: information, education and support on clonality testing [internet]. J Hematopathol DOI 10.1007/s12308-011-0120-x. Disponible en: http://www.moleculardiagnostics.be/images/3SCM_Langerak_article1.pdf [ Links ]
17. Melotti CZ, Amary MFC, Sotto MN, Diss T, Sanches JA. Polymerase chain reaction-based clonality analysis of cutaneous B-cell lymphoproliferative processes. Clinics (Sao Paulo). 2010 Jan;65(1):53-60. [ Links ]
18. Harris S, Bruggemann M, Groenen PJTA, Schuuring E, Langerak AW, Hodges E. Clonality analysis in lymphoproliferative disease using the BIOMED-2 multiplex PCR protocols: experience from the EuroClonality group EQA scheme. J Hematop. 2012 Mar;5(1-2):91-8. [ Links ]
19. van den Beemd R, Boor PP, van Lochem EG, Hop WC, Langerak AW, Wolvers-Tettero IL, et al. Flow cytometric analysis of the Vbeta repertoire in healthy controls. Cytometry. 2000 Aug;40(4):336-45. [ Links ]
20. Ribera J, Zamora L, Juncà J, Rodríguez I, Marcé S, Cabezón M, et al. Usefullness of IGH/TCR PCR studies in lymphoproliferative disorders with inconclusive clonality by flow cytometry. Cytometry B Clin Cytom. 2013 Jul 25; 21118(b):10.1002. [ Links ]
21. Kalina T, Flores-Montero J, van der Velden VHJ, Martin- Ayuso M, Böttcher S, Ritgen M, et al. EuroFlow standardization of flow cytometer instrument settings and immunophenotyping protocols. Leukemia. 2012 Sep;26(9):1986-2010. [ Links ]
22. Lima M, Almeida J, Santos AH, dos Anjos Teixeira M, Alguero MC, Queirós ML, et al. Immunophenotypic analysis of the TCR-Vbeta repertoire in 98 persistent expansions of CD3(+)/TCR-alphabeta(+) large granular lymphocytes: utility in assessing clonality and insights into the pathogenesis of the disease. Am J Pathol. 2001 Nov;159(5):1861-8. [ Links ]
23. Groenen PJTA, Langerak AW, van Dongen JJM, van Krieken JHJM. Pitfalls in TCR gene clonality testing: teaching cases. J Hematop. 2008 Sep;1(2):97-109. [ Links ]
24. Ortiz YM, Arias LF, Alvarez CM, García LF. Memory phenotype and polyfunctional T cells in kidney transplant patients. Transpl Immunol. 2013 Mar;28(2- 3):127-37. [ Links ]
25. Vakiani E, Basso K, Klein U, Mansukhani MM, Narayan G, Smith PM, et al. Genetic and phenotypic analysis of B-cell post-transplant lymphoproliferative disorders provides insights into disease biology. Hematol Oncol. 2008 Dec;26(4):199-211. [ Links ]
26. Bonzheim I, Fröhlich F, Adam P, Colak S, Metzler G, Quintanilla-Martinez L, et al. A comparative analysis of protocols for detection of T cell clonality in formalin- fixed, paraffin-embedded tissue—implications for practical use. J Hematop. 2011 Dec 10;5(1-2):7-16. [ Links ]
27. Paireder S, Werner B, Bailer J, Werther W, Schmid E, Patzak B, et al. Comparison of protocols for DNA extraction from long-term preserved formalin fixed tissues. Anal Biochem. 2013 Aug;439(2):152-60. [ Links ]
28. Ghorbian S, Jahanzad I, Javadi GR, Sakhinia E. Evaluation diagnostic usefulness of immunoglobulin light chains (Igκ, Igλ) and incomplete IGH D-J clonal gene rearrangements in patients with B-cell non- Hodgkin lymphomas using BIOMED-2 protocol. Clin Transl Oncol. 2014 Nov;16(11):1006-11. DOI 10.1007/s12094-014-1188-4. [ Links ]
29. van Dongen JJM, Orfao A. EuroFlow: Resetting leukemia and lymphoma immunophenotyping. Basis for companion diagnostics and personalized medicine. Leukemia. 2012 Sep;26(9):1899-907. [ Links ]
30. Medeiros LJ, Carr J. Overview of the role of molecular methods in the diagnosis of malignant lymphomas. Arch Pathol Lab Med. 1999 Dec;123(12):1189-207. [ Links ]
31. Kosari F, Shishehbor F, Saffar H, Sadeghipour A. PCRbased clonality analysis in diffuse large B-cell lymphoma using BIOMED-2 primers of IgH (FR3) on formalin- fixed paraffin-embedded tissue. Arch Iran Med. 2013 Sep;16(9):526-9. [ Links ]
32. Hebeda KM, Van Altena MC, Rombout P, Van Krieken JHJM, Groenen PJTA. PCR clonality detection in Hodgkin lymphoma. J Hematop. 2009 Mar;2(1):34-41. [ Links ]
33. Beaufils N, Ben Lassoued A, Essaydi A, Dales J-P, Formisano- Tréziny C, Bonnet N, et al. Analysis of T-cell receptor-γ gene rearrangements using heteroduplex analysis by high-resolution microcapillary electrophoresis. Leuk Res. 2012 Sep;36(9):1119-23. [ Links ]
34. Boone E, Verhaaf B, Langerak AW. PCR-based analysis of rearranged immunoglobulin or T-cell receptor genes by GeneScan analysis or heteroduplex analysis for clonality assessment in lymphoma diagnostics. Methods Mol Biol. 2013 Jan;971:65-91. [ Links ]
35. Yang H, Xu C, Tang Y, Wan C, Liu W, Wang L. The significance of multiplex PCR/heteroduplex analysis-based TCR-γ gene rearrangement combined with laser-capture microdissection in the diagnosis of early mycosis fungoides. J Cutan Pathol. 2012 Mar;39(3):337-46. [ Links ]
36. Evans PAS, Pott C, Groenen PJTA, Salles G, Davi F, Berger F, et al. Significantly improved PCR-based clonality testing in B-cell malignancies by use of multiple immunoglobulin gene targets. Report of the BIOMED-2 Concerted Action BHM4-CT98-3936. Leukemia. 2007 Feb;21(2):207-14. [ Links ]
37. Langerak AW, Groenen PJTA, Brüggemann M, Beldjord K, Bellan C, Bonello L, et al. EuroClonality/BIOMED- 2 guidelines for interpretation and reporting of Ig/TCR clonality testing in suspected lymphoproliferations. Leukemia. 2012 Oct;26(10):2159-71. [ Links ]
38. Bosaleh A, Denninghoff V, García A, Rescia C, Avagnina A, Elsner B. Rearreglos de genes de cadenas pesadas de las inmunoglobulinas en las gammapatías monoclonales. Medicina (B Aires). 2005;65(3):219-25. [ Links ]
39. Piña-Oviedo S, Fend F, Kramer M, Fournier F, Farca A, Ortiz-Hidalgo C. [Diagnosis of early gastric marginal zone lymphoma (MALT lymphoma) in endoscopic biopsies. Report of a case that demonstrates the utility of immunohistochemistry and the molecular analysis]. Rev Gastroenterol México. 2008 Jan;73(3):172-6. [ Links ]
40. Novoa V, Núñez NA, Carballo OG, Lessa CF. Inmunofenotipos aberrantes en leucemias agudas en una población hospitalaria de Buenos Aires. Medicina (B Aires). 2013;73(1):9-16. [ Links ]
41. Quesnel B, Preudhomme C. [Residual disease: the hematologist's point of view]. Bull Cancer. 2001 Jun;88(6):571-5. [ Links ]
42. Puig N, Sarasquete ME, Balanzategui A, Martínez J, Paiva B, García H, et al. Critical evaluation of ASO RQ-PCR for minimal residual disease evaluation in multiple myeloma. A comparative analysis with flow cytometry. Leukemia. 2014 Feb;28(2):391-7. [ Links ]
43. Cavé H, van der Werff ten Bosch J, Suciu S, Guidal C, Waterkeyn C, Otten J, et al. Clinical significance of minimal residual disease in childhood acute lymphoblastic leukemia. European Organization for Research and Treatment of Cancer--Childhood Leukemia Cooperative Group. N Engl J Med. 1998 Aug;339(9):591-8. [ Links ]
44. Garand R, Beldjord K, Cavé H, Fossat C, Arnoux I, Asnafi V, et al. Flow cytometry and IG/TCR quantitative PCR for minimal residual disease quantitation in acute lymphoblastic leukemia: a French multicenter prospective study on behalf of the FRALLE, EORTC and GRAALL. Leukemia. 2013 Feb;27(2):370-6. [ Links ]
45. van Dongen JJ, Seriu T, Panzer-Grümayer ER, Biondi A, Pongers-Willemse MJ, Corral L, et al. Prognostic value of minimal residual disease in acute lymphoblastic leukaemia in childhood. Lancet. 1998 Nov;352(9142):1731-8. [ Links ]
46. Brüggemann M, Schrauder A, Raff T, Pfeifer H, Dworzak M, Ottmann OG, et al. Standardized MRD quantification in European ALL trials: proceedings of the Second International Symposium on MRD assessment in Kiel, Germany, 18-20 September 2008. Leukemia. 2010 Mar;24(3):521-35. [ Links ]
47. Allen RC, Zoghbi HY, Moseley AB, Rosenblatt HM, Belmont JW. Methylation of HpaII and HhaI sites near the polymorphic CAG repeat in the human androgenreceptor gene correlates with X chromosome inactivation. Am J Hum Genet. 1992 Dec;51(6):1229-39. [ Links ]
48. Carrel L, Willard HF. X-inactivation profile reveals extensive variability in X-linked gene expression in females. Nature. 2005 Mar 17;434(7031):400-4. [ Links ]
49. Gong L, Ren K-X, Li Y-H, Liu X-Y, Zhang W-D, Yao L, et al. Determination of clonal status of pulmonary sclerosing hemangioma with X-chromosome inactivation mosaicism and polymorphism of phosphoglycerate kinase and androgen receptor genes. Med Oncol. 2011 Sep;28(3):913-8. [ Links ]
50. Mossner M, Nolte F, Hütter G, Reins J, Klaumünzer M, Nowak V et al. Skewed X-inactivation patterns in ageing healthy and myelodysplastic haematopoiesis determined by a pyrosequencing based transcriptional clonality assay. J Med Genet.2013 Feb; 50(2): 108-17. [ Links ]