INTRODUCCIÓN
Hasta hace unos pocos años la alteración del genoma de un organismo era vista como un proceso complejo y difícil. Sin embargo, es conocido que organismos procariotas utilizan sistemas de respuesta inmune innata que les permite modificar la información genética, ya sea por escisión de la secuencia o modificación de grupos metilo en la secuencia de ADN. Las enzimas de las bacterias encargadas de llevar a cabo estas modificaciones en el ADN foráneo fueron descubiertas por Werner Arber, Daniel Nathans y Hamilton Smith, quienes ganaron el premio nobel de Medicina en 1978 y lo llamaron el Sistema de restricción modificación (1).
La enzima de restricción corta cualquier ADN foráneo que contenga un sitio de reconocimiento específico en su secuencia, protegiendo a la bacteria de la inclusión de ese ADN dentro de su genoma. Por otro lado, el genoma de un bacteriófago (virus que infectan a las bacterias) con sitios de restricción metilados (o sin estos sitios específicos), ya no será escindido por la actividad de estas enzimas y la infección del fago continuará, lo que conducirá a la muerte celular y a la liberación de nuevos viriones. Los sistemas de restricción modificación han sido clasificados en cuatro tipos principales: los tipos II son los más conocidos y ampliamente utilizados en biología molecular. Estas enzimas (que generalmente son secuencias palindrómicas), llamadas tipo II, cortan dentro o cerca del sitio de reconocimiento, para producir extremos 5’-fosfato o 3’-hidroxilo (1).
Los mecanismos de manipulación genética consistían en nucleasas (enzimas de restricción tipo II) de dedos de zinc (ZFN, zinc-finger nucleases) y nucleasas tipo activadores de transcripción (TALEN, transcription activator like effector nucleases), que realizan cortes en la cadena doble de ADN (2).Para el reconocimiento de la secuencia de ADN las primeras poseen proteínas con sitios de unión que reconocen de 9 a 18 pares de bases nitrogenadas, y las segundas utilizan series de aminoácidos que identifican una única base nitrogenada (2).
Una vez se realiza el corte en el ADN, estos mecanismos modifican la información genética al estimular vías de reparación del ADN tal como la unión de extremos no homólogos o la reparación dirigida por homología. Con la primera se pueden producir inserciones o deleciones pequeñas que generalmente cambian el marco de lectura que inactiva la función del gen, mientras que por la segunda vía se podría lograr la inserción de secuencias de ADN o sustituciones de un solo nucleótido en sitios específicos (2). Posteriormente, con el descubrimiento de los sistemas asociados a CRISPR/Cas, se plantea la edición genética generando retos no solo técnicos, sino también éticos y de transferencia de conocimiento para su aplicación práctica (2,3).
Los sistemas CRISPR/Cas también actúan como un sistema inmune adaptativo bacteriano frente a los virus. Se identificó originalmente en 1987 en Escherichia coli, cuando se describieron secuencias repetitivas de hasta 29 nucleótidos (CRISPR) separados por secuencias “espaciadoras” que, posteriormente, se descubrió que eran idénticas a las secuencias de ARN viral (4). Esto llevó a reconocer que las bacterias guardan una “memoria genética” sobre las infecciones víricas que les permite reconocer y destruir el mismo patógeno en el futuro (3,4).
CRISPR/Cas es el acrónimo en inglés de clustered regularly interspaced short palindromic repeats (en español: repeticiones cortas agrupadas regularmente espaciadas), mientras que Cas - Cas9, Cas12a y Cas-cade-Cas3 (5) se refieren a las proteínas asociadas con CRISPR; es decir, las enzimas que realizan los cortes en las secuencias de ADN y permiten la edición de la información genética (Figura 1) (6). Los sistemas CRIS-PR/Cas usan este mecanismo bacteriano de inmunidad adaptativa, desarrollado para dar protección contra infecciones virales, al reconocer secuencias específicas de ADN y cortarlas (6).
La investigación en la industria de alimentos fue un área de estudio inicial porque existía la preocupación por las infecciones virales en las cepas bacterianas usadas para la producción de yogur y queso. Estos primeros investigadores en sistemas CRISPR/Cas9 no tenían en mente ninguna aplicación de uso directo en humanos (7). El investigador Francisco Mojica propuso el termino CRISPR para esta familia de secuencias repetitivas. Este fue acuñado en el año 2002 por Jansen et al. (8). Por mucho tiempo los sistemas CRISPR/Cas9 fueron de interés para los microbiólogos, hasta que en el 2012 los grupos de investigación de Doudna y Charpentier realizaron experimentos en células vivas que les permitieron mejorar el sistema al usar Cas9 y declarar: “Nuestro estudio revela una familia de endonucleasas que utilizan los ARN dobles para escindir el ADN en secuencias específicas y pone de relieve el potencial para explotar el sistema para la edición programable del genoma mediada por ARN” (3,7). En el 2013, Zhang et al., demostraron la edición exitosa del ADN en células eucarióticas, incluyendo a las humanas, usando CRISPR/Cas9 (9).
MECANISMO DE ACCIÓN
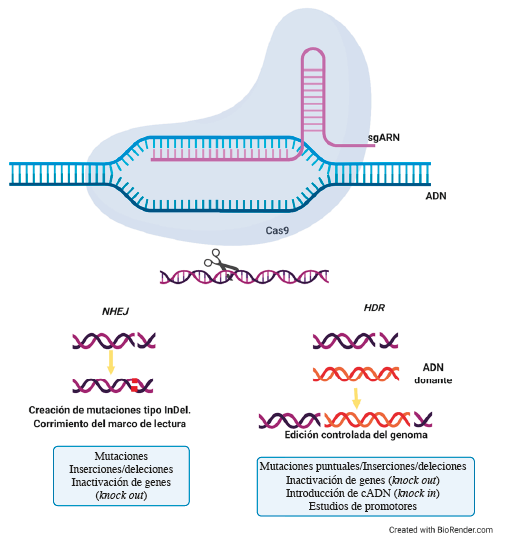
En general, se considera un mecanismo relativamente sencillo que usa una endonucleasa. Cas es una enzima de restricción que corta el ADN en un lugar específico previamente determinado por una cadena corta de ARN, denominada ARN guía (sgARN). Esta molécula de ARN establece la secuencia blanca y sirve para que la enzima se posicione y realice el corte (10). Es entonces cuando la maquinaria de reparación celular entra en acción por dos vías posibles: recombinación no homóloga (Non-homologous end joining, NHEJ) que consiste en unir los fragmentos de manera imperfecta de tal forma que logre alterarse la función del gen blanco o, por recombinación homóloga (Homology directed repair, HDR), en la cual se copia una plantilla de ADN incorporado a la enzima Cas para llenar el espacio faltante en la secuencia empleada (10,11) (Figura 2).
Cada una de las posibles vías de reparación tiene una utilidad práctica. Mientras la NHEJ suele ser usada para estudios de función génica a través de la inactivación (knock out) de secuencias codificantes o para la disrupción de marcos de lectura normales, la HDR se emplea para modificar el genoma al introducir nueva información a la secuencia de ADN (9). De esta manera, si se provee un templado de ADN sería posible introducir una copia de cualquier información en la secuencia deseada, permitiendo corregir desde una mutación puntual hasta todo un gen defectuoso (10) (Figura 2).
Fuente: creación propia con BioRender.com
Figura 2 Mecanismos de acción CRISPR/Cas9. El sgARN dirige la endonucleasa Cas9 para escindir ambas cadenas de ADN en una secuencia específica. La escisión del ADN se produce 3 pares de bases cadena arriba de un motivo adyacente (PAM). Después de la ruptura de doble cadena el genoma se repara mediante mecanismos de reparación de ADN. Se usa el sistema CRISPR/Cas9 se pueden realizar modificaciones específicas del genoma, como la introducción de pequeñas inserciones y deleciones (InDel) mediadas a través de la ruta de recombinación no homóloga o, unión de extremos no homólogos (NHEJ), relativamente, propensa a errores o la edición dirigida por la vía de reparación por homología o por recombinación homóloga (HDR).
Todo lo anterior permite establecer diferencias entre el método de ADN recombinante y esta tecnología; ya que, a pesar de que ambos permiten la manipulación de ADN usando mecanismos biológicos (de origen viral y bacteriano, respectivamente), el primero resulta de un proceso de transfección celular en el que se incorpora material genético externo (viral, bacteriano, vegetal, sintético, entre otros) al interior del huésped a través de vectores como los plásmidos, con el fin de poder evaluar la expresión de genes o permitir la producción de proteínas, como es el caso de los antígenos para vacunas u hormonas como la insulina (12).
El segundo, la tecnología CRISPR/Cas, busca realizar una modificación en el ADN celular de forma directa, de tal forma, que el cambio logre ser “definitivo” en la célula intervenida. Esto representa un gran salto no solo en el análisis funcional de genes, sino también en la producción proteica y en terapia génica (10).
Cabe resaltar que se ha descrito que los sistemas de tipo restricción modificación y CRISPR/Cas son mutuamente incluyentes; además, que el uso de dos estrategias de defensa diferentes podría ayudar a inclinar la balanza en favor de la bacteria en la carrera armamentista coevolutiva contra fagos virulentos (1).
PERSPECTIVAS DEL USO DE LA TECNOLOGÍA CRISPR/CAS
En la actualidad se explora el uso de esta técnica en múltiples campos de la medicina. Por ejemplo, en el caso de la infectología, puntualmente en enfermedades como el VIH, se busca eliminar el virus latente en las células T de memoria que persisten a pesar del uso de la terapia antirretroviral (11,13). Esto se lograría al tomar secuencias virales integradas al ADN del huésped e inmunizando estas células contra la reactivación del material genético viral. Otros investigadores han logrado modular correceptores del virus, como CCR5, en células hematopoyéticas y progenitoras, obteniendo una eficacia hasta del 30 % al momento de conferirles inmunidad celular (11). Se ha planteado incluso la posibilidad de realizar trasplantes de células madre pluripotenciales o de médula ósea genéticamente diseñadas para erradicar la infección por VIH (14). Existen también reportes de su implementación en infecciones como la del virus de Epstein-Barr, donde se ha conseguido detener su proliferación, y en el virus de papiloma humano, usando como genes blanco E6 y E7 con el fin de promover el arresto del ciclo celular y la apoptosis de células infectadas (13).
Otra de las aplicaciones es la edición de embriones en etapas tempranas con la inoculación directa del sistema CRISPR/Cas, logrando así modificar por completo su genoma; esto realizado incluso desde las células germinales con el fin de perpetuar dichos cambios por varias generaciones, lo que, eventualmente, permitiría la intervención temprana en enfermedades genéticas de carácter hereditario (11). Se ha documentado la edición de variantes patogénicas en el gen que codifica para la proteína distrofina en embriones murinos, responsable de la distrofia muscular de Duchenne y Becker, obteniendo una corrección parcial del defecto en el modelo animal (10). En un estudio llevado a cabo por Yin, et al., para la corrección de la tirosinemia tipo I, causada por el déficit en la actividad de la enzima fumarilacetoacetato hidrolasa, se reportó la reparación del alelo mutado que permitió reducir la toxicidad hepatocelular y aumentar la ganancia de peso en los ratones de laboratorio con la enfermedad (11,15).
Otro ejemplo de las aplicaciones de CRISPR/Cas es la modificación de mutaciones en el gen PCSK9, asociado con bajos niveles de lipoproteínas de baja densidad (LDL). Se reportó que el uso de esta tecnología en ratones para inducir la pérdida de función en el locus del gen, permite un aumento en los niveles de expresión de los receptores hepáticos de LDL y la caída de colesterol plasmático, constituyendo así un potencial procedimiento terapéutico para el manejo de la hipercolesterolemia familiar (15).
Con esta tecnología se ha logrado introducir variantes patogénicas puntuales en las células madre embrionarias, que permiten perpetuar modificaciones de diversa índole dentro de su información genética y de las células hijas; de esta forma, se ha conseguido crear modelos celulares para estudios en laboratorio con mayor rapidez, traduciéndose en una ventaja en el campo de la investigación (6). Incluso ha sido útil en la creación de modelos animales que sean portadores de enfermedades como el cáncer, al lograr modificar la expresión de genes de supresión tumoral u oncogénicos, repercutiendo de esta manera también en el desarrollo de trabajos en laboratorio que empleen estudios in vivo, por ejemplo, en enfermedades como la leucemia o el cáncer de colon (6,16).
La diversidad de estudios que se llevan actualmente a cabo en el terreno de la medicina se resume en la Tabla 1.
Tabla 1 Estudios clínicos con uso de la tecnología CRISPR/Cas9 en patologías mendelianas, oncológicas o infecciosas
| Tipo de edición | Gen/enfermedad | Estudios actuales | Referencia |
|---|---|---|---|
| Introducción | β-Talasemia (BT) | Corrección de mutaciones en el gen beta-talasemia en células madre pluripotentes específicas de pacientes usando CRPR/Cas9 y PiggyBac | 17 |
| Modificación | Distrofia muscular | Prevención de la distrofia muscular en ratones a través de la edición de ADN en líneas germinales mediante CRISPR/Cas9 | 18 |
| Modificación | Fibrosis quística | Reparación funcional de CFTR en organelos de células madre intestinales de pacientes con fibrosis quística mediante el uso de CRISPR/Cas9 | 19 |
| Modificación | Adenocarcinoma de pulmón | Knock en ratones mediante CRSPR/Cas9 para la edición de genes en cáncer. | 20 |
| Eliminación | Virus de Inmunodeficiencia humana (VIH). | Uso del sistema CRISPR/Cas9 para irrumpir en el provirus latente de VIH-1 | 21 |
| Eliminación | Virus de Papiloma Humano (VPH) | Supresión in vitro e in vivo del crecimiento de células positivas para cáncer de cérvix por VPH mediante CRISPR/Cas9. | 22 |
| Modificación | Malaria | Eficiencia en la edición del genoma plasmodium falciparum mediado por CRISPR/Cas9 | 23 |
DIFICULTADES EN LA IMPLEMENTACIÓN DE CRISPR/CAS
Aún existen retos y limitaciones para el uso de esta tecnología. En primera instancia, el transporte seguro y eficiente de los componentes de CRISPR/Cas al medio celular debe ser optimizado, ya que al parecer es altamente dependiente del tipo de célula u organismo en el cual va a ser usado (24). Hasta el momento se ha probado con el uso de sistemas virales y no virales, pero todos ellos presentan en mayor o menor medida ciertas consideraciones, tales como el aumento de frecuencia de mutagénesis aleatoria, activación o silenciamiento de loci adyacentes a los del sitio blanco, activaciones oncogénicas e, incluso, barreras celulares que obstaculizan la acción del sistema CRISPR/Cas (24). Los adenovirus parecen ser la opción más prometedora gracias a su alta efectividad y baja toxicidad e inmunogenicidad. Sin embargo, aún no se han arrojado resultados concluyentes al respecto (11,25). Un estudio reciente ha evidenciado la inmunidad adaptativa preexistente en humanos a los ortólogos de la proteína Cas9 asociada, proveniente de Staphylococcus aureus y Streptococcus pyogenes; lo anterior es secundario a la constante exposición del hombre a estos microorganismos, hecho que podría explicar el hallazgo de anticuerpos contra estas proteínas, convirtiéndolo en un punto importante al momento de iniciar los ensayos clínicos con esta tecnología (26).
Un rasgo relevante es la baja eficiencia del mecanismo HDR para la reparación celular en comparación con NHEJ, esto limita este tipo de terapia a solo la represión génica y disminuye su utilidad en cuanto a la edición e inserción de secuencias dentro del genoma, que es uno de los mayores intereses y objetivos de esta tecnología (11). Otro reto para superar es el hecho de que la reparación celular HDR necesita de condiciones especiales en la célula (comocongelar las fases S y G2 del ciclo celular), lo cual dificulta su aplicación al momento de introducir material al genoma. Se han logrado avances que continúan en estudio para eliminar enzimas que inhiben esta última fase o, con el uso de PITCH (precise integration into the target chromosome), que inducen la vía HDR en células postmitóticas (27).
Por otro lado, es importante tener en cuenta que los sistemas CRISPR/Cas suelen inducir mutaciones no solo en su blanco, sino en zonas off target (modificaciones no deseadas fuera de la secuencia blanco). Esto, si bien es útil para las bacterias por permitirles reconocer con mayor eficacia el ADN viral invasor hipervariable, puede generar mutaciones y efectos no deseados en humanos, los cuales aún han de ser evaluados a profundidad (25).
En cuanto al cáncer, al ser esta una enfermedad en principio poligénica con perfiles de aberración genética heterogéneos entre pacientes y estadios tumorales, es difícil crear terapias de manipulación y corrección de genes, al tiempo que se evite que las células no editadas con material defectuoso proliferen con una mayor velocidad respecto de las células editadas, lo que se traduce, rápidamente, en el fracaso de ellas (26). Dicho esto, es necesario asegurar la alta eficiencia de la técnica desarrollando mejores vectores de transporte y optimizando el diseño de ARN guías más específicos al igual que endonucleasas más potentes que Cas9 (28).
Si bien, CRISPR/Cas es una tecnología que ofrece muchas ventajas al momento de editar genes y, por consiguiente, es candidato para el uso en terapia génica con el fin de corregir muchos defectos genéticos, aún existe un gran vacío en cuanto a los rasgos susceptibles de edición además de la seguridad y eficacia en contraposición con los resultados off target que podría desencadenar (27). Hay que tener en cuenta también las variaciones genéticas entre individuos, lo cual podría también afectar el rendimiento de la tecnología, pues se requeriría una mayor especificidad y un diseño, quizás individual, para evitar efectos deletéreos. Algunas respuestas a este problema estarían en la creación de algoritmos óptimos y predictivos de secuencias de ARN guías, con un riesgo mínimo de modificaciones no deseadas, teniendo en cuenta la alta similitud de algunas regiones en el genoma (29). Lo anterior va de la mano con modificaciones a la endonucleasa Cas9 para que esta realice cortes de una sola banda (single-stranded breaks [SSB]), dando lugar a una reparación por NHEJ, creación de fusiones con otro tipo de endonucleasas como FokI, y situación de control de la concentración de la enzima para mejorar la especificidad del sistema (29).
Todo esto conlleva los dilemas éticos que plantea su uso en humanos y en líneas germinales. En este sentido, en diciembre de 2015 la comunidad científica llevo a cabo el evento International Summit on Human Gene Editing, el cual contó con la participación de delegados de Estados Unidos, China y Reino Unido en las áreas de ciencias, derecho, ingeniería, bioética, filosofía y medicina con el fin de discutir las precauciones para su implementación en seres humanos. Se llegó a la conclusión de que se necesita establecer normas éticas, legales y un seguimiento apropiado para la investigación básica y clínica de edición de genes en humanos, de su uso en líneas somáticas con mayores restricciones, en las líneas celulares germinales o embrionarias, hasta obtener información suficiente acerca de la seguridad y eficacia de la técnica (30).
Recientemente, el Comité de ética de la agencia de investigación Biomédica de Francia (INSERM) sentó su postura al respecto y recomendó la prohibición de todas las modificaciones genéticas de línea germinal, proponiendo además la creación de un comité europeo de expertos en diferentes disciplinas para evaluar el alcance, la eficacia y la seguridad de CRISPR/Cas y, de otro que se encargaría del seguimiento de las partes interesadas para promover el debate abierto sobre los aspectos sociales de estas tecnologías (30).
Por último, los aplicantes para el registro de patente de CRISPR/Cas9 han tenido bastantes problemas entre sí. En el 2012, las investigadoras Jennifer Doudna y Emmanuelle Charpentier de la Universidad de Berkeley usaron este sistema para realizar cortes en la estructura del ADN en sitios específicos. Sin embargo, en el 2013, Feng Zhang, del instituto Broad del MIT (Massachusetts Institute of Technology), reportó su utilidad en células eucarióticas, incluidas las humanas. Desde que se presentaron estos avances se han realizado más de 800 solicitudes de patente para CRISPR/Cas9 (31). A pesar de que el grupo de Berkeley la solicitó primero, al de Zhang se le concedió antes, tras lo cual, este pidió declarar el acto de sus contrapartes como una interferencia de patente. Con esto se dio inicio con un proceso para establecer quién llegó a la invención de esta tecnología antes que los demás (31). El argumento de Zhang es que él fue el primero en reportarlo en células eucariotas, punto clave si se tiene en cuenta que la aplicación más lucrativa de CRISPR/Cas9 a futuro, sea, probablemente, en salud humana. Adicionalmente, muchas compañías de biotecnologías se han involucrado en esta situación apoyando a una de las dos partes (31).
AVANCES EN EL DESARROLLO DE LA TECNOLOGÍA DE EDICIÓN DE GENES CON SISTEMAS CRISPR/CAS
Con el fin de evitar los efectos indeseados, se han desarrollado métodos alternativos al usar enzimas que realizan rupturas de una hebra en lugar de una doble y, de esta forma, incrementar su especificidad en el reemplazo de las endonucleasas originales. Se ha implementado el uso de ARN guías de menor tamaño, nuevos vectores de transporte para introducir la enzima junto al ARN guía de formas más eficientes, y el reemplazo de Cas9 por otras nucleasas con mayor especificidad (24). Por otro lado, se ha descrito el uso de CRISPR/Cas como herramienta útil en la modulación de expresión de genes, activando o reprimiendo su transcripción (32).
Huang et al., reportaron que las cinco naciones que lideran la investigación en edición genética son Estados Unidos, China, Alemania, Reino Unido y Japón. Además, mostraron que las publicaciones de investigación en edición genética con sistemas CRISPR/Cas continúa creciendo, mientras que, desde el 2016, se observa una disminución en las publicaciones de otras tecnologías como la ZFN y TALEN (33).
En junio de 2016 el National Institutes of Health aprobó el primer ensayo clínico con CRISPR/Cas9 en seres humanos para terapias oncológicas en linfocitos T, el primer paso para el estudio in vivo de otras enfermedades a futuro (34). Se hizo una búsqueda rápida de ensayos clínicos usando esta tecnología en el sitio clinicaltrials.gov con el criterio “CRISPR - Cas9” y, hasta el 25 de octubre de 2019, se encontraron 11 estudios registrados. De estos, 9 se encuentran en reclutamiento, 1 en espera de iniciarlo, otro fue retirado y se desconoce el estado de 1 de ellos. En la Tabla 2 se resumen algunos de los estudios encontrados hasta la fecha.
Tabla 2 Aplicaciones actuales de la tecnología CRISPR/Cas9 en ensayos clínicos
| Ensayo clínico | País | Estado actual | Clinicaltrials.gov, identificador |
|---|---|---|---|
| Seguridad y eficacia de TALEN y CRISPR/Cas9 en el tratamiento de neoplasia intraepitelial cervical relacionada con VPH | Guangdong, China | Desconocido | NCT03057912 |
| Seguridad del trasplante de células CD34 positivas con CRISPR CCRS en sujetos infectados por VIH con neoplasias hematológicas | Beijing, China | En reclutamiento | NCT03164135 |
| Estudio de la supresión de los genes PD-1 y TRC en células CAR-T dirigidas contra mesotelina mediado por CRSPR/Cas9 en pacientes con múltiple tumores sólidos positivos para mesotelina. | Beijing, China | En reclutamiento | NCT03545815 |
| Estudio de seguridad y eficacia donde se evalúa CTX001 en sujetos con beta-talasemia pendiente de transfusión. | Regensburg, Alemania | En reclutamiento | NCT03655678 |
Fuente: creación propia. Criterios de búsqueda: Crispr/Cas9
También en el 2016 se publicó en la revista Nature una investigación de China sobre el uso de linfocitos modificados. En esta se inactivaba el gen que codifica para la proteína PD-1, involucrada en la potenciación de la respuesta inmunológica y destrucción de células tumorales en pacientes con cáncer de pulmón metastásico de células no pequeñas con recaída o refractarios al manejo con quimioterapia. El estudio actualmente se encuentra en seguimiento de efectos adversos, pero hasta el momento no ha arrojado reportes negativos (30).
En noviembre del 2018 se generó un gran escándalo en el mundo que evidenció la necesidad urgente de discutir y regular el uso de tecnologías de edición de genes en humanos. En China He Jiankui anunció el nacimiento de dos gemelas en las cuales habían realizado edición del gen CCR5 y, por lo tanto, serían las primeras humanas con edición genética en línea germinal. La proteína codificada por el gen CCR5 participa en la entrada del virus del VIH en las células inmunes y producir una deleción podría dar resistencia a la infección. La comunidad científica permanece desde entonces expectante a información respecto a si realmente se realizó este experimento, los efectos biológicos resultantes y la salud de las niñas (35-37).
Por último, en octubre del 2019 sale a la luz pública el método Prime Editing (PE) como una modificación del sistema CRISPR/Cas9, en el cual se pretende disminuir los efectos off target indeseados. El PE realiza cortes en una sola hebra -hasta el momento- de ADN a través de la enzima Cas9 modificada (se fusionó con una retrotranscriptasa en su extremo C-terminal), de forma tal que al introducir un ARN guía para PE (Prime Editing guide RNA o pegRNA) que también sirva de molde, se haga la reparación de la hebra. Posteriormente, la hebra reparada serviría de molde para la no editada usando la maquinaria de reparación propia de la célula blanco. Este método fue probado en células humanas y de ratón, se estima que puede corregir el “89 % de las 75.122 variantes patogénicas humanas reportada en ClinVar”. Sin embargo, es una técnica muy reciente que necesita de investigación adicional para ser perfeccionada (38).
CONCLUSIÓN
El avance en las tecnologías de edición de genes basadas en los sistemas CRISPR/Cas ha sido vertiginoso en los últimos años, debido, principalmente, a las posibilidades terapéuticas que se plantea para su uso en humanos. Esto genera cada vez mayor interés para el desarrollo de ensayos clínicos y una carrera entre los grupos científicos con mayor inversión económica para investigación básica y clínica que genere más conocimiento y optimización de esta tecnología. Es necesario conocer mejor los aspectos de seguridad de este tipo de intervenciones, ya que existe la posibilidad de presentarse eventos adversos serios.
Por otro lado, el uso de la edición de genes en humanos plantea dilemas éticos. Se ha abierto un importante debate a nivel mundial que ha llevado a que las asociaciones médico-científicas emitan activamente comunicados dirigidos a gobiernos y a la comunidad general, promoviendo la necesidad de una regulación de estas innovaciones en los países (39).
Quizás sea necesario considerar la creación de redes internacionales de conocimiento y de investigación que permitan la generación de espacios de discusión entre los expertos, para así aunar fuerzas en la investigación (incluyendo la participación de investigadores latinoamericanos) y compartir rápidamente el conocimiento generado. Igualmente, las asociaciones médico-científicas nacionales e internacionales, a través de una aproximación transdisciplinaria, están llamadas a permanecer atentas al avance de la investigación en la edición de genes y hacer un ejercicio permanente de pensamiento crítico que logre dar información clara y suficiente a la sociedad para que se construya el marco normativo que regule la transferencia de conocimiento y el desarrollo de la investigación en este campo. Finalmente, vale la pena resaltar que es indispensable incorporar en este análisis al pensamiento reflexivo del enfoque de la bioética, para así enfrentar los retos que plantean los avances científicos de eventual gran impacto en la humanidad y que podrán generar grandes expectativas y controversia en la sociedad.