Introducción
En diciembre de 2019 se evidenció la aparición de una serie de casos de neumonía en la ciudad de Wuhan, capital de la provincia de Hubei, China; algunos de los cuales fueron inicialmente relacionados al mercado mayorista Huanan de mariscos, pescados y animales vivos, este es el lugar probable de origen del agente causal de esta enfermedad 1. Este nuevo microorganismo perteneciente a la subfamilia de los Beta-coronavirus, fue completamente secuenciado por científicos chinos el 7 de enero, y nombrado como nuevo coronavirus 2019 (2019-nCoV, por sus siglas en inglés) por la Organización Mundial de la Salud (OMS) el 12 de enero de 2020; posteriormente el grupo de estudio de coronavirus del Comité Internacional de Taxonomía de Virus (ICTV, por sus siglas en inglés) asignó a este agente como Coronavirus 2 del síndrome respiratorio agudo severo (SARS-CoV-2, por sus siglas en inglés) y la enfermedad que produce fue denominado por la OMS el 11 de febrero como COVID-19 (es el acrónimo en inglés, enfermedad por coronavirus de 2019) 2), (3. La rápida propagación de SARS-CoV-2 en los diversos continentes; con un elevado número de personas infectadas y fallecidas, provocó que la OMS declare a la COVID-19 como pandemia global el 11 de marzo 4.
Características de los coronavirus
Los coronavirus (CoV) son virus cuyo genoma consiste en una única molécula de ácido ribonucleico (ARN) en sentido positivo, no segmentados, envueltos, son redondos y a veces pleomórficos, a los coronavirus se les dio su nombre en función de su característica en forma de corona bajo un microscopio electrónico, diámetros de 80-120 nm y con un genoma que varía de 26 a 32 kilobases, es el genoma de ARN viral más grande 5), (6.
El virión tiene una nucleocápside compuesta de ARN genómico y la proteína nucleocápside (N) fosforilada, que está cubierta dentro de las bicapas fosfolipídicas y está cubierta por dos tipos de espiga: el recortador de glucoproteína de espiga (S) que se encuentra en todos los CoV, y la hemaglutinina-estereasa (HE) que existe en algunos CoV. La proteína de membrana (glicoproteína transmembrana de tipo III o M) es la proteína más abundante de la partícula viral, y la proteína de la envoltura (E) que se encuentra entre las proteínas S en la envoltura del virus 7.
Los coronavirus son miembros de la subfamilia Orthocoronavirinae de la familia Coronaviridae, y del orden Nidovirales (ICTV). La subfamilia de los CoV se divide de forma genotípica y serológica en cuatro géneros los CoV alfa (α), beta (β), gamma (γ) y delta (δ). El β-CoV se puede clasificar en cuatro linajes virales, a saber, el linaje A-D 7), (8. Las infecciones humanas son causadas por α y β-CoV, pero también estos virus infectan a mamíferos y aves. Los β-CoV son endémicos en todo el mundo y representan del 10% al 30% de infecciones del tracto respiratorio superior en adultos 9.
Existen siete 7 tipos de coronavirus que han sido identificados por el Centro y control de prevención de enfermedades (CDC, por sus siglas en inglés), que incluyen 2 grupos. Los coronavirus humanos (HCoV, por sus siglas en inglés) comunes: 229E, NL63 (α-coronavirus), OC43, HKU1 (β-coronavirus) y otros coronavirus humanos: Coronavirus causante del Síndrome Respiratorio del Medio Oriente (MERSCoV, por sus siglas en inglés), Coronavirus causante del Síndrome respiratorio Agudo Severo (SARS-CoV, por sus siglas en inglés) y Coronavirus 2 causante del Síndrome respiratorio Agudo Severo (SARS-CoV-2, por sus siglas en inglés), anteriormente denominado 2019-nCoV; estos tres últimos pertenecen al grupo de β-CoV 9),(10.
Los cuatro CoV (229E, NL63, OC43 y HKU1) son prevalentes y típicamente causan síntomas de resfriado común en individuos inmunocompetentes, y algunas veces pueden ocasionar infecciones graves en poblaciones de edades extremas 11. Las otras cepas, el SARS-CoV y MERS-CoV, son de origen zoonótico y se relacionan con enfermedades a veces fatales. El SARSCoV fue el agente causal de los brotes de síndrome respiratorio agudo severo en 2002 y 2003 en la provincia de Guangdong, China; MERS-CoV fue el patógeno responsable de brotes de enfermedades respiratorias graves en 2012 en el Medio Oriente 12. Dada la alta prevalencia y amplia distribución de los coronavirus, la gran diversidad genética y la recombinación frecuente de sus genomas, y el aumento de las actividades de interfaz entre humanos y animales, es probable que emerjan periódicamente nuevos coronavirus en humanos, debido a infecciones frecuentes entre especies y eventos de contagio ocasionales 8), (10), (13.
Tras la secuenciación del genoma del SARS-CoV-2 y β-CoV/RaTG/2013, ambos virus están en el mismo grupo y mostró un 96,2% de identidad de secuencia del genoma global, lo que sugiere que el CoV de murciélago y el SARS-CoV-2 podrían compartir el mismo ancestro común14. Estudios recientes destacaron que los genes SARS-CoV-2 comparten <80% de identidad de nucleótidos y 89,10% de similitud de nucleótidos con genes SARS-CoV 15), (16.
Epidemiología
La infección aguda atípica del tracto respiratorio estalló por primera vez en la ciudad Wuhan, provincia de Hubei de China, desde el 12 de diciembre de 2019. El 31 de diciembre de 2019, la comisión Municipal de Salud y Sanidad de Wuhan informó sobre un grupo de 27 casos de neumonía de etiología desconocida, con una exposición común a un mercado mayorista de marisco, pescado y animales vivos, como aves de corral, murciélagos, marmotas y serpientes (denominado mercado húmedo). El 7 de enero de 2020, las autoridades chinas identificaron como agente causante del brote a un nuevo coronavirus que posteriormente fue denominado SARS-CoV-2 17), (18.
Los CoV ocasionan infecciones en los seres humanos y en una variedad de animales, incluyendo aves y mamíferos como camellos, gatos, cerdos y murciélagos. Se trata de una enfermedad zoonótica, lo que significa que pueden transmitirse de los animales a los humanos 19), (20.
Varios estudios sugieren que el murciélago puede ser el reservorio potencial de SARS-CoV-2, y el pangolín el hospedero intermediario, sugiriendo que este es el mecanismo de transmisión al hombre, lo que produjo que COVID-19 se extienda en toda China y varios países a nivel mundial, por lo cual la OMS declaró este brote una emergencia de salud pública de interés internacional el 30 de enero de 2020 21), (22.
En 2003 SARS-CoV presentó una tasa de letalidad del 10% y desde entonces no se volvió a detectar en humanos. Por otra parte, se notificó que en 2013 MERSCoV alcanzó el 34% de letalidad, y hasta el 11 de mayo de 2020 en Europa la tasa de letalidad de SARS-CoV-2 fue de 4,5% 23), (24.
La transmisión de persona a persona de SARS-CoV-2 fue evidenciada por los primeros estudios efectuados en China, donde establecen una media del periodo de incubación de 5 días, con un rango de 1-14 días 25), (26), (27. La transmisión por microgotas de la vía aérea, y el contacto con fómites contaminados fue dilucidada por las investigaciones de Holbrook, et al 28. Existe controversia sobre si SARS-CoV-2 pueda transmitirse de una madre infectada a su producto antes del nacimiento 29. Aunque la transmisión vertical no ha sido demostrada en algunos estudios efectuados en distintos países (Irán, Estados Unidos y China) 30), (31), (32. Recientemente el estudio de Long, et al. determinaron que la mediana de duración de la eliminación de SARS-CoV-2 (intervalo entre el primer y el último hisopado nasofaríngeo positivo) en individuos asintomáticos fue de 19 días; aunque no definieron la carga viral respiratoria asociada con infección 33.
Características clínicas y complicaciones ocasionadas por SARS-CoV-2
Los coronavirus que tienen capacidad infecciosa en el humano son siete, de estos los HCov 229E, NL63, OC43, y HKU1 son endémicos globalmente causando infecciones del tracto respiratorio superior no severas; siendo las infecciones zoonóticas producidas por SARSCoV, MERS-CoV y SARS-CoV-2 las que producen cuadros severos de neumonía que conducen a distrés respiratorio agudo, shock séptico, fallo multiorgánico y la muerte en los individuos afectados 34), (35.
El nuevo patógeno SARS-CoV-2 tiene una elevada tasa de transmisión de persona a persona, los valores reproductivos básicos (R0) de COVID-19, es de 2 a 2,5, lo que indica que un paciente puede transmitir a otras dos o tres personas 36; siendo la mayoría de los individuos infectados asintomáticos; ciertamente, en medio de la pandemia de COVID-19, los grupos vulnerables son personas sin hogar o desamparadas, sino también personas de un gradiente de grupos socioeconómicos que podrían tener dificultades para hacer frente de forma financiera, mental o físicamente con la crisis; presentando cuadros más severos los ancianos, personas con comorbilidades asociadas: hipertensión arterial, enfermedades cardiovasculares, diabetes, enfermedad
Tabla 1 Grupos vulnerables.
| Características | Richardson, et al.41 (n=5700) | Zhang, et al.42 (n=1464) |
| País (ciudad) | EEUU (Nueva York) | China (Wuhan) |
| Edad Mediana (IQR*) | 63 (52-75) | 64 (49-70) |
| Sexo Femenino, n (%) | 2263 (39,7) | 728 (49,7) |
| Masculino, n (%) | 3437 (60,3) | 736 (50,3) |
| Comorbilidades, n (%) Hipertensión | 3026 (56,6) | 306 (20,9) |
| Enfermedades Cardiovasculares | 966 (18) | 211 (14,4) |
| Diabetes | 1808 (33,8) | 211 (14,4) |
| Tumores malignos | 320 (6) | 17 (1,2) |
| Enfermedades Crónicas respiratorias (EPOC**/Asma y otras) | 920 (17,3) | 50 (3,4) |
| Enfermedad renal crónica | 454 (8,5) | 27 (1,8) |
| Enfermedad hepática crónica | 30 (0,6) | 36 (2,5) |
| Enfermedad cerebrovascular | N/R | 47 (3,2) |
| Inmunodepresión (VIH/transplante) | 98 (0,9) | N/R*** |
| Enfermedad metabólica (obesidad y otras) | 2528 (60,7) | N/R |
pulmonar obstructiva, enfermedad renal crónica, enfermedad hepática crónica y con diversos grados de inmunodepresión producto de la malnutrición, enfermedades oncohematológicas, tratamientos con agentes inmunosupresores, trauma, cirugía y enfermedades infecciosas 37), (38), (39), (40. La mayoría de los estudios coinciden que la hipertensión arterial y la diabetes son los principales factores que se asocian a cuadros clínicos severos de COVID-19 y la obesidad es un factor de riesgo reconocido en Nueva York 41), (42.
Las manifestaciones más comunes son la fiebre, tos seca, mialgia, fatiga, anorexia y falta de aire; otros síntomas y signos de menor frecuencia son la cefalea, dolor de garganta, rinorrea, expectoración, hemoptisis, dolor torácico, dolor abdominal, náuseas, vómitos y diarrea 40),(43), (44), (45), (46), (47. Las manifestaciones y complicaciones neurológicas derivadas de la afectación del Sistema Nervioso Central son el mareo, cefalea, deterioro de la conciencia, encefalopatía, encefalitis aguda, encefalopatía necrotizante hemorrágica aguda, accidente cerebrovascular, mielitis transversa, epilepsia y ataxia 48), (49. Por otra parte, las repercusiones clínicas originadas por invasión del Sistema Nervioso Periférico son la anosmia, hiposmia, ageusia, hipogeusia, neuralgia y síndrome de Guillain-Barré 50), (51. Sin embargo, las complicaciones neurológicas surgen como un área cada vez más reconocida de morbilidad y mortalidad. En una cohorte de 179 pacientes con neumonía por SARSCoV-2, la enfermedad cerebrovascular preexistente también se asoció con una mayor mortalidad 52. Una revisión sistemática y metanálisis también identificaron un riesgo 2,5 veces mayor de infección grave entre los pacientes con un accidente cerebrovascular previo 53.
También se ha identificado como síntomas extrapulmonares de COVID-19 diversas lesiones cutáneas en el estudio efectuado por Galán, et al. en España, siendo los más frecuentes el exantema maculopapular o macular, erupción papulovesicular, pápulas dolorosas de color rojo púrpura acral con o sin vesículas (pseudo-sabañones) y la urticaria 54.
Las complicaciones asociadas al SARS-CoV-2 varían de acuerdo a los grupos etarios y comorbilidades asociadas a los pacientes, las cuales pueden repercutir en la inmunocompetencia de los individuos afectados. Las complicaciones más frecuentes observadas fueron el síndrome de distrés respiratorio agudo, arritmias, fallo cardíaco agudo y shock séptico; aunque está reportado otras alteraciones como el fallo renal agudo, fallo respiratorio, infección bacteriana secundaria, miocarditis aguda, infarto agudo de miocardio, trombosis venosa profunda, embolia pulmonar y coagulación intravascular diseminada (CID) 42), (43), (44), (45), (46), (47), (55), (56.
El estudio retrospectivo de Zhou, et al. identificaron varios factores de alta probabilidad de complicaciones, y muerte en particular en individuos de edad avanzada, niveles elevados de dímero-D, y una elevada puntuación de la evaluación del fallo orgánico secuencial (SOFA, por sus siglas en inglés) 57. La susceptibilidad a padecer infecciones virales y complicaciones en adultos mayores, se debe en parte a la inmunosenescencia, que es consecuencia de los procesos intrínsecos que ocurren en el sistema inmune relacionados con el envejecimiento 58), (59. Estas características están relacionadas principalmente con la disminución en el número y actividad de las células de la inmunidad innata; involución tímica a partir de la pubertad que provoca un escaso repertorio de linfocitos vírgenes; con la senescencia replicativa que con conduce a un predominio de células de memoria y linfocitos exhaustos la cual es agravada por la estimulación antigénica crónica, principalmente a la infección por citomegalovirus (CMV) que finalmente lleva al desgaste progresivo del repertorio de linfocitos 59), (60.
Las manifestaciones clínicas producidas por SARSCoV-2 en niños son sustancialmente menos agresivas que en adultos, lo cual también ha sido descrito para SARS-CoV en el brote epidémico de 2002 61. Los niños infectados pueden encontrarse asintomáticos o presentar fiebre, tos seca, fatiga, dolor de garganta congestión nasal, rinorrea, estornudos y síntomas gastrointestinales como dolor abdominal, náuseas, vómitos y diarrea con buen pronóstico, generalmente logrando la remisión del cuadro dentro de la primera o segunda semana del comienzo de la enfermedad 62.
Replicación y Patogénesis de SARS-CoV-2
La entrada de SARS-CoV-2 a las células del hospedero está mediada por las subunidades S1 y S2 de la glicoproteína S 63. La subunidad S1 contiene el motivo de unión al receptor (RBM, por sus siglas en inglés) que permite la unión del virión a su receptor, enzima convertidora de angiotensina 2 (ACE-2, por sus siglas en inglés) una carboxipeptidasa de membrana y S2 contiene los motivos estructurales que consisten en regiones de repeticiones de siete aminoácidos (HR1 y HR2, por sus siglas en inglés) implicadas en la fusión virus-membrana plasmática de las células epiteliales del hospedero del tracto respiratorio y digestivo, resultando en la endocitosis y liberación del genoma viral en el citoplasma celular 63), (64), (65. Para la fusión de SARS-CoV-2 con la membrana de la célula es importante la escisión proteolítica en límite de la unión de la S1/S2 o S2` del homotrímero S, por parte de la serinas proteasas transmembras 2 (TMPRSS2) asociadas a la superficie celular y catepsinas L y P que permiten la separación entre el RBM y el dominio de fusión 63), (67.
Una vez en el citoplasma de la célula del hospedero el genoma viral ARN monocatenario (+) no envuelto, comienza la traducción de dos largas réplicas de poliproteínas denominados pp1a y pp1ab, las cuales son escindidas por las proteasas tipo papaína (PLpro), proteasa tipo serina y la proteasa principal generando las proteínas no estructurales (nsp1-nsp16) y el complejo replicasa-transcriptasa responsable de la replicación y transcripción del ARN subgenómicos 67), (68. Estos segmentos ribonucleicos actúan como ARNm, permitiendo la traducción de proteínas accesorias y proteínas estructurales S, E, M y N del virión con la participación de las subunidades ribosomales; estas últimas son introducidas en el retículo endoplasmático (RE) y el aparato de Golgi (AG) donde el genoma viral es encapsulado por las proteínas N, finalmente se acoplan las demás proteínas estructurales y estos son liberados de los compartimentos RE-AG en forma de viriones maduros 67), (69. Por último, estas vesículas que contienen los viriones se fusionan con la membrana plasmática y se libera el SARS-CoV-2 maduro 11), (70.
La nueva evidencia planteada por Angeletti, et al. sugiere que la presión selectiva positiva, ha logrado modificaciones puntuales en aminoácidos de las proteínas no estructurales nsp2, y nsp3 que le confiere a SARS-CoV-2 mayor capacidad de propagación e infección en relación a SARS-CoV 71. Esto puede ser relevante, ya que nsp3 está compuesto por múltiples dominios como PLpro que participa en la entrada del virus a la célula del hospedero, pero además nsp3 es un componente esencial del complejo replicasatranscriptasa, que es vital para su ciclo de vida 72. Los diversos estudios realizados indican que SARS-CoV-2 tiene un efecto citopático directo en las células que infecta, a través mecanismos no establecidos, aunque su tasa de replicación elevada está implicada en la misma 70), (73.
Respuesta inmune contra SARS-CoV-2
Respuesta inmune innata
Las células epiteliales del tracto respiratorio son las primeras en ser infectadas y modular la respuesta inicial, ya que cuentan con receptores de reconocimiento de patrones (RRP) como los receptores tipo Toll (TLR-7 y 8), los receptores del gen I inducible por ácido retinoico (RIG-I, por sus siglas en inglés) y proteína 5 asociada a la diferenciación de melanoma (MDA5, por sus siglas en inglés) ambos de la familia de receptores tipo RIG-I, los cuales se encargan de reconocer el genoma de SARS-CoV-2 dentro la célula e iniciar la activación de los factores de transcripción IRF3, IRF7 y NF-kB 74), (75. Esto conduce a la liberación de interferones de tipo I
(INFα e INFβ) y mediadores inflamatorios como IL6, TNF-α, G-SCF y GM-SCF, pero además induce la producción de quimiocinas CXCL-8, CXCL-10 y CCL5 que facultan el reclutamiento de neutrófilos, células NK, monocitos y linfocitos T al tracto respiratorio 76. Las células epiteliales de la vía aérea producen factores antivirales como lactoferrina, proteínas surfactantes (SP-A y SP-D), β-defensinas humanas 1-4 y catelicidinas LL-37 que pueden interferir en ciclo vital de SARS-CoV-2 76), (77), (78. Por otra parte, el daño de las células endoteliales, la activación directa por C3a, C5a, IL-1, IL-6, TNF-α y SARS-CoV-2 de estas células en el parénquima pulmonar inducen la liberación de mediadores proinflamatorios, moléculas de adhesión (ICAM-1, VCAM-1, selectina P y E) y quimiocinas como CXCL-9, CXCL-10 y CCL-2 que favorecen la migración de células de la inmunidad innata y adquirida 79.
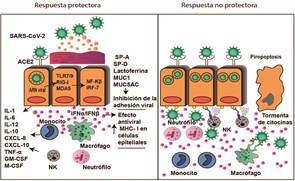
Los macrófagos alveolares, macrófagos diferenciados y neutrófilos reclutados, se encargan de la fagocitosis de células infectadas, cuerpos apoptóticos que contienen virus, partículas virales y viriones; liberan citocinas proinflamatorias (IL-1, IL-6, IL-8, IL12, IL-18, INFα, INFβ, TNFα, G-CSF y GM-CSF), prostaglandinas, leucotrienos, enzimas proteolíticas e hidrolíticas, péptidos antimicrobianos (defensinas y catelicidinas), especies reactivas del oxígeno, óxido nítrico y diversidad de quimiocinas que se encargan del eliminación viral; aunque también contribuyen al daño alveolar difuso y edema pulmonar 80. A pesar de la abundante infiltración de monocitos, neutrófilos y linfocitos, la respuesta inmune no es eficiente, ya que no existe linfocitos T altamente específicos contra SARSCoV-2 que puedan eliminar a las células infectadas y eviten la propagación del virus 81), (82. La acumulación de estas células en el pulmón provoca elevada producción citocinas proinflamatorias, que conducen a la tormenta de citocinas caracterizada por la liberación incontrolada de IL-1β, IL-2, IL-6, IL-8, IL-10, TNFα, G-CSF, GM- CSF y MCP-1 (CCL-2) que provocan SDRA, fallo multiorgánico y elevación de parámetros inflamatorios 83. La replicación activa y liberación de viriones maduros conduce a la activación del inflamasoma, que provoca la piroptosis en las infectadas con liberación de patrones moleculares asociados a daño (DAMPs, por sus siglas en inglés) a la circulación que son reconocidos por las células endoteliales, monocitos y macrófagos agravando la tormenta de citocinas 84. (Figura 1) También la IL-6 puede unirse a la forma soluble de IL-6R (sIL-6R), formando un complejo con un dímero gp130, este complejo se deposita en la membrana de las células endoteliales favoreciendo la tormenta de citocinas que involucra mediadores inflamatorios mencionados 85), (86.
Figura 1. Respuesta inmune innata.
Respuesta protectora: inhibición de la adhesión viral por medio de SP-A, SP-D, lactoferrina, MUC1 y MUC5AC; secreción de citocinas y quimiocinas que atraen y activan células de la inmunidad innata, el macrófago secreta IFNα/β, se incrementa la expresión de las MHC-I a nivel del macrófago y otras células. Respuesta no protectora: las células de inmunidad secretan grandes cantidades de citocinas y quimiocinas como la IL-1, IL-6, IL-12, IL-10, CXCL-8, CXCL-10, TNF-α, GM-CFS, M-CSF (tormenta de citocinas); las células NK eliminan las células infectadas; los macrófagos inician la fagocitosis, todos estos mecanismos facilitan e incrementan el estado inflamatorio que provocan la piropoptosis.
Las células NK son las principales células de la inmunidad innata que se encargan de la eliminación viral, y ejercen su acción a través de la liberación de mediadores citotóxicos como granzima B, perforinas, y serglicinas que activan la vía mitocondrial de la apoptosis en la célula infectada; expresan ligandos de receptores de muerte (Ligando del receptor de TNF, FASL y TRAIL) y liberan TNFα e INFγ 87), (88), (89. Esta última citocina induce una eficaz activación de los macrófagos, diferenciación de linfocitos T CD4+ vírgenes en células Th1 y promueve una respuesta por linfocitos T citotóxicos, por lo cual es esencial en la eliminación viral 90. Sin embargo, se ha identificado un descenso significativo de células NK en pacientes con cuadros severos infectados con SARS-CoV-2, lo que sugiere el deterioro de la respuesta inmune antiviral 91.
Las células dendríticas CD141+ presentes en la lámina propia del tracto respiratorio pueden ser activadas por el reconocimiento y endocitosis de las partículas virales de SARS-CoV-2 o por estimulación de citocinas proinflamatorias 75), (92. Estas células comienzan a madurar y migrar a los ganglios linfáticos regionales, donde activan eficazmente a los linfocitos T vírgenes CD4+ y CD8+ 75), (93.
Respuesta inmune adaptativa
Respuesta inmune humoral
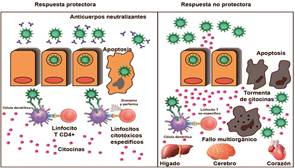
La respuesta humoral está mediada por los linfocitos B, que reconocen directamente los antígenos virales de SARS-cov-2, se activan y diferencian en células plasmáticas y células B de memoria 94. Las primeras producen las inmunoglobulinas de la clase M, G, y A las cuales limitan la diseminación de los viriones en las células no infectadas, a través de mecanismos como la neutralización, opsonización, activación del complemento y la citotoxicidad celular dependiente de anticuerpos (ADCC, por sus siglas en inglés) 94), (95. No está claro la relevancia de la respuesta humoral en la contención y eliminación de SARS -cov-2, pero los hallazgos de Zhao, et al. Demuestran que la mediana del tiempo de seroconversión para igm y igg fue de 12 y 14 días respectivamente, con una frecuencia de seroconversión en 173 individuos se detectó en el 82,7% igm (143/173) y solo el 64,7% de igg (112/173) 96. Sin embargo, las investigaciones de Wang, et al. Identificaron descensos significativos de los linfocitos B en sangre periférica de pacientes con neumonía moderada y severa, que pudiera sugerir un papel preponderante en la contención viral 97. La importancia de la respuesta humoral también fue identificada por el estudio de Zhang, et al. Donde encontraron igg específica contra el RBM de la glicoproteína S de SARS-cov-2, que limitó la propagación viral en niños infectados 98. (Figura 2)
Respuesta inmune celular
Los linfocitos T cooperadores (Th) CD4+ en particular el subtipo Th-1, son importante para la eliminación de las células infectadas, estas células se encargan de la producción de IL-2, IL-12 e INFγ, siendo la IL-2 vital para la proliferación y diferenciación de los linfocitos B y T CD8+ 99. Los niveles elevados de IL-6 asociado TGF-β polariza la diferenciación de los linfocitos Th vírgenes al subtipo Th-17 productor de IL-17A, IL-17F, IL-22 e IL-21 que agravan el proceso inflamatorio a nivel pulmonar y sistémico en pacientes con COVID-19 100.
La respuesta de linfocitos T citotóxicos (CTLs) es esencial para la completa resolución de la infección por SARS-CoV-2, ya que por distintos mecanismos que inducen la apoptosis de las células infectadas logra la remisión completa de los procesos infecciosos virales 101. Los linfocitos T citotóxicos logran la eliminación de las células infectadas a través de mecanismos dependientes del contacto celular, donde intervienen la perforina, granzimas A, B, serglicina y FASL, los cuales activan las vías intrínseca y extrínseca de la apoptosis 102), (103. Pero además los CTLs secretan citocinas como el TNFα e INFγ, este último estimula a las células dendríticas a expresar IL-12 e induce la diferenciación de linfocitos T CD8+ vírgenes en células citotóxicas 102), (104.
Por los múltiples estudios realizados se ha logrado identificar una disminución significativa de las poblaciones de linfocitos T CD4+ y CD8+ en sangre periférica, principalmente en pacientes con cuadros clínicos moderados y severos de COVID-19 91), (97), (105.
Respuesta protectora: Neutralización viral mediada por IgG e IgA. La presentación de antígeno por las células dendríticas a los linfocitos T CD4+ y CD8+, esta última célula es específica para SARS-CoV-2, así eliminar a las células infectadas. Respuesta no protectora: La replicación viral continúa, incrementa la secreción de citocinas por los linfocitos T no específicos y ocurre la apoptosis en un mayor número de células, que todo esto conllevan a un fallo multiorgánico.
Tratamiento de la COVID-19
No existe un tratamiento antiviral específico recomendado para la COVID-19, y actualmente no existe vacuna disponible. El tratamiento es sintomático y la oxigenoterapia representa la principal intervención de tratamiento para pacientes con infección grave 106. La ventilación mecánica puede ser necesaria en casos de insuficiencia respiratoria refractaria a la oxigenoterapia 107. El tratamiento de rescate con plasma convaleciente e inmunoglobulina G por vía intravenosa (IgGIV) se administra a algunos casos críticos según sus condiciones 108), (109), (110.
Se pueden desarrollar varias opciones para controlar o prevenir infecciones emergentes de SARS-CoV-2, incluidas vacunas, anticuerpos monoclonales, terapias
basadas en oligonucleótidos, péptidos, terapias de interferón 111. Sin embargo, es probable que las nuevas intervenciones requieran meses o años para desarrollarse. Dada la urgencia del brote de SARSCoV-2, nos centramos aquí en el potencial de reutilizar los agentes antivirales existentes aprobados o en desarrollo para tratar infecciones causadas por el virus de la inmunodeficiencia humana (VIH), el virus de la hepatitis B (VHB), el virus de la hepatitis C (VHC) y la influenza, según la experiencia terapéutica con otras dos infecciones causadas por coronavirus humanos: SARS-CoV y MERS-CoV 112), (113.
Tabla 2 Fármacos aprobados y ensayos clínicos contra el SARS-CoV-2.
| Drogas | Enfermedades en que se aprueba Mecanismo de acción su uso | Ref. |
| Lopinavir/Ritonavir | Inhibe las proteasas de VIH-1 y VIH-2. También VIH/SIDA, SARS-CoV y MERS- inhibe a la Proteasa de cisteína tipo 3-quimiotripsina CoV (3CLpro) | 114, 115 |
| Cloroquina e Hidroxicloroquina | Malaria y enfermedades Incremento del pH endosomal y de fagolisosoma autoinmunes | 116, 117 |
| Remdesivir | Análogo de nucleósido que inhibe a la ARN polimerasa dependiente de ARN viral, interfiriendo Ébola ensayo clínico fase II/III con la replicación viral | 118,119,120 |
| Tocilizumab | Tormenta de citocinas asociado Bloquea al receptor de IL-6, inhibiendo la al tratamiento con CAR-T, artritis transducción de señales reumatoide | 121, 122 |
| Ruxolitinib | Inhibidor selectivo de las quinasas asociadas a Janus (JAK1 y JAK2). Estas quinasas median la Tormenta de citocinas transducción de señales de citocinas | 123 |
| Siltuximab | Se une de forma específica a la IL-6 y así evita la Enfermedad de Castleman unión de esta citocina a sus receptores multicéntrica | 124, 125 |
| Baricitinib | Inhibe las Janus quinasas (JAK1 y JAK2) de forma Artritis reumatoide transitoria y reversible | 126 |
| Interferón α-2B | Induce en las células un estado de resistencia a Papilomatosis respiratoria infecciones virales, y modula la fase efectora del recurrente, condiloma acuminado, sistema inmune para neutralizar los virus o eliminar hepatitis B y C. células infectadas | 127 |
| Ivermectina | Inhibe la importación de la integrasa, la ivermectina Antiparasitario: ectoparasitosis, se adhiere al heterodímero α/β1 importina (IMP) contra nemátodos y oncocercosis | 128, 129 |
| Colchicina | Inhibe el inflamasoma en dos niveles: inhibe la activación del receptor P2x7 y la polimerización de Gota y fiebre mediterránea familiar ASC, inhibiendo así la interacción entre dominios similares a la pirina. | 130 |
Conclusión
La infección por SARS-CoV-2 es un problema de salud global con repercusiones socioeconómicas a nivel mundial. Los orígenes de este agente no están totalmente establecidos, ya que no se ha identificado al huésped intermediario en su modo de transmisión al hombre. Las manifestaciones clínicas dependen del estatus inmunológico del individuo infectado, las comorbilidades asociadas, el grupo etario y las particularidades patogénicas de SARS-CoV-2. Por otro lado, la inmunosenescencia del adulto mayor, las enfermedades crónicas y cualquier factor que conduzca a una inmunodepresión se asocian a complicaciones en pacientes con COVID-19. En las primeras etapas de la infección de SARS-CoV-2 la respuesta inmune innata se encarga de la contención y eliminación viral mediada por neutrófilos, monocitos, macrófagos, mastocitos, células NK y el patrón inflamatorio generado a nivel pulmonar y sistémico. Este mismo estado inflamatorio genera daño en el parénquima pulmonar, y en diversos órganos asociado al efecto citopático directo de SARSCoV-2. Para la completa resolución de este proceso se requiere, una respuesta inmune adaptativa altamente específica y robusta contra este agente.
Los linfocitos Th-1 y CTLs son esenciales para la completa eliminación de SARS-CoV-2 en las células infectadas. Pero se ha descrito en pacientes graves bajos niveles de linfocitos B, linfocitos T CD4+ y CD8+, aunque se ha identificado un aumento del subtipo Th-17 que agrava el proceso inflamatorio sistémico. No existen estudios acerca del papel de los linfocitos NKT, linfocitos T γδ, células linfoides innatas, células dendríticas plasmocitoides, linfocitos T reguladores, y otros por lo que la dinámica de la respuesta inmune frente a SARS-CoV-2 no está esclarecida. Actualmente no existe tratamiento específico para COVID-19, ya que existe resultados contradictorios en los ensayos clínicos que evaluaron algunos fármacos.