Introducción
Según la Organización Mundial de la Salud, las enfermedades transmitidas por vectores representan el 17 % de todas las enfermedades infecciosas en el mundo, generan más de 700 000 muertes cada año, tienen la mayor carga en áreas tropicales y subtropicales, y afectan a las poblaciones más pobres1. Entre estas se encuentran las enfermedades arbovirales, que continúan siendo un problema de salud pública mundial2 y son ocasionadas por virus que se transmiten entre hospederos vertebrados por medio de artrópodos, como mosquitos, garrapatas, entre otros. El dengue continúa siendo la enfermedad arboviral de mayor frecuencia en el mundo. Sin embargo, la reemergencia y emergencia de otras enfermedades como fiebre amarilla, chikungunya y zika, todas estas transmitidas por mosquitos Aedes spp, acentúa que las enfermedades arbovirales siguen siendo un problema de salud en el siglo XXI y que se requiere reevaluar las prioridades de investigación y las intervenciones de salud pública contra estas enfermedades3.
En Colombia, desde su reemergencia en la década de los 70, el dengue ha tenido un comportamiento endemo-epidémico, con epidemias en la última década en 2010, 2013, 2016 y 2019 y alrededor del 80 % de los municipios del país reportan casos de dengue al sistema de vigilancia epidemiológica. Además, el país ha transitado por la emergencia de chikungunya en 2014 y de zika en 20154; y se ha evaluado que 87,6 % de las localidades colombianas tienen presencia del vector Aedes aegypti5. También, se ha estimado que estas tres enfermedades causaron 491 629,2 años de vida ajustados por discapacidad (AVAD) entre 2013 y 2016, siendo superior a la carga ocasionada en algunos años de este periodo por la tuberculosis y VIH/SIDA6.
Estas enfermedades pueden prevenirse a través de la combinación de diferentes estrategias de salud pública3, entre estas se encuentran la vigilancia epidemiológica y la vigilancia vectorial, así como el desarrollo de modelos predictivos de incidencia que utilicen aproximaciones de diversas disciplinas como la epidemiología y el aprendizaje automático2. El valor de la vigilancia radica en la entrega oportuna de datos útiles, la flexibilidad de los sistemas al incremento en las necesidades de información, así como en el uso apropiado de las tecnologías para difundirla oportunamente. Por tanto, la vigilancia en salud pública puede beneficiarse de los avances en ciencias de la información, de las tecnologías, y del aumento de fuentes y bases de datos7.
En este sentido, emerge un área interdisciplinar denominada epidemiología computacional, que utiliza modelos computacionales y big data para entender y controlar la difusión espacio-temporal de una enfermedad en la población8. De manera que se está optando por desarrollar tecnologías en bases de datos, así como aplicar técnicas de analítica como minería de datos y aprendizaje automático, que permiten manejar información estructurada y no estructurada, construir modelos predictivos con variedad de técnicas basados en datos históricos, y apoyar la toma de decisiones junto con herramientas y técnicas de visualización9-11. Por lo anterior, el objetivo de la presente revisión fue identificar los métodos de aprendizaje automático utilizados con el fin de predecir el comportamiento epidemiológico, entendido este como la frecuencia de ocurrencia de casos incidentes de enfermedades arbovirales en un periodo de tiempo específico, así como documentar las diferentes variables predictoras usadas.
Metodología
Pregunta de investigación: ¿Cuáles son los métodos de aprendizaje automático que se han utilizado para predecir el comportamiento epidemiológico de las enfermedades arbovirales?
Fuentes de información y estrategia de búsqueda: los documentos se obtuvieron por medio de EMBASE y PubMed haciendo uso de la siguiente ecuación: (("predictive model*" OR predict* OR forecast) AND ("computational epidemiology" OR "descriptive analyt*" OR "predictive analyt*" OR "machine learning" OR "deep learning" OR "supervised classifi*" OR "supervised* algorithms" OR "supervised learning" OR "unsupervised classifi*")) AND ("arboviral diseas*" OR arbovirus* OR "vector-borne diseas*" OR "transmitted by vector*" OR dengue OR zika or chikungunya).
Además de los artículos obtenidos a través de la ecuación, se incluyeron algunos por medio del principio de bola de nieve.
Criterios de elegibilidad: se incluyeron artículos que utilizaron el aprendizaje automático para la predicción del comportamiento epidemiológico de enfermedades arbovirales. Se excluyeron los artículos enfocados en la predicción de desenlaces individuales y de clases de virus, o que no aportaran información de interés a esta revisión. No se realizó filtro por idioma o por fecha.
Síntesis de la información: se adoptó el flujo de trabajo PRISMA12. La ecuación de búsqueda arrojó 413 artículos. Se agregaron 6 artículos utilizando el principio de bola de nieve. Se obtuvieron 346 artículos después de la eliminación de duplicados. Inicialmente, se realizó un filtro por lectura del título y resumen de cada artículo, conservando 96 artículos relevantes. Después, la lectura de texto completo permitió seleccionar 41 artículos que cumplieron con los criterios de elegibilidad (Figura 1). Además, se realizó un análisis bibliométrico utilizando la nube de palabras, la co-ocurrencia entre las palabras clave, las coautorías y las revistas en las cuales se publicaron los artículos seleccionados.
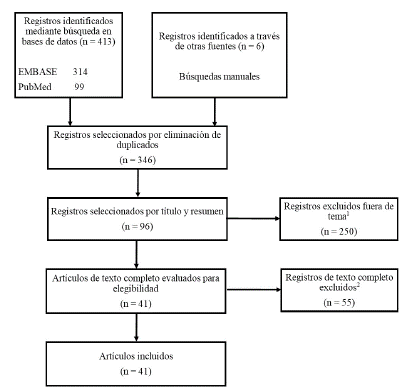
1. Exclusiones fuera de tema: Estudios del genoma o proteínas del virus, ontologías y taxonomías, estudios de inhibidores, estudios de predicción a nivel individual (diagnóstico de enfermedad o necesidad de hospitalización, predicción de la gravedad de la enfermedad), modelación del vector o predicción de la abundancia del vector, desarrollo de vacunas o de fármacos, redes de información (de salud), predicción del tipo de arbovirus o parásitos, diseño de sistemas de vacunación, enfermedades diferentes a las transmitidas por vectores.
2. Exclusiones de texto completo: No contenían la aplicación de métodos de aprendizaje automático. Artículos o estudios en los que el modelo se entrenó para mejorar los resultados de los sistemas de gestión de la atención médica. Se excluyeron los artículos cuyo objeto de estudio fuese diferente a la predicción del número de casos, nivel de incidencia o la probabilidad de ocurrencia de la enfermedad arboviral. Se excluyeron los artículos sin acceso a texto completo con las licencias institucionales, o estudios que fuesen revisiones de literatura.
Figura 1 Flujograma de la búsqueda de la literatura realizada.
Resultados
Análisis bibliométrico
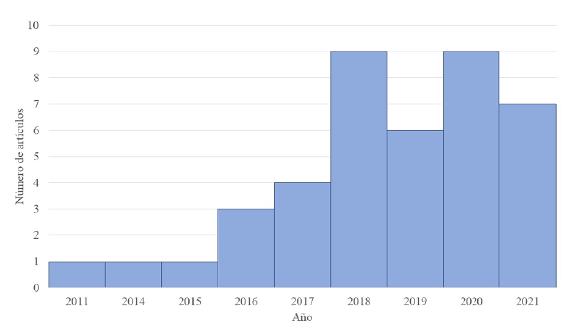
Los 41 artículos seleccionados a partir de la búsqueda se publicaron en la última década, se observó un incremento en la publicación a partir de 2016 (Figura 2). El 88,3 % de los autores participó en un artículo, mientras que el 8,9 % participó en dos artículos y el 2,8 % restante en tres artículos. En cuanto a la revista de publicación, la más frecuente fue PLoS Neglected Tropical Diseases (24,4 %), seguida por PLoS One (3,3 %) y BMC Infectious Diseases (3,3 %). Adicionalmente, en Scientific Reports, Acta Tropica, BMC Medicine y Epidemiology and Infection se publicaron dos artículos en cada una.
En relación con los intervalos de tiempo considerados en los artículos, 1 estudio usó casos informados diariamente, 33 estudios casos semanales, 1 estudio usó casos mensuales y 6 estudios no especificaron el intervalo de tiempo. Además, la serie de tiempo más larga fue de 27 años, mientras que la más corta fue de 1 año, con un promedio de 8 años y una mediana de 5 años. En 24 estudios se utilizaron series de tiempo de menos de 10 años, mientras que en 11 estudios la serie de tiempo fue de 10 años o más.
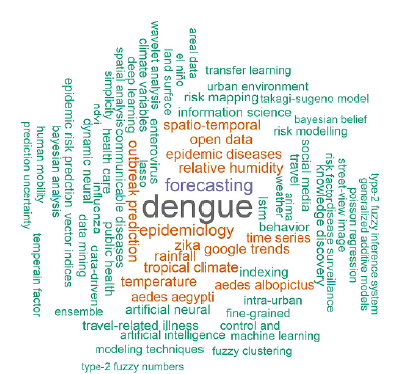
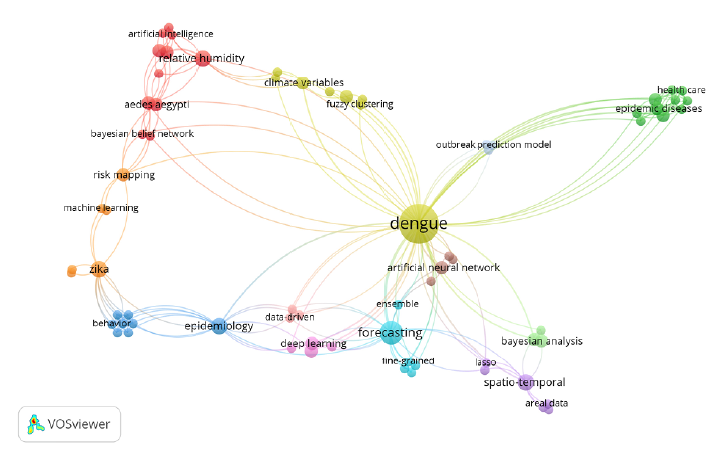
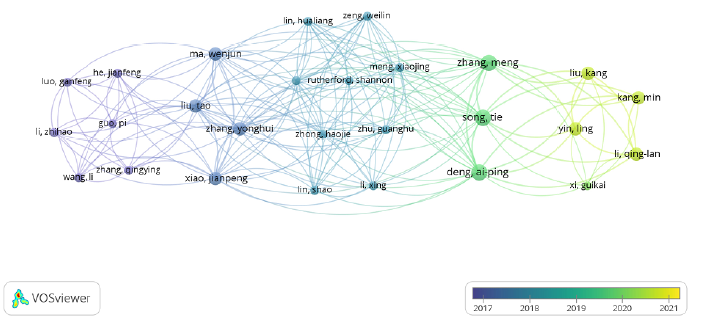
Por otro lado, la palabra clave más frecuente fue dengue, seguida de pronóstico (forecasting), epidemiología (epidemiology) y zika. Después, figuraron los factores asociados al comportamiento epidemiológico de las arbovirosis como humedad relativa, temperatura y el vector (Aedes aegypti, Aedes albopictus). Llama la atención que los métodos analíticos aparecen en el grupo de palabras menos frecuentes (Figura 3). Al analizar la co-ocurrencia entre palabras clave se evidenció como nodo principal a dengue relacionado con diversos métodos analíticos como redes neuronales artificiales, LASSO, Fuzzy clustering, redes bayesianas, entre otros. Además, el dengue se relacionó con los factores asociados al comportamiento epidemiológico; en contraste, zika se relacionó con machine learning, mapas de riesgo y epidemiología (Figura 4). Los autores con más artículos fueron Lee Ching Ng y Jayanthi Rajarethinam, que participaron en cuatro, y Ai-ping, Deng, Tie Song, Meng Zhang, Alex Cook, Shaohong, Liang, Xu Liu y Grace Yap que participaron, cada uno, en tres artículos. Al analizar la coautoría se observó mayor interrelación entre los autores del periodo 2018 - 2019, con una posterior concentración en 2020 (Figura 5). Es importante aclarar que algunos de los autores con mayor cantidad de artículos, no figuran en la gráfica de coautorías por que publicaron con los mismos coautores, y esta gráfica refleja la cantidad de coautores diferentes con los que ha publicado el investigador, mas no la cantidad de artículos.
Herramientas para la vigilancia de enfermedades arbovirales como fuentes de datos para el uso de aprendizaje automático
La vigilancia epidemiológica consiste en "la recopilación, el análisis y la interpretación, sistemática y continua de datos de salud esenciales para la planificación, implementación y evaluación de la práctica de salud pública, estrechamente integrada con la diseminación oportuna de estos datos a quienes necesitan conocerlos"13. Dentro de las herramientas para la vigilancia, además de la información sobre los casos de la enfermedad, los datos de la vigilancia entomológica son muy importantes porque pueden facilitar la comprensión de la ecología de los vectores en un área determinada y permiten determinar el efecto de las estrategias de intervención anti-vectorial14,15.
Otra herramienta para la vigilancia son los sistemas de información geográfica, que ayudan a identificar casos y exposiciones, tendencias espaciales, así como a correlacionar diferentes conjuntos de datos y probar hipótesis estadísticas. Actualmente, el desarrollo de sistemas cada vez más sofisticados ayuda a los profesionales de la salud pública a monitorear y responder a los desafíos de salud16. Por ejemplo, las herramientas para georreferenciar permiten analizar las relaciones espaciales entre áreas con altos niveles de infestación de mosquitos y los sitios óptimos para el desarrollo larvario, y ahorran tiempo en la identificación de áreas problemáticas para que los trabajadores de salud pública realicen intervenciones de control17. La epidemiología molecular es otra herramienta que se ocupa de comprender la distribución y las relaciones de variantes genéticas, serotipos u otras agrupaciones moleculares de los patógenos, visualizándolas con árboles filogenéticos o dendrogramas18. Los análisis espacio-temporales de los genomas se centran en el mapeo y la predicción del intercambio de linajes de virus entre ubicaciones, para reconstruir las vías de introducción y propagación19. Además, estas herramientas ayudan a caracterizar el número de introducciones de un patógeno que conducen a la transmisión en una nueva ubicación, cuantificar el riesgo de transmisión entre especies y a inferir factores ecológicos de transmisión20.
Por otra parte, en la literatura sobre salud pública se evidencia el incremento de estudios que utilizan el análisis de redes sociales como otra herramienta para la vigilancia; los propósitos se centran en la identificación de las características comunes de los infectados, la comunicación visual o el mapeo de los casos para una mejor comprensión de los brotes, y la identificación de posibles vías de transmisión y características geográficas21,22,23. Los estudios han encontrado que los usuarios discuten públicamente los síntomas y comportamientos utilizados para prevenir dolencias24,25. Sin embargo, se conoce que el uso de las redes sociales tiene un sesgo demográfico. Por ejemplo, en Brasil utilizaron tweets de eventos de dengue geoetiquetados para explorar las tendencias de notificación espacio-temporales, y los relacionaron con los casos confirmados. Sus resultados mostraron que el volumen de tweets fue significativamente menor que el volumen de casos de dengue, y lo asociaron a la desigualdad y bajo ingreso de estas poblaciones, donde existe poca cultura para el uso de ciertas tecnologías en el área de la salud26.
La teledetección es otra herramienta para la vigilancia, puesto que los satélites diariamente recopilan grandes cantidades de datos y con diferentes tipos de medición que se pueden utilizar para desarrollar modelos predictivos para la propagación de enfermedades transmitidas por vectores27. Por ejemplo, se han hallado correlaciones entre el riesgo de transmisión del virus del Nilo Occidental y los cambios en la floración primaveral, la temperatura y la humedad, observados por satélites en EE. UU28. Además, se pueden obtener diversos tipos de indicadores e informar sobre los hábitats y riesgos de enfermedades transmitidas por mosquitos, garrapatas, moscas negras y flebótomos29. También se ha reportado que, en los países donde la vigilancia es escasa o está fracasando, el uso de datos de teledetección ha contribuido a la predicción de enfermedades emergentes transmitidas por vectores, permitiendo fomentar y focalizar medidas preventivas, como el control de vectores en las zonas afectadas27,30.
Todas estas herramientas generan información que, junto con el recuento de casos de la vigilancia, ha sido utilizada para tratar de explicar o predecir el comportamiento epidemiológico de las enfermedades arbovirales e intentar disminuir su impacto con el uso de diferentes metodologías de análisis.
Métodos de aprendizaje automático usados para predecir el comportamiento epidemiológico de enfermedades arbovirales
En los 41 artículos incluidos en esta revisión se encontraron 16 métodos de aprendizaje automático diferentes. La red neuronal artificial fue el más utilizado (n = 12), seguido de las máquinas de vectores de soporte (n = 9), las redes bayesianas (n = 8), los bosques aleatorios (n = 8), la regresión LASSO (n = 7), la media móvil integrada autorregresiva o media móvil integrada autorregresiva estacional (ARIMA/SARIMA, por sus siglas en inglés) (n=7) y el modelo aditivo generalizado (GAM, por sus siglas en inglés) (n = 6) (Tabla 1).
Tabla 1 Métodos de aprendizaje automático que se han utilizado para predecir el comportamiento epidemiológico de enfermedades arbovirales
| Método | Fuente | Cantidad de estudios |
|---|---|---|
| Red Neuronal Artificial | Salim et al. 202143; Liu et al. 202164; Mussumeci & Coelho, 202044; Bomfim et al. 202044; Zhao et al. 202039; Polwiang, 202057; Ho et al., 202067; Xu et al. 202045; Liu et al. 202040; Akhtar et al. 201947; Jiang et al. 201842; Baquero et al. 201832 | 12 |
| SVM | Nejad & Varathan, 202160; McGough et al.202161; Liu et al. 202164; Xu et al. 202045; Liu et al. 202040; Stolerman et al. 201952; Guo et al. 201731; Nsoesie et al. 201626; Althouse et al. 201122 | 9 |
| Redes Bayesianas | Salim et al. 202143; Nejad & Varathan, 202160; Akter et al. 202135; Aswi, et al. 201934; Baquero et al. 201832; Martínez-Bello et al. 201737; Ho et al. 201736; Nsoesie et al. 201626 | 8 |
| Random Forest | Mussumeci & Coelho, 202044; Benedum et al. 202062; Zhao et al. 202039; Carvajal et al. 201863; Jiang et al. 201842; Ong et al. 201856; Xiao et al. 201868; Liu et al. 201665 | 8 |
| Regresión LASSO | Mussumeci & Coelho, 202044; Liu et al. 202040; Chen et al. 201853; Wu et al. 201830; Guo et al. 201731; McGough et al. 201725; Shi et al. 20 1 654 | 7 |
| ARIMA // SARIMA | Benedum et al. 202062; Bomfim et al. 202046; Zhao et al. 202039; Polwiang, 202057; Carvajal et al. 201863; Baquero et al. 201832; Teng et al. 201723 | 7 |
| GAM | Xu et al. 202045; Jain et al. 201933; Carvajal et al. 201863; Xiao et al. 201868; Baquero et al. 201832; Guo et al. 201731 | 6 |
| Árboles de decisión | Salim et al. 202143; Wu & Kao, 202151; Nejad & Varathan, 202160; Ho et al. 202067; Campbell et al. 201555 | 5 |
| GBM | Xu et al. 202045; Tuladhar et al. 201958; Carvajal et al. 201863; Jiang et al. 201842 | 4 |
| Regresión Logística | Ho et al. 202067; Daughton & Paul, 201924; Benedum et al. 201815; Althouse et al. 201122 | 4 |
| NBM | Liu et al. 2021 64; Guo et al. 201731; Althouse et al. 201122 | 3 |
| Modelos Difusos | Adak & Jana, 202150; Torres et al. 201449 | 2 |
| GLM | Yuan et al. 202059; Ho et al., 202067 | 2 |
| SEIR Modelo Mecanicista | Bomfim et al. 202046 | 1 |
| Regresión de Poisson | Polwiang, 202057 | 1 |
| Máxima Entropía | Nsoesie et al. 201626 | 1 |
SVM = Support Vector Machines (Máquina de vectores de soporte)
GLM = Generalized Linear Models (Modelos lineales generalizados)
GBM = Gradient Boosting Machine (Máquina de aumento de gradiente)
GAM = Generalized Additive Model (Modelo aditivo generalizado)
NBM = Negative Binomial Regression Model (Modelo de regresión binomial negativa)
Algunos de estos estudios utilizaron varios modelos para predecir el comportamiento epidemiológico y los compararon para determinar cuál de ellos tenía el mejor desempeño. Por ejemplo, Guo et al. evaluaron seis algoritmos para predecir la incidencia de dengue en China y encontraron que el modelo GAM obtuvo los valores del promedio del error cuadrático medio (RMSE) más altos, mientras que el modelo de regresión de vectores de soporte (SVR) los tuvo más bajos en comparación con los modelos de regresión binomial negativa (NBM), la máquina de aumento de gradiente (GBM) y la regresión LASSO (least absolute shrinkage and selection operator), por tanto, el SVR fue considerado el modelo de mejor desempeño31. Por otro lado, en São Paulo, Baquero et al. realizaron un estudio comparativo de las predicciones del dengue utilizando diferentes métodos. Ellos encontraron que el GAM fue el más preciso, pues predijo correctamente grandes epidemias, incluidos los picos de 2014 y 2015, con un mejor desempeño que los modelos de red neuronal artificial (ANN) y SARIMA32.
Una de las aplicaciones más recientes fue realizada por Jain et al, quienes por medio de datos meteorológicos, clínicos, socioeconómicos y de vigilancia, desarrollaron modelos GAM para ajustar las relaciones entre los predictores (con un rezago de un mes) y el desenlace de brote de dengue hemorrágico. La evaluación de la capacidad de discriminación del modelo final contra el umbral constante específico de Bangkok y el umbral móvil de la epidemia de la OMS logró una especificidad del 92,6 %, sensibilidad del 87 %, un valor predictivo positivo del 95,7 % y un valor predictivo negativo del 78,8 %33.
Adicional a los modelos predictivos, se encuentran los modelos espaciales bayesianos, los cuales son usados para el análisis espacial de enfermedades porque pueden reducir la varianza estimada de la variable respuesta, particularmente para regiones con poblaciones pequeñas. También incorporan una gama amplia de componentes de varianza en diferentes niveles en el modelo y es más fácil obtener una evaluación más completa de la incertidumbre de la predicción basada en la máxima verosimilitud34. Por ejemplo, Akter et al. utilizaron modelos de regresión de Poisson multivariados con enfoque bayesiano y estructura previa condicional autorregresiva para analizar la variación espacial de las notificaciones de dengue en relación con la variabilidad climática y los factores socioecológicos en la zona climática tropical de Queensland, Australia35. En otro estudio, Ho et al. presentaron una red de creencias bayesianas (BBN) para evaluar el riesgo potencial de aparición y distribución del virus dengue en Australia Occidental, mientras que Martínez-Bello et al. aplicaron modelos espacio-temporales bayesianos jerárquicos para evaluar el riesgo relativo del dengue en Colombia36,37.
Es importante resaltar que el aprendizaje automático es una herramienta que combina la estadística con la informática mediante un uso eficiente de los conjuntos de datos masivos y de modelos no lineales con alta dimensionalidad38,39,40. Además, este se diferencia del modelo estadístico tradicional (por ejemplo, modelos de regresión) en que hay menos supuestos sobre la distribución subyacente de los datos y las relaciones entre las variables41,42. Por ejemplo, Salim et al. utilizaron varios modelos de aprendizaje automático para predecir los brotes de dengue, a saber: árboles de decisión (CART), ANN, SVM (Lineal, Polynomial, RBF) y Red Naíve Bayes (TAN). La variable de brote de dengue (1 = brote y 0 = sin brote) se creó con base en el número de casos de dengue notificados y se utilizaron 1300 registros. Estos modelos fueron similares en el sentido de que lograron obtener la probabilidad de la variable objetivo binaria e identificaron los predictores importantes43.
En Brasil, Mussumeci & Coelho compararon un modelo de red neuronal de memoria a largo y corto plazo (LSTM), un modelo de regresión de bosque aleatorio (RF) y regresión LASSO para predecir la incidencia de dengue en 790 ciudades brasileñas. Además, considerando que es muy difícil pronosticar con precisión la incidencia semanal en una ciudad utilizando solo sus propios datos históricos y que la transmisión de dengue tiene un componente espacial relacionado con la movilidad de las personas entre ciudades, utilizaron series de los predictores agrupando ciudades vecinas. Para esto definieron el grupo de ciudades dentro de un mismo estado con base en las distancias de correlación y para cada ciudad se ajustaron y probaron los modelos. Los datos históricos correspondieron a series de tiempo semanal (442 semanas en total) de incidencia de dengue, temperatura, humedad relativa, presión atmosférica y tuits sobre el dengue de cada grupo de ciudades. Los modelos LSTM y RF tuvieron los errores de predicción más bajos, sin embargo, LSTM pudo aproximar mejor la distribución empírica de la incidencia semanal del dengue y fue el menos sesgado de los tres modelos que sobrestimaron ligeramente las incidencias muy bajas de periodos interepidémicos mientras que subestimaron los picos epidémicos44.
Xu et al. propusieron un modelo basado en LSTM para predecir de manera eficiente los casos de dengue en 20 ciudades de China continental. Sus resultados mostraron que el LSTM redujo el promedio del RMSE de las predicciones de un 24,91 % a un 12,99 % y el RMSE promedio de las predicciones en el período del brote de un 26,82 % a 15,09 %, en comparación con GAM, SVR y GBM45. De manera similar, en un estudio en Fortaleza, Brasil, el LSTM pronosticó con precisión los casos de dengue, y la inclusión de datos de movilidad humana mejoró sustancialmente su desempeño46.
Adicionalmente, Akhtar et al. aplicaron un modelo de red neuronal dinámica basada en modelos autorregresivos no lineales con entradas exógenas conocidas como redes neuronales NARX, haciendo uso de datos epidemiológicos, volúmenes de viajes aéreos de pasajeros, idoneidad del hábitat del vector Ae. Aegypti, y datos socioeconómicos y de población. Su modelo se basó en un clasificador de riesgo binario, es decir, clasificar una región k como de alto o bajo riesgo en el tiempo t + N. El modelo se entrenó y probó utilizando los datos disponibles hasta la semana (X - N). Por ejemplo, la predicción con 12 semanas de anticipación para la semana 40 se realizó usando los datos disponibles hasta la semana 28. La precisión del modelo estuvo por encima del 80 °%47.
Además, el sistema de inferencia difusa (FIS), una de las técnicas de inteligencia artificial más populares y aceptadas, se ha convertido recientemente en una potente herramienta para desarrollar y analizar muchos sistemas del mundo real. FIS consta de funciones de pertenencia, operador de lógica difusa y algunas reglas definidas por el usuario que están en formato "si-entonces"48. Al respecto, Torres et al. emplearon un enfoque metodológico que combinó análisis multirresolución y FIS para evaluar su capacidad predictiva respecto al número de casos de dengue y dengue severo en un horizonte de tres años en Colombia. Acorde con sus resultados, la técnica logró un rendimiento significativamente superior al obtenido con las técnicas tradicionales de modelado difuso utilizadas hasta el momento. De esta forma, la similitud entre los datos originales y la señal aproximada aumentó del 21,13 % al 90,06 % y del 18,90 % al 76,83 % en el caso del dengue y el dengue severo, respectivamente49.
En una aplicación más reciente, Adak y Jana desarrollaron un modelo matemático utilizando un FIS tipo 2 para predecir las condiciones adecuadas para un brote de dengue, en el cual consideraron como variables de entrada los datos climatológicos (Temperatura, Precipitaciones y Humedad relativa) y como variable de salida la "Probabilidad del dengue". Para cada una de las variables se definieron rangos, mediante los cuales se asignaron las funciones de pertenencia que se tomaron como 'baja', 'media' y 'alta', consideraron todas las combinaciones posibles de las variables de entrada para asegurar la estabilidad del modelo. Los autores desarrollaron tres sistemas, el primer (R2 = 0,8169) y segundo sistema (R 2= 0,6832) funcionaron satisfactoriamente para predecir la probabilidad de ocurrencia del dengue en contraste con el tercer sistema (R2 = 0,6271), donde los valores predichos sobrestimaron los observados50.
Finalmente, la extracción de conocimientos de múltiples fuentes de datos abiertos en el campo de las enfermedades epidémicas se ha vuelto cada vez más relevante para la gestión de la salud pública, particularmente en relación con enfermedades causadas por algunos virus como Dengue, por lo que Wu y Kao en su estudio aplicaron un enfoque de minería de datos en un modelo de predicción en el cual consideraron datos de Google Trends. Sin embargo, al evaluar su efecto y desempeño en el modelo predictivo, no encontraron que esta información mejorara la predicción del dengue51.
Discusión
Algunas enfermedades arbovirales tienen un comportamiento epidemiológico estacional, es decir presentan un patrón regular de aumento de casos en ciertos periodos del año. Por consiguiente, el manejo eficaz de enfermedades estacionales depende del despliegue oportuno de medidas de control antes de la temporada de alta transmisión. Dado que la temporada epidémica varía de un año a otro, la disponibilidad de pronósticos precisos de incidencia puede ser decisiva para lograr el control de tales enfermedades o la preparación de los sistemas de salud para atender los casos que se presenten de tal forma que se disminuya la morbimortalidad44,52. Se han realizado múltiples investigaciones en diferentes partes del mundo para estudiar variables asociadas al comportamiento epidemiológico de las enfermedades arbovirales y se han tenido en cuenta diferentes fuentes de información y factores. Dentro de los factores estudiados se encuentran las condiciones socioeconómicas, el tamaño de la población, los comportamientos relacionados con el almacenamiento de agua, la urbanización, los índices vectoriales, la movilidad humana53,54,55, datos entomológicos56 y variables climatológicas como la temperatura, las precipitaciones, la humedad, y la velocidad del viento, entre otras46,50,54,57-63.
Adicionalmente, la posibilidad de utilizar información de múltiples fuentes de datos abiertos con el objetivo de predecir enfermedades epidémicas51,64,65, cuyas soluciones propuestas se han enfocado en modelos de predicción que incluyen estadísticas descriptivas, relaciones casuales, modelos de regresión y análisis de correlación66, han llevado a que la epidemiología computacional haya surgido como alternativa para analizar gran cantidad de datos provenientes de múltiples fuentes de información. Dentro de los métodos emergentes se resaltan los modelos de red neuronal artificial y máquina de soporte vectorial como los más utilizados. Adicionalmente, la enfermedad arboviral más frecuentemente estudiada fue el dengue y la mayoría de los estudios se desarrollaron en Asia.
Aunque en diferentes partes del mundo se han desarrollado diversos modelos predictivos de enfermedades arbovirales utilizando la información de vigilancia, se ha reportado que las técnicas de modelado estadístico más utilizadas en los estudios de enfermedades arbovirales como el dengue son la regresión de Poisson, la NBM58,67, ARIMA y GAM68. Los modelos ARIMA y GAM son modelos de referencia estándar para asociar factores ambientales con la enfermedad y una herramienta para el análisis de predicción de series de tiempo43. Sin embargo, aunque estas técnicas de modelado estadístico se utilizan ampliamente, tienen ciertas desventajas como el manejo de los valores perdidos, la sensibilidad de los valores atípicos y la multicolinealidad63.
Otras alternativas que se han utilizado para predecir el comportamiento epidemiológico de las enfermedades arbovirales son los modelos espaciales bayesianos y las técnicas basadas en inteligencia artificial. Los primeros pueden reducir la varianza estimada de la variable respuesta, e incorporar componentes de la varianza en diferentes niveles34, y las segundas tienen un enfoque no lineal, con menos supuestos sobre la distribución de los datos y las relaciones entre las variables41. Las anteriores características han permitido que estos modelos puedan considerar diversas fuentes de información y puedan tener una mejor predicción del evento. Sin embargo, las máquinas de vectores de soporte (SVM) podrían ser muy lentas para tareas a gran escala. También, a pesar del buen desempeño de los sistemas de pronóstico de redes neuronales, el modelo de red neuronal requiere una gran cantidad de datos y esfuerzo para ajustar sus hiperparámetros con el fin de evitar el sobreajuste. Por lo tanto, se necesita computación de alto rendimiento para el entrenamiento de los modelos.
En relación con las limitaciones para el uso de los métodos respecto a las fuentes de información, una de estas es la notificación "insuficiente", esto es, los casos de dengue en los estudios están significativamente subregistrados, especialmente porque los casos leves se subnotifican y los asintomáticos no se diagnostican. Esto sesga la asociación con las variables predictoras estudiadas y ocasiona que los modelos subestimen la cantidad real de infecciones. Adicionalmente, en varios estudios, los recuentos oficiales de casos en algunas semanas eran inferiores a 50, mientras que en otras semanas epidémicas los casos superaban los 1000. Por lo tanto, los modelos no tuvieron la capacidad de predecir con exactitud el número de casos, sobreestimándolos en las semanas bajas y subestimándolos en las semanas altas. También se reconoce como limitación el periodo a predecir, puesto que la exactitud disminuye a media que la ventana de pronóstico es más larga.
En cuanto a las variables predictoras, una de las limitaciones es la omisión de algunas de estas por falta de información que obliga a ajustar estadísticamente los modelos, lo cual afecta su calidad y rendimiento. Dentro de estas variables se encuentran la densidad vectorial, la biología del vector, el movimiento humano, los servicios de salud, entre otras. Otra limitación para los estudios cuya base de información fueron las búsquedas en Internet sobre la enfermedad, es que las tasas de uso de Internet y de búsqueda de información cambiaron con el tiempo, y esto dificultó la estimación de los parámetros en los modelos. Adicionalmente, los patrones de búsqueda pueden reflejar la cobertura de los medios y el conocimiento de la situación epidemiológica por parte de la población puede no coincidir con la dinámica de la enfermedad que se está rastreando. Además, los diferentes países y ciudades tienen distintas maneras de reportar las noticias sobre estos eventos. De esta forma, los medios locales en regiones endémicas para enfermedades arbovirales pueden reaccionar de manera diferente a los brotes que en regiones no endémicas. Por lo tanto, la atención de los medios tiene el potencial de influir dramáticamente en las predicciones semanales.
Finalmente, la presente revisión se enfocó en identificar los métodos de aprendizaje automático usados para predecir el comportamiento epidemiológico de enfermedades arbovirales y documentar las variables predictoras utilizadas. Sin embargo, dentro de las limitaciones de esta revisión se reconoce que solo se utilizaron dos bases de datos de publicaciones científicas para la búsqueda de los artículos y que no se realizó búsqueda de literatura gris. De este modo, pudieron no haberse incluido algunos artículos relevantes para la revisión.
Conclusiones
En la última década se ha incrementado la publicación de trabajos de investigación que pretenden predecir el comportamiento epidemiológico de las enfermedades arbovirales utilizando diversos métodos de aprendizaje automático para apoyar a los tomadores de decisiones en salud pública.
Los diversos métodos permiten incorporar no solo las series de tiempo de los casos y las variables climatológicas, si no también otras fuentes de información incluida la movilidad de los humanos, y variables de datos abiertos como las consultas en la web y los comentarios en redes sociales65. El rendimiento predictivo de estos métodos puede variar dependiendo del área geográfica, la longitud de las series de tiempo, el periodo de la predicción, la enfermedad objeto de predicción y las variables predictoras utilizadas. De tal forma, la predicción puede ser precisa solo para el área en particular donde se realizó el estudio, no pudiéndose extender su aplicación a otras áreas o regiones. Por consiguiente, se requiere evaluar la precisión entre diversos modelos para seleccionar el que tenga mejor rendimiento de generalización del evento de interés en el área geográfica específica.