Introducción
La infección persistente del epitelio cervical con Virus del Papiloma Humano de Alto Riesgo (VPH-AR) causa cáncer de cuello uterino (CCU)1. Cada año se diagnostican a nivel mundial más de 570 000 casos nuevos y es la segunda causa de muerte por cáncer, ya que ocasiona más de 300 000 muertes al año) (2-4. En Colombia la prevención del CCU se centra en acciones de prevención primaria (vacunación) y secundaria (tamización). La citología cérvico uterina es la herramienta utilizada hace más de 30 años para detectar lesiones premalignas y remitir posteriormente a tratamiento5. Sin embargo, en el momento de diagnosticar lesiones escamosas intraepiteliales de alto grado (HSIL), este método alcanza un porcentaje de falsos negativos entre el 42 y el 61 % y una sensibilidad del 50 %6. Por esta razón, se han desarrollado técnicas moleculares, las cuales han demostrado mayor sensibilidad en la detección de la infección por VPH y además permiten la genotipificación del virus7. Las pruebas moleculares detectan la presencia de ADN viral de estos genotipos en muestras obtenidas del cérvix, de esta manera se puede identificar a las mujeres en riesgo y prevenir el desarrollo de este cáncer.
Muchos protocolos se basan en la amplificación de ADN mediante PCR y utilizan diferentes cebadores que se dirigen principalmente hacia el gen L1 del virus, que posteriormente permite la diferenciación en genotipos por medio de sondas específicas que hibridizan los productos amplificados de la PCR. Sin embargo, existen regiones diana de mayor significancia clínica y epidemiológica como el oncogén E7, indispensable en el proceso cancerígeno de cuello uterino y presente durante todas las etapas de la progresión de la enfermedad, a diferencia del gen L1 que puede interrumpirse durante la integración del ADN viral en el genoma del hospedador hasta en el 11 % de los casos8,9. Estos datos sugieren que la PCR con cebadores consenso de L1 puede conducir a una pérdida significativa de detección de casos, mientras que la PCR con cebadores específicos para E7 puede proporcionar resultados más precisos y confiables, pues reduce la competencia para la amplificación del ADN cuando hay múltiples infecciones por VPH, mejorando notablemente la especificidad y sensibilidad del ensayo (Tabla 1). Por lo anterior, el objetivo del presente estudio fue estandarizar una PCR en tiempo real del oncogén E7 (E7-qPCR) para genotipificación y cuantificación de 6 genotipos de VPH-AR.
Tabla 1 Estudios comparativos para la detección molecular de VPH-AR
| Ensayo | Resultado | Referencia |
|---|---|---|
| PCR con cebadores consenso de L1 y PCR con cebadores específicos de genotipo E6/E7 | El 10,9 % de las infecciones por VPH-AR no son detectadas por PCR de L1, mientras que por PCR de E6/E7 sí es posible | Depuydt et al., 20079 |
| PCR con cebadores consenso de L1 | Omite el 8,3 % de las infecciones por VPH-16 y el 27,9 % de infecciones por VPH-18 | Tjalma, Depuydt, 201310 |
| PCR con cebadores de la región L1 en comparación con cebadores específicos para las regiones E6/E7 | En las infecciones por VPH-16 el 0,3 % de las NIC-2 y el 3,94 % de NIC-3 fueron negativas por L1 y positivas por E6/E7 | Roberts et al., 200611 |
| Sensibilidad relativa (SR) de la PCR | Cebadores consenso: 80,7 % PCR genotipo-específica: 97,8 % | Sahiner et al., 201412 |
| Frecuencia de falsos negativos para la PCR con cebadores consenso de L1 | 17,4 °% en infecciones únicas, 23,3 % en infecciones múltiples y 34,6 % en infecciones múltiples con tres o más genotipos de VPH. | Sahiner et al., 201412 |
| Concordancia entre la PCR de L1 y la de E7 | Los resultados discordantes entre la PCR de L1 y la de E7 permitieron detectar un 15,8 % de falsos negativos por parte de la PCR de L1 | Peña-López et al., 201913 |
| Variabilidad genética en los oncogenes E6 y E7 de VPH-52 | 96,6 % de las variantes de VPH-52 pertenecen al linaje B, que parece estar definido únicamente por G350T, A379G (K93R) en el gen E6 y por C751T, A801G en el gen E7. | Yang et al., 202214 |
| Variabilidad genética de VPH-16 y -52 | Para VPH-16, las mutaciones más prevalentes fueron T266A (27/27) y N181T (7/27) para L1, D32E para E6 y S63F para E7. Para VPH-52, todos los cambios de nucleótidos fueron mutaciones sinónimas en los genes L1 (excepto L5S) y E7. | Wang, et al., 202215 |
| Variabilidad genética de VPH-18 y -58 | En las secuencias de VPH-18 E6-E7-L1, se observaron cambios de 38 nucleótidos con 10/38 mutaciones no sinónimas (5 en el gen E6, 1 en el gen E7 y 4 en el gen L1). En las secuencias de VPH-58 E6-E7-L1, se observaron 53 cambios de nucleótidos con 23/53 mutaciones no sinónimas (3 en el gen E6, 5 en el gen E7 y 15 en el gen L1). | Wang, et al., 202316 |
Metodología
Amplificación ORF E6/E7: se seleccionaron 6 muestras con infección única para cada uno de los genotipos de VPH-AR elegidos que se encuentran dentro de los más frecuentes en el mundo (VPH-16, -18, -31, -33, -35 y -45). Estas muestras habían sido previamente analizadas por amplificación del gen L1 por PCR con subsecuente hibridación reversa con la técnica HPV Direct Flow CHIP. Se amplificaron los marcos de lectura abiertos de E6/E7 de los 6 genotipos de VPH-AR con cebadores específicos de genotipo utilizando la PCR Master Mix DreamTaq Green, 2X de Thermo Scientific (Massachusetts, USA) y el termociclador T100 de Bio-Rad (California, USA). La PCR se realizó con 150 ng de ADN en un volumen final de 20 µL y siguiendo el programa de amplificación descrito a continuación: 1 ciclo de desnaturalización inicial de 2 min a 95 °C; 40 ciclos de desnaturalización a 95 °C por 15 s, alineamiento a 58 °C por 20 s y extensión a 72 °C por 1 min; finalmente, un ciclo de extensión a 72 °C por 7 min y conservación a 4 °C. Se corrieron 15 µL de los productos de amplificación en un gel con concentración de agarosa al 2,0 % y los 5 µL restantes se conservaron a -20 °C. Posteriormente, se observó la separación de los fragmentos de ADN en el transiluminador de Accuris Instruments (Nueva Jersey, USA).
Clonación del ORF de E6/E7: Se utilizó el plásmido pMinit 2.0 del kit de clonación de NEB (Massachusetts, USA) según las especificaciones del fabricante. Posteriormente, se transformaron 50 µL de las bacterias competentes Escherichia coli TOP10F con 10 µL de la ligación por choque térmico y se seleccionaron 5-10 colonias transformantes las cuales se incubaron en 5 mL de caldo Luria Bertani-LB con ampicilina (100 µg/mL) a 37 °C y en agitación durante la noche. Se realizó una miniprep utilizando el kit Invisorb® Spin Plasmid Mini Two de Invitek molecular (Berlín, Alemania) a partir de 4 mL de suspensión bacteriana, y se cuantificó la concentración de ADN plasmídico y el radio de absorbancia a 260/280 nm en el espectrofotómetro NanoDrop 2000 de Thermo Scientific (Massachusetts, USA).
Confirmación de clonación del ORF de E6/E7: se hizo una digestión enzimática del plásmido pMinit 2.0 + ORF de E6/E7 con la enzima de restricción EcoRI-H de NEB (Massachusetts, USA), durante 20 minutos a 37 °C. Luego se realizó una electroforesis en gel de agarosa al 1 % con HydraGreen ACTGene (Nueva Jersey, USA) y se visualizó el ADN en el transiluminador de Accuris Instruments (Nueva Jersey, USA). Posteriormente, se confirmó por PCR convencional la ligación del inserto y, finalmente, para corroborar las identidades de los amplicones obtenidos, se secuenciaron por el método de Sanger (Macrogen, Korea, Seúl).
qPCR en Tiempo Real: se diseñaron cebadores específicos para fragmentos entre 138 y 239 pb del oncogén E7 de los 6 genotipos de VPH-AR y se optimizaron las condiciones de la qPCR utilizando el sistema convencional comercial SsoFast EvaGreen supermix de Bio-Rad (California, USA). Las curvas de estándares se construyeron por duplicado a partir de un ciclo umbral (threshold cycle, Ct) con 5 a 7 diluciones seriadas de los plásmidos de cada genotipo (107 a 101 copias). Uno de los controles negativos incluyó todos los componentes de la PCR sin ADN plantilla, y el otro incluyó el vector de clonación vacío. La amplificación, la detección de la señal y el análisis de datos se realizaron en el equipo CFX96 de Bio-Rad (California, USA).
Resultados y análisis
A partir de las muestras seleccionadas positivas para cada uno de los genotipos de VPH-AR se obtuvieron amplificaciones específicas de los ORF E6/E7 de los genotipos de VPH-16, -18, -31, -33, -35 y -45 (Figura 1A). Con 1 µL de estos productos amplificados se ligaron los ORF de E6/E7 en el plásmido pMinit 2.0 y se confirmó la clonación mediante digestión enzimática dando como resultado bandas entre 800 y 900 pb y entre 2500 y 3000 pb correspondientes a los ORF E6/E7 y al vector de clonación, respectivamente (Figura 1B). Posteriormente, se probaron los plásmidos ligados con los cebadores y sondas dirigidos a E7 específicos de genotipo en una PCR punto final, obteniéndose productos específicos de los 6 genotipos de VPH-AR. Finalmente, los resultados de la secuenciación mostraron un porcentaje de identidad del 99 % para cada uno de estos genotipos (datos no mostrados).
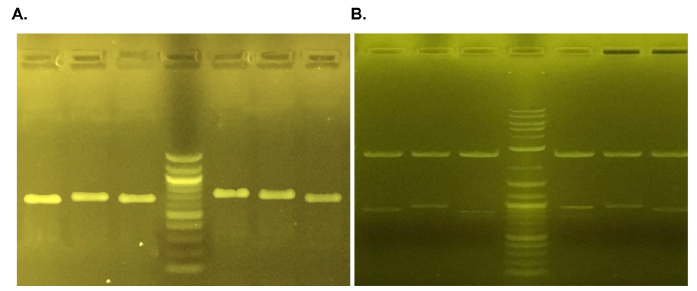
Figura 1 A. y B. Amplificación ORF E6/E7 y digestión enzimática. Carriles 1-3: bandas correspondientes a los ORF de E6/E7 de los genotipos VPH-16 (776 pb), VPH-18 (803 pb) y VPH-31 (749 pb); Carril 4: marcador de peso molecular; Carriles 5-7: Bandas correspondientes a los genotipos VPH-45 (806 pb), VPH-33 (758 pb) y VPH-35 (752 pb).
Para la estandarización de la PCR en tiempo real se optimizaron las condiciones de amplificación individualmente, se hicieron gradientes de temperatura y se escogió 57 °C como la mejor temperatura de alineamiento. Se ajustaron otros parámetros dando como resultado la siguiente mezcla final: 5 µL de SsoFast EvaGreen supermix (2x), 0,6 µL de cebadores 300 nM, 3,4 µL de agua grado biología molecular y 1 µL de la respectiva dilución; y el siguiente programa de amplificación: 1) 95 °C por 30 s, 2) 95 °C por 5 s, 3) 57 °C por 5 s, 4) volver al paso 2,39 veces, 5) Curva de Melt de 65 a 95 °C incrementando 0,5 °C cada 5 s.
Se obtuvieron curvas de amplificación sigmoidales, límites de detección desde 102 copias, ciclos de cuantificación desde 20 ciclos, eficiencias entre el 90 y 110 %, pendientes de la curva alrededor de -3,4 a -3,1, R2 > 0,97 y curvas de Melt que revelan productos específicos con picos de fusión entre 81-84 °C (Figura 2). De esta forma, se estandarizó la PCR en tiempo real del oncogén E7, que permite no solamente la detección de VPH-16, -18, -31, -33, -35 y -45 si no también la cuantificación de estos seis genotipos de VPH-AR. Teniendo en cuenta la evidencia que ha demostrado que las pruebas moleculares basadas en cebadores específicos para las regiones E6/E7 son más sensibles que las basadas en la región L1, puesto que reducen la frecuencia de falsos negativos (Tabla 1), se espera que la prueba estandarizada, una vez se valide, aumente la detección de la infecciones en nuestra población por estos seis genotipos de VPH-AR.
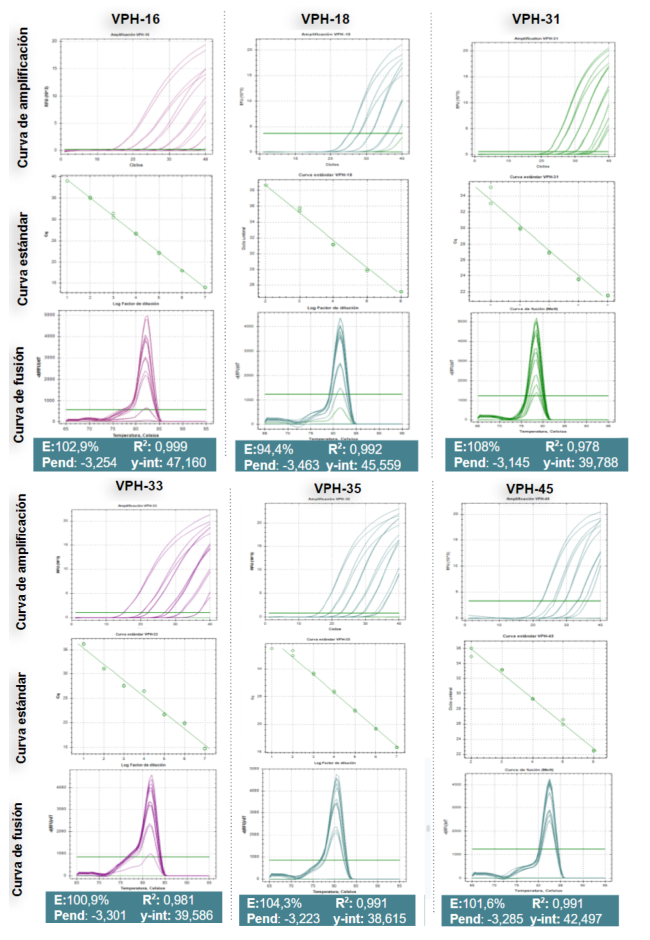
Figura 2 Curvas de amplificación, curvas estándar y curvas de fusión con diluciones de los plásmidos. Rango lineal hasta el valor R2 > 0,97 para el sistema Eva Green.
Además, se ha establecido que la importancia de cuantificar el título viral de VPH-AR radica en la asociación que existe entre la carga viral alta y la evolución de las lesiones preneoplásicas del cuello uterino17. Adicionalmente, la integración viral también está involucrada en el proceso de malignización de las células cervicales18. En este sentido, la ausencia de L1 y el incremento de E7 constituyen marcadores de integración y predicción de CCU.
Por otra parte, en infecciones con múltiples genotipos de VPH, el perfil de carga viral específico de cada genotipo podría distinguir las infecciones por VPH en progresión de aquellas en regresión. Por consiguiente, la E7-qPCR permite determinar la carga viral específica de 6 genotipos de VPH-AR, dando a este ensayo potencial aplicabilidad en la estratificación del riesgo, en la determinación de la persistencia de infección por VPH-AR y en el seguimiento post-tratamiento.
Conclusiones
Se estandarizó la PCR en tiempo real del oncogén E7 para genotipificación y cuantificación de 6 genotipos de VPH-AR altamente frecuentes. Esta puede ser una herramienta diagnóstica específica, rápida y de alto desempeño, que permitiría procesar un número elevado de muestras y contribuiría en la prevención del CCU. El siguiente paso será multiplexar esta prueba usando tecnología de sondas marcadas con fluoróforos y ampliar la cobertura de detección a 14 genotipos de VPH-AR para brindar un diagnóstico más sensible y específico.















