Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de Ciencias
Print version ISSN 0121-1935
rev. cienc. vol.19 no.1 Cali Jan./June 2015
Frecuencias polimórficas del gen de Apolipoproteína E
Polymorphic Frequencies of the Apolypoprotein E Genein Valle del Cauca, Colombia
Nelson Rivera Vivian
Departamento de Biología, Universidad del Valle, Cali - Colombia
E-mail: rivera.nelson@correounivalle.edu.co
Andrea Perdomo
Departamento de Biología, Universidad del Valle, Cali - Colombia
E-mail: vivian.perdomo@correounivalle.edu.co
Guillermo Barreto
Departamento de Biología, Universidad del Valle, Cali - Colombia
E-mail: guillermo.barreto@correounivalle.edu.co
Recived: November 7, 2014
Accepted: March 18, 2015
Resumen
El polimorfismo del gen de la Apolipoproteína E (ApoE) ha mostrado ser un factor implicado en laetiología de patologías cardiovasculares y neurodegenerativas; sus distintas isoformas son codificadaspor tres alelos comunes, E2, E3 y E4, siendo este último el principal factor de riesgo para llegar a sufrir la enfermedad de Alzheimer tipo esporádico o de inicio tardío. Considerando que el origen étnico y poblacional han mostrado ser factores determinantes del genotipo de este gen, y teniendo en cuenta la contribución amerindia, afrodescendiente y europea a la población del Valle del Cauca, se definió como objetivo de esta investigación conocer la distribución de las frecuencias alélicas y genotípicas para el gen ApoE en este Departamento y comparar estos resultados con los reportados para otras poblaciones de Colombia. Para lograrlo se analizaron 183 individuos mestizos sanos del Valle del Cauca mediante SSP-PCR. Fueron observadas las siguientes frecuencias alélicas: E2=3.0%,E3=84.7% y E4=12.3% y genotípicas: E3E3=71.0%, E3E4=21.9%, E2E3=5.5%, E2E4=0.5%,E4E4=1.1% y E2E2=0%. La población en estudio mostró frecuencias significativamente diferentes a las reportadas para poblaciones afrodescendientes de Colombia, mientras que para la mayoríade poblaciones amerindias analizadas estas fueron muy parecidas. Por otra parte, no se reportaron diferencias significativas con la población de Bogotá ni con la región del centro-oriente de Colombia, caso contrario ocurrió con la población del Quindío. En conclusión, la estructura y composición genética poblacional asociadas a procesos microevolutivos parecen ser factores determinantes en la distribución de polimorfismos de ApoE en Colombia. Los resultados de este trabajo sobre las frecuencias ApoE para el Valle del Cauca abren perspectivas orientadas a analizar mayores rangos de distribución alélica para confirmar si el incremento de alelos E4 podría ser un estimativo de riesgo en la población para desarrollo de patologías como el Alzheimer, y a su vez, si la baja presencia del alelo E2 puede ser un indicativo de ausencia de riesgo.
Palabras clave: Apolipoproteína E, polimorfismo APOE, frecuencias alélicas y genotípicas gen APOE, enfermedad de Alzheimer.
Abstract
The polymorphism of Apolipoprotein E (ApoE) has shown to be a factor involved in theethiology of cardiovascular and neurodegenerative pathologies. Different isoforms are encoded by three common alleles, E2, E3 and E4. E4 is the main risk factor for the sporadic or late onset type of Alzheimer disease. Because ethnic and racial origins have shown to be determinants for its genotype and considering the Amerindian, Afrodescendent and European contributions to populations in the Valle del Cauca, the purpose of this research was to establish the allelic distribution and genotype frequencies for ApoE gene in Valle del Cauca populations and compare our results with those from other Colombian populations. For this purpose, 183 unrelated mestizo individuals from Valle of Cauca were analysed by PCR-SSP. We observed the following allele frequencies: E2 = 3.0%, E3 = 84.7% and E4 = 12.3 and the genotype frequencies: E3E3 = 71.0%, E3E4 = 21.9%, E2E3 = 5.5%, E2E4= 0.5%, E4E4 = 1.1% and E2E2 = 0%. The studied population showed significantly different frequencies from those reported for Afrodescendent populations from Colombia, while in the most of Amerindian populations studied these frequencies were similar. Moreover, no significant differences were observed with the population from Bogotá or that from the Central-Eastern region of Colombia. In contrast, significant differences were found whit the Quindio´s population. In conclusion, the population genetic structure and composition associated with microevolutionary processes seem to be determinant factors in the distribution of ApoE polymorphisms in Colombia. These results open perspectives to analyse wider ranges of allelic distribution to confirm whether the increase of the E4 allele could be an indicative of population risk for developing diseases such as Alzheimer's and the rare presence of E2 allele in the population and indicative of a lower risk.
Keywords: Apolypoprotein E, ApoE polymorphism, ApoE gene allelic and genotypic frequencies, Alzheimer disease en el Valle del Cauca, Colombia.
1. Introducción
La Apolipoproteína E (ApoE) es una glicoproteína de 3,4 kD sintetizada principalmenteen el hígado y el cerebro (en astrocitos y microglías). Participa en diversos procesosbiológicos entre los que se incluyen el transporte y metabolismo de lípidos, la utilización intracelular del colesterol, el crecimiento celular, la inmunoregulación, y en el crecimiento, plasticidad, mantenimiento y reparación neuronal (Raffai et al., 2001). Esta proteína se compone de 299 aminoácidos plegados en dos dominios estructurales con funciones distintas; un dominio de interacción con el receptor de lipoproteínas de baja densidad (LDL por sus siglas en inglés) (residuos 1 al 191), y un dominio responsable de la interacción de ApoE con lípidos y lipoproteínas de baja densidad (residuos 216 al 299) (Mahley et al. 2006).
Existen tres isoformas comunes de ApoE, conocidas como ApoE2, ApoE3 y ApoE4, codificadas por los alelos E2, E3 y E4 del gen ApoE (19q13.2). Las diferencias alélicas se deben a uno o dos SNPs en el cuarto exón del gen, ocasionando el cambio de los codones paracisteína (CGC) y arginina (TGC) en las posiciones 112 y 158 de la proteína respectivamente (Hixson & Vernier, 1990; Strittmatter et al., 1993; Chou et al., 2005). ApoE3 presenta cisteína en la posición 112 y arginina en la posición 158, mientrasque ApoE2 tiene cisteínas en ambas posiciones y ApoE4 argininas en las mismas (Hixson, 1990; Chou et al., 2005). Estas isoformas son metabólicamente distintas y difieren en su afinidad por lipoproteínas, y también en la capacidad con la cual se unen a los receptores LDL (Fullerton et al. 2000).
La ApoE3 es considerada tanto metabólica como fisiológicamente la isoforma normal de la proteína mientras que sus formas alternas son asociadas a una variedad de desórdenes patológicos [21]. ApoE2 tiene una baja afinidad a los receptores LDL y está asociada con hiperlipoproteinemia tipo III; mientras que ApoE4 lo está con niveles elevados de LDLy colesterol en plasma, y es considerada un factor de predisposición a enfermedadescardiovasculares y desórdenes neurodegenerativos como el Alzheimer (Raffai et al. 2001).
Como consecuencia de su importancia biológica se han realizado estudios determinandolas frecuencias alélicas del gen ApoE en diversas regiones del mundo, las cuales varían según la estructura genética poblacional. En Latinoamérica, cuya población es étnicamenteheterogénea, los resultados obtenidos en países como Argentina y Chile no son comparablescon la población colombiana (Jacquier et al. 2001), sugiriéndose inclusive la existencia de variabilidad en las frecuencias alélicas y genotípicas del gen APOE entre grupos étnicos o poblaciones dentro del mismo país (Arboleda et al. 2001).
En este trabajo se determinó la frecuencia de las diferentes variantes polimórficas del gen ApoE en una muestra representativa de la población mestiza del Valle del Cauca y se comparó con las reportadas para otras regiones de Colombia. Estos resultados servirán de base en futuros estudios de diversidad genética del gen ApoE en Colombia y permitirán en la práctica clínica tener una valoración más precisa del riesgo de Alzheimer en la población del Valle del Cauca.
3. Metodología
El presente estudio contó con la participación de 183 individuos mestizos sanos (62 mujeres y 121 hombres) con edades entre 30 y 50 años sin parentescos familiares y pertenecientes a la población del Valle del Cauca. Los participantes en este estudiofueron reclutados entre grupos de trabajadores y empleados de diferentes instituciones. Los potenciales participantes fueron informados, grupal o individualmente, sobre elpropósito del estudio y lo que se requería de ellos para su participación. Los individuos que aceptaron participar asistieron a una entrevista semiestructurada (realizada por el Grupo de Investigación Clínica en Psicología, Neuropsicología y Neuropsiquiatría, Instituto de Psicología, Universidad del Valle) para determinar la presencia o ausencia de enfermedad mental, neurológica o sistémica con implicaciones cognitivas y determinar la inclusión o exclusión en el estudio. A aquellos individuos que pasaron el filtro de la entrevista y firmaron el consentimiento de participación voluntaria en esta investigación se les tomó una muestra de sangre para determinar su genotipo ApoE.
El método de "Salting-Out" de Miller et al. 1988 (1988) se empleó para la extracción del ADN de cada una de las muestras. La identificación de los genotipos E3E3, E2E2, E4E4, E3E2, E3E4, E2E4 del gen ApoE se llevó a cabo mediante la técnica "Sequence Specific Primer (SSP)-PCR", descrito por Pantelidis et al. (2003).
La reacción de SSP-PCR se llevó a cabo en un volumen de 15ul, conteniendoADN genómico (50 ng), Taq ADN polimerasa (0,3 U), dNTPs (0,03 mM de cada uno), cebador alélico E2, E3 o E4 (0,67 uM), cebador control (0,33 uM), MgCl2 (4 mM) y una concentración final de Buffer 1X de reacción de PCR.
La reacción de amplificación se realizó en un equipo Veriti® 96-Well Thermal Cycle usando un protocolo de PCR touchdown con temperaturas de anillamiento altas paraasegurar la especificidad en la amplificación como está descrito por Pantelidis et al. [10].Los fragmentos generados por la PCR fueron visualizados en geles de poliacrilamida al 6% teñido con nitrato de plata.
La determinación de las frecuencias de ApoE se realizó mediante conteo directo de los diferentes alelos (fragmentos) generados en la PCR. Los intervalos de confianza (I.C.) para las frecuencias alélicas fueron determinados según la fórmula propuesta por Nielsen et al. 1976, citado por Gerdes 2002 [11] con α = 0.05. Mediante el programa Arlequín versión 3.5 se evaluó Equilibrio Hardy-Weinberg para este locus. Para evidenciar la existencia de diferencias significativas entre las frecuencias encontradas en otras poblaciones deColombia y las frecuencias encontradas para el Valle del Cauca se realizó la prueba exacta de diferenciación poblacional de Raymond & Rousset (1995), mediante el programa Arlequínversión 3.5 (Excoffier & Lischer, 2010).
Las diferencias en las frecuencias de ApoE entre todas las poblaciones comparadas fueron calculadas con el estadístico Fst mediante el programa GenAlEx6.2 [13]. Estas relaciones de similitud fueron representadas gráficamente (dendrograma) mediante elmétodo UPGMA con el programa Phylip© (Phylogeny Inference Package) versión 3.69 (Felsenstein 1989) y TreeView© versión 1.6.6 (Peakall & Smouse, 2006).
3. Resultados
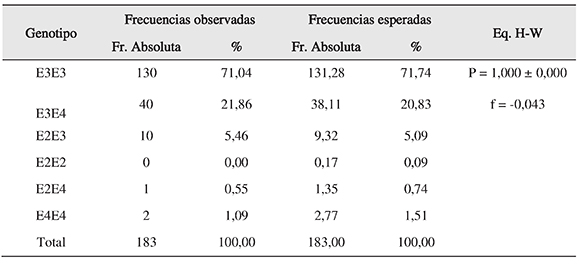
Las frecuencias genotípicas y alélicas del gen ApoE en la población del Valle del Caucase presentan en las tablas 1 y 2. El genotipo E3/E3 fue el más frecuente (71.04%), seguido por los genotipos E3/E4 (21,86%), E2/E3 (5,46%), E4/E4 (1,09%) y E2/E4 (0,55%). El genotipo E2/E2 no fue observado (Tabla 1). El alelo E3 fue el más frecuente (84,70%), seguido por E4 (12,30%) y E2 (3,00%), el menos frecuente (Tabla 2). Las frecuencias genotípicas observadas fueron similares a las esperadas bajo equilibrio Hardy-Weinberg (P = 1,000 ± 0,000), indicando que efectos como la endogamia o exogamia no están influenciandosignificativamente la distribución de ApoE en esta población (f = -0.043) (Tabla 1).
Tabla 1. Frecuencias genotípicas de ApoE en una muestra poblacional de mestizos del Valle del Cauca
Tabla 2. Frecuencias alélicas de ApoE en población mestiza del Valle del Cauca
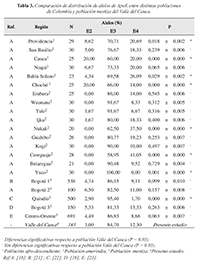
No se observaron diferencias significativas entre las frecuencias de ApoE encontradas para esta población y las poblaciones mestizas de Bogotá y el centro-oriente de Colombia, como síocurrió con la población mestiza de Quindío (Tabla 3). Las poblaciones afro-descendientes de Nuquíy San Basilio de Palenque no presentaron diferencias significativas,mientras que Providencia, Cauca, Bahía Solano y Chocó sí(Tabla 3). Por otra parte, la mayoría de las poblaciones amerindias no presentaron diferencias significativas respecto al Valle del Cauca (Emberá, Waunana, Tule, Ijka, Guahibo, Butaregua y Kogí), y solo las frecuencias de ApoE de Nukak, Coreguaje y Yuco fueron estadísticamente diferentes (Tabla 3).
Tabla 3. Comparación de distribución de alelos de ApoE entre distintas poblaciones de Colombia y población mestiza del Valle del Cauca.
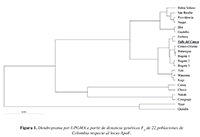
Las diferencias y similitudes entre las distintas poblaciones colombianas respecto a las frecuencias del locus ApoE son ilustradas en la figura 1. Se puede observar que las poblaciones más similares a la población mestiza del Valle del Cauca son las poblaciones mestizas de Bogotá y el centro-oriente de Colombia, junto con las etnias amerindiasButaregua y Embera, seguido de un cluster conformado por tres poblaciones indígenas(Waunana, Kogí, y Tule). Quindío a pesar de ser una población mestiza se encontró en un cluster separado del resto de poblaciones de Colombia, junto a Yuco. Otro clustercomprendió la mayoría de poblaciones afro-descendientes analizadas (Providencia, Nuquí,San Basilio de Palenque y Bahía Solano) y las poblaciones amerindias de Ijka y Guahibo. Las agrupaciones de Chocó - Cauca y Nukak – Coreguaje mostraron ser las más distintas del resto de poblaciones.
Figura 1. Dendrograma por UPGMA a partir de distancia genéticas Fst de 22 poblaciones de Colombia respecto al locus ApoE
4. Discusión
Es conocido que a partir de los siglos XV y XVI se da una importante inmigración española y africana a Colombia, sin embargo, actualmente, las poblaciones de Colombia sonrelativamente estables (Jacquieret al., 2001). Nuestro estudio confirmó la estabilidad poblacional colombianapor la existencia de equilibrio Hardy-Weinberg para el locus ApoE en una muestrapoblacional del Valle del Cauca. Asímismo, el índice de fijación (f) obtenido presentó un valor muy cercano a cero, indicando la ocurrencia de panmixia en esta población respecto al locus ApoE.
En genes asociados a patologías es común encontrar desviaciones del equilibrio H-W, debido al efecto de los alelos patológicos sobre la supervivencia y/o la reproducción de la especie (Salanti et al., 2005). Sin embargo, en el caso de ApoE no se aprecia este comportamiento,observándose equilibro para este locus en la mayoría de poblaciones que hasta el momento han sido analizadas.
La alta frecuencia del alelo E3 en esta población fue similar a las reportadas previamentepara otras poblaciones de Colombia, principalmente las poblaciones mestizas del centro (Bogotá) y centro-oriente de Colombia (Cundinamarca, Boyacá, Meta, Santander y Norte de Santander). Lo anterior pudo deberse al gran flujo génico existente entre poblaciones del centro y sur-occidente colombiano como lo muestran Rondón et al. (2008) sugiriendola posibilidad que estas comunidades conformen un mismo grupo poblacional (Rondón et al., 2008). La población del Quindío por su parte presentó diferencias significativas con relación a la población mestiza del Valle del Cauca. Posiblemente el flujo génico en esta población no ha sido suficiente como para homogenizar las frecuencias ApoE con el resto de poblaciones de esta región, y en consecuencia sus diferencias sean el resultado de la mezcla diferencial de etnias y/o de procesos microevolutivos una vez se dio el proceso de colonización.
La baja frecuencia del alelo E2 en las poblaciones mestizas de Colombia puede ser el resultado de su componente genético nativo americano. Según Wang et al. (2008)en promedio las poblaciones mestizas de Colombia tienen un componente genéticoprincipalmente amerindio (49.45%) y europeo (46.18%), y en menor proporción africano (4.43%) (Wang et al., 2008). Esto podría explicar porque la mayoría de poblaciones nativas americanas comparadas no presentaron diferencias significativas con la del Valle del Cauca. Sin embargo, algunas de estas poblaciones fueron agrupadas en clústeres lejanos al resto depoblaciones amerindias y mestizas, posiblemente ocasionado por procesos microevolutivosque llevaron a que sus frecuencias fuesen más similares a otros grupos poblacionales, comopor ejemplo las poblaciones de Nukak y Coreguaje que pueden ser influenciadas por el aislamiento genético, cuellos de botella y ventajas selectivas de su genotipo y el tipo de dieta (Jaramillo-Correa et al., 2001).
Con relación a los alelos ApoE la mayoría de las poblaciones afrodescendientespresentaron diferencias significativas cuando comparadas con la población mestiza del Valledel Cauca. Esta observación puede ser explicada si consideramos: 1. La alta frecuencia del alelo E4 en la población afrodescendiente en comparación a la población del Valle del Cauca, y, 2. El aumento del alelo E2 en dos de estas poblaciones afrodescendientes (Cauca y Chocó). Esta etnia, originaria del continente africano, fue traída a América como esclavos principalmente entre los siglos XVI y XIX (Rondón et al., 2008). Por lo tanto, las frecuencias en estas poblaciones se asemejan más a las del continente africano que las que presentan otras poblaciones americanas incluyendo las colombianas. No obstante, procesos demiscegenación pueden contribuir a la disminución de diferencias en las frecuencias de ApoE en estas poblaciones, razón por la cual San Basilio de Palenque y Nuquíno fueron estadísticamente distintas al Valle del Cauca, como también puede ser el resultado de un artefacto del muestreo o el bajo tamaño muestral.
Finalmente, el determinar cómo se encuentran distribuidos los distintos polimorfismos de ApoE en una población es de especial importancia en términos de su asociación con diversas patologías, entre estas, enfermedades cardiovasculares y neurodegenerativascomo el Alzheimer (Mahfouz et al., 2006). El alelo E4 es conocido como un importante factor genético de susceptibilidad para el desarrollo de la enfermedad de Alzheimer de tipo tardío, el cual en interacción con otros factores biológicos, genéticos y ambientales pueden determinar su aparición y desarrollo (Al-Khedhairy, 2004), mientras que el alelo E2 presenta un efecto contrario (Mahfouz et al., 2006).
Aunque se tiene evidencia que no siempre es necesario ser portador del alelo E4para desarrollar la enfermedad de Alzheimer, los individuos heterocigotos y homocigotos presentan un riesgo más alto de desarrollarla (OD = 7.4 y 19.6 respectivamente (Jacquier et al., 2001; Arboleda et al., 2001). En esta población una cantidad importante de individuos fueron portadores de al menos un alelo E4 (12,3%) y muy pocos portadores de alelos E2 (3,0%), lo que señala al gen ApoE como un factor genético potencialmente importante para el desarrollo de esta patología en los individuos de esta población.
5. Conclusión
A pesar de que las enfermedades multifactoriales como el Alzheimer son el resultado de complejas interacciones entre genes y factores medioambientales, el análisis de alelos de importancia clínica como ApoE proporciona información valiosa para la comprensión del desarrollo de esta patología en una determinada población.
Los resultados obtendios indican que la estructura y composición genética poblacional asociados a procesos microevolutivos parecen ser factores determinantes en la distribución de polimorfismos de ApoE en Colombia y proporcionan datos para una mejor comprensión de la variación geográfica, étnica y poblacional del gen ApoE en nuestro país.
Referencias
Al-Khedhairy, A.A. (2004). Apolipoprotein E polymorphism in Saudis. Molecular Biology Reports, 31(4), 257-260. [ Links ]
Arboleda, G.H., Yunis, J.J., Pardo, R., Gómez, C.M., Hedmont, D., Arango, G. et al. (2001). Apolipoprotein E genotyping in a sample of Colombian patients with Alzheimer's disease. Neuroscience Letters, 305(2), 135-138. [ Links ]
Callas, N., Poveda, E., Baracaldo, C.M., Hernández, P., Castillo, C., & Guerra, M. (2007). Polimofismo genético de la apolipoproteína E en un grupo de Escolares del Centro-oriente colombiano: Comparación con las concentraciones plasmáticas de lípidos y apolipoproteínas. Biomédica, 27(4), 526-536. [ Links ]
Chou, C.J., Lin, Y.L., Huang, Y.C., Sheu, S.Y., Lin, T.H., Tsay, H.J. et al. (2005). Structural variation in human apolipoprotein E3 and E4:Secondary structure, tertiary structure, and size distribution. Biophysical Journal, 88(1), 455-466. [ Links ]
Excoffier, L., & Lischer, H.E.L. (2010). Arlequin suite ver 3.5: a new series of programsto perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 10(3), 564-567. [ Links ]
Forero, D.A., Pinzón, J., Arboleda, G.H., Yunis, J.J., Alvarez, C., Cataño, N. et al. (2006). Analysis of Common Polymorphisms in Angiotensin-converting Enzyme and Apolipoprotein E Genes and Human Longevity in Colombia. Archives of Medical Research, 37(7), 890-894. [ Links ]
Fullerton, S.M., Clark, A.G., Weiss, K.M., Nickerson, D.A., Taylor, S.L., Stengard, J.H. et al. (2000). Apolipoprotein E Variation at the Sequence Haplotype Level: Implications for the Origin and Maintenance of a Major Human Polymorphism. The American Journal of Human Genetics, 67(4), 881-900. [ Links ]
Gerdes, L.U. (2002). Some Epidemiological Aspects of The Common GeneticPolymorphism of Apolipoprotein E. Tesis de post-grado, The Faculty of Health Sciences, University of Aarhus, Denmark, Dinamarca. [ Links ]
Hixson, J.E., & Vernier, D.T. (1990). Restriction isotyping of human apolipoprotein E by gene amplification and cleavage with HhaI. Journal of Lipid Research, 31(3), 545-548. [ Links ]
Jacquier, M., Arango, D., Villareal, E., Torres, O., Serrano, M.L., Cruts, M. et al. (2001). ApoEe4 and Alzheimer's disease positive association in a Colombian clinical series and review of the Latin-American studies. Arquivos de Neuro-Psiquiatria, 59(1), 11-17. [ Links ]
Jaramillo-Correa, J.P., Keyeux, G., Ruiz-Garcia, M., Rodas, C., & Bernal, J. (2001). Population Genetic Analysis of the Genes ApoE, ApoB (3'VNTR) and ACE in SomeBlack and Amerindian Communities from Colombia. Human Heredity, 52(1), 14-33. [ Links ]
Landázuri, P., Loango, N., Gallego, M.L., & Restrepo, B. (2009). Diferencias de sexo, edad y lípidos plasmáticos asociadas al polimorfismo de la apolipoproteína E en un grupo de escolares de Quindío, Colombia. Biomédica, 29(3), 381-391. [ Links ]
Mahfouz, R.A., Sabbagh, A.S., Zahed, L.F, Mahfoud, Z.R., Kalmoni, R.F., Otrock, Z.K. et al. (2006). Apolipoprotein E Gene Polymorphism and Allele Frequencies in the Lebanese Population. Molecular Biology Reports, 33(2), 145-149. [ Links ]
Mahley, R.W., Weisgraber, K.H., & HUANG, Y. (2006). Apolipoprotein E4: A causative factor and therapeutic target in neuropathology, including Alzheimer's disease. Proceedings of the National Academy of Sciences of the United States of America, 103(15), 5644-5651. [ Links ]
Miller, S.A., Dykes, D.D., & Polesky, H.F. (1988). A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Research, 16(3), 12-15. [ Links ]
Pantelidis, P., Lambert-Hammill, M., & Wierzbicki, A.S. (2003). Simple Sequencespecific-Primer-PCR Method to Identify the Three Main Apolipoprotein E Haplotypes. Clinical Chemistry, 49(11), 1945-1948. [ Links ]
Peakall, R., & Smouse, P.E. (2006). GENALEX 6: genetic analysis in Excel. Populationgenetic software for teaching and research. Molecular Ecology Notes, 6 (1), 288-295. [ Links ]
Raffai, R.L., Dong, L.M., Farese, R.V., & Weisgraber, K.H. (2001). Introduction of human apolipoprotein E4 "domain interaction" into mouse apolipoproteína E. Proceedings of the National Academy of Sciences of the United States of America, 98(20), 11587-11591. [ Links ]
Raymond, M., & Rousset, F. (1995). An exact test for population differentiation. Evolution, 49(6), 1280-1283. [ Links ]
Rondón, F., Osorio, J.C., Peña, A.V., Garcés, H.A., y Barreto, G. (2008). Diversidad genética en poblaciones humanas de dos regiones colombianas. Colombia Médica, 39(Supl 2), 52-60. [ Links ]
Salanti, G., Amountza, G., Ntzani, E. E., & Ioannidis, J. P. (2005). Hardy-Weinberg equilibrium in genetic association studies: an empirical evaluation of reporting, deviations, and power. European journal of human genetics, 13(7), 840-848. [ Links ]
Strittmatter, W.J., Weisgraber, K.H., Huang, D.Y., Dong, L.M., Salvesen, G.S., Pericak-Vance, M. et al. (1993). Binding of human apolipoprotein E to synthetic amyloid B peptide: Isoformspecific effects and implication for late-onset Alzheimer disease. Proceedingsof the National Academy of Sciences, USA, 90(17), 8098-8102. [ Links ]
Wang, S., Ray, N., Rojas, W., Parra, M.V., Bedoya, G., Gallo, C. et al. (2008). Geographic Patterns of Genome Admixture in Latin American Mestizos. PLoS GenetICS, 4(3), e1000037. [ Links ]

Revista de Ciencias por Universidad del Valle se encuentra bajo una licencia Creative Commons Reconocimiento 4.0.