1 Introducción
El petróleo es considerado como uno de los contaminantes más perjudiciales para el medio ambiente. Se ha informado que los derrames accidentales del crudo han causado la muerte de miles de especies de animales y plantas. En el caso del suelo, la contaminación por hidrocarburos impide el intercambio gaseoso con la atmósfera, afectando diferentes procesos fisicoquímicos, como la evaporación y la capilaridad, que, dependiendo del tipo de hidrocarburo, temperatura, humedad, textura del suelo y cantidad vertida pueden convertirse en procesos lentos, ocasionando toxicidad en los suelos y aguas contaminadas. En el caso de la contaminación en ambientes acuáticos, el hidrocarburo flota por diferencia de densidad, impidiendo la entrada de la luz y el intercambio gaseoso, iniciando la solubilización de compuestos hidrosolubles y afectando diferentes comunidades de organismos vivos 1.
A nivel global, son muchos los derrames de hidrocarburos, algunos de forma accidental como el desastre del buque Exxon Valdés entre Alaska y Canadá el 24 de marzo de 1989 y muchos otros, de manera provocada, tal es el caso de Colombia, en donde el transporte de crudo y sus derivados se ha visto afectado considerablemente durante los últimos 20 años por una permanente actividad terrorista contra los oleoductos e instalaciones petroleras. Este tipo de actividades ocasionaron el derramamiento de cerca de dos millones de barriles de petróleo; 7.6 veces el petróleo que se derramó en el desastre del buque Exxon Valdés 1.
Por la gravedad de la situación y en favor de la búsqueda de soluciones que permitan contrarrestar los graves efectos que acarrea el derrame de crudo sobre el ambiente natural, se han probado métodos convencionales de remoción de crudo de lugares contaminados, entre ellos, el empleo de dispersantes químicos. Sin embargo, estos pueden causar un mayor impacto ecológico que el mismo derrame, por su toxicidad y dificultad de degradación. Una estrategia alternativa para mitigar el impacto ambiental generado por la contaminación es la biorremediación; biotecnología emergente, que aprovecha la capacidad metabólica de los microorganismos (levaduras, bacterias, hongos, microalgas), plantas y sistemas biológicos (enzimas) para degradar, bio-transformar, o formar subproductos menos tóxicos. Básicamente la biorremediación permite acelerar los procesos bio- degradativos que de forma natural ocurren en los ecosistemas contaminados 2.
Desde la última década del siglo XX, se estableció que los microorganismos pueden degradar distintas formas de hidrocarburos, por su alta diversidad y capacidad de adaptación metabólica, que les confiere distintos mecanismos de acción sobre los contaminantes 3, de manera que individualmente, o en consorcios, estos pueden degradar sustratos y metabolitos que pueden generarse a partir de su utilización 4. Por lo tanto, técnicas como la biorremediación permiten aprovechar el potencial de los microorganismos para efectuar procesos de descontaminación natural. El objetivo del presente trabajo fue determinar la capacidad bio-degradadora de microorganismos nativos de suelo contaminado con hidrocarburos para remover el queroseno, subproducto que se obtiene de la destilación del petróleo.
2 Materiales y métodos
2.1 Muestreo
Se tomaron muestras de suelo en un taller de reparación de bicicletas ubicado en el centro de la ciudad de Cali (Colombia) (3°26’58.6”N 76°30’15.9”O), en el cual emplean gasolina y ACPM para limpieza de las bici-partes impregnadas de lubricantes de tipo oleaginoso. Considerando un área total aproximada de 12 m2, se tomaron tres submuestras de 10 g de suelo superficial a 5 cm de profundidad, en un trayecto lineal con una distancia entre punto y punto de 50 cm; las submuestras fueron integradas en una sola muestra, que se almacenó en una bolsa estéril y se trasladó inmediatamente hasta el laboratorio de docencia de microbiología del Departamento de Biología de la Universidad del Valle.
2.2 Aislamiento de microrganismos
Se pesó 1.0 g de la muestra de suelo y se diluyó en un matraz Erlenmeyer de 250 mL con 100 mL de medio mínimo de sales (MMS) que contenía (Ca (NO3)2 :60 mg/L, NaHCO3: 125 mg/L, KNO3: 70 mg/L, NH4Cl: 70 mg/L, KH2PO4: 100 mg/L, FeSO4.7H2O: 10 mg/L, MnCl.H2O: 7 mg/L, ZnSO4:1.5 mg/L) queroseno 1% v/v y extracto de levadura al 0.4%, (modificado de Narváez) 5, luego se incubó en agitación constante (shaker) a 150 rpm y temperatura ambiente por 48 horas. Posteriormente, se transfirió 1 mL del cultivo anterior a otro matraz que contenía 100 mL de MMS sin extracto de levadura y se incubó a las condiciones descritas. Seguidamente, se procedió al aislamiento de los microorganismos por el método de siembra por estrías en MMS solidificado con agar bacteriológico (Sharlau) suplementado con 1% de queroseno como fuente de carbono 6. Los cultivos se mantuvieron en incubación por 7 días a 30° C, considerando las condiciones ambientales adecuadas para el crecimiento tanto de hongos como de bacterias.
2.3 Selección de cepas microbianas para ensayos de degradación
La selección de los microorganismos se realizó mediante pruebas Concentración Mínima Inhibitoria (CMI) en matraz Erlenmeyer de 250 mL que contenían 100 mL de MMS con 10% de inóculo, obtenido a partir de un cultivo puro de 12 a 14 h, de cada una de las cepas aisladas preliminarmente, a concentraciones de 2%, 6% y 10% v/v de queroseno 5. Cada ensayo se realizó por triplicado, a 30° C, bajo agitación constante a 125 rpm (New Bruswick Scientific Series 126) y el crecimiento celular se determinó por densidad óptica a 600 nm en un espectrofotómetro (Thermo Spectronic Genesys 20, digital UV VIS) 2, tomando una muestra diaria de 1 mL del cultivo durante 14 días.
2.4 Caracterización bioquímica de la cepa bacteriana con mayor tolerancia al queroseno
La cepa bacteriana denominada preliminarmente como SH3, de mayor tolerancia al queroseno, se identificó y caracterizó bioquímicamente utilizando el kit BBL CRYSTAL™ Enteric/Nonfermenter (E/NF) Identification (ID), siguiendo las indicaciones de uso del fabricante (Becton, Dickinson and Company. ©2015 BD); adicionalmente, se realizó la prueba de Indol y Catalasa.
2.5 Identificación molecular de la cepa con mayor tolerancia al queroseno
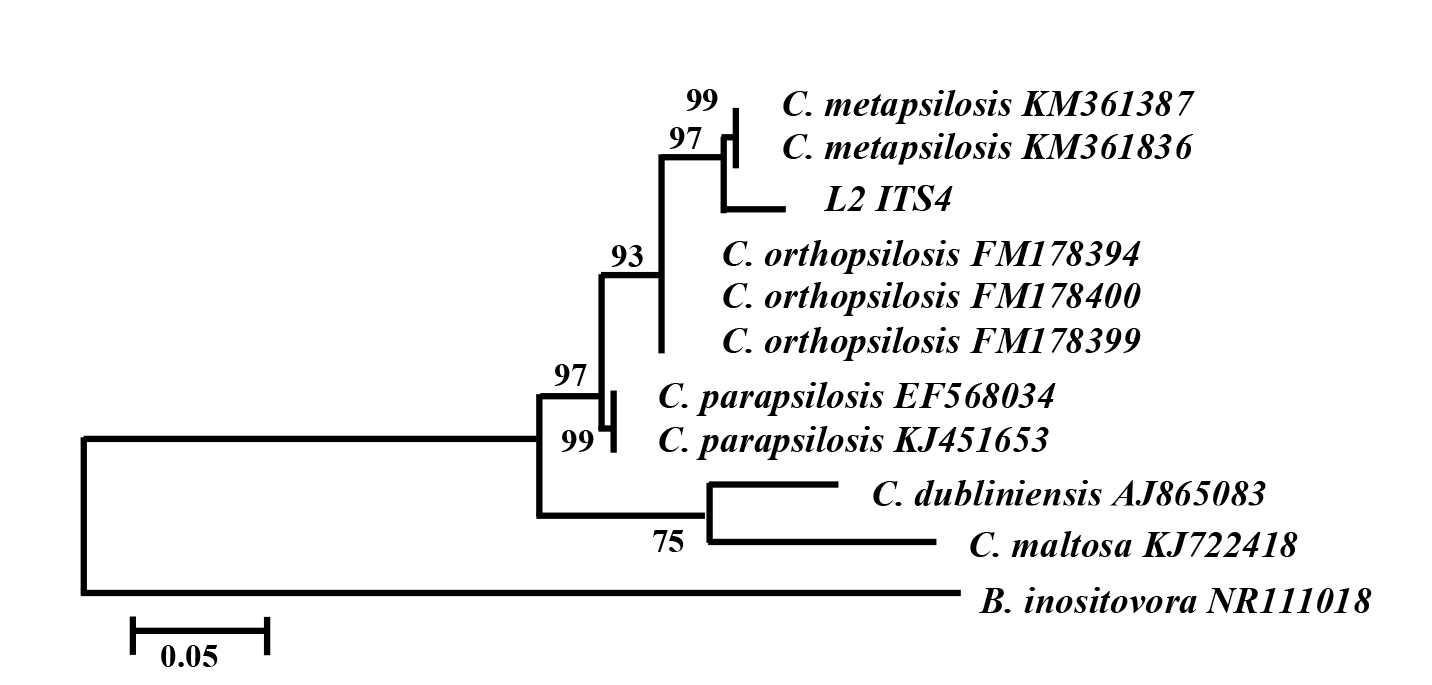
La identificación molecular se realizó sobre el microorganismo con mayor potencial hidrocarburoclástica que en este caso correspondió a la levadura (SH1). Para la extracción de ADN genómico se utilizó el kit ultra clean microbial DNA isolation (MOBIO LAB), garantizando buena calidad del producto para la PCR. El ADN extraído se amplificó utilizando los cebadores universales: ITS 5’TCCGTAGGTGAACCTGCGG3’, e ITS4 5’TCCTCCGCTTATTGATATGC3’; el coctel de ligación se elaboró con base en las siguientes concentraciones finales: Buffer (1X), MgCl2 (1.75 mM), DNTp´s (20mM), cebadores (0.4 mM cada uno). La amplificación se realizó en un termociclador (Applied Biosystems™ Veriti Thermal Cycler) bajo las siguientes condiciones modificadas de Adaskaveg y Hartin 7: una fase de desnaturalización a 94º C por 5 minutos, seguido de 35 ciclos con fases de desnaturalización a 94º C por 1 minuto, seguido de hibridación a 54º C 45 segundos y extensión a 72º C por 40 segundos; finalizando la PCR con una fase de extensión a 72º C de 1 minuto.
Las secuencias se determinaron mediante procedimientos estándares realizados por la compañía Macrogen (EEUU). Las secuencias parciales de la región ITS fueron editadas con el programa Chromas LITE versión 2.1; Las secuencias obtenidas fueron comparadas directamente con las secuencias depositadas en la base de datos del Gen Bank y alineadas con el programa Bioedit Sequence Alignment Editor 8. La clasificación filogenética fue inferida mediante el método de máxima verosimilitud utilizando el software MEGA versión 6.0, el modelo de substitución nucleotídica fue seleccionado por el criterio de Akaike en ModelTest versión 2.1.3 9; el soporte estadístico de los nodos se estimó mediante el bootstrap de máxima verosimilitud con 1000 repeticiones.
2.6 Ensayos de biodegradación de queroseno
Se realizaron en matraz Erlenmeyer de 250 mL, durante 67 días de incubación en MMS con 10% de inóculo microbiano, a un volumen final de 100 mL, a concentraciones de 2%, 6% y 10% de queroseno para B. cepacia y 6% y 10% para C. metapsilosis, se incluyó un control abiótico al 2% de queroseno. El queroseno se recuperó por medio de una extracción líquido-líquido con hexano, el cual arrastró el hidrocarburo hacia la parte superior del Erlenmeyer agitando vigorosamente de forma manual por un minuto y retirándolo con un gotero de punta fina a un vaso de precipitado. A la anterior mezcla se añadió Sulfato de sodio anhidro, para retirar el excedente de agua proveniente del medio; de la mezcla resultante se tomaron 1.5 mL, los cuales se traspasaron a viales de vidrio para las mediciones en un cromatógrafo de gases con detector selectivo de masas (GC/ MS) HP 6890/5973, el cual operó con las siguientes condiciones: temperatura inicial 40° C (2 min), temperatura final de 280° C (5 min), inyección Splitles de 20.0 a flujo constante de 1 mL/min. Las mediciones se centraron en la abundancia relativa de los hidrocarburos alifáticos comprendidos entre el C10 y el C16 presentes en el queroseno.
3 Resultados y discusión
3.1 Descripción e identificación de las cepas microbianas nativas con capacidad degradadora de queroseno
Se aislaron cuatro cepas bacterianas, denominadas SH2, SH3 y SH5 y una levadura, denominada SH1, a partir del medio MMS con queroseno, cuyas características morfológicas se resumen en la Tabla 1.
Tabla 1 Caracterización morfológica de los aislados microbianos.
| Cepa | Identificación | Gram | Características |
|---|---|---|---|
| Levadura SH1 | Candida metapsilosis. | Células levaduriformes, de colonias convexas de color blanco | |
| Bacteria SH2 | No identificada | (-) | Coco Bacilos, de colonias convexas, beige crema y con iridiscencia |
| Bacteria SH3 | Burkholderia cepacia. | (-) | Bacilos, de colonias planas, amarillas traslucidas con producción de pigmento verde azulado |
| Bacteria SH5 | No identificada | (-) | Cocos, de colonias convexas, color amarillo crema, brillantes y mucosas |
Las características metabólicas reveladas por el kit BBL Crystal™ permitieron ubicar a la cepa SH3 como Burkoldheria cepacia, lo cual se corrobora con las descripciones realizadas por Pallenori 10, la cual inicialmente había sido descrita como Pseudomonas cepacia y cuyas características son compartidas con los miembros del género Pseudomonas. Como características de la especie, se describe la capacidad de utilizar una gran variedad de compuestos de cadenas carbonadas, en especial la habilidad para crecer a expensas de azúcares como la arabinosa, la fructosa, la celobiosa y los polialcoholes como butilenglicol, hidroxibenzoato y aminas como la treonina, ornitina y triptamina; que son fuente importante de carbono y energía para su crecimiento.
La inferencia de la historia filogenética de la levadura SH1 se realizó con base en el mejor árbol generado por el método de máxima verosimilitud (Figura 1); el modelo de sustitución nucleotídica utilizado fue HKY +I+G con 0.6980 como parámetro alfa de la distribución gamma, 0.300 como la proporción de sitios invariantes, las frecuencias nucleotídicas fueron T=0.3482, C=0.1933, A=0.2692 y G=0.1893. Los análisis combinados de alineamientos realizados en la base de datos y la reconstrucción filogenética sugieren que la cepa estudiada tendría una relación filogenética muy estrecha con C. metapsilosis con una probabilidad aproximada del 97%.
3.2 Crecimiento microbiano con queroseno
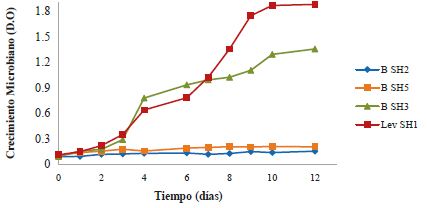
La prueba de CMI mostró que Candida metapsilosis (SH1) y Burkholderia cepacia (SH3) utilizaron el queroseno como fuente de carbono para el crecimiento celular, según los resultados de la curva de crecimiento realizada en MMS con queroseno al 10%, como única fuente de carbono (Figura 2), por lo cual, dichos microorganismos, fueron seleccionados para el ensayo de degradación del hidrocarburo de prueba.
Las cepas SH2 y SH3 por su parte, no fueron seleccionadas ni identificadas debido a su baja tolerancia ante la prueba de CMI. Es evidente que la carga de queroseno en un ambiente natural crea una fuerza de selección que ejerce un efecto negativo sobre el crecimiento de los microorganismos menos adaptados. Este comportamiento se observó en las cepas en estudio, cuyo crecimiento se vio inhibido a concentraciones de queroseno del 10%. Este comportamiento puede ser entendido si se considera que el queroseno es una mezcla de hidrocarburos muy complejos, con altos pesos moleculares y/o con grupos funcionales tales como los halógenos, sustancias tóxicas para diversos microorganismos (11). Aunque en mínima cantidad, se encuentran hidrocarburos aromáticos policíclicos, originados del benceno y el tolueno, que se han reportado como tóxicos para los microorganismos 12 , 13. La toxicidad es debida a la acumulación de contaminantes disueltos en la capa lipofílica de la membrana celular de los microorganismos, que, al ser fraccionados, tienen un modo de acción narcótica no específica o inhibitoria, asociada con cambios en membranas, enzimas y proteínas 14.
Por otro lado, los microorganismos que toleran elevadas concentraciones de hidrocarburos han desarrollado mecanismos para mantener la integridad de sus membranas ante un flujo excesivo de hidrocarburos, tales como: el incremento en la rigidez de la membrana por descenso en el contenido de ácidos grasos insaturados, la alteración en la conformación cis/trans de los fosfolípidos, la presencia de estructuras o enzimas capaces de metabolizar o degradar el contaminante y sistemas de exclusión homólogos a los empleados por las bacterias en la resistencia a antibióticos 15 , 16. Estos mecanismos explicarían la resistencia de las cepas SH1 y SH3 al queroseno, a pesar del posible efecto tóxico sin aparente inhibición.
3.3 Ensayos de degradación de queroseno
En la Figura 3, se muestra el cambio de abundancias relativas de los hidrocarburos alifáticos analizados. Para mejor compresión, obsérvese que los tratamientos cuyas concentraciones de queroseno fueron 2%, 6% y 10% se denotan como 2N, 6N y 10N respectivamente, en relación al control Q2s. La degradación de hidrocarburos ocurrió de manera similar en los dos microorganismos, mostrando una mayor disminución de las áreas relativas del tetradecano, pentadecano y hexadecano, con una tendencia contraria para las especies de decano y undecano. Estos resultados permitieron comprobar que la presencia de microorganismos acelera el proceso de fragmentación de hidrocarburos de mayor masa molecular hacia especies menos complejas.
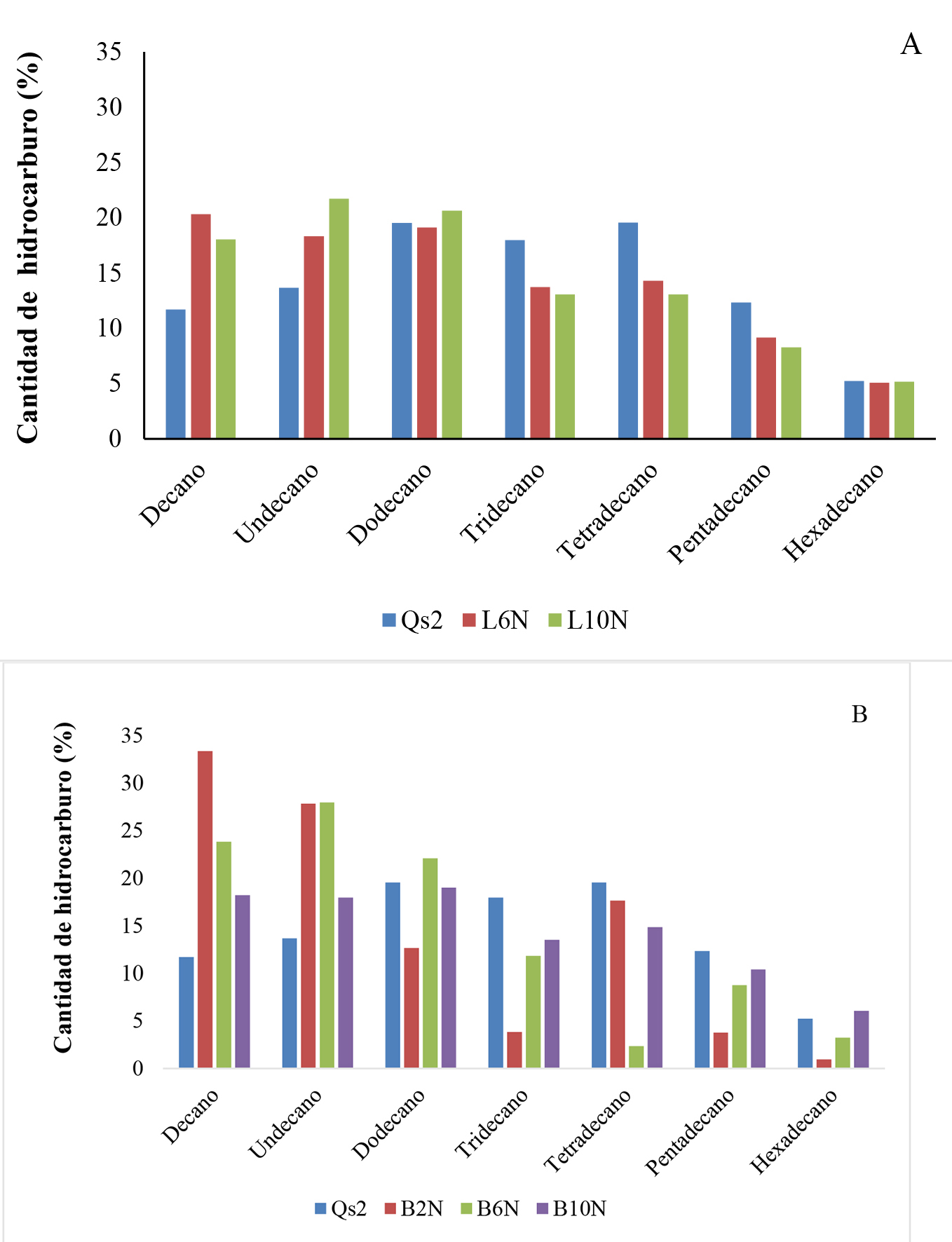
Figura 3 . Cantidad relativa de hidrocarburos alifáticos del queroseno (C10-C16). A) Candida metapsilosis, B) Burkholderiacepacia. Qs2: Control 2%; 2N, 6N, 10N: Concentración de queroseno (2%, 6% y 10%).
En este sentido, B. cepacia mostró mayor fragmentación de hidrocarburos, causando un aumento significativo de compuestos de cadenas más cortas. Esta fragmentación es más evidente al 2%, donde se observa la degradación de compuestos más largos, inducida posiblemente, por enzimas específicas para la biodegradación del queroseno 17. La tendencia de degradación de hidrocarburos de cadenas largas presentes en el queroseno fue descrita por González 18, presentando la afinidad de Stenotrophomonas sp., y Pseudomonas sp., para metabolizar hidrocarburos de cadenas entre 19 y 27 carbonos.
Sanchez-Yañez 19 sugiere que B. cepacia posee resistencia y capacidad de operar incluso a altas concentraciones de hidrocarburo, con una importante afinidad por los compuestos de cadenas carbonadas largas, lo cual podría revelar posibles aplicaciones industriales, como el aumento de octanaje de los combustibles a base del petróleo. Es probable que sus mecanismos de acción estén relacionados con la especialización de enzimas extracelulares inducibles como las lipasas. Estas enzimas se expresan durante la fase exponencial de crecimiento 15 , 20, aunque al ser inducibles se expresan también cuando el sustrato blanco está presente y las células las requieren para poder utilizarlo 18, que en el caso de los microorganismos expuestos a queroseno se expresa inmediatamente. Cabe resaltar que B cepacia, presentó dificultad para la degradación del hexadecano, similarmente a lo observado en miembros relacionados filogenéticamente, como el género Pseudomonas, sin embargo, para miembros de este género la situación ha sido superada significativamente con la adición de ácidos orgánicos como: ácido glutárico, ácido succínico, ácido malónico, ácido oxálico o ácido acético, en combinación de cualquiera de estos ácidos con el hexadecano 21.
En lo que respecta a C. metapsilosis, ha sido reportada como una levadura de importancia clínica con amplia distribución en todos los continentes, siendo aislada de abscesos, líquidos de lavado alveolar, bronquial y líquido articular 22. En varios estudios se reporta como perteneciente al complejo de C. parapsilosis, donde sería una de las cepas causantes de candidiasis e infecciones del torrente sanguíneo 22 , 23. Sus usos biotecnológicos, alimenticios o agropecuarios son desconocidos 13. En este sentido, los resultados de este estudio se constituirían en el primer reporte que evidencia la utilidad de C. metapsilosis con capacidad para metabolizar queroseno, contribuyendo así al conocimiento de microorganismos silvestres potencialmente útiles en procesos de biorremediación de hidrocarburos presentes en sitios contaminados con petróleo y sus derivados.
4 Conclusiones
Los microorganismos nativos C. metapsilosis SH1 y B. cepacia SH3 toleraron concentraciones de queroseno desde el 2% al 10%, demostrando habilidades para crecer eficientemente en presencia del hidrocarburo de prueba como única fuente de carbono y habilidades para fragmentar alcanos de cadenas carbonadas largas hacia especies menos complejas. Se propone el C. metapsilosis como microorganismo potencialmente útil para la biorremediación de ambientes contaminados por hidrocarburos.