Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Facultad de Odontología Universidad de Antioquia
Print version ISSN 0121-246X
Rev Fac Odontol Univ Antioq vol.26 no.1 Medellín July/Dec. 2014
REVISIÓN DE TEMA
ROL DE LA VÍA DE SEÑALIZACIÓN NOTCH DURANTE EL DESARROLLO DE ESTRUCTURAS CRANEOFACIALES
Belfran Alcides Carbonell Medina1
1 Odontólogo, especialista en docencia universitaria, candidato a magister en Odontología, Instituto de Genética, Universidad Nacional de Colombia. Correo electrónico: bacarbonellm@unal.edu.co
RECIBIDO: ENERO 22/2012-ACEPTADO:NOVIEMBRE 19/2013
Carbonell BA. Rol de la vía de señalización NOTCH durante el desarrollo de estructuras craneofaciales. Rev Fac Odontol Univ Antioq 2014; 26(1): 164-179.
RESUMEN
INTRODUCCIÓN: la vía de señalización NOTCH es un mecanismo de señalización célula-célula conservado evolutivamente entre las especies, el cual es indispensable para un correcto desarrollo embrionario, mediando una variedad de procesos celulares como proliferación, diferenciación, apoptosis, transformación epitelio- mesénquimal, migración, angiogénesis, mantenimiento de células madre y definición de destino celular. Varios genes componentes de esta vía han sido implicados en el desarrollo de estructuras craneofaciales. El 80% de los pacientes con síndrome de Alagille, presentan mutaciones en el gen que codifica para el receptor Jagged1 (Jag1), acompañado de hipoplasia del tercio medio facial y de craneosinostosis esporádica. Ratones con mutaciones homocigotas en el gen Jagged2 (Jag2) presentan paladar hendido, como resultado de fusiones ectópicas entre la lengua y los procesos palatinos. Por otro lado, mutaciones inducidas en el gen Hes1 generan defectos en el desarrollo de estructuras craneofaciales, derivadas de las células de la cresta neural craneal (CCNC) que incluyen: paladar hendido, agenesia del hueso frontal, malformación de base craneal y disminución en el tamaño del maxilar superior e inferior. Recientes estudios han evidenciado alteraciones durante la morfogénesis dental de ratones mutantes Jagged2-/-, acompañada de defectos en la citodiferenciación de ameloblastos y deficiente deposición de matriz de esmalte. Estos estudios muestran cómo la vía de señalización NOTCH está implicada en el desarrollo de una variedad de estructuras craneofaciales como paladar, dientes, maxilares y cráneo. Por esta razón, el propósito del presente artículo es presentar una revisión de las diferentes funciones de la vía NOTCH durante el desarrollo de estas estructuras craneofaciales, y de las alteraciones resultantes cuando existen mutaciones en algunos genes componentes de la vía NOTCH, como Jagged2, Jagged1, Hes1, Notch1 y Notch2.
Palabras clave: vía NOTCH, desarrollo craneofacial, palatogénesis, Jagged1.
INTRODUCCIÓN
El complejo craneofacial está formado por un conjunto de estructuras que incluyen, a groso modo, el viscerocráneo (cara) y el neurocraneo.1 El desarrollo craneofacial es quizás uno de los procesos más complejos durante la embriogénesis, ya que requiere una amplia variedad de interacciones entre los diversos tejidos embrionarios. 2, 3 Los tejidos que dan origen a las estructuras craneofaciales en vertebrados, son derivados de ectodermo, mesodermo, endodermo y, adicionalmente, de las células de la cresta neural craneal (CCNC).3, 4 Durante la embriogénesis, señales provenientes del ectodermo y el endodermo contribuyen recíprocamente para regular procesos celulares como proliferación, supervivencia, migración y diferenciación del mesénquima facial, a través de interacciones epitelio- mesénquima.5, 6 El mesénquima facial está compuesto tanto por células de la cresta neural craneal (CCNC) como por células del mesodermo paraxial cefálico.7
El mesodermo paraxial cefálico da origen a la musculatura voluntaria facial y células endoteliales, mientras que las CCNC dan origen al tejido conectivo craneofacial, como dentina, pulpa dental, cartílagos faciales, huesos del viscerocráneo, y región anterior de la base craneal.3, 5, 7 Varias vías de señalización han sido implicadas durante el desarrollo del complejo craneofacial, dentro de las más estudiadas tenemos BMP( Bone Morphogenetic protein), SHH(Sonic Hedgehog), WNT (Wingless), TGF-beta (Transforming Growth Factor Beta), FGF (Fibroblast Growth Factor) y más recientemente se ha vinculado la vía de señalización NOTCH.8-11 La vía de señalización celular NOTCH está implicada durante la etapa embrionaria y posnatal en varios procesos celulares, como proliferación, diferenciación, apoptosis, mantenimiento de células madre indiferenciadas y decisión de destino celular.12 Mutaciones inducidas en varios genescomponentes de la vía de señalización NOTCH como Jagged2 y Hes1, en modelos animales como el ratón, han mostrado alteraciones en el desarrollo de varias estructuras craneofaciales como paladar, dientes, maxilares, base y bóveda craneal.13-15
Dentro de las alteraciones craneofaciales más sobresalientes, se han reportado: paladar hendido, alteraciones morfológicas y deficiente formación de matrizde dentina, agenesia de huesos frontales, disminución del tamaño de los maxilares y cierre prematuro de las fisuras craneales, generando un fenotipo de craneosinostosis, involucrándose en esta última alteración la participación del gen Twist1.10, 13-15 Adicionalmente, entre el 70 y el 80% de los pacientes con síndrome de Alagille, reportan mutaciones en el gen Jagged1, el cual codifica un ligando perteneciente a la vía NOTCH.16 Este síndrome se caracteriza por alteraciones multiorgánicas y craneofaciales, que incluyen frente ancha, barbilla puntiaguda, punta de la nariz bulbosa, apariencia facial de triángulo invertido y craneosinostosis ocasional.17-19 Este artículo se hizo con el objetivo de revisar el rol de varios genes componentes de la vía de señalización NOTCH, como Notch1, Notch2, Jagged1, Jagged2 y Hes1, durante el desarrollo de estructuras craneofaciales como paladar, dientes, cráneo y maxilares.
Generalidades de la vía de señalización NOTCH
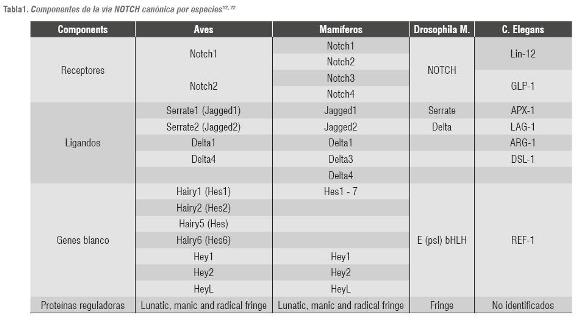
La vía de señalización canónica de NOTCH es un mecanismo de señalización célula-célula conservado evolutivamente, el cual participa en una variedad de procesos celulares como: especificación de destino celular, proliferación, apoptosis, adhesión, transformación epitelio-mesénquimal, migración, angiogénesis, mantenimiento de células madre yhomeostasis de tejidos adultos.12, 20, 21 La vía de señalización canónica de NOTCH está integrada por diversos componentes, entre los cuales podemos citar: receptores NOTCH, ligandos (DSL), genes diana (genes de la familia bHLH, Hesy Hey) y otras proteínas reguladoras de la vía descritas en la tabla 1.
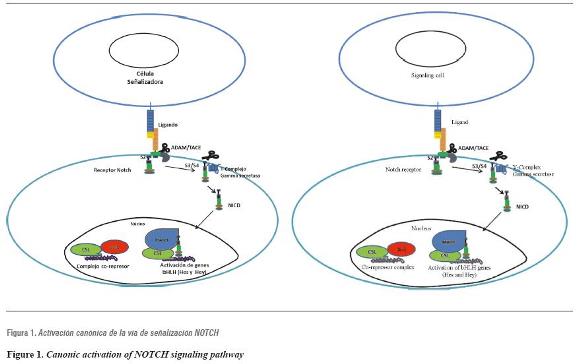
En mamíferos, como humanos y ratones, se han descrito cuatro receptores NOTCH (Notch1, Notch2, Notch3 y Notch4), cinco ligandos (Jag1, Jag2, Delta1, Delta3 y Delta4) y varios genes diana, dentro de los más estudiados se incluyen Hes1, Hes5 y Hey1.23 El receptor de la vía NOTCH es una proteína transmembranal que recibe señales de ligandos transmembranales que son expresados en células vecinas.22 El contacto directo entre el receptor y el ligando desencadena una serie de eventos proteolíticos a nivel del receptor NOTCH, provocando que el dominio intracelular del receptor se tras lo que al núcleo donde activa la transcripción de los genes diana Hes y Hey (figura 1).22 Las señales que son transducidas por estos receptores tienen un papel central en diversas etapas del desarrollo embrionario.24 De manera significativa, tanto las deficiencias y anormales aumentos de señalización NOTCH se han asociado con anomalías de desarrollo y cáncer.18, 25-28
El contacto entre el ligando de una célula señalizadora (célula en la parte superior) y el receptor NOTCH de una célula vecina (célula inferior), genera una serie de eventos proteolíticos a nivel de los dominios extracelular e intracelular del receptor NOTCH, provocando que el dominio intracelular NOTCH (NICD), se trasloque al núcleo, activando la transcripción de los genes de la familia bHLH, Hes y Hey, a través de la interacción con el factor de trascripción CSL y el coactivador MAML.
Vía NOTCH en el desarrollo del paladar secundario
El desarrollo del paladar secundario toma lugar alrededor del estadio E12-E15 en ratones, correspondiente al periodo comprendido entre la 8.a y la 12.a semana de gestación en humanos.27 Durante este periodo, los procesos palatinos que emergen de los bordes internos de las prominencias maxilares, crecen verticalmente a lado y lado de la lengua, posteriormente se elevan y se posicionan horizontalmente sobre el dorso de la lengua, para finalmente contactar uno con otro y fusionarse, dando lugar a una división entre la cavidad oral y la cavidad nasal.28 Cada uno de estos pasos durante la palatogénesis son altamente regulados por varias vías de señalización, y un fallo durante el crecimiento, elevación, contacto o fusión de los procesos palatinos, genera una fisura palatina.27
Durante el desarrollo normal del paladar, se ha detectado la expresión de varios genes componentes de la vía de señalización NOTCH.10 Los genes que codifican para los receptores Notch2 y Notch3, se expresan en el mesénquima lingual, procesos maxilares, mandibulares y palatinos desde E12.5 hasta E14.5. Contrario a este patrón de expresión mesénquimal, Jag2 y Notch1 son coexpresados predominantemente en el epitelio lingual, maxilar, palatino y epitelio lateral mandibular.10Ratones con mutaciones homocigotas Jag2-/-, exhiben paladar hendido acompañado de fusiones ectópicas entre la región dorsal de la lengua y los procesos palatinos.13
El fenotipo de paladar hendido en estos mutantes es atribuido a estas fusiones ectópicas que no permiten una elevación adecuada de los procesos palatinos y, por consiguiente, la génesis de la hendidura palatina.10, 13
Fusiones patológicas entre el paladar y la lengua han sido descritas en humanos.29, 30 Sin embargo, los mecanismos celulares y moleculares involucrados en este proceso patológico, no están totalmente claros. Casey, en el 2006, identificó en el modelo de ratón altos niveles del receptor Notch1 activado durante el proceso de diferenciación de las células del periderma lingual, maxilar, palatino y mandibular, desde E11.5 hasta E14.5.10 Los ratones mutantes Jag2sm/sm homocigotos, además de mostrar paladar hendido y fusiones entre la lengua y los procesos palatinos, presentan fusiones aberrantes entre los procesos maxilares y mandibulares, acompañado de reducción de los niveles activados de Notch1 durante la formación del periderma oral, con desorganización y pérdida de la morfología plana de las células peridérmicas que recubren el paladar y la lengua.10
Lo anterior sugiere a Jag2 como el ligando responsable de la activación de Notch1 y de la diferenciación del periderma oral. Por lo tanto, la señalización Jag2-Notch1 parece ser tempo-espacialmente regulada durante el desarrollo temprano del paladar, para prevenir prematuras adhesiones entre los procesos palatinos con el resto del epitelio oral en contacto, y así facilitar la elevación, contacto, adhesión y fusión de los procesos palatinos.10 Adicionalmente, otros autores han mostrado que la vía de señalización NOTCH, mediada por Jag2 junto a IRF6 (transcription factor interferón regulatory factor 6), funcionan como vías de señalización convergentes durante el proceso de diferenciación del epitelio oral, durante el periodo de palatogénesis.31
Otro de los genes pertenecientes a la vía de señalización NOTCH implicados en el desarrollo de estructuras craneofaciales como el paladar, es el gen Hes1.15 Ratones con mutaciones homocigotas Hes1-/-,presentan alteraciones durante el proceso de palatogénesis secundaria, caracterizado por un crecimiento deficiente y una prematura elevación y reorientación horizontal de las crestas palatinas, lo cual genera un paladar hendido, dado que el deficiente tamaño de las crestas palatinas no permite que estas puedan contactar y fusionarse en la mayoría de los mutantes. 15 Adicionalmente, estos ratones mutantes exhiben defectos en el desarrollo de la base craneal relacionados con agenesia del hueso esfenoides. Los mecanismos celulares y moleculares específicos involucrados en el fenotipo de estos mutantes aún es desconocido. Sin embargo, los autores sugieren que una diferenciación temprana de las células de la cresta neural craneal con una reducida proliferación, pudo haber inducido la elevación prematura y deficiente tamaño de las crestas palatinas.15
Vía de señalización NOTCH durante la odontogénesis: Jagged2 regula la diferenciación y morfogénesis dental.
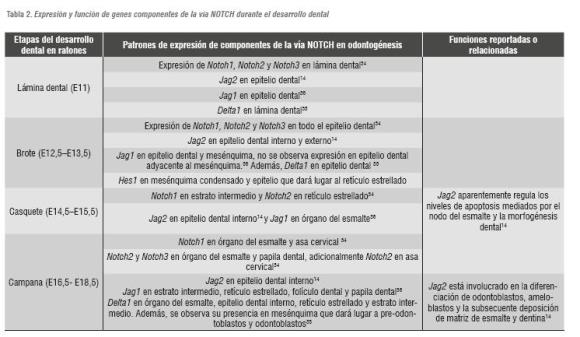
Además de la implicación de la vía NOTCH en el desarrollo de estructuras como paladar, varios componentes de esta vía de señalización han sido identificados durante el desarrollo dental de ratones. Varios estudios han evidenciado que los componentes de la vía de señalización NOTCH se expresan durante el desarrollo dental de ratones. La expresión de Notch1, Notch2, Notch332 Dll133, Jag134 y Jag235, 36 prefiguran en el desarrollo dental en la subdivisión de regiones ameloblásticas y no ameloblásticas, en las etapas iniciales del desarrollo dental (tabla 2). Esto se hace evidente durante la etapa de citodiferenciación, en la que varios receptores NOTCH y varios de sus ligandos, muestran patrones de expresión complementarios: la expresión de Notch1 se limita al estrato intermedio, mientras que Dll1 y Jag2 están expresados en la capa adyacente al epitelio dental interno (figura 2).33, 35, 36
Del mismo modo, en el mesénquima dental, Dll1 se expresa en odontoblastos en proceso de diferenciación, mientras que los genes NOTCH se expresan predominantemente en la capa sub-odontoblástica.34 Estos resultados sugieren que los receptores y ligandos de la vía NOTCH participan en los procesos de diferenciación celular durante el desarrollo dental. Recientes estudios han analizado la expresión, regulación y función del gen Jag2 en desarrollo dental de ratones.14 Estos estudios han mostrado que Jag2 se expresa en las células epiteliales, que darán lugar a la producción de esmalte (ameloblastos) durantelas primeras etapas del desarrollo dental.
Así mismo, en experimentos de recombinación tisular, se evidenció que la expresión de Jag2 en el epitelio está regulada por señales derivadas del mesénquima.14 Cultivos in vitro de explantes de epitelio dental muestran cómo la aplicación local FGF estimula la expresión de Jag2, mientras que la aplicación de BMP genera un efecto contrario. Lo anterior indica que durante el desarrollo dental, la expresión del gen Jag2 en el mesénquima es controlado por FGF y BMP desde el mesénquima. Ratones mutantes homocigotos Jag2 presentan una variedad de anomalías dentales.14 En molares, la morfología de la corona es deforme, con cúspides adicionales y en incisivos la citodiferenciación de ameloblastos y la deposición de matriz de esmalte son inhibidas.
Estos resultados demuestran que la vía NOTCH, mediada por Jag2, es indispensable para una correcta odontogénesis.14, 37, 38 Estudios recientes han permitido evidenciar una nueva función de la vía NOTCH mediada por el receptor NOTCH1 y una de sus proteínas efectoras Hes1, en el desarrollo el asa cervical. Mediante el uso de un inhibidor de la vía NOTCH, conocido como DAPT en modelos murinos in vitro e in vivo, se demostró que el bloqueo de la expresión del gen Hes1 dio lugar a un incremento en los niveles de apoptosis y una disminución en la proliferación de células madre del asa cervical.39 Estos resultados indican que la vía NOTCH a través de Hes1 controla la supervivencia de las células madre epiteliales del asa cervical en desarrollo.39
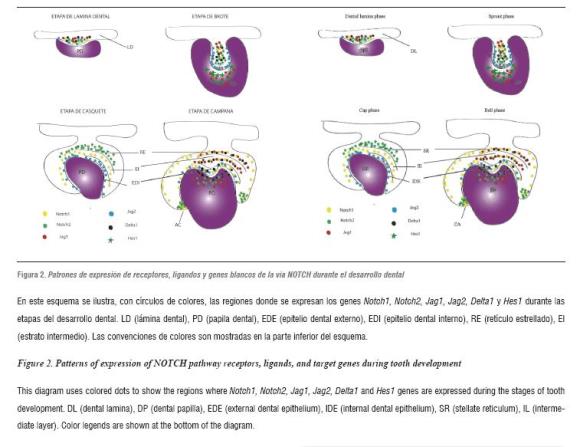
En este esquema se ilustra, con círculos de colores, las regiones donde se expresan los genes Notch1, Notch2, Jag1, Jag2, Delta1 y Hes1 durante las etapas del desarrollo dental. LD (lámina dental), PD (papila dental), EDE (epitelio dental externo), EDI (epitelio dental interno), RE (retículo estrellado), EI (estrato intermedio). Las convenciones de colores son mostradas en la parte inferior del esquema.
Implicación de la vía NOTCH en el fenotipo craneofacial de pacientes con síndrome de Alagille y Hajdu Cheney
El síndrome de Alagille es un trastorno autosómico dominante que afecta el sistema hepático, cardíaco, esquelético, renal, oftalmológico y el desarrollo facial.17, 40 El síndrome de Alagille es causado predominantemente por una haploinsuficiencia del gen Jagged1.16, 19, 41 Sin embargo, mutaciones en el gen Notch2 han sido identificadas en un subgrupo de pacientes con este síndrome. 18, 42, 43 El síndrome de Alagille se caracteriza por alteraciones craneofaciales que incluyen: frente ancha, barbilla puntiaguda, punta de la nariz bulbosa, hipoplasia del tercio medio facial que da la apariencia facial de triángulo invertido y craneosinostosis ocasional.17-19, 40, 44 El clásico rasgo facial de V invertida es encontrado en un 95% de los pacientes que son diagnosticados, basado en el fenotipo de conducto intrabiliar hepático.40, 45 Estos rasgos faciales sugieren que Jagged1 participa en la morfogénesis del tercio medio facial.
Varios modelos animales, como ratón y zebrafish, han sido utilizados para modelar las características del síndrome de Alagille, dentro de éstas las craneofaciales.46-48
El uso del modelo de ratón ha permitido identificar cuál es la función que desarrolla Jagged1 durante la morfogénesis facial. La delección de Jagged1 en células de la cresta neural craneal usando un ratón Wnt1-cre; Jag1 Flox/ Floxpermitió recapitular el fenotipo de hipoplasia del tercio medio facial de pacientes con síndrome de Alagille.49 La etiología de la hipoplasia del tercio medio facial en estos ratones, fue una consecuencia de una proliferación celular reducida de las CCNC en el tercio medio facial, aberrante vasculogénesis y deficiente producción de matriz extracelular en los procesos palatinos, asociados con un crecimiento anormal de la región facial.49 Específicamente, se evidenció tamaño disminuido del maxilar superior y deficiente elongación de los procesos palatinos.
Con respecto al desarrollo de la bóveda craneal, Jagged1 ha sido implicado en el mantenimiento indiferenciado de células pre ontogénicas inmaduras durante el desarrollo de las suturas craneales, garantizando así un desarrollo armonioso entre el crecimiento del cerebro y el cierre de las suturas en modelos de ratón.50 Estos estudios han evidenciado que Jagged1 funciona como un gen blanco del factor de transcripción TWIST1 para regular la expresión de genes como Β-catenina, Smad 1/3/8 y PrK1/2, implicados en la diferenciación de osteoblastos de la bóveda craneal. 50 De esta forma, Jagged1 mantiene indiferenciadas las células precursoras osteoblásticas y, por consiguiente, la permanencia de las suturas craneales. Mutaciones en Jagged1 resultan entonces en un cierre prematuro de las suturas coronales, generando así un fenotipo de craneosinostosis como el observado de manera esporádica en el síndrome de Alagille.44, 50
Otro de los síndromes con alteraciones en el desarrollo de estructuras craneofaciales, es el síndrome de Hajdu- Cheney.51, 52 Este síndrome presenta una herencia autosómica dominante, aunque se pueden encontrar casos esporádicos. Los pacientes con este síndrome presentan una mutación puntual en el exón 34 del gen Notch2, lo que genera un defecto en la síntesis del dominio PEST de la región intracelular de la proteína Notch2.53 El dominio PEST está involucrado en la ubiquitinación y degradación dela porción intracelular del receptor NOTCH, por lo tanto, la ausencia de este dominio permite que la activación de la vía NOTCH no sea regulada adecuadamente.22
El fenotipo craneofacial de los pacientes con síndrome de Hajdu-Cheney, incluye las siguientes características: Dismorfismo facial, micrognatismo, deficiente cierre de suturas craneales y formación de huesos wormianos.51 Hasta la fecha no se ha evidenciado la función que cumple el gen Notch2 durante el desarrollo de las estructuras craneofaciales. Por lo tanto, es necesario que, mediante el uso de modelos animales, se investiguen los mecanismos celulares y moleculares alterados durante el desarrollo de las estructuras craneofaciales afectadas en este síndrome.
CONCLUSIONES
Los estudios actuales sobre alteraciones en genes componentes de la vía NOTCH y su implicación en el desarrollo de estructuras craneofaciales, como paladar, bóveda craneal, base craneal y dientes, se limita a un número restringido de genes que incluyen Jagged2, Jagged1, Notch1, Notch2 y Hes1. Sin embargo, los mecanismos celulares y moleculares implicados en cada alteración del desarrollo resultante de la pérdida de función de cada uno de estos genes, aún no es clara. Por lo que es necesario que, mediante el uso de modelos animales como ratón u otros modelos que incluyen Zebrafish y pollo, se investigue la expresión de cada uno de los genes componentes de la vía NOTCH en cada etapa del desarrollo y, por medio de estudios de pérdida y ganancia de función génica, se establezca la función que desarrolla cada gen, tanto en etapas tempranas como tardías del desarrollo craneofacial.
Por otro lado, además de la necesidad de continuar ahondando en los aspectos celulares y moleculares de la vía NOTCH en el desarrollo craneofacial, se hace necesario hacerestudios genéticos en poblaciones afectadas con defectos craneofaciales, como paladar hendido y defectos de asimetría facial, en donde se investigue la presencia de mutaciones en varios de los genes de la vía NOTCH que han sido relacionados con estos defectos. Teniendo en cuenta los vacíos aún existentes referentes a la función de la vía NOTCH en el desarrollo craneofacial, es posible plantear los siguientes interrogantes que nos permitan orientar un proceso investigativo: ¿Cuál es la prevalencia de mutaciones en el gen Hes1 y Jagged2 en pacientes con paladar hendido? ¿Además de los pacientes con Alagille, en qué otros pacientes con asimetría facial se pueden identificar mutaciones en genes componentes de la vía NOTCH, además de Jagged1 y Notch2?
REFERENCIAS
1. Gross JB, Hanken J. Review of fate-mapping studies of osteogenic cranial neural crest in vertebrates. Deve Biol 2008; 317(2): 389-400. [ Links ]
2. Marcucio RS, Cordero DR, Hu D, Helms JA. Molecular interactions coordinating the development of the forebrain and face. Dev Biol 2005; 284(1): 48-61. [ Links ]
3. Szabo-Rogers HL, Smithers LE, Yakob W, Liu KJ. New directions in craniofacial morphogenesis. Dev Biol 2010; 341(1): 84-94. [ Links ]
4. Chambers D, McGonnell IM. Neural crest: facing the facts of head development. Trends Genet 2002; 18(8): 381-384. [ Links ]
5. Le Douarin NM, Brito JM, Creuzet S. Role of the neural crest in face and brain development. Brain Rev 2007; 55(2): 237-247. [ Links ]
6. Helms JA, Cordero D, Tapadia MD. New insights into craniofacial morphogenesis. Development. 2005; 132(5): 851-861. [ Links ]
7. Cordero DR, Brugmann S, Chu Y, Bajpai R, Jame M, Helms JA. Cranial neural crest cells on the move: their roles in craniofacial development. Am J Med Genet 2011; 155A(2): 270-279. [ Links ]
8. Nie X, Luukko K, Kettunen P. BMP signalling in craniofacial development. Int J Dev Biol 2006; 50(6): 511- 521. [ Links ]
9. Paiva KB, Silva-Valenzuela Md, Massironi SM, Ko GM, Siqueira FM, Nunes FD. Differential Shh, Bmp and Wnt gene expressions during craniofacial development in mice. Acta histochem 2010; 112(5): 508-517. [ Links ]
10. Casey LM, Lan Y, Cho ES, Maltby KM, Gridley T, Jiang R. Jag2-Notch1 signaling regulates oral epithelial differentiation and palate development. Dev Dyn 2006; 235(7): 1830-1844. [ Links ]
11. Loomes KM, Stevens SA, OBrien ML, Gonzalez DM, Ryan MJ, Segalov M et al. Dll3 and Notch1 genetic interactions model axial segmental and craniofacial malformations of human birth defects. Dev Dyn 2007; 236(10): 2943-2951. [ Links ]
12. Artavanis-Tsakonas S, Rand MD, Lake RJ. Notch signaling: cell fate control and signal integration in development. Science 1999; 284(5415): 770-776. [ Links ]
13. Jiang R, Lan Y, Chapman HD, Shawber C, Norton CR, Serreze DV et al. Defects in limb, craniofacial, and thymic development in Jagged2 mutant mice. Genes Dev 1998; 12(7): 1046-1057. [ Links ]
14. Mitsiadis TA, Graf D, Luder H, Gridley T, Bluteau G. BMPs and FGFs target Notch signalling via Jagged2 to regulate tooth morphogenesis and cytodifferentiation. Development 2010; 137(18): 3025-3035. [ Links ]
15. Akimoto M, Kameda Y, Arai Y, Miura M, Nishimaki T, Takeda A et al. Hes1 is required for the development of craniofacial structures derived from ectomesenchymal neural crest cells. J Craniofac Surg 2010; 21(5): 1443- 1449. [ Links ]
16. Warthen DM, Moore EC, Kamath BM, Morrissette JJ, Sanchez-Lara PA, Piccoli DA et al. Jagged1 (JAG1) mutations in Alagille syndrome: increasing the mutation detection rate. Hum Mutat 2006; 27(5): 436-443. [ Links ]
17. Emerick KM, Rand EB, Goldmuntz E, Krantz ID, Spinner NB, Piccoli DA. Features of Alagille syndrome in 92 patients: frequency and relation to prognosis. Hepatology 1999; 29(3): 822-829. [ Links ]
18. McDaniell R, Warthen DM, Sanchez-Lara PA, Pai A, Krantz ID, Piccoli DA et al. Notch2 mutations cause Alagille syndrome, a heterogeneous disorder of the notch signaling pathway. Am J Hum Genet 2006; 79(1): 169- 173. [ Links ]
19. Penton AL, Leonard LD, Spinner NB. Notch signaling in human development and disease. Semin Cell Dev Biol 2012; 23(4): 450-457. [ Links ]
20. Bolos V, Grego-Bessa J, de la Pompa JL. Notch signaling in development and cancer. Endocr Rev 2007; 28(3): 339- 363. [ Links ]
21. Pan Y, Liu Z, Shen J, Kopan R. Notch1 and 2 cooperate in limb ectoderm to receive an early Jagged2 signal regulating interdigital apoptosis. Dev Biol 2005; 286(2): 472-482. [ Links ]
22. Fiuza UM, Arias AM. Cell and molecular biology of Notch. J Endocrinol 2007; 194(3): 459-474. [ Links ]
23. Gordon WR, Arnett KL, Blacklow SC. The molecular logic of Notch signaling a structural and biochemical perspective. J Cell Sci 2008; 121(Pt 19): 3109-3119. [ Links ]
24. Artavanis-Tsakonas S, Rand MD, Lake RJ. Notch signaling: cell fate control and signal integration in development. Science 1999; 284(5415): 770-776. [ Links ]
25. Aster JC, Pear WS, Blacklow SC. Notch signaling in leukemia. Annu Rev Pathol 2008; 3: 587-613. [ Links ]
26. Aster JC. Deregulated NOTCH signaling in acute T-cell lymphoblastic leukemia/lymphoma: new insights, questions, and opportunities. Int J Hematol 2005; 82(4): 295-301. [ Links ]
27. Gritli-Linde A. Molecular control of secondary palate development. Dev Biol 2007; 301(2): 309-326. [ Links ]
28. Dudas M, Li WY, Kim J, Yang A, Kaartinen V. Palatal fusion - where do the midline cells go? A review on cleft palate, a major human birth defect. Acta histochem 2007; 109(1): 1-14. [ Links ]
29. Din SU. Atypical tongue-tie due to congenital tonguepalate fusion. J Coll Physicians Surg Pak 2003; 13(8): 459- 460. [ Links ]
30. Humphrey T. Palatopharyngeal fusion in a human fetus and its relation to cleft palate formation. Ala J Med Sci 1970; 7(4): 398-426. [ Links ]
31. Richardson RJ, Dixon J, Jiang R, Dixon MJ. Integration of IRF6 and Jagged2 signalling is essential for controlling palatal adhesion and fusion competence. Hum Mol Genet 2009; 18(14): 2632-2642. [ Links ]
32. Mitsiadis TA, Lardelli M, Lendahl U, Thesleff I. Expression of Notch 1, 2 and 3 is regulated by epithelial-mesenchymal interactions and retinoic acid in the developing mouse tooth and associated with determination of ameloblast cell fate. J Cell Biol 1995; 130(2): 407-418. [ Links ]
33. Mitsiadis TA, Hirsinger E, Lendahl U, Goridis C. Deltanotch signaling in odontogenesis: correlation with cytodifferentiation and evidence for feedback regulation. Dev Biol 1998; 204(2): 420-431. [ Links ]
34. Mitsiadis TA, Henrique D, Thesleff I, Lendahl U. Mouse Serrate-1 (Jagged-1): expression in the developing tooth is regulated by epithelial-mesenchymal interactions and fibroblast growth factor-4. Development 1997; 124(8): 1473-1483. [ Links ]
35. Mitsiadis TA, Regaudiat L, Gridley T. Role of the Notch signalling pathway in tooth morphogenesis. Arch Oral Biol 2005; 50(2): 137-140. [ Links ]
36. Valsecchi C, Ghezzi C, Ballabio A, Rugarli EI. JAGGED2: a putative Notch ligand expressed in the apical ectodermal ridge and in sites of epithelial-mesenchymal interactions. Mech Dev 1997; 69(1-2): 203-207. [ Links ]
37. Harada H, Kettunen P, Jung HS, Mustonen T, Wang YA, Thesleff I. Localization of putative stem cells in dental epithelium and their association with Notch and FGF signaling. J Cell Biol 1999; 147(1): 105-120. [ Links ]
38. Mustonen T, Tummers M, Mikami T, Itoh N, Zhang N, Gridley T et al. Lunatic fringe, FGF, and BMP regulate the Notch pathway during epithelial morphogenesis of teeth. Deve Biol 2002; 248(2): 281-293. [ Links ]
39. Felszeghy S, Suomalainen M, Thesleff I. Notch signalling is required for the survival of epithelial stem cells in the continuously growing incisor. Differentiation 2010; 80(4- 5): 241-248. [ Links ]
40. Kamath BM, Loomes KM, Oakey RJ, Emerick KE, Conversano T, Spinner NB et al. Facial features in Alagille syndrome: specific or cholestasis facies? Am J Med Genet 2002; 112(2): 163-170. [ Links ]
41. Yuan ZR, Kohsaka T, Ikegaya T, Suzuki T, Okano S, Abe J et al. Mutational analysis of the Jagged 1 gene in Alagille syndrome families. Hum Mol Genet 1998; 7(9): 1363- 1369. [ Links ]
42. Kamath BM, Bauer RC, Loomes KM, Chao G, Gerfen J, Hutchinson A et al. NOTCH2 mutations in Alagille syndrome. J Med Genet 2012; 49(2): 138-144. [ Links ]
43. Turnpenny PD, Ellard S. Alagille syndrome: pathogenesis, diagnosis and management. Eur J Hum Genet 2012; 20(3): 251-257. [ Links ]
44. Kamath BM, Stolle C, Bason L, Colliton RP, Piccoli DA, Spinner NB et al. Craniosynostosis in Alagille syndrome. Am J Hum Genet 2002; 112(2): 176-180. [ Links ]
45. Piccoli DA, Spinner NB. Alagille syndrome and the Jagged1 gene. Semin Liver Dis 2001; 21(4): 525-534. [ Links ]
46. Lorent K, Yeo SY, Oda T, Chandrasekharappa S, Chitnis A, Matthews RP et al. Inhibition of Jagged-mediated Notch signaling disrupts zebrafish biliary development and generates multi-organ defects compatible with an Alagille syndrome phenocopy. Development 2004; 131(22): 5753- 5766. [ Links ]
47. McCright B, Lozier J, Gridley T. A mouse model of Alagille syndrome: Notch2 as a genetic modifier of Jag1 haploinsufficiency. Development 2002; 129(4): 1075- 1082. [ Links ]
48. Zuniga E, Stellabotte F, Crump JG. Jagged-Notch signaling ensures dorsal skeletal identity in the vertebrate face. Development 2010; 137(11): 1843-1852. [ Links ]
49. Humphreys R, Zheng W, Prince LS, Qu X, Brown C, Loomes K et al. Cranial neural crest ablation of Jagged1 recapitulates the craniofacial phenotype of Alagille syndrome patients. Hum Mol Genet 2012; 21(6): 1374- 1383. [ Links ]
50. Yen HY, Ting MC, Maxson RE. Jagged1 functions downstream of Twist1 in the specification of the coronal suture and the formation of a boundary between osteogenic and non-osteogenic cells. Dev Biol 2010; 347(2): 258-270. [ Links ]
51. Zanotti S, Canalis E. Notch signaling in skeletal health and disease. Eur J Endocrinol 2013; 168(6): R95-103. [ Links ]
52. Zanotti S, Canalis E. Notch regulation of bone development and remodeling and related skeletal disorders. Calcif Tissue Int 2012; 90(2): 69-75. [ Links ]
53. Isidor B, Lindenbaum P, Pichon O, Bézieau S, Dina C, Jacquemont S et al. Truncating mutations in the last exon of NOTCH2 cause a rare skeletal disorder with osteoporosis. Nat Genet 2011; 43(4): 306-308. [ Links ]
54. Mucchielli ML, Mitsiadis TA. Correlation of asymmetric Notch2 expression and mouse incisor rotation. Mech Dev 2000; 91(1-2): 379-382. [ Links ]











 text in
text in