Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
ORINOQUIA
versión On-line ISSN 0121-3709
Orinoquia vol.18 supl.1 Meta dic. 2014
ARTÍCULO ORIGINAL/ORIGINAL ARTICLE
Caracterización genética de Ichthyoelephas longirostris de los ríos La Miel y Ranchería usando marcadores microsatelites
Genetic characterization of Ichthyoelephas longirostris La Miel and Ranchería Rivers using markers microsatellites
Caracterização genética do Ichthyoelephas longirostris dos ríos La Miel e Ranchería utilizando marcadores microssatélites
Yuly A. Perdomo-Aguirre1*
Wilson R. Cruz-Flor1*
Luisa P. Espinosa-León2*
Mauricio Carrillo-Avila3*
1 Estudiante de Licenciatura en Ciencias Naturales
2 Tecnóloga en Acuicultura Continental
3 Biólogo Marino, MSc, PhD
* Laboratório de Biodiversidad Molecular y Citogenética, Facultad de Ciencias Exáctas, Universidad Surcolombiana, Neiva, Huila, Colombia Email: jayaps_29@hotmail.com
Recibido: octubre 30 de 2014. Aceptado: noviembre 28 de 2014
Resumen
El Ichthyoelephas longirostris comúnmente conocido como el Pataló es una especie endémica de la cuenca del río Magdalena y del río Ranchería. Posee una fuerte presión pesquera comercial y de consumo. Según el libro rojo de peces dulceacuícolas de Colombia actualmente se encuentra reportada como una especie amenazada debido a las fuertes alteraciones de su hábitat producidas principalmente por contaminación y erosión. Teniendo en cuenta su importancia y la falta de información genética reportada, en este estudio se analizaron seis marcadores microsatélites para determinar el grado de variabilidad y estructura genético-poblacional del Pataló de los ríos La Miel y Ranchería. Para las dos localidades la heterozigosidad esperada varió de 0,6182 a 0,546, la heterozigosidad observada entre 0,6812 a 0,454, la riqueza alélica entre 6,760 y 4,640 y la diversidad génica encontrada estuvo entre 0,631 y 0,540 respectivamente. El índice de endogamia FIS evidenció un exceso de heterocigotos dado por su valor medio negativo. Los análisis realizados indican que existen dos poblaciones diferentes de Pataló con una estructuración genética moderada, las cuales presentan un buen estado genético. Estos resultados son de gran valor por ser el primer estudio de caracterización genética realizado en esta especie y con el cual se podrán tomar medidas de control y manejo especialmente cuando ya se están implementando acciones de repoblamiento y se está trabajando en su reproducción en cautiverio.
Palabras claves: Peces de agua dulce, estructura poblacional, conservación, variabilidad genética.
Abstract
The Ichthyoelephas longirostris, commonly known as Pataló, is an endemic species from the Magdalena River and Rancheria River in Colombia. It is facing a b commercial and consumer fishing pressure. According to the red book of freshwater fishes of Colombia, it is identified as an endangered species due to b changes in its habitat, mainly caused by pollution and erosion. In this research, given its importance and the lack of genetic information reported, six microsatellite markers were analyzed to determine the degree of genetic variability and population structure of Pataló from La Miel and Rancheria rivers. For the two locations the expected heterozygosity ranged from 0.6182 to 0.546, the observed heterozygosity from 0.6812 to 0.454, allelic richness between 6.760 and 4.640, and the gene diversity ranged between 0.631 and 0.540, respectively. The Fis inbreeding index showed an excess of heterozygotes given its average negative value. The analysis howed two distinct populations of Pataló with moderate genetic structure, which exhibit a good genetic status. These results are of great value, given that this is the first study of genetic characterization conducted in this species. Based on these results control and management measures can be taken, relevant for repopulating actions being implemented with regard to reproduction in captivity.
Key words: Freshwater fish, population structure, conservation, genetic variability.
Resumo
O Ichthyoelephas longirostris é uma espécie conhecida como Pataló a qual é endêmica do rio Magdalena e do Rio Ranchería. Tem uma forte pressão comercial e para o consumo. De acordo com o libro vermelho de espécies de agua doce do Colômbia atualmente encontra-se classificada como ameaçada devido às fortes alterações no seu habitat causadas principalmente pela poluição e erosão. Dada a sua importância e a falta de informação genética reportada, neste estudo seis marcadores microssatélites foram analisados para determinar o grau de variabilidade genética e estrutura populacional do Pataló dos ríos La Miel e Ranchería. Para ambos os locais a heterozigosidade esperada variou de 0,6182-0,546, a heterozigosidade observada entre 0,68120,454, a riqueza alélica entre 0,6760 e 0,4640 e a diversidade genética encontrada entre 0,631 e 0,540 respectivamente. O índice de endogamia Fis mostrou um excesso de heterozigotos dado pelo seu valor médio negativo. As análises indicam que existem duas dieferentes populações de Pataló com estrutura genética moderada, que apresentam um bom estado genético. Estes resultados são de grande valor como o primeiro estudo de caracterização genética realizada nesta espécie e como qual podem-se tomar medidas de controle e manejo, especialmente quando já estão implementando ações de repovoamento e trabalhando em reprodução em cativeiro.
Palavras chave: peixes de agua doce, estrutura populacional, conservação, variabilidade genética.
Introducción
La cuenca del Magdalena comprende 256.622 Km2, corresponde al 22.5 % de la superficie del país, representando el escenario de las principales pesquerías de aguas continentales, ya que en ella se concentra la mayor población dedicada a esta actividad, la cual está estimada en aproximadamente 30 mil pescadores artesanales (Dahl, 1971 y Beltrán et al., 2000). El río La Miel es un tributario de la cuenca del Magdalena localizado al oriente de la Cordillera Central de los Andes colombianos, representando la principal fuente hídrica del departamento de Caldas (Arango y Montes-RJM. 2009).
Otra fuente fluvial de interés es el rio Ranchería que es uno de los más importantes del departamento de la Guajira. Nace en la laguna de chirigua que está ubicada en la Sierra Nevada de Santa Marta a 3700 m de altura sobre el nivel del mar y desemboca directamente en el mar Caribe (Polania, et al., 2006; Mojica, et al., 2006). El Pataló, Ichthyoelephas longirostris (Steindachner, 1879), es una especie con distribución en la cuenca Magdalénica y el río Ranchería (Patiño, 1973; RománValencia y Ortiz-Muñoz, 2001; Mojica et al., 2002). Es considerado un pez detritívoro que se alimenta principalmente de Navicula spp. y de Synedra spp. (Bacillariaceae) (Román-Valencia, 1993) que se desarrollan en los ríos de aguas claras y limpias. Debido a su hábito alimenticio las poblaciones son seriamente afectadas por la contaminación, el incremento de la erosión debido a la deforestación, explotaciones mineras principalmente del oro y la presión pesquera dado a su gran aceptación para el consumo debido a la calidad de su carne principalmente en la cuenca alta del río Magdalena debajo de la represa de Betania; que en algunos casos utilizan para su captura métodos destructivos de pesca como la dinamita: lo que ha conllevado a la desaparición en algunas áreas (Román-Valencia, 1993, Román-Valencia y Ortiz-Muñoz, 2001, Mojica et al., 2012; Castro y Vari, 2004). Según Patiño (1973), la especie se reproduce en aguas torrentosas, migrando en cardúmenes.
A pesar de existir pocos estudios de historia natural y pesquerías, esta especie se encuentra catalogada en peligro a nivel nacional y en peligro crítico para la cuenca del río Ranchería (Mojica, et al., 2012).Esta condición de especie amenazada hace importante dirigir esfuerzos para realizar estudios de su biología, reproducción en cautiverio, y caracterización genética de poblaciones naturales con el fin de diseñar políticas y programas de manejo y conservación de la especie.
Es por esta razón que estudios de la variabilidad genética de las poblaciones silvestres de este pez son muy importantes para el reconocimiento de las unidades genético-poblacionales existentes que puedan contribuir en programas de conservación que consideren el pool génico de la especie.
Para estos estudios los marcadores tipos microsatélites surgen como una herramienta importante en el auxilio de estas investigaciones. Los microsatélites son secuencias cortas de 1-6 bases repetidas en tándem encontradas en todos los genomas de los eucariotos y procariotos analizados hasta el momento. Están presentes en las regiones codificantes y no codificantes y son caracterizados por un alto grado de polimorfismo, además de constituir un comportamiento co-dominante y presentar un alto número de alelos. El origen de tal polimorfismo está todavía en debate pero el mecanismo más probable parece estar relacionado a eventos de slippage durante la replicación del ADN (Schlotterer y Tautz 1992).
El principal inconveniente de la utilización de marcadores microsatélites es que ellos necesitan ser aislados para la mayoría de las especies a ser examinadas por primera vez. Sin embargo, la presencia de regiones flanqueadoras altamente conservadas han sido relatadas para algunos locus microsatélites en ballenas (Schlotterer, et al., 1991), tortugas (Fitzsimmons, et al., 1995) y peces (Rico et al., 1996). Con relación a estas amplificaciones heterólogas Barbará et al., (2007), en un análisis de trabajos publicados que reportan amplificaciones heterólogas, mencionan que la transferibilidad de los marcadores microsatélites entre especies es desigualmente distribuido entre los taxones. El éxito de la transferibilidad es evaluado en términos de su polimorfismo. Pruebas positivas de transferibilidad de marcadores polimórficos pueden ser esperados para muchos grupos de animales entre y dentro géneros y hasta mismo entre diferentes familia (> 40% éxito de transferencia en mamíferos, > 25% en peces, y > 10% en aves a este nivel). Zane, et al., (2002) mencionan que es posible obtener un 50% de éxito en La tasa de amplificación heteróloga para la detección de polimorfismo en especies que divergieron de 10 a 20 millones de años atrás.
Teniendo en cuenta lo anterior, como no existen marcadores microsatélites aislados para el Pataló, en el presente estudio se utilizaron marcadores microsatélites aislados para Prochilodus sp, especies que pertenecen a la misma familia, esperando generar herramientas que permitan asegurar un mejor manejo de esta especie contribuyendo de manera efectiva para su mantenimiento y conservación.
Materiales y métodos
Toma de muestras
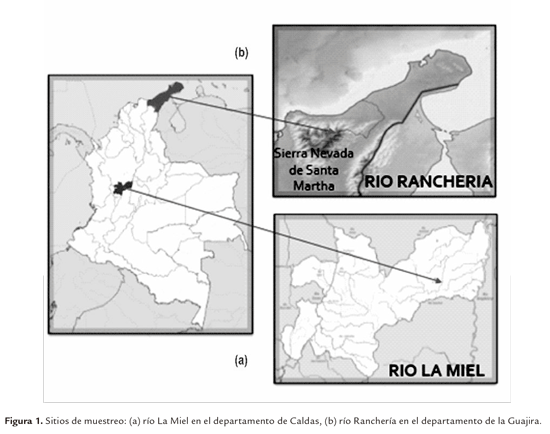
Para la caracterización genético-poblacional se tomó una muestra de tejido muscular de 30 ejemplares de Pataló del río Ranchería obtenidas de la faena diaria de los pescadores de la región (figura1). El tejido muscular se almacenó en etanol al 96% y fue conservado a una temperatura de aproximadamente -32°C. Por otro la lado, las muestras con los respectivos datos estadísticos del río La Miel fueron proporcionadas por el proyecto "Evaluación del estado del Pataló (Ichthyoelephas longirostris) en el embalse Amaní" AUNAP - Fundación Humedales. CONVENIO AUNAP- HUMEDALES N° 040 del 2013.
Extracción y cuantificación del ADN
El ADN fue extraído en el laboratorio de "Biodiversidad Molecular y Citogenética" de la Facultad de Ciencias Exactas y Naturales de la Universidad Surcolombiana. Se utilizó de 50 a 100 mg de tejido muscular, debidamente conservado en etanol al 96% usando el Kit comercial Wizard SV Genomic ADN Purification System de Promega® según recomendación del fabricante. La calidad del ADN de las muestras fue verificada mediante la técnica de electroforesis en gel de agarosa al 1% sumergido en tampón Tris-Borato-EDTA (1x). La cuantificación del ADN se realizó con la ayuda del THERMO A CIENTIFIC NANODROP 2000 del laboratorio de Inmunología de la Facultad de Salud de la Universidad Surcolombiana.
Amplificación de los microsatélites
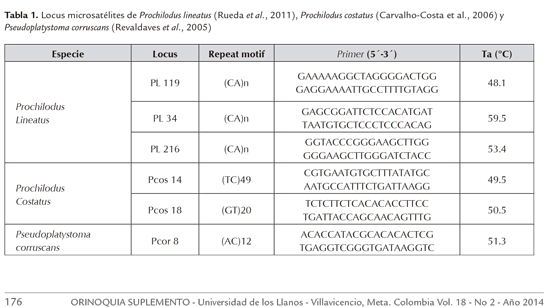
La diversidad genética fue analizada usando 6 loci microsatélites previamente aislados y caracterizados para Prochilodus lineatus por Rueda et al., (2011), Prochilodus costatus por Carvalho-Costa et al., (2006) y Pseudoplatystoma corruscans por Revaldaves et al., (2005) (tabla 1). Los loci microsatélites fueron amplificadas vía PCR con un volumen final de 30 µl conteniendo: 10-50 ng de ADN, tampón 1x (Tris-HCl 200 mM, pH 8,4 y KCl 500 mM), 1.4 - 1.5 mM MgCl2, 10 pmol de cada primer, 0.25 mM de dNTP, y 0.5 U de Taq ADN polimerasa (Invitrogen Life Technologies). Las condiciones de PCR fueron las siguientes: una desnaturalización inicial de 95°C por 5 min, seguido de 35 ciclos de 30s a 94°C, 30s a temperatura de annealing, 30s a 72°C; y una extensión final de 72°C por 20 min. Las reacciones fueron corridas en un termociclador MultiGene OptiMax Thermal Cycler. Las reacciones de genotipage fueron realizadas en el instituto de Genética de la Universidad de Antioquia utilizando un analizador ABI 3100.
Análisis estadísticos
Con el fin de evaluar la variabilidad genética se calcularon datos sobre el equilibrio de Hardy-Weinberg para cada locus o globalmente con la población como un todo utilizando el test de probabilidad; también se calcularon las frecuencias alélicas, además de los valores no embiesados de la heterozigosidad esperada y observada (Nei 1987), utilizando el programa GENEPOP 3.4. (Raymond y Rousset 1995). Las estimativas de riqueza alélica (Petit et al., 1998), los valores de diversidad génica (Nei, 1987), así como la presencia de desequilibrio de ligación entre los locus (para cada par de poblaciones o globalmente) fueron obtenidos por medio del programa FSTAT (Goudet, 2001).
En todos los análisis múltiples efectuados se consideró la corrección secuencial de Bonferroni (Rice, 1989). El grado de subdivisión poblacional fue probado por los índices de fijación de Wright (1978) Fis para cada locus, con la utilización del programa FSTAT 2.9.1 (Goudet, 2001). La proporción de valores de Fis presentes en esta distribución que sean mayores que el valor observado (PL) nos indica si hay o no un déficit de heterozigotos.
Al contrario, la proporción de valores de Fis menores que el valor observado (PS) indica si hay exceso de heterocigotos. Estos valores son indicativos del grado de endogamia de la población.
La diferenciación genética entre las poblaciones fue estimada por medio del Fst (Weir y Cockerham, 1984) que calcula las diferencias poblacionales en comparaciones par-par utilizando la estimativa del valor de theta (θ). Tal programa se basa en el modelo de alelos infinitos (Infinite Alleles Mutation) (Kimura y Crow, 1964). La significancia de los valores encontrados se evaluó por medio de las aleatorizaciones implementadas en el propio programa.
Resultados
Los resultados fueron analizadas por poblaciones de Pataló, los cuales se agruparon por lugar de origen de captura; siendo estas poblaciones originarias del río La Miel y Ranchería.
Caracterización intrapoblacional de la población del río La Miel
En los individuos del río La Miel los microsatélites analizados presentaron un elevado polimorfismo. El número de alelos varió entre 12 (P119) y 4 (Pcos18). Para todos los individuos fue obtenido un total de 44 alelos (media de 7.3 alelos/locus). La heterozigosidad esperada (He) varió de 0.2425 (PL34) a 0.8542 (PL119) con una media de 0.6182 para toda la población y la heterozigosidad observada (Ho) varió de 0.1111 (Pl34) a 1.000 (Pcos18) con una media general de 0.6812.
El índice de endogamia Fis evidenció un pequeño exceso de heterocigotos dado por su valor medio negativo, sin embargo, no se encontraron diferencias significativas lo cual fue verificado por el valor de PL para un nivel de significancia de 95%. La riqueza alélica (Ra) varió de 3.194 (Pl34) a 8.199 (Pl119) con media de 5.028 alelos. La diversidad génica (DG) encontrada estuvo entre 0.245 (Pl34) a 0.852 (Pl119) (tabla 2).
Caracterización intrapoblacional de la población del río Ranchería
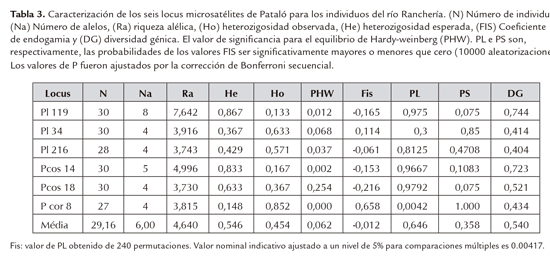
En los individuos del río Ranchería los microsatélites analizados presentaron un elevado polimorfismo. El número de alelos varió entre 8 (P119) y 4 (Pcos18, PL34, Pl216, Pcor8). Para todos los individuos fue obtenido un total de 29 alelos (media de 6.00 alelos/locus). La heterozigosidad esperada (He) varió de 0.148 (Pcor8) a 0.867 (PL119) con una media de 0.546 para toda la población y a heterozigosidad observada (Ho) vario de 0.133 (Pl119) a 0.852 (Pcos18) con una media general de 0.454.
El índice de endogamia Fis evidenció un pequeño exceso de heterocigotos dado por su valor medio negativo, sin embargo, no se encontraron diferencias significativas lo cual fue verificado por el valor de PL para un nivel de significancia de 95%. La riqueza alélica (Ra) varió de 3.730 (Pcos18) a 7.642 (Pl119) con media de 4.640 alelos. La diversidad génica (DG) encontrada estuvo entre 0.404 (Pl216) a 0.744 (Pl119) (tabla 3).
Caracterización interpoblacional
Los valores de diferenciación génica para las dos poblaciones mostraron que existe una diferenciación
moderada entre ellas. Iguales resultaros fueron encontrados para los valores de diferenciación genotípica. Fue observada una moderada diferenciación genética cuando las poblaciones fueron comparadas mediante le índice de fijación Fst (Weir e Cockerham, 1984) calculado mediante comparaciones par-par, el cual fue significativo para un nivel de probabilidad de 5% (Tabla 4). Estos resultados del Fst muestran que existen diferencias poblacionales entre las poblaciones.
Discusión
Los locus microsatélites caracterizados y utilizados en este trabajo, fueron eficientes en la amplificación de las poblaciones de Pataló, mostrando una eficiencia de aproximadamente el 50% en la pruebas de amplificación cruzada. Con estos datos fue posible realizar la caracterización de las diferentes poblaciones estudiadas. Espinosa-León y Carrillo-Ávila (2013), mencionan con relación a las amplificaciones heterólogas, que Bárbara, et al., (2007) reportaban que la transferilidad de los marcadores microsatélites entre especies es desigualmente distribuida entre los taxones y que el alto éxito de la amplificación dentro y entre los géneros, en muchos grupos de animales y plantas, indican un gran potencial para la utilización de microsatélites y sus regiones flanqueadoras, esperándose aproximadamente un 70% de éxito cuando son utilizados, en el caso de los peces, entre especies dentro del mismo género.
Las poblaciones del Pataló del río La Miel y Ranchería presentaron un alto grado de polimorfismo encontrando un número total de alelos de 44 y 29 respectivamente, (media de 7,33 y 6,0 alelos por locus) con heterozigosidades esperadas medias entre 0,6182 y 0,546 para las dos poblaciones. Estos valores encontrados de heterozigosidades medias esperadas son similares a los valores medios de 0,54 heterozigosidades y 7,5 alelos reportados por De Woody e Avise, (2000) para otras especies de peces de agua dulce.
Es importante tener en cuenta que el número de muestras analizadas es pequeña, 28 individuos para el río La Miel y 29 para el río Ranchería, por lo que posiblemente el número de alelos incremente con un mayor número de muestras.
Los resultados obtenidos indican que las dos poblaciones no presentan altos niveles de endogamia lo que se ve reflejado en los valores de Fis encontrados en cada una de las poblaciones analizadas. Saura et al., (2006), mencionan que el monitoreo de la variabilidad genética y de la endogamia es importante para la conservación de la capacidad de adaptación de las especies. Según Wang et al., (2002), un aumento del 10% de la endogamia puede resultar una reducción de la capacidad de supervivencia en alrededor de 3 a 15%. Por otro lado, Cena, et al., (2006), constataron una correlación negativa entre la endogamia y el crecimiento.
La reducción de la variabilidad genética puede promover mayor sensibilidad a las variaciones ambientales y eventualmente puede provocar la extinción de una especie. Por lo tanto, su mantenimiento tiene gran importancia para la conservación de las especies y es necesario para que los individuos enfrenten positivamente las variaciones ambientales.
Las dos poblaciones de Pataló presentaron una desviación del equilibrio de Hardy- Weinberg en el loci Pcos8, aspecto que se ve revelado por de índice de endogamia Fis (Wright, 1978).
En este estudio fue posible detectar una significativa estructuración genética entre las poblaciones cuando fueron considerados todos los diferentes parámetros de la diversidad genética (riqueza alélica, heterozigosidades, diversidades génicas y genotípicas), hecho verificado por el valor de Fst.
El valor encontrado de Fst fue de 0,0525 el cual fue significativo; en donde, según Wright (1978) las dos poblaciones estudiadas presentan una moderada diferenciación genética, es decir que las dos poblaciones analizadas pertenecen a dos grupos diferentes.
El índice Fst además de ser el más utilizado para estimar la distancia genética entre poblaciones de peces (O´Connell y Wright, 1997), es el más indicado en estudios que contengan muestras de tamaño moderado y pequeño (menor que 50) y utilicen menos de 10 locus, y también, cuando el número de migrantes es alto, lo que torna la migración como el factor más importante que las mutaciones (Gaggiotti, et al., 1999).
La moderada estructuración poblacional puede ser explicada debido a la distribución geográfica de las dos cuencas. El río La Miel hace parte única de la cuenca del río Magdalena que nace en la cordillera Central de Los Andes de Colombia en la laguna de La Magdalena, atraviesa gran parte del país hasta desembocar directamente en el mar Caribe. Por otro lado, el río Ranchería nace en la laguna de Chirigua ubicada en la Sierra Nevada de Santa Marta y desemboca en el mismo mar. Al haber una barrera geográfica evidente entre los afluentes hídricos, se impide el flujo génico entre las dos poblaciones.
Sánchez, (2007) menciona que aunque en menor intensidad, casos de estructuración poblacional han sido reportados para peces brasileros de agua dulce. Ejemplos de estructuración son los reportados para las especies Prochilodus argenteus (Hatanaka y Galetti, 2003, Hatanaka, et al., 2006) y Brycon orthotaenia (Wasko y Galetti, 2002) de la cuenca del río São Francisco.
En el caso de la especie Brycon opalinus, endémica de la cuenca del río Paraíba do Sul, análisis de ADN mitocondrial y de marcadores microsatélites revelaron también que existe una diferenciación poblacional en la cuenca (Hilsdorf, et al., 2002; Barroso, et al., 2005).
En un estudio con Brycon hilarii, marcadores RAPD identificaron diferencias significativas entre muestras colectadas en la región del Pantanal brasilero (Sánchez y Galetti, 2006). Sánchez (2007) con marcadores microsatélites encontró diferencias entre por lo menos dos poblaciones de B. hilarii. Benites (2008) identificó una fuerte diferenciación genética entre 11 poblaciones de Pseudoplatystoma corruscans de las cuencas de los ríos Paraná y Paraguay utilizando marcadores microsatélites.
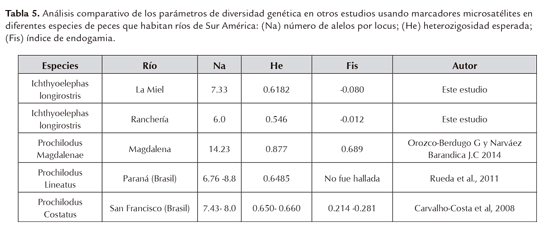
Los resultados aquí obtenidos muestran un buen estado genético de las poblaciones estudiadas, donde a pesar de estar catalogada como amenazada, los valores de su diversidad genética se encontraron similares a los reportados para un grupo de peces pertenecientes a la misma familia; como por ejemplo Prochilodus magdalenae (Orozco-Berdugo G y Narváez Barandica J.C, 2014), Prochilodus lineatus, (Rueda, et al., 2011), Prochilodus costatus (Carvalho-Costa et al., 2008) (tabla 5).
Consideraciones para la conservación del Pataló (Ichthyoelephas longirostris)
Este tipo de trabajos son muy importantes porque ayudan a identificar las unidades básicas representadas por individuos genéticamente más homogéneos que se reproducen aleatoriamente (o próximo a la aleatoriedad) en un área en particular (Laikre, et al., 2005) y ayudan a la implementación de programas de manejo y conservación de cada una de las unidades genéticas identificadas con miras a la conservación de los diferentes hábitats por medio de políticas de preservación de la variabilidad genética como un todo y no solo de las especies vulnerables o en peligro de extinción.
Cualquier acción para recuperar las poblaciones debe considerar métodos científicos de monitoreo, como las metodologías más adecuadas (Povh et al., 2008). Los estudios con marcadores moleculares se pueden utilizar la caracterización genética de las poblaciones nativas de peces y lotes de peces, para identificar especies y para estudiar los efectos de las variaciones ambientales sobre la variabilidad genética.
Agradecimientos
Los autores desean agradecerle a la Vicerrectoría de Investigaciones y Proyección Social de la Universidad Surcolombiana por financiar parte del proyecto. A la AUNAP y a la fundación Humedales que por medio del convenio AUNAP- HUMEDALES N° 040 del 2013 del proyecto "Evaluación del estado del Pataló (Ichthyoelephas longirostris)en el embalse Amaní" proporcionaron los resultados estadísticos de las muestras de Pataló del río La Miel. Gracias al Dr. Carlos Hernández por el apoyo y la orientación.
Referencias
Arango L, Montes-R JM. Caracterización entomológica parcial de la cuenca del río La Miel en el departamento de Caldas (Colombia). Boletín científico centro de museos museo de historian. 2009;13(2):249-268. [ Links ]
Bárbara TC, Palma-Silva GM, Paggi F, Bered MF, Fay, Lexer C. Crossspecies transfer of nuclear microsatellite markers: potential and limitations. Mol Ecol. ;16(18):3759-3767.doi: 10.1111/j.1365294X.2007.03439.x [ Links ] Barroso RM, Hilsdorf AWS, Moreira HLM, Cabello PH, Traub-Cseko YM. Genetic diversity of wild and cultured populations of Brycon opalinus (Cuvier, 1819) (Characiforme, Characidae, Bryconiae) using microsatellites. Aquaculture. ;247:51-65. [ Links ]
Beltrán IC, Estrada M, Valderrama M. 2000. Plan de Ordenamiento: manejo y aprovechamiento sostenible pesquero y acuícola en la cuenca del Río Magdalena. Instituto Nacional de Pesca y Acuicultura- INPA. Santafé de Bogotá, D.C. [ Links ]
Benites C. 2008. Caracterização genética do pintado Pseudoplatystoma corruscans, (Spix & Agassiz, 1829) (Siluriformes: Pimelodidae) da Bacia Paraná-Paraguai, por marcadores moleculares do tipo microsatélite. Tese doutorado, Centro de Aquicultura da Universidade Estadual Paulista. [ Links ]
Castro MC, Vari R. Detritivores of the South American Fish Family Prochilodontidae (Teleostei: Ostariophysi: Characiformes): A Phylogenetic and Revisionary Study. Smith Contr Zool. 2004; 622:1-187 [ Links ]
Carvalho-Costa LF, Hatanaka T, Galetti PM. Isolation and characterization of polymorphic microsatellite markers in the migratory freshwater fish Prochilodus costatus. Mol Ecol Notes. 2006; 6(3):818-819. doi: 10.1111/j.1471-8286.2006.01356.x [ Links ]
Carvalho-Costa LF, Hatanaka T, Galetti PM. Evidence of lack of population substructuring in the Brazilian freshwater fish Prochilodus costatus. GenetiesMol Biol.2008; 31(Suppl 1):377380. [ Links ]
Cena CJ, Morgan GE, Malette MD, Heath DD. Inbreeding, outbreeding and environmental effects on genetic diversity in 46 walleye (Sander vitreus) populations. Mol Ecol. 2006; 15:303-320. [ Links ]
De Woody JA, Avise JC. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. Journal do fish Biology. 2000; 56:461-473. [ Links ]
Espinosa-León LP, Carrillo-Ávila M. 2013. Estado de conservación de la población del bagre rayado Pseudoplatystoma magdaleniatum en la cuenca magdalénica, valoración de implementaciones ambientales y socioeconómicas y definición de escenarios de ordenación pesquera y sostenibilidad. Informe técnico, componente genético. [ Links ]
Fitzsimmons NN, Morits C, Moore SS. Conservation and dynamics of microsatellite loci over 300-million years of marine turtle evolution. Mol Biol Evol. 1995; 12:432-440. [ Links ]
Gaggiotti EO, Lange O, Rassman K, Gliddon C. A comparison of two indirect methods for estimating average levels of gene flow using microsatellite data. Mol Ecol. 1999; 8:1513-1520. [ Links ]
Goudet J. 2001. FSTAT, a program to estimate and test gene diversities and fixation indices (version 2.9.3). Available from http://www.unil.ch/izea/softwares/fstat.html. Updated from Goudet (1995). [ Links ]
Hatanaka T, Galleti PM. RAPD marker indicate the occurrence of structured populations in a migratory fish species. Genet Mol Biol. 2003; 26(1):19-25. [ Links ]
Hatanaka T, Silva FH, Galetti PM. Population substructuring in a migratory freshwater fish Prochilodus argenteus (Characiformes, Prochilodontidae) from the São Francisco River. Genetic. 2006; 126:153-159. [ Links ]
Hilsdorf AW, Azeredo - Espin AM, Krieger MH, Krieger JE. Mitochondrial DNA diversity in wild and cultured populations of Brycon opalinus (curvier, 1819) (Characiformer, Characidae, Bryconinae) form the Paraiba do sul Basin, Brazil. Aquaculture. 2002; 214:81-91. [ Links ]
Kimura M, Crow F. The number of alleles that can be maintained in a finite population. Genetics. 1964; 49:725-738. [ Links ]
Laikre L, Palm S, Ryman N. Genetic population structure of fishes: implications for coastal zone management. Ambio. 2005; 34(2):111-119. [ Links ]
Mojica JI, Castellanos C, Usma S, Álvarez R. 2002. Libro rojo de peces dulceacuícolas de Colombia. La serie de Libros Rojos de Colombia. Instituto de Ciencias Naturales Universidad Nacional de Colombia, Ministerio del Medio Ambiente, Bogotá, Colombia. [ Links ]
Mojica J, Castellanos C, Sánchez P, Díaz C. Peces de la cuenca del rio ranchería, la guajira Colombia. Biota Colombia. ;7(1):129142. [ Links ]
Mojica JI, Castellanos C, Usma S, Álvarez R. 2012. Libro rojo de peces dulceacuícolas de Colombia. La serie de Libros Rojos de Colombia. Instituto de Ciencias Naturales Universidad Nacional de Colombia, Ministerio del Medio Ambiente, Bogotá, Colombia. [ Links ]
Nei M. 1987. Molecular Evolutionary Genetics. Columbia University Press, New York. [ Links ]
O´Connell M, Wright JM. Microsatellite DNA in fishes. Rev Fish Biol Fisher. 1997; 7:331-363. [ Links ]
Orozco-Berdugo G, Narváez- Barandica JC. Genetic diversity and population structure of bocachico Prochilodus magdalenea (Pisces, Prochilontidae) in the Magdalena River basin and its tributaries, Colombia. Genet Mol Biol. 2014; 37(1):37-45. [ Links ]
Patiño A. Cultivo experimental de peces en estanques. Cedespia. 1973; 2(5):75-128. [ Links ]
Petit RJ, El Mousadik A, Pons O. Identifying Populations For Conservation On The Basis Of Genetic Markers. Conserv Biol. 1998; 12:844-855. [ Links ]
Polania J, Orosco C, Ángel L. Delta del rio ranchería (la guaria, Colombia): caudal salinidad y transporte de sólidos y sus posibles influencias sobre la composición y estructura de los manglares. Actual Biol. 2006; 28(84):27-37 [ Links ]
Povh JA, Lopera Barrero NM, Ribeiro RP, Lupchinski E, Gomes PC, López TS. Monitoreo genético en programas de repoblamiento de peces mediante marcadores moleculares. Cien Inv Agr. 2008; 35(1):5-15. [ Links ]
Raymond M, Rousset. GENEPOP (version 1.2): A population genetic software for exact test and ecumenism. J Hered. 1995; 86:248249. [ Links ]
Revaldaves E, Pereira LH, Foresti F, Oliveira C. Isolation and characterization of microsatellite loci in Pseudoplatystoma corruscans (Siluriformes: Pimelodidae) and cross- species amplification. Mol Ecol Notes. 2005; 5:463-465. DOI:10.1111/ j.14718286.2005.00883x [ Links ]
Rice WR. Analyzing tables of statistical tests. Evolution. 1989; 43:223-225. [ Links ]
Rico C, Rico I, Hewitt G. 470 millions years of conservation of microsa-tellite loci among fish species. Proc R Soc Lond B. 1996; 263:549-557. [ Links ]
Román C. 1993. Historia natural del jetudo, Ichthyoelephas longirostris (Steindachner 1989) (Pisces: prochilodontidae) en la cuenca del rio la vieja, alto Magdalena, Colombia. [ Links ]
Roman-Valencia C, Ortiz-Muñoz V. Sobre la reproducción de Ichthyoelephas longirostris (Pisces, Prochilodontidae) en la cuenca del Rio Magdalena, Colombia. Dahlia Revista de Asociacion Colombiana de Ictiologia. 2001; 4:33-35. [ Links ]
Rueda E, Sommer J, Scarabotti P, Markariani R, Ortí G. Isolation and characterization of polymorphic microsatellite loci in the migratory freshwater fish Prochilodus lineatus (Characiformes, Prochilodontidae). Conserv Genet Resource. 2011; 3:681-684. [ Links ]
Sánchez A, Galetti PM. Microsatellite Loci isolated in the Freswater fish Brycon hilarii. Mol Ecol Notes. 2006; 6:1045-1046. [ Links ]
Sánchez A. 2007. Estructura genética poblacional de Brycon hilarii (characidae) da sub-bacia do Rio Miranda, e seu significado para programas de conservação. Tese Doutorado. Universidade Federal de São Carlos. [ Links ]
Saura M, Caballero P, Caballero A, Morán P. Genetic variation in restored Atlantic salmon (Salmo salar L.) populations in the Ulla and Lérez rivers, Galicia, Spain. ICES Journal of Marine Science. 2006; 63:1290-1296. [ Links ]
Schlötterer C, B. Amos y D. Tautz. (1991). Conservation of polymorphic simple sequence loci in cetacean species. Nature, 354:6365. [ Links ]
Schlötterer C, Amos B, Tautz D. 1992. Slippage synthesis of simple sequence DNA. Nuclei Acids Research, 20: 211-215. [ Links ]
Wang S, Hard JJ, Utter F. 2002. Salmonid inbreeding: a review. Reviews in Fisheries Biology and Fisheries, 11:301-319. [ Links ]
Wasko AP, Galetti PM. 2002 RAPD analysis in the Neotropical fish Brycon lundii: Genetic diversity and its implications for the conservation of the species. Hydrobiology, 474:131-137. [ Links ]
Weir BS, Cockerham CC. 1984. Estimating F-statistics for the analysis of population structure. Evolution, 38: 1358-1370. [ Links ]
Wright S.1978. Variability within and among natural populations. v.4. The University of Chicago Press. [ Links ]
Zane LL, Bargelloni, Patarnello T. 2002. Strategies for microsatellite isolation: a review. Molecular Ecology, 11: 1-16. [ Links ]