Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
ORINOQUIA
On-line version ISSN 0121-3709
Orinoquia vol.18 supl.1 Meta Dec. 2014
ARTÍCULO ORIGINAL/ORIGINAL ARTICLE
Variedad bacteriana en cultivos piscícolas y su resistencia a antibacterianos
Bacterial variety of fish farms and resistance to antibacterial
Variedade bacteriana em cultivos piscícolas e sua resistência aos antibacterianos
Maritza Parrado1
María Clemencia Salas2
Gilma Hernández-Arévalo3
Patricia Ortega4
Martha I. Yossa5
1 MVZ, Instituto de Acuicultura de los Llanos-IALL
2 Microbiol, ESP Aquaprimavera Ltda.
3 MVZ, ESP, MSc, Grupo de investigación en alimentación y nutrición de organismos acuáticos GRANAC
4 Zootec
5 Zootec, MSc, Dr, Universidad de los Llanos, km 12 vía Puerto López, Villavicencio, Meta Email: granac.iall@gmail.com
Recibido: octubre 30 de 2014. Aceptado: noviembre 24 de 2014
Resumen
Para determinar la variedad y resistencia frente a antibacterianos, durante un periodo productivo en el cual no se utilizaron antibióticos, en dos piscícolas del departamento del Meta, se obtuvieron muestras de agua y sedimento de donde se aislaron bacterias pertenecientes al grupo de heterótrofas Gram negativas. Las muestras se sembraron en medios selectivos Agar TCBS, McConkey, Citófaga, Cetrimide y Plate Count Agar para hacer recuentos bacterianos con las Técnicas de Análisis Microbiológico de la ICMSF y el Standart Methods. Las colonias diferentes de cada medio se repicaron para identificación bioquímica con kit comercial BBL-cristal®, y para el estudio de resistencia a seis antibacterianos se empleó el procedimiento descrito por Kirby-Bauer. La multi-resistencia, se estimó con el índice resistencia múltiple a antibióticos (MAR), y el índice de abundancia relativa (IAR) se calculó de acuerdo con Latorre (2007). Se identificaron 19 cepas bacterianas, seis correspondieron a Klebsiella pneumoniae, cinco a Enterobacter sp y cloacae, y las demás a Pseudomonas fluorescens y aerouginosa, Chryseobacterium sp. y gleum, Sphingobacterium multivorum, Serratia marcescens y Tetrathiobacter mimigardefordensis. Todas las cepas aisladas fueron resistentes a Eritromicina y el mayor índice de multi- resistencia lo presentó Enterobacter cloacae con 1.00 y 0.8. El mayor IAR correspondió a enterobacterias (≥800ufc/g). El encontrar seis cepas de Klebsiella pneumonidae y cinco de Enterobacter spcondiferentes índices de resistencia en los dos ambientes analizados, indica la gran variabilidad de estas especies. La resistencia de todos los microorganismos a Eritromicina, y el alto IMR de Enterobacter, demuestran que el uso de antibióticos en acuicultura, sumado a la alta concentración de enterobacterias puede tener efectos ambientales adversos que podrían repercutir en la salud pública.
Palabras clave: cepas bacterianas, estanques, microorganismos, MAR, IAR.
Abstract
Bacterial aerobic gram negative heterotrophic community was isolated from water and sediment to determine the variety and antibacterial resistance during a productive period in which no antibiotics were used in two fish farms of Meta country. Samples were plated on selective medium TCBS Agar, McConkey, Citófaga, Cetrimide and Plate Count Agar toperform bacterial counts with the Technical Analysis of Microbiological ICMSF and the Standard Methods. The different colonies were replicated to each medium biochemical identification BBL-Cristal® commercial kit, and for the study of the six antibacterial resistance was used the procedure described by Kirby -Bauer. The multi - resistance was estimated by the multiple antibiotic resistance index (MAR) and the relative abundance index (RAI) was calculated according to Latorre (2007).19 bacterial strains were identified, six were for Klebsiella pneumoniaeand five Enterobacter sp cloacae, and others: aerouginosa Pseudomonas and fluorescens, Chryseobacterium sp. and gleum, Sphingobacterium multivorum, Serratia marcescens and Tetrathiobacter mimigardefordensis. All isolates were resistant to erythromycin and the highest rate of multi- resistant Enterobacter cloacae presented him with 1.00 and 0.8. The biggestRAI accounted enterobacteria (≥800 cfu/g). Finding six strains of Klebsiella pneumonidae five Enterobacter sp with different resistance rates in both environments analyzed, indicates the great variability of these species. The resistance of all microorganisms to erythromycin, and high MAR Enterobacter show that the use of antibiotics in aquaculture coupled with the high concentration of enterobacter may have adverse environmental impacts that could affect public health.
Key words: bacterial strains, ponds fish, microorganismos, MAR, RAI.
Resumo
A comunidade bacteriana heterótrofa aeróbica gram negativa, foi isolada de água e sedimento para determinar a variedade resistência a antibacteriano se em duas granjas piscícolas do estado do Meta, durante um período produtivo no qual não foram utilizados antibióticos. As amostras de água e sedimento foram sermeadas em meios seletivos de Agar TCBS, McConkey, Citófaga, Cetrimide e Plate Count Agar para fazer as contagens bacterianas com as Técnicas de Análises Microbiológico da ICMSF e o Standard Methods. As colônias diferentes de cada meio foram replicadas para identificação bioquímica com kit comercial BBL-cristal®, e para o estudo da resistência os seis antibacterianos foi empregado o procedimento descrito por Kirby-Bauer. A multirresistência, foi estimada pelo Índice resistência múltipla a antibióticos (IMAR) e o Índice de abundância relativa (IAR), foi calculado de acordo com Latorre (2007). Foram identificadas 19 cepas bacterianas, seis corresponderam a Klebsiella pneumoniae, cinco a Enterobacter sp y cloacae, e as demais a Pseudomonasfluorescens e aerouginosa, Chryseobacterium sp. e gleum, Sphingobacterium multivorum, Serratia marcescens e Tetrathiobacter mimigardefordensis. Todas as cepas isoladas foram resistentes a Eritromicina e o maior índice de multirresistência apresentou Enterobacter cloacae com 1.00 e 0.8. O maior IAR correspondeu às enterobacterias (≥800 ufc/g). As seis cepas de Klebsiella pneumonidae e cinco de Enterobacter sp comdiferentes índices de resistência encontrada em todos os ambientes analisados, indica a grande variabilidade destas espécies. A resistência de todos os micro-organismos a Eritromicina, e o alto IMR de Enterobacter demostram que o uso de antibióticos na aquicultura aliado a alta concentração de enterobacterias pode ter efeitos ambientais adversos e repercutir na saúde pública.
Palavras chave: cepas bacterianas, viveiros, micro-organismos, MAR, IAR.
Introducción
En los ambientes acuáticos, la variedad bacteriana juega un papel importante en la descomposición de la materia orgánica (MO) y el reciclaje de nutrientes (Tšertova et al., 2013); sin embargo, la acuicultura intensiva ha generado ambientes artificiales que involucran la adición de abonos orgánicos, disminución en las tasas de recambio de agua, aumento en la densidad de siembra y administración de altas cantidades de alimento; factores que influyen en la formación de MO que se acumula en el sedimento, favoreciendo así, el crecimiento de una abundante variedad microbiana tanto patógena como oportunista (Gastalho et al., 2014; Negrete et al., 2004; Ranjard et al., 2000). Esta dinámica poblacional puede ser alterada por el suministro de altas cantidades de antibióticos al agua (Martínez, 2008).
Estudios han demostrado la presencia de patógenos bacterianos en peces incluyendo Aeromonas hydrophila, A. salmonicida, Edwarsiella tarda, Pasteurella piscicida, Vibrio alguillarum y Yersinia ruckeri, entre otras (Jha et al., 2008; Sarter, 2007; Hatha, 2005).
En Colombia, según Eslava (2008), se han presentado brotes por Flavobacterium columnare, Aeromona hydrophila y Edwardsiella tarda; Iregui et al., (2004) reportó Streptococcus sp en tilapia. Igualmente, Hernández et al., (2009) reportaron Streptococcus sp en tilapias con pesos entre 20 y 150 g, Penagos et al., (2008) reportaron Mycobacterium sp en peces ornamentales, mencionando, además, que la deficiencia en el diagnóstico y las prácticas inapropiadas de manejo de los peces ponen en riesgo la salud pública.
Desde los años 70, en granjas piscícolas se vienen usando antimicrobianos (químicos y antibióticos) sin restricción, para controlar la proliferación de microorganismos y tratar infecciones producidas por patógenos bacterianos de peces, incluyendo aquellos no biodegradables que persisten en el ambiente acuático. Además, se utilizan como promotores de crecimiento incorporados en el alimento o administrados directamente en el agua, generando una carga alta de residuos en el sistema productivo (Petersen et al., 2002). Uno de los primeros en utilizarse fue sulfonamida, que se le adicionó trimetoprim a finales de los años 80 (Negrete et al., 2004; Lunden y Bylund, 2000). Entre los antibióticos más empleados en acuicultura está la oxitetraciclina, que persiste en el sedimento de 50 a 64 días después de haber sido suministrado con el alimento (Luna et al., 2003); sus residuos se han encontrado en sedimento superficial y sub-superficial a una distancia de hasta 30 metros, en cultivos en jaulas (Capone et al., 1996). Eso puede afectar el crecimiento y la supervivencia del erizo de mar al destruir bacterias importantes en el metabolismo del nitrógeno (Campbell et al., 2001).
Tales prácticas en el ambiente acuícola han generado un medio para la selección de especies bacterianas resistentes a antimicrobianos. Aquello posibilita una alta frecuencia de intercambio de elementos genéticos movibles, codificadores de la resistencia, entre ellos plásmidos R conjugativos, integrones y cassettes, considerados como potenciales donadores (Ishida et al., 2010). También se ha incrementado el número de bacterias resistentes a antibióticos, lo que obliga a plantear estrategias claras y precisas para minimizar la resistencia bacteriana (Alanis, 2006; Negrete et al., 2004; Fernández et al., 2003).
Variados estudios en peces y ambientes acuáticos en India, Vietnam, Egipto, Argentina, Chile y otros países, han corroborado la presencia de bacterias resistentes a múltiples antibióticos como cloranfenicol, polimyxina B, oxitetraciclina, ciprofloxacina, streptomicina, gentamicina, amoxicilina, ampicilina, novoviocina, nitrofurantoína, trimetoprim-sulfa y ácido nalidixico (Ishida et al., 2010; Sarter et al., 2007; Hathaa et al., 2005; Lösch et al., 2005; Miranda et al., 2003; Teuber, 2001). En la actualidad, el mayor problema es la resistencia de un mismo microorganismo a múltiples medicamentos y la transferencia horizontal de resistencia entre diferentes especies bacterianas, lo que dificulta el tratamiento de enfermedades infecciosas. Este trabajo contribuye al conocimiento de la diversidad de especies bacterianas cultivables en sistemas piscícolas y la resistencia a seis de los antibióticos utilizados, para establecer su grado de resistencia o multi-resistencia.
Materiales y métodos
El estudio se hizo el primer semestre del 2011 en dos piscícolas. Una comercial (granja A), en el municipio de Castilla la Nueva (Meta), con manejo de aguas verdes, recirculación, aplicación de probióticos y aireación. Otra psicícola experimental (granja B), en el Instituto de Acuicultura de los Llanos de la Universidad de los Llanos, en Villavicencio, con agua proveniente de pozo profundo, sin recirculación ni suministro de aditivos. En cada granja fueron seleccionados al azar, tres estanques donde se hicieron tres muestreos bimensuales en tres puntos de cada uno (entrada, centro y salida).
Muestras de agua y sedimento
Las muestras de agua se recolectaron en frascos estériles de 100 ml a una profundidad de 15 cm. Las muestras de sedimento se recolectaron desde un bote con un tubo PVC estéril de 15 cm de largo y 1.5 cm de diámetro, acoplado a un tubo de mayor longitud para llegar hasta el fondo del estanque. Las muestras se trabajaron según la metodología adaptada de la International Commission on Microbiological Specifications for Foods (ICMSF, 2002) y el Standard Methods (2005), haciendo diluciones (103 y 104) en agua peptonada estéril (APE). Se sembraron alícuotas de 0.1 ml de las diferentes diluciones en los medios selectivos Agar Cetrimide, McConkey, TCBS y Citófaga. Las diluciones del sedimento se hicieron a partir de 1 g de muestra.
Recuento total de microorganismos mesófilos aerobios
Para el recuento total de mesófilos aerobios, de las mismas diluciones de agua y sedimento, se sembraron alícuotas en Plate Count Agar. Se incubaron a 30°C durante 48 horas, para el recuento de las colonias que se reportó como UFC por unidad de muestra, siguiendo las Técnicas de Análisis Microbiológico de la ICMSF (2002) y el Standard Methods (2005).
Aislamiento e identificación de las colonias
Las colonias aisladas se clasificaron de acuerdo con la forma característica, tamaño, estructura, superficie, borde, color y opacidad; a las que aparecieron diferentes en cada uno de los medios, se procedió a identificar la morfología por tinción con Gram y se hicieron pruebas de reacción, oxidasa e indol. Una vez identificadas se repicaron en medio selectivo agar McConkey, se incubaron a 30°C durante 24 horas, obteniendo así los cultivos de las colonias para las pruebas bioquímicas con kit comercial (BBL-cristal®); como muestra control se empleó E. coli ATCC 25922.
Prueba de sensibilidad antibacteriana
Se empleó el método descrito por Kirby-Bauer (1966), se hizo suspensión de bacterias en solución salina fisiológica 0.85%, patrón de turbidez equivalente a 0.5 de la escala de Mac-Farland; se sembró en medio Mueller-Hilton y se utilizaron sensidiscos comerciales (OXOID® y BBL®) de oxitetraciclina (OT), trimetoprimsulfametoxisol (SXT), nitrofurantoína (F/M), ciprofloxacina (CIP), eritromicina (E) y ácido nalidixico (NA),(CLSI 2010).
Los resultados se determinaron midiendo el diámetro del halo de inhibición de crecimiento y se compararon con los referentes establecidos para cada antibiótico, de acuerdo con los estándares internacionales (CLSI 2010). Para muestra control se empleó E. coli ATCC 25922 y se determinó la multi-resistencia, utilizando el índice de resistencia múltiple a antibióticos (MAR) desarrollando la siguiente fórmula:
Índice MAR = a/b
Donde:
a = número de antibióticos a los que fue resistente b = número de antibióticos a los que se expone
Índice de Abundancia relativa
Una vez cuantificadas las bacterias en los medios selectivos, se determinó el índice de abundancia relativa (IAR), de acuerdo a lo señalado por Latorre (2007), empleando la expresión:
IAR = 100 * Log10n/Log10N
Dónde:
n = conteo en el medio de cultivo
N = recuento total
Para este análisis, se descartaron las muestras con límite de cuantificación <10 UFC/ml y >4000 UFC/ml.
Análisis de resultados
Para el análisis de recuentos bacterianos y en virtud de la cantidad de datos fuera del límite de cuantificación, se optó por un análisis de frecuencias relativas. Para cada medio de cultivo y muestra (agua-sedimento) se establecieron 4 escalas de valores, se determinó el número de conteos bacterianos perteneciente a cada escala y dicha frecuencia se expresó en términos relativos. El análisis se desarrolló atendiendo a las variables de clasificación de mayor interés (granja, estanque, sitio de muestreo). El establecimiento de los rangos propendió por reflejar la distribución de los datos, incluyendo categorías para los datos inferiores y superiores al límite de cuantificación. Para el caso del recuento total en sedimento, esta variable fue analizada mediante ANDEVA, previa transformación (Log 10); se empleó la prueba de comparación de medias de Tukey (α = 0.05).
Resultados Recuento Bacteriano
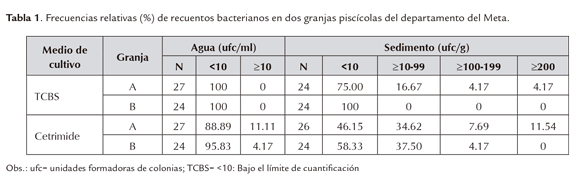
En la tabla 1, se observa que en Agar -Tiosulfato-Citrato-Sales biliares-Sacarosa (TCBS), selectivo para Vibrio, el 100% de las muestras de agua analizadas en las dos granjas presentaron valores inferiores a 10ufc/ ml. Luego de la identificación bioquímica en sedimento, las pocas colonias encontradas correspondieron a Klebsiella neumoníae. Entre tanto, en Agar Cetrimide, selectivo para Pseudomonas, los resultados en agua determinaron que la mayor proporción de las muestras están por debajo del límite de cuantificación (≤10 ufc/ml), para las dos granjas. De manera similar, una alta proporción de las muestras en las dos granjas se ubicaron por debajo del límite de cuantificación en sedimento; sin embargo, la distribución incluyó muestras en la categoría ≥200 ufc/mg, en la granja A.
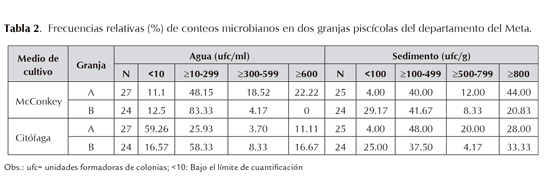
En Agar McConkey, selectivo para enterobacterias, se obtuvieron los mayores recuentos en las dos granjas (≥600 ufc/ml en agua e incontables en sedimento, ≥800 ufc/ml). Ver tabla 2. Sin embargo, la distribución diferencial evidencia que la granja A tiene los mayores recuentos en agua y sedimento. En el medio de cultivo Citofaga, selectivo para Aeromonas y Flavobacterium, la mayor proporción de las muestras 25.93 y 58.33% estuvieron en el rango (≥10-299 ufc/ml), con los mayores recuentos, tanto en este rango como en los máximos en la granja B (Tabla 2).
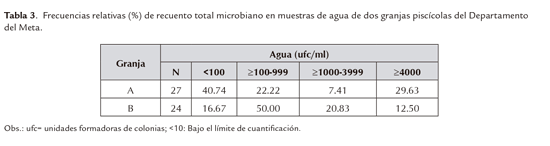
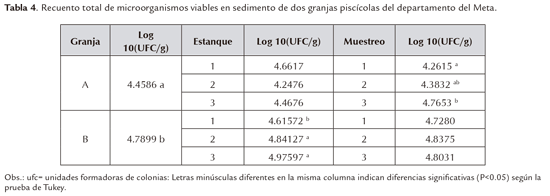
Los resultados del recuento total se presentan en las tablas 3 y 4. En agua, el rango máximo de conteo se presentó en la granja A, con 29.63%. En sedimento, sitio donde se obtuvo el mayor número de organismos viables, se observó mayor concentración de bacterias en la granja B. Con este hallazgo se procedió a valorar si al interior de cada granja se presentaron diferencias entre los estanques muestreados, lo cual fue verificado en la granja B, donde el estanque 1 fue significativamente diferente (P≤0.05) del 2 y 3 (Tabla 4). En cuanto a muestreos, en la granja A, la concentración de bacterias creció progresivamente, siendo mayor en el muestreo 3, con diferencia significativa P<0.05 (Tabla 4).
Bacterias identificadas
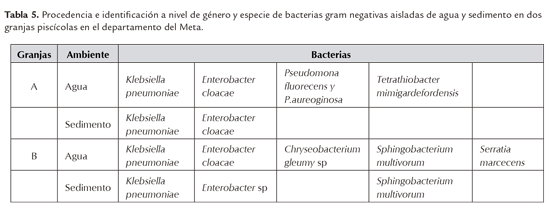
Fueron encontradas 18 cepas (Tabla 5) y se presentó mayor diversidad en agua que en sedimento.
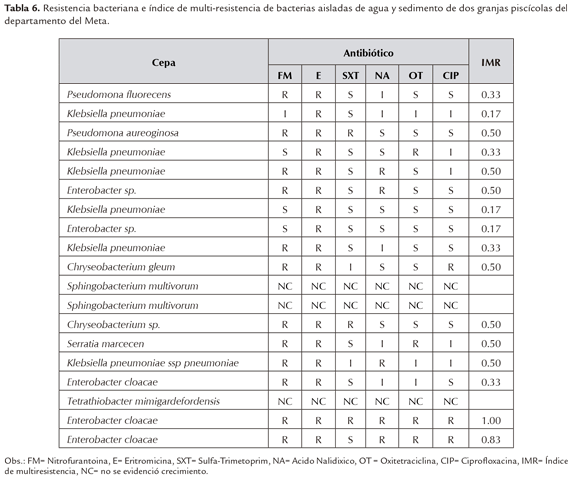
Resistencia Bacteriana
Todas las especies evaluadas fueron resistentes a eritromicina, seguida por nitrofurantoína. La menor resistencia se presentó a sulfa-trimetoprim y ciprofloxacina. Igualmente, se registraron diferentes niveles de resistencia a los antibióticos evaluados. El mayor IMR se presentó en las especies de Enterobacter cloacae (1.00 y 0.83).
Índice deAbundancia Relativa
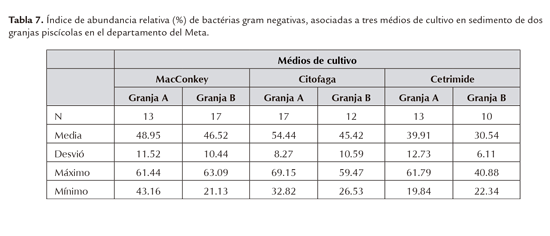
El mayor IAR en sedimento correspondió, en su orden, a flavobacteriaceas, enterobacterias y pseudomonaceas (Tabla 6); mientras que en agua, el mayor IAR correspondió a enterobacterias (79.66±20.39 y 67.89±25.93) en las granjas A y B respectivamente, seguido de flavobacterias (54.32±13.51 y 63.56±17.65). El mayor y menor IAR en sedimento se dio en el medio de cultivo Citófaga y Cetrimide para las granjas A y B, respectivamente (Tabla 7).
Discusión
Los recuentos en medios selectivos indicaron que la mayor concentración de bacterias en el ambiente piscícola (agua y sedimento) corresponde a enterobacterias y puede deberse al sistema de manejo que se da a los estanques al abonarlos con heces de animales, además de las excretas de los mismos peces y la carga bacteriana que pueda traer los afluentes; la materia fecal es una de las formas de contaminación alimentaria, debido a la facilidad con que los alimentos la pueden adquirir (Arias y Antillón 2000). Marin et al., (2009), al analizar tilapia fresca en Costa Rica, encontraron que el 74 y 58% de las muestras superaron los valores permitidos de coliformes totales y fecales respectivamente.
En este estudio se encontró que el recuento total de microorganismos viables fue menor en agua que en sedimento en las dos granjas e igual a lo reportado por Øvreås et al., (2003). En todos los medios, el mayor porcentaje de conteos máximos se presentaron en la granja A y hubo además diferencia significativa P≤0.05 entre muestreos, lo que refleja aumento respecto al tiempo. Estos resultados se deben principalmente a factores de manejo (utilización de aireadores, alta densidad de siembra, administración de probióticos, además de los factores ambientales), que crean un hábitat definido para la selección de bacterias. La granja B solo presentó recuentos mayores a los de la granja A en sedimento. Posiblemente, esto se debió a la cantidad de materia orgánica sedimentada, pues no tener prácticas periódicas de manejo disminuye la dinámica del ambiente, y crea un nicho para mayor presencia de patógenos u oportunistas; quizá por la misma razón no hubo diferencia significativa entre muestreos.
Respecto a la identificación y resistencia a antimicrobianos, encontrar cinco cepas de Klebsiella pneumonidaee, igual número de Enterobacter sp con diferentes índices de resistencia y en todos los ambientes analizados, demuestra la gran variabilidad de estas especies. Esta variabilidad puede ser reflejo de los cambios en los menores (Martínez, 2008, Calderón et al., 2003). Estudios moleculares han reportado múltiples genes de resistencia a varios antibióticos. Hong-Xia et al., (2014) encontraron multi-resistencia en cepas de Salmonella sp, y Zhang et al., (2013) en cepas de E. Coli; ambos estudios hechos en acuicultura China. RodríguezMartínez et al., (2012) describió la presencia de los genes de resistencia qnrS2 y blaVIM-1 en dos aislados diferentes de K. pneumoniae y K. Oxitoca, en un mismo plásmido transferible.Grace et al., (2013) encontraron un complejo de integrones en plásmidos de multi-resistencia en Citrobacter freundii y sugieren que los genes de resistencia de los organismos patógenos se pueden transferir a bacterias nativas, contribuyendo así al pool ambiental de resistencia a antibióticos.
Una especie de Enterobacter cloacae fue la que tuvo mayor índice de multi-resistencia. La presenta ante los seis antibióticos evaluados y todos los microorganismos aislados fueron resistentes a eritromicina. Esto corrobora el inminente riesgo asociado al uso de antibióticos en la producción animal, lo que ha obligado a implementar sistemas de regulación más estrictos sobre el uso profiláctico de antibióticos y la presencia de residuos en productos de acuicultura (Defroirdt et al., 2011). Igual de preocupante resultó la multi-resistencia de todos los aislados; y aunque ahora último se han evaluado varias alternativas de biocontrol, esos métodos todavía están en fase investigativa para la piscicultura (Defroirdt et al., 2011). Por otro lado, la presencia de multi-resistencia bacteriana en alimentos de origen animal en países exportadores sugiere la transferencia de un país a otro (Hong-Xia et al., 2014).
De acuerdo con el IAR, el mayor porcentaje de bacterias en sedimento se presentó en el medio citófaga (flavobacterias y aeromonas) en las dos granjas, significando un riesgo para la salud de los peces. Pulkkinen et al., (2010) reportaron un incremento en la ocurrencia de la enfermedad producida por Flavobacterium columnaris en alevinos de salmón, además de cambios evolutivos en la virulencia, que incrementan la severidad de los síntomas. Tamibién sugieren que las granjas de peces proveen un ambiente que promueve la circulación de cepas más virulentas, por la intensificación de los cultivos, sumado al aumento de la temperatura. Igual reporte hizo Pridgeon y Klesius (2011) al analizar tres aislados de Aeromona hydrophila en bagre de canal en Estados Unidos. El IAR de enterobacterias (McConkey) fue mayor en agua y concuerda con lo reportado por Lima et al., (2006), quienes aislaron estos microorganismos en mayor proporción de contenido estomacal, superficie de los peces, filetes frescos y congelados de tilapia, agua y ración. En sedimento también se presentó un alto porcentaje corroborando los resultados de los recuentos diferenciales.
Conclusión
El estudio reflejó la variedad y variabilidad de los microorganismos, la alta concentración de enterobacterias en aguas productivas, la resistencia de todos los microorganismos aislados a eritromicina y un alto índice de resistencia múltiple a antibióticos por parte de Enterobacter sp y las demás bacterias. El uso de antibióticos en acuicultura tiene efectos adversos que repercuten en el medio ambiente, en la salud de los peces y de los seres humanos.
Agradecimientos
Al Ministerio de Agricultura y Medio Ambiente, por financiar el proyecto Dinámica de nutrientes, convenio 057/2007; Universidad de los Llanos Orientales y Aquaprimavera Ltda, por la Cofinanciación y apoyo logístico.
Referencias
Alanis A. Resistance to Antibiotics Are We in the Post-Antibiotic Era?. Arch Med Res. 2006; 36: 697-705 [ Links ]
Arias ML, Antillón F. Contaminación microbiológica de los alimentos en Costa Rica. Una revisión de 10 años. Rev Biomed. 2000;11: 113-122 [ Links ]
Calderón R, Sacsaquispe R, Pasterán FG, Galas M, Soto J, Riveros J, Valencia A, Silva A, Suárez M, Montoya Y. Caracterización molecular de Klebsiella pneumoniae y Enterobacter cloacae productoras de ß-lactamasas de espectro extendido tipo SHV-5 en una unidad de cuidados intensivos neonatal de Lima. Rev Peru med exp salud pública. 2003; 20:121-127. [ Links ]
Campbell D, Pantazis P, Kelly M. Impact and residence time of oxytetracycline in the sea urchin, Psammechinus miliaris, a potential aquaculture specie. Aquaculture. 2001;202:73-87. [ Links ]
Capone D, Weston D, Miller V, Shoemaker C. Antibacterial residues in marine sediments and invertebrates following chemotherapy in aquaculture. Aquaculture. 1996;145:55-75. [ Links ]
Defoirdt T, Sorgeloos P, Bossier P. Alternatives to antibiotics for the control of bacterial disease in aquaculture. Current Opinion in Microbiology. 2011;14: 227-372. [ Links ]
Eaton AD, Clesceri LS, Greenberg AE. 2005. Standard methods for the examination of water and wastewater. ed. American Public Health Association. Washington, DC. [ Links ]
Eslava Mocha PR. Una aproximación sistémica a las enfermedades de peces dulceacuícolas de Colombia, XIV jornada de Acuicultura- Instituto de Acuicultura Universidad de los LLanos (IALL). 2008;14:16-33. [ Links ]
Fernández F, López H, Ponce M, Machado B. Resistencia bacteriana. Rev cubana medmilit. 2003;32:44-48. [ Links ]
Gastalho S, da Silva G, Ramos F. Uso de antibióticos em aquacultura e resistência bacteriana: impacto em saúde pública. Acta Farmacêutica Portuguesa. 2014;3:29-45. [ Links ]
Grace Y, Waldan K, Julian D, Vivian M. Complex integrons containing qnrB4-ampC (blaDHA-1) in plasmids of multidrug-resistant Citrobacter freundii from wastewate, Can J Microbiol. 2013;59:110-116. [ Links ]
Hathaa M, Vivekanandhanb G, Christol A. Antibiotic resistance pattern of motile aeromonads from farm raised fresh water fish. Int J Food Microbiol. 2005;98: 131-134. [ Links ]
Hernández E, Figueroa J, Iregui C. Streptococcosis on a red tilapia, Oreochromis sp., farm: a case study. J Fish Dis. 2009;32:247- 252. [ Links ]
Hong-Xia Jiang,Li Song, Ji Liu, Xiao-Hua Zhang, Yan-Na Ren, WenHui Zhang, Jing-Yuan Zhang, Ya-Hong Liu, Mark A Webber, David O O, Zhen-Ling Zeng, Piddock JV.Multiple transmissible genes encoding fluoroquinolone and third-generation cephalosporin resistance co-located in non-typhoidal Salmonella isolated from food-producing animals in China. Int J Antimicrob. 2014;43:242-247. [ Links ]
International Commission on Microbiological Specifications for Foods ICMSF. 2002. Microorganisms in Foods, Microbiological Testing in Food Safety Management. 7 ed. Kluwer Academic/ Plenum Pub, NY, p. 362. [ Links ]
Iregui C, Comas J, Hernández EA, Jiménez AP, Peña LC, Pulido A, Rey AL, Rodríguez M. Primer Mapa epidemiológico de las lesiones y enfermedades de los peces en Colombia. Universidad Nacional de Colombia, Ministerio de Agricultura y Desarrollo Rural, Bogotá-Colombia. 2004;1:9-45 [ Links ]
Ishida Y, Ahmed AM, Mahfouz NB, Kimura T, El-Khodery SA, Moawad, A.A, Shimamoto, T. Molecular analysis of antimicrobial resistance in gram-negative bacteria isolated from fish farms in Egypt- journal of veterinary medical science/the japanese society of veterinary science. 2010;72:727-734. [ Links ]
Jha P, Barat S, Nayak C. Fish Production, Water Quality and Bacteriological Parameters of Koi Carp Ponds Under Live-food and Manure Based Management Regimes, Zoological Research. 2008;29:165-173. [ Links ]
Kirby W, Bauer AW. Antibiotic suseptibility testing by standardized single disk method. Am J pathol. 1966;45:493-496. [ Links ]
Klesius PH. Molecular identification and virulence of three Aeromonas hydrophila isolates cultured from infected channel catfish during a disease outbreak in west Alabama (USA) in: 2009. Dis Aquat Organ. 2011;94:249-253. [ Links ]
Lima RMS, Figueiredo HCP, Faria FCD, Picolli RH, Bueno-Filho JS, Logato, P V R. Resistência a antimicrobianos de bactérias oriundas de ambiente de criação e filés de tilápias do Nilo (Oreochromis niloticus). Cienc Agrotec. 2006;30:126-132. [ Links ]
Lösch LS, Merino LA, Alonso J M. 2005. Resistencia antimicrobiana en cepas de Pseudomona aeruginosa aisladas de fuentes de agua de la provincia del Chaco (Argentina). Universidad Nacional del Nordest. comunicaciones científicas y tecnológicas. [ Links ]
Luna D, De León J, Vallejo A, Velásquez G. 2003. Evaluación de la resistencia bacteriana frente a tres antibióticos usados en la maduración del camaron marino (Litopenaeusvannamei). Universidad de Córdoba, Facultad de Medicina Veterinaria y Zootecnia, Departamento de Acuicultura. 2003;8:2-334. [ Links ]
Lunden T, Bylund G. The influence of in vitro and in vivo exposure to antibiotics on mitogen-induced proliferation of lymphoid cells in rainbow trout (Oncorhynchusmykiss). Fish Shellfish Immunol. 2000;10:395-404. [ Links ]
Marín C, Fonseca C, Arias S, Villegas I,.García A, Ishihara H. Carga bacteriana de los peces Cynoscion squamipinnis (Perciformes: Scianidae) y Lutjanus gutattus (Perciformes: Lutjanidae) en la cadena de comercialización, Costa Rica. Rev Biol Trop. 2009;57:1-2. [ Links ]
Martínez JL. Antibiotics and antibiotic resistancegenes in natural environments. Science. 2008;321:365-367. [ Links ]
Miranda CD, Kehrenberg C, Ulep C, Schwarz S, Roberts C. Diversity of Tetracycline Resistance Genes in Bacteria from Chilean Salmon Farms. Antimicrob. Agents Chemother. 2003;47:883888 [ Links ]
Negrete P, Romero J, Arredondo J. Antibiotic resitance and presence of plasmids in: Aeromonas hydrophila, Vibrio fluvialis, and Vibrio furnissi isolated from Carassius auratus auratus. Vet Mex. 2004;35(1):21-30 [ Links ]
Øvreås L, Daae FL, Torsvik V, Rodríguez-Valera F. Characterization of microbial diversity in hypersaline environments by melting profiles and reassociation kinetics in combination with terminal restriction fragment length polymorphism (T-RFLP). Microbial ecology. 2003;46(3):291-301. [ Links ]
Petersen A, Andersen J, Kaewmak T, Somsiri T, Dalsgaard A. Impact of http://www.sciencemag.org SCIENCE VOL 296 10 MAY 2002 Integrated Fish Farming on Antimicrobial Resistance in a Pond Environment Appl Environ Microbiol. 2002;68:6036- 6042. [ Links ]
Pridgeon JW, Find all citations by this author (default). [ Links ] Pulkkinen, K, Suomalainen LR., Read AF, Ebert D, Rintamäki P, Valtonen ET. Intensive fish farming and the evolution of pathogen virulence: the case of columnaris disease in Finland. Proceedings of the Royal Society B: Biological Sciences. 2010;277:593-600. [ Links ]
Ranjard L, Poly F, Nazaret S. Monitoring complex bacterial communities using culture-independent molecular techniques: application to soil environment. Res Microbiol. 2000;151:167-177. [ Links ]
Rodríguez-Martínez JM, Conejo MC, Díaz de Alba P, López-Cerero L, Fernandez-Echauri P, Pascual A. Asociación en un mismo plásmido de blaVIM-1 y qnrS2 en Klebsiella pneumoniae y Klebsiella oxytoca aisladas en Sevilla. Enferm Infecc Microbiol Clin. 2012;30:246-248 [ Links ]
Sarter S, Nguyen H, Hung L, Lazard J, Montet D. Antibiotic resistance in Gram-negative bacteria isolated from farmed catfish. Food Control. 2007;18:1391-1396. [ Links ]
Tešertova N, Kisand A, Baty F, Kisand V. Homogeneus microbial diversity in the upper sediment layers of a shallow lake.Aquatic Microbial Ecology. 2013;70:77-85. [ Links ]
Teuber M. 2001, Veterinary use and antibiotic, resistance Current Opinion in Microbiology. 2001;4:493-499. [ Links ]
Zhang RQ, Ying GG, Su HC, Zhou LJ, Liu YS. Antibiotic resistance and genetic diversity of Escherichia coli isolates from traditional and integrated aquaculture in South China. J Environ Sci Health B. 2013;48:999-1013. [ Links ]