Introducción
El eje hipotalámico-hipofisiario-gonadal controla todos los estados de la reproducción. El hipotálamo produce hormona liberadora de gonadotropinas (GnRH), la cual se moviliza a la hipófisis anterior y estimula la secreción de hormona folículo estimulante (Senger, 2012) y de hormona luteinizante (LH) (Casarini et al., 2011).
La LH es una hormona Glucoproteica heterodimérica cuya vida media es de unos 20 minutos (Gay et al., 1970). Está estructuralmente conformada por dos cadenas, una α, la cual comparte con otras hormonas como la FSH (McArdle y Roberson, 2015) y una β que le confiere la especificidad biológica (Cadagan y Towlson, 2017).
En bovinos y ovinos, los estados finales del desarrollo folicular requieren de pulsos de LH (Rawan et al., 2015); si la secreción de progesterona permanece alta, los pulsos de LH no se darán más rápidamente y el folículo se atresiará, induciendo una nueva onda folicular (Ginther, 2000).
La habilidad de las gonadotropinas para modular el crecimiento folicular depende no sólo de los niveles circulantes de estas hormonas, sino también de la expresión de receptores específicos que se encuentran embebidos en la membrana de las células de la granulosa y de la teca interna del folículo ovárico Richards y Hedin, (1988); Rawan et al., (2015).
El gen del LHR es un gen altamente conservado en vertebrados, se encuentra en peces (Clelland y Peng, 2009; Liu et al., 2011), aves (Johnson et al., 1996) y mamíferos y en el bovino está ubicado en el cromosoma 11, identificado con código GeneID: 281900. (Genbank 2020). Este gen se expresa en las gónadas junto con el receptor de la FSH (FSHR), mientras que receptor el de la GnRH lo hace en la hipófisis anterior, está sujeto a corte y empalme alternativo (splicing alternativo), dando origen a diferentes variantes del mRNA del gen (Ma et al., 2012); en bovinos se han reportado variantes que carecen del exón 10 , deleción parcial del exón 11 (Robert et al., 2003; Nogueira et al., 2007) y del exón 3 (Nogueira et al., 2007).
Aunque las propiedades de unión y funcionales de esas variantes no han sido completamente dilucidadas, se ha observado que la variante con deleción parcial del exón 11 traduce en proteína pero ésta no atraviesa la membrana celular (Kawate, 2004), por lo tanto, es razonable asumir que las diferencias en la respuesta a la LH estén relacionada con la expresión de los LHR (Mamluk et al., 1998) ya que en humanos por ejemplo, la función de los LHR se pierde cuando se presentan mutaciones en los exones 8 y 10 e igualmente, ante una deleción del exón 10, no hay respuesta a la acción de la LH aunque si a la hCG (Dickinson et al., 2009).
Este trabajo tuvo como objetivo comparar entre la raza criolla colombiana Romosinuano y la raza Brahman los posibles polimorfismos del ARNm del gen que codifica para el receptor de la hormona luteinizante, como potenciales indicadores del potencial ovulatorio.
Materiales y métodos
El trabajo contó con la aprobación del comité de bioética de la Facultad de Medicina Veterinaria y de Zootecnia de la Universidad Nacional de Colombia y se desarrolló en el Centro Universitario Regional del Norte (CURDN) de la Universidad del Tolima, ubicado en el kilómetro 80 de la carretera que de Ibagué conduce al municipio de Armero-Guayabal, con temperatura media de 28.2 °C, precipitación anual media de 1.791 mm, humedad relativa de 71 % y Altitud entre 275 - 550 m.s.n.m., clasificado como zona de vida: Bosque seco tropical (bs-T) (Holdridge, 1967). Los procedimientos moleculares se realizaron en las instalaciones del laboratorio de biología molecular de la Facultad de Medicina Veterinaria y Zootecnia de la Universidad del Tolima.
Se tomaron 5 vacas de la raza Romosinuano (RS) y 5 Brahman (Br) con edad entre tres y cinco años, que habían registrado por lo menos un parto, clínicamente sanas y cíclicas, las cuales permanecieron en potreros establecidos en pasto Ángleton (Dichanthium aristatum) y contaron con sal mineralizada y agua a voluntad.
Para verificar ciclicidad ovárica, a las 10 hembras se les realizó diariamente seguimiento ecográfico durante tres ciclos estrales, usando la técnica descrita por Pierson y Ginther (1988), mediante la utilización de un equipo Aquila, con sonda dual de 6.0 y 8.0 MHz. (Pie Medical. http://www.piemedicalimaging.com/), registrando las imágenes en una computadora portátil mediante el uso de una tarjeta exportadora importadora de video Pinacle®. La detección de estros se realizó por observación directa con la ayuda de un macho con desviación lateral del prepucio, entre las 05:30 y las 06:00 horas, las 09:30 y las 10:00, las 13:30 y las 14:00, las 17:30 y las 18:00 y entre las 21:30 y las 22:00.
A partir de la aparición de cada uno de los celos, se realizó ecografía cada 4 horas para precisar el lapso transcurrido entre el inicio de los signos de celo y la ovulación; el tiempo de ovulación se definió como el tiempo promedio de no observación del folículo a la ecografía, menos dos horas (Larsson, 1987).
Durante el tercer estro, se verificó por ultrasonografía la presencia del folículo preovulatorio y se extirpó el ovario que contenía dicho folículo, mediante abordaje lateral derecho, siguiendo la técnica descrita por Peiró et al.,(2009).
Para el procedimiento quirúrgico las vacas se sedaron con xilacina aplicada por vía intravenosa (IV) a una posología de 0,05 a 0,1 mg/kg, ketamina (IV) a una posología de 0.5 mg/kg y se practicaron bloqueos paravertebral y locales con lidocaína al 2%.
Una vez obtenidos los ovarios, se realizó un corte sobre el estigma del folículo preovulatorio para seccionarlo en dos partes iguales:
A una de las partes se le retiró delicadamente la pared folicular mediante el uso de una pinza de disección sin garra, ésta se transportó al laboratorio en RNA Later® a 4 °C y se conservó a -20 °C hasta su procesamiento.
Del tejido folicular se extrajo el RNA total usando kit comercial AMBION PureLink™ RNA Mini Kit® siguiendo las instrucciones del fabricante. Una vez obtenido el RNA, se realizó la determinación de su concentración mediante el uso de espectrofotómetro NanoDropTM 2000 Thermo Scientific. La pureza del RNA se estableció haciendo uso de la relación 260 : 280.
A partir de 2 µg del RNA total, se sintetizó un ácido desoxirribonucleico complementario (ADNc) utilizando el Kit de síntesis de ADNc con transcriptasa reversa SuperScriptTM II, ThermoFisher Scientific (Invitrogen-USA). El ADNc obtenido se usó como plantilla para identificar la expresión génica. Inicialmente, se evaluó la calidad de sintesis del ADNc mediante la amplificación del gen de la β-actina como gen housekeeping de control interno. El PCR de punto final se realizó mediante los siguientes pasos: pre-desnaturalización a 95°C por 3 minutos (min), desnaturalización a 95°C por 30 segundos (seg), anillamiento a 55ºC por 30 seg, extensión a 72°C por 30 seg, en 35 ciclos y un paso de extensión final a 72°C por 5 min, en el termociclador T100 de Bio-Rad. Posterior a la amplificación, el producto de la reacción se visualizó mediante electroforesis en gel de agarosa al 2% con bromuro de etidio como intercalante, corrido a 100 voltios por 50 min en el equipo electroforético Power-Pac HC (Bio-Rad -USA), siguiendo los protocolos estandarizados del laboratorio de Inmunología y Biología Molecular de la Universidad del Tolima.
Para detectar la expresión de los segmentos de interés LHR-A y LHR-B del gen para el receptor de LH en las células foliculares, se estableció la presencia del mRNA mediante ejecución de reacción en cadena de la polimerasa con transcriptasa reversa (RT-PCR) usando SuperScriptTM II ThermoFisher Scientific y amplificación mediante PCR de punto final.
Para la realización de la RT-PCR se siguieron los procedimientos descritos por Nogueira (2007), se utilizó un cebador directo para un fragmento de gran parte de dominio extracelular (LHR-A) en el exón 2 (5`-TCAAAGTAATCCCATCTCAAGC-3`) y uno inverso en el exón 9 (5`-CTCGTTTGTTGGCAAGTTTCT-3`); y los cebadores directo (5`-AAACTTGCCAACAAACGA-3`) e inverso (5`-ATAGCAAGTCTTGTCCAGGA-3`) en los exones 9 y 11 respectivamente, para el dominio de toda la región de transmembrana (LHR-B), del gen del receptor. Figura 1.
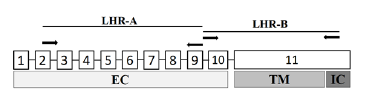
Figura 1 Representación de la ubicación de los cebadores (flechas) para la PCR de los fragmentos LHR-A y LHR-B. Los recuadros numerados representan los exones. Se muestran los dominios de la proteína (EC, extracelular TM región de transmembrana e IC, dominio intracelular. Nogueira et al., (2007).
Para la PCR de punto final para los fragmentos LHR-A y LHR-B de la región codificante del LHR se siguieron los siguientes pasos: pre-desnaturalización a 95°C por 10 minutos (min), desnaturalización a 95°C por 1 min, anillamiento a 64°C por 1 min, extensión a 72°C por 1 min, en 35 ciclos y un paso de extensión final a 72°C por 7 min. Posterior a la amplificación, el producto de la reacción se reveló mediante electroforesis en gel de agarosa al 1% con bromuro de Etidio como intercalante, con un corrido a 100 voltios por 60 minutos.
Los productos amplificados por PCR de punto final fueron visualizados mediante el uso de geles de agarosa al 1%, aplicando 100v durante 60 minutos.
Las bandas observadas en los geles fueron cortadas, purificadas y enviadas a MACROGEN (Corea) para establecer mediante secuenciación del cADN del gen que codifica para el receptor, los polimorfismos de los LHR-mRNA presentes en las muestras.
Las secuencias recibidas se analizaron haciendo uso del software Geneious R8 y se usó el BLAST (Basic Local Alignment Search Tool) para compararlas con otras secuencias que han sido previamente reportadas en el National Center for Biotechnology Information (NCBI), BTU20504 y XM_019969469 para Bos taurus y Bos indicus respectivamente. https://blast.ncbi.nlm.nih.gov/Blast.cgi
Resultados
La visualización mediante geles de agarosa de los productos de amplificación de los cDNA para los fragmentos LHR-A y LHR-B se presentan en la figura 2.
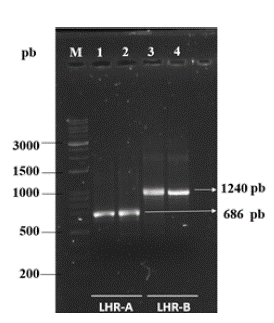
Figura 2 Electroforesis de los fragmentos LHR-A y LHR-B, de vacas de las razas Romosinuano (RS) y Brahman (Br). Carriles 1 y 3: RS, 2 y 4: Br.
Las bandas sugirieron la presencia de un solo polimorfismo para los fragmentos LHR-A y LHR-B, obtenidas a partir de tejido de folículos preovulatorios tanto en la raza RS como en la raza Br. Esta situación fue corroborada por las secuencias obtenidas, ya que para el caso de las hembras de la raza RS el tamaño de los fragmentos codificantes LHR-A y LHR-B del receptor de la hormona luteinizante presentaron una longitud de 630 y de 792 pares de bases respectivamente y para las hembras de la raza Brahman una longitud de 511 y 751 respectivamente.
En la figura 3 se representa la región codificante del gen para el receptor de la hormona luteinizante y la alineación de la secuencias consensus extraídas a partir de los resultados de secuenciación de los fragmentos LHR-A y LHR-B de la raza Romosinuano, contra la secuencias de LHR del GenBank de BTU20504 de Bos taurus.

Figura 3 Representación gráfica de los fragmentos LHR-A y LHR-B secuenciados para la raza RS, sobre la región codificante U20504 (Genbank). Software Geneious R8.
Como se puede observar, la secuencia codificante de los fragmentos LHR-A y LHR-B de hembras de la raza RS de 630 y de 792 pares de bases al ser comparados en el Blast contra la secuencia de referencia LHR en el Genbank de Bos Taurus BTU20504, presentaron una completa similitud (100%) entre secuencias. Tabla 1.
Tabla1. Porcentaje de similitud entre la secuencia para los fragmentos LHR-A y LHR-B para la raza RS y la secuencia de referencia U20504
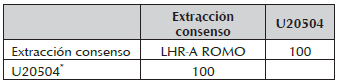
*Genbank
Parámetro obtenido mediante software Geneious R8.
La comparación entre las secuencias obtenidas para los fragmentos LHR-A y LHR-B de la región codificante del gen del receptor para LH de la raza RS con la secuencia de referencia del Genbank U20504, se presentan en las figuras 4 y 5.
Figura 4 Comparación entre la secuencia de la región codificante del fragmento LHR-A RS y la secuencia de referencia Bos taurus U20540 del Genebank. Identities = 630/630 (100%), Gaps = 0/630 (0%).Software Genious R8.

Figura 5 Comparación entre la secuencia de la región codificante del fragmento LHR-B RS y la secuencia de referencia Bos taurus U20540 del Genebank. Identities = 792/2413 (32%), Gaps = 1620/2413 (67%).
En la figura 6 se observa la representación de la secuencia codificante de los fragmentos LHR-A y LHR-B obtenidos para el receptor de la hormona luteinizante a partir de tejido de folículos preovulatorios de hembras de la raza Br, éstos presentaron respectivamente una longitud de 511 y 751 pares de bases, se estableció mediante Blast que los fragmentos del LHR A y B presentaron alta similitud con la secuencia del gen de LHR XM_019969469 de Bos indicus en el Genbank (98 y 99%).

Figura 6 Representación gráfica de los fragmentos LHR-A y LHR-B secuenciados para la raza Br, sobre la región codificante XM_019969469 (Genbank). Software Geneious R8.
Las comparaciones entre las secuencias obtenidas para los fragmentos LHR-A y LHR-B de la región codificante del gen del receptor para LH de la raza Brahman contra la secuencia de referencia del XM_019969469 del Genbank, se presentan en las figuras 7 y 8.
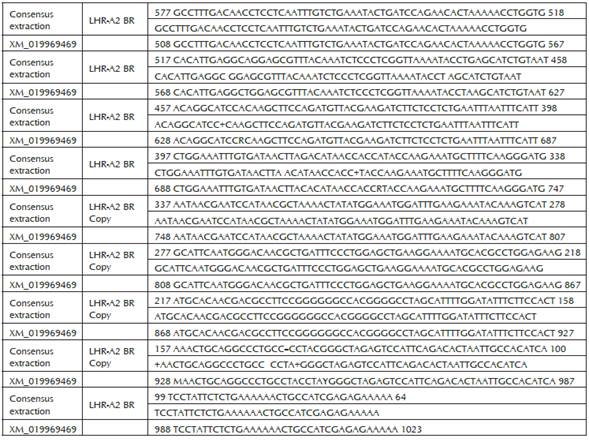
Figura 7 Comparación entre la secuencia de la región codificante del fragmento LHR-A Br y la secuencia de referencia Bos indicus >XM_019969469 del Genebank. Identities = 507/516 (98%), Gaps = 2/516 (0%).
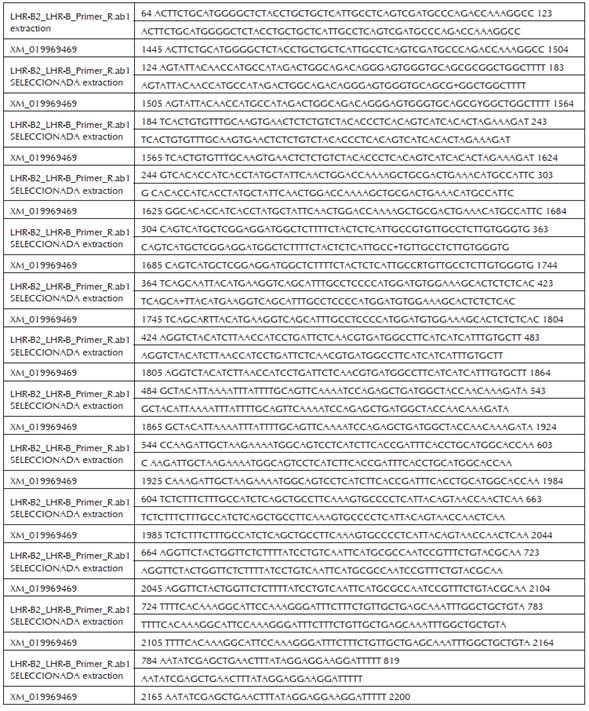
Figura 8 Comparación entre la secuencia de la región codificante del fragmento LHR-B Br y la secuencia de referencia Bos indicus >XM_019969469 del Genebank. Identities = 751/756 (99%), Gaps = 0/756 (0%).
Como se puede observar, la secuencia codificante obtenida en presente trabajo de los fragmentos LHR-A y LHR-B de hembras de la raza Brahman de 511 y de 751 pares de bases al ser comparados en el Blast contra la secuencia de referencia LHR en el Genbank XM_019969469, presentaron una alta similitud (98-99% respectivamente) entre secuencias.
Discusión
El análisis comparativo de la secuencia codificante para los fragmentos LHR-A y LHR-B obtenidos el receptor de la hormona luteinizante a partir de tejido de folículos preovulatorios de hembras de la raza Romosinuano y Brahman, contra las secuencia de referencia del Genbank B20504 y XM_019969469 de Bos indicus, se realizó para identificar posibles variantes que confieran diferencias en el potencial ovulatorio y por ende diferencias en su desempeño reproductivo, observables en la tasas de fertilidad de las dos razas.
En ambas razas las secuencias obtenidas para los fragmentos LHR-A y LHR-B corresponden a segmentos de las secuencias de longitud completa reportadas en las bases de datos.
Para la raza RS el fragmento LHR-A presentó una longitud de 630 bp, se observó que existe una identidad desde el nucleótido 18 de la secuencia consensus con el 232 de la BTU 20504, hasta el nucleótido 647 con el 861 de las secuencias consensus y BTU 20504 respectivamente, es decir en los 630 nucleótidos perteneciente a una región del dominio extracelular del gen, lo que corresponde con lo reportado por Robert et al., (2003), como clon A y por Nogueira et al., (2007), como LHR-A1 para la secuencia de la fracción codificante completa de la región extracelular, es decir sin adiciones ni deleciones, condición que permitiría un desempeño reproductivo normal, sustentado por la regularidad de los ciclos en cuanto a la presentación de los estros y la ovulación de los animales estudiados de la raza Romosinuano, ya que en la observación ultrasonográfica de los ovarios durante los tres ciclos previos a la obtención de los tejidos no se presentaron estros anovulatorios ni ovulaciones sin exhibición de signos de estro.
El fragmento LHR-B obtenido en este trabajo presentó una longitud de 792 bp y corresponde a parte del de los dominios de transmembrana e intracelular del receptor. Se observó que existe identidad desde el nucleótido 72 de la secuencia consensus con el 1226 de la BTU 20504, hasta el nucleótido 864 con el 2018 de las secuencias consensus y BTU 20504 respectivamente, lo que concuerda con lo reportado por Robert et al., (2003), como clon A y por Nogueira et al., (2007), como LHR-B3, para la secuencias de las fracciones codificantes completa de la región de transmembrana y parte intracelular.
De igual manera en lo referente a la raza Brahman, el fragmento LHR-A presentó una longitud de 511 bp, se observó que existe una identidad del 98% desde el un cleótido 577 de la secuencia consensus con el 508 de la XM_019969469, hasta el nucleótido 64 con el 1023 de las secuencias consensus y XM_019969469 respectivamente, es decir en 507/516 nucleótidos perteneciente a una región del dominio extracelular del gen, lo que corresponde de igual manera con la secuencia de longitud completa reportada por Robert et al., (2003), como clon A y por Nogueira et al., (2007), como LHR-A1 para la secuencia de la fracción codificante completa de la región extracelular.
El fragmento LHR-B obtenido en este trabajo presentó una longitud de 751 bp y corresponde a parte del de los dominios de transmembrana e intracelular del receptor. Se observó que existe identidad desde el nucleótido 64 de la secuencia consensus con el 1445 de la XM_019969469, hasta el nucleótido 819 con el 2200 de las secuencias consensus y XM_019969469 respectivamente, lo que concuerda con lo reportado por Robert et al., (2003) como clon A y por Nogueira et al., (2007) como LHR-B3, para la secuencias de las fracciones codificantes completa de la región de transmembrana y parte intracelular.
Al parecer en las secuencias estudiadas para los dos fragmentos en razas RS y Br no se presentaron inserciones ni deleciones que comprometieran la acción del ligando. Tampoco se presentaron polimorfismos de nucleótido simple como los reportados por Vásquez et al., (2014) o Hastings et al., (2006) asociados a la fertilidad en ganado lechero.
En conclusión se puede expresar que aunque el gen objeto de este trabajo es apenas uno de los muchos que probablemente afecten la fertilidad de las hembras bovinas, los fragmentos obtenidos para la región codificante de los dominios extracelular y transmembrana del receptor para la hormona luteinizante en tejido folicular de las razas Romosinuano y Brahman permiten suponer que ante la presencia de las secuencias completas del mismo y sin adiciones, los animales podrían presentar un desempeño reproductivo dentro de los parámetros fisiológicos normales.
Convendría realizar trabajos encaminados a la cuantificación de la expresión del gen y la determinación de los componentes celulares que lo expresan comparando animales de buen historial reproductivo con animales que presenten alteraciones en su normal ciclicidad ovárica o con problemas de fertilidad.















