Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Vitae
Print version ISSN 0121-4004
Vitae vol.15 no.1 Medellín Jan./June 2008
REVISIONES
EDICIÓN POR APOBEC, UN NUEVO MECANISMO DE RESISTENCIA A LA INFECCIÓN POR EL VIH-1
EDITION BY APOBEC, A NEW RESISTANCE MECHANISM TO HIV-1 INFECTION
Claudia PATIÑO.1*; Silvio URCUQUI-INCHIMA.1
1 Grupo Inmunovirología, Universidad de Antioquia. A.A.: 1226 Medellín Colombia
RESUMEN
El HIV/SIDA afecta a más de 40 millones de personas en todo el mundo. Los altos costos de los antiretrovirales, y la resistencia a estos fármacos han obligado a la búsqueda de nuevas moléculas bioactivas enfocadas a bloquear proteínas celulares o virales, o moléculas que participen en el ciclo de replicación del virus, y que permitan aumentar la supervivencia y la calidad de vida de las personas infectadas. Recientemente se describieron ciertos factores celulares con actividad anti-VIH-1, los cuales por sus características, constituyen una familia de proteínas celulares conocidas como APOBEC. Los miembros de esta familia son enzimas deaminasas que modifican citosinas por uracilos en DNA/RNA celulares o extraños. La subfamilia de proteínas APOBEC3 es la más estudiada, ya que está implicada en la respuesta inmune innata, es decir, presentan actividad antiviral contra diferentes virus, entre ellos, el HIV-1. APOBEC3, no sólo es capaz de editar el genoma viral, sino que participa en diferentes etapas del ciclo replicativo. En esta revisión se discuten hasta donde es posible, los diferentes mecanismos en los cuales esta subfamilia de proteínas participa activamente en la respuesta antiviral, mediante la inhibición de la replicación de virus.
Palabras clave: Resistencia, Deaminasas celulares, APOBEC3G, hA3G, VIH-1
ABSTRACT
HIV/AIDS affects more of 40 million people in the world. The high costs of the antiretroviral compounds, and the resistance to these medicines has led to intense search for new antiviral, focused on substances to be actives white proteins or other molecules that participates in the virus's cycle, in order to increase the survival and the quality of life of infected patients. Recently, certain cellular factors were described by their anti-HIV- 1 activity. According to their characteristics, they constitute a family of cellular proteins known as APOBEC. The members of this family are deaminases enzymes that modify cytosine to uracilo in cellular or foreign DNA/RNA. The sub-family APOBEC 3 is the most intensely studied, since some of they members exhibited antiviral activity against viruses such as HIV-1. APOBEC3, not only after edition of the viral genome, but with the blockaded of it's replication in different stages of his cycle. This review attempts to analyse the APOBEC family of proteins to understand the mechanisms by which they offer resistance to certain viruses particularly to HIV-1, where still more studies need to be performed to understand how this virus partially or completely escapes inhibition by these proteins.
Key Words: Resistance, Cellular deaminases, APOBEC3G, hA3G, HIV-1
INTRODUCCIÓN
El síndrome de inmunodeficiencia adquirida (SIDA) ocasionado por la infección con el virus de inmunodeficiencia humana tipo 1 (VIH-1), es el resultado de la interrelación de diferentes factores, tanto celulares como virales. Multiples estudios y la utilización de algunos modelos, han permitido identificar y caracterizar ciertas proteínas celulares que actúan como factores celulares anti-VIH-1; es decir, actúan negativamente en las diferentes etapas del ciclo replicativo del virus. En ese sentido, miembros de la familia de proteínas APOBEC han adquirido gran importancia en los últimos 4 años, por su capacidad de limitar la replicación del virus en células normalmente permisivas tales como macrófagos y linfocitos. Sin embargo, este mecanismo de respuesta antiviral celular es contrarrestado por la proteína Vif, la cual es codificada por el genoma del VIH-1. En la presente revisión se hace una discusión de los conocimientos disponibles actualmente sobre la actividad antiviral de APOBEC con una orientación específica hacia VIH-1, por ser un fenómeno que ha permitido avanzar en la comprensión de la relación que existe entre proteínas virales y celulares.
GENERALIDADES DEL VIH-1
El Virus de la Inmunodeficiencia Humana (VIH) es un retrovirus que pertenece a la subfamilia de los Lentivirus. Invade directamente los linfocitos T CD4+, macrófagos, monocitos, causando un deterioro progresivo del sistema inmune; además también infecta células dendríticas de mucosas (células de Langerhans) y células nerviosas de la microglía (1).
Actualmente se han identificado dos subtipos virales: VIH-1, aislado en 1983, distribuido mundialmente, es el responsable de la pandemia (SIDA) del siglo XX; por otro lado, el VIH-2, aislado en 1986, presente principalmente en Asia y África Occidental (2).
Estructuralmente el virus es una partícula icosahédrica de aproximadamente 100 nm, presenta dos capas proteicas y una lipídica. El genoma viral consta de dos cadenas de RNA lineal de sentido positivo que codifica tres proteínas estructurales (Gag, Pol y Env), dos proteínas reguladores (Tat y Rev), y cinco proteínas accesorias (Nef, Vpu, Vif, Vpr, Vpx). El RNA viral se encuentra asociado a tres enzimas virales: proteasa (PR), integrasa (IN) y transcriptasa reversa (TR). Tanto el RNA como las enzimas se encuentran protegidas por la primera capa proteica llamada nucleocápside, ésta a su vez está envuelta por la segunda capa proteica o cápside, y finalmente está la envoltura lipídica tomada de la célula hospedera al momento de salir por gemación (2).
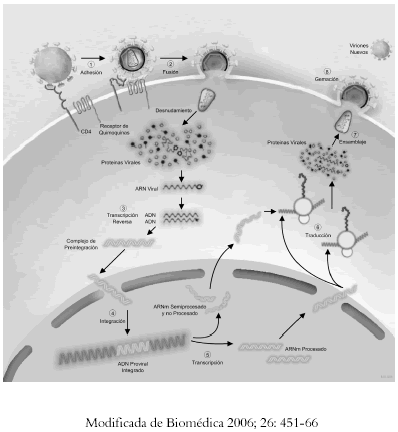
En resumen, la replicación del VIH- 1 se desarrolla en las siguientes etapas (Véase figura 1):
1. Adherencia: Es la primera etapa del ciclo replicativo. Se presenta cuando las glicoproteínas gp120 y gp41 reconocen el receptor y co-receptores (CD4 y CXCR4/CCR5, respectivamente) de las células blanco.
2. Penetración: Luego del reconocimiento se fusiona la envoltura viral con la membrana celular, permitiendo la entrada de la cápside.
3. Transcripción inversa: Gracias a un RNA de transferencia celular (tRNA), que es usado como cebador y tomando el RNA viral como plantilla, la TR forma un DNA de doble cadena (cDNA) a partir del RNA viral. Este fenómeno incluye la formación de los LTR (del ingles "long terminal repeat") en cada extremo del genoma, que corresponden a regiones largas no codificantes indispensables para la integración del DNA al genoma celular.
4. Integración: Después de sintetizado el cDNA viral, es translocado al núcleo como un complejo de preintegración, y gracias a la acción de la IN, es integrado en el genoma celular (provirus).
5. Transcripción: Gracias a la maquinaria celular, se producen tres tipos de mRNA (procesados, semiprocesados y sin procesar) que son exportados al citoplasma, donde son reconocidos por los ribosomas y traducidos en las respectivas proteínas.
6. Ensamblaje: Las proteínas estructurales se ensamblan y empaquetan las dos cadenas de RNA proviral, enzimas y proteínas virales, además de un grupo de proteínas celulares.
7. Salida: El virión inmaduro sale por gemación de la célula infectada y adquiere la envoltura, en la cual se encuentran incrustadas las glicoproteínas virales (gp120 y gp41).
8. El último paso es la maduración del virión, el cual se produce luego de la gemación y del procesamiento de las proteínas precursoras virales, por la PR viral; este proceso es necesario para que la nueva progenie viral sea infecciosa.
Figura 1. Ciclo de replicación del VIH-1
En el proceso replicativo del VIH-1, las proteínas reguladoras Tat y Rev juegan un papel muy importante. La primera es la responsable de incrementar la tasa transcripcional del genoma viral, gracias a su capacidad de interactuar con factores transcripcionales celulares y formar un complejo transcripcional. Por su parte, Rev es la responsable de exportar los mRNA semiprocesados y sin procesar; es decir, transcritos en cuya secuencia de nucleótidos se conservan parcial o totalmente los intrones (3).
NUEVAS DIANAS CONTRA LA INFECCIÓN POR VIH-1
El SIDA es una de las enfermedades más estudiadas en la actualidad debido a las consecuencias de la pandemia; cada día mueren más pacientes y el número de personas infectadas por el VIH-1 aumenta.
Actualmente hay cuatro clases de medicamentos ampliamente distribuidos (4, 5), los cuales están dirigidos contra pasos específicos de la replicación viral:
• Inhibidores de fusión: se adhieren a la cubierta de proteína del virus inhibiendo la fusión de membranas.
• Inhibidores de transcriptasa reversa análogos de nucleótidos: interrumpen el proceso de replicación inhibiendo la elongación de los transcriptos.
• Inhibidores de transcriptasa reversa no análogos: interactúan con la TR y la inactivan.
• Inhibidores de proteasa: bloquean esta enzima inhibiendo el procesamiento de proteínas virales.
La terapia antirretroviral ha mejorado la calidad de vida y la supervivencia de las personas infectadas; sin embargo, la amenaza continua de resistencia a los antiretrovirales ha obligado a la búsqueda de nuevos medicamentos que sean capaces de suprimir por completo la carga viral sin efectos adversos, permitiendo la restauración completa del sistema inmune. Esto significa nuevos retos para la comunidad científica, y un mejor entendimiento de las interrelaciones tanto entre proteínas virales y celulares, en especial, en las implicadas en la respuesta anti-VIH-1, como entre las mismas proteínas celulares.
El conocimiento del ciclo replicativo del VIH-1, y el descubrimiento de nuevos factores celulares indispensables para una replicación exitosa, han permitido un cambio de paradigma generando nuevas estrategias terapéuticas orientadas a bloquear receptores de entrada, factores celulares y en general, cualquier estructura o molécula que participe en la replicación del virus y en los mecanismos de defensa celular.
Entre las nuevas estrategias se encuentran las estátinas, compuestos que inhiben la entrada del virus, bloqueando la unión de la molécula de adhesión intercelular ICAM-1 a su receptor fisiológico LFA-1 (del ingles lymphocyte function-associated antigen 1) (6).
Desde el 2004 se están realizando estudios sobre una proteína llamada Prostrátina, que es capaz de internalizar el receptor CD4, protegiendo la célula de la infección (7); actualmente se piensa en esta proteína como una posibilidad terapéutica ya que no es tóxica y no genera procesos tumorigénicos. Sin embargo, aún falta mucho por investigar sobre ella. También se están evaluando inhibidores de CXCR4 como AMD3100 y KRH-1636 (fase de investigación clínica) (8). Es importante tener en cuenta que ante este tipo de estrategias, los tratamientos pueden intervenir con el buen funcionamiento del sistema inmune; por esto se están buscando inhibidores intracelulares que participen de alguna manera en el ciclo de replicación, como la Ciclofilina A, que podría alterar el proceso de entrada, al interactuar con la proteína de la cápside (9). Los inhibidores de la Ciclofilina, las ciclosporinas, aunque tienen efecto inmunosupresor, son buenos candidatos para tratamientos futuros. Igualmente se están evaluando inhibidores de la integrasa viral, como S-1360 (9).
En los últimos 4 años, la comunidad científica y las compañías farmacéuticas han centrado una buena parte de la investigación en ciertos factores celulares con actividad anti-VIH, que suprimen la infecciosidad del mismo, la cual a su vez, se ha descrito, es bloqueada por la proteína viral vif. Nos referimos a los miembros de la familia de proteínas APOBEC (del ingles apolipoprotein B mRNA editing enzyme, catalytic polypeptide), las cuales, con base en algunos reportes, se plantea participan activamente en la respuesta antiviral. A continuación describiremos las características más importantes de esta familia de proteínas, su papel en la respuesta anti-VIH-1 y el mecanismo que utiliza el virus para contrarrestar su efecto antiviral. APOBEC se podría considerar como un nuevo mecanismo de resistencia celular, ya que a diferencia del interferón (tipo 1 o 2) que es inducido en respuesta a la misma infección, APOBEC es una proteína que se encuentra en la célula y es encapsidada en el proceso de ensamblaje de la partícula viral, de tal manera que su acción se manifiesta tan pronto la TR inicia su función.
DEAMINASAS CELULARES Y LA FAMILIA APOBEC
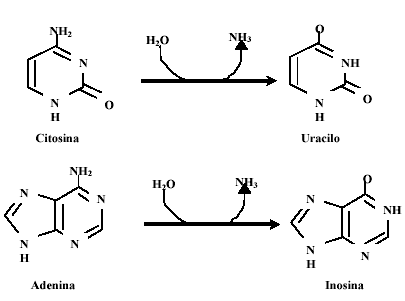
Las deaminasas celulares son un gran grupo de proteínas que participan en diversos procesos metabólicos relacionados directamente con los nucleótidos. Estas enzimas reconocen y modifican DNA/RNA híbridos, o DNA de cadena sencilla y son capaces de editar tanto DNA como RNA, afectando diversas funciones fisiológicas celulares. Las ediciones más frecuentes son de Adenina a Inosina (A→I) o de citosina a uracilo (C→U), alterando la capacidad codificante del mRNA (Véase figura 2). Las deaminasas celulares de Citosina editan este nucleótido a Uracilos en DNA/RNA extraños, lo cual actúa como un sistema de defensa innato de las células (10). Es decir, durante el proceso de la transcripción, los nuevos transcriptos en presencia de APOBEC presentan diversas mutaciones.
Figura 2. La deaminación de citosina y adenina es catalizada por la enzima APOBEC. El tautomerismo es producido por la adicción de agua y la eliminación de un grupo amino.
APOBEC es una megafamilia de deaminasas de (deoxi) citidinas, constituida por las subfamilias APOBEC1, APOBEC2, APOBEC3 (A a H), APOBEC4 y AID (11). Una característica importante es que esta familia de proteínas sólo se ha descrito en vertebrados. Los integrantes de esta familia se caracterizan porque presentan un dominio catalítico, el cual contiene un dominio de unión a cinc, con una secuencia consenso His-X-Glu-X23-28-Pro-Cys-X2-4 (La X representa un aminoácido cualquiera) (12).
APOBEC1 fue la primera proteína identificada de esta familia. Es expresada en enterocitos y está implicada en el metabolismo de lípidos. Actúa como subunidad catalítica de una enzima que edita el RNA y convierte la Citidina en la posición 6666 a Uracilo, lo que origina un codón de parada, que detiene prematuramente la síntesis de proteínas desde el mRNA de la Apoliproteína B, produciendo una forma funcional truncada de ésta (13). Simultáneamente, también se describieron las deaminasas inducidas por activación (AID), presentes exclusivamente en los linfocitos B.
Las AID cumplen un papel importante en la inmunidad adaptativa, ya que la deaminación de CU es fundamental para la diversificación de inmunoglobulinas dependientes antígenos; es decir, para dar origen a anticuerpos con alta afinidad (14, 15). En las células B este gen se caracteriza por la presencia de regiones repetitivas ricas en Guanina, lo que induce la alta afinidad por los anticuerpos (16).
De APOBEC2 (también conocida como ARCD-1), se desconoce su función; se ha descrito que edita citosinas libres, mRNA y DNA naciente (17, 18). Existe evidencia que muestra que ratones con deficiencia en APOBEC2 y APOBEC3, no muestran deterioro en su desarrollo normal, en la supervivencia, ni en su fertilidad (18).
La última proteína descrita de esta superfamilia, es APOBEC4 (19); fue identificada gracias a estudios filogenéticos y análisis computacionales; en éstos se ubicó el gen que codifica esta proteína en el cromosoma 12q23, a diferencia de APOBEC1, en el que se encuentra codificada en el locus 13 del cromosoma 12 (13). Aún no se conoce su función ni las células que la expresan, pero se ha aislado de mamíferos, ratones y gallinas (19).
Subfamilia de proteínas APOBEC3
El gen que codifica APOBEC3 se ha ubicado en el locus 13.1 del cromosoma 22; en algunos mamíferos el gen codifica una sola APOBEC, pero en humanos y en algunos primates, codifica nueve proteínas diferentes (denominadas por su abreviación hA3A - hA3H además hA3DE) (Véase Tabla 1). Se desconoce el significado de las diferentes proteínas codificadas por ese gen, pero se ha sugerido que puede ser consecuencia de la presión evolutiva, generada por la aparición de los virus y la necesidad de neutralizarlos (20).
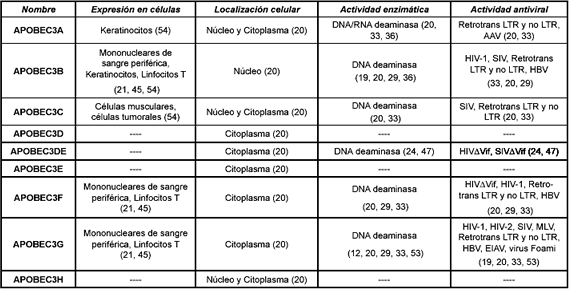
Tabla 1. Características de la subfamilia APOBEC3 (el número entre paréntesis, indica la referencia).
La secuencia consenso His-X-Glu-X23-28-Pro-Cys-X2-4, que representa un sitio activo deaminasa citidina (CDA), está presente en todos los miembros de la familia APOBEC3, pero hA3B, hA3C, hA3F y hA3G en lugar de uno, presentan dos sitios CDA, y se ha sugerido que son los que confieren a esas cuatro proteínas, su capacidad antiviral (21).
Recientemente se determinó por microscopía de fluorescencia, que hA3A, hA3C y hA3H colocalizan en el núcleo y citoplasma; sin embargo, hA3B se encuentra exclusivamente en el núcleo y hA3DE, hA3F y hA3G, exclusivamente en citoplasma (22). Esto podría explicar de cierta forma, la fuerte actividad antiretroviral de hA3F y hA3G (Véase Tabla 1).
Hasta el momento se conoce que hA3G se expresa en linfocitos T, macrófagos, células dendríticas mieloides, células dendríticas plasmacitoides, y en las líneas celulares CEM (ATCC: CCL-199, células T linfoblastoides) y H9 (ATCC: HTB-176, linfocitos T cutáneos). Todas ellas expresan el receptor CD4 y por ende son susceptibles a la infección por VIH-1 (23). Además se han estimulado las líneas celulares HepG2, Huh-7, y hepatocitos primarios humanos con el fin de evaluar la acción antiviral dirigida contra el Virus de la Hepatitis (24).
hA3G existe en dos formas diferentes: una con bajo peso molecular (LMM), la cual está asociada a la restricción del VIH-1; y otra de alto peso molecular, que carece completamente de actividad enzimática y por ende, de actividad antiviral (23, 25).
RESISTENCIA ANTIVIRAL MEDIADA POR APOBEC3
La replicación del genoma viral está regulada por proteínas virales y por factores celulares: una coordinación entre los dos tipos de componentes es fundamental para el éxito de la infección. De igual manera, la dispersión del virus, la inducción de una enfermedad (patogénesis), o el proceso de transmisión a nuevos hospederos, son controlados por factores celulares y regulados por proteínas virales. Esto muestra en consecuencia, que existe una dinámica en la coevolución virus/célula (hospedero).
Como se discutirá a continuación, existen nuevas evidencias que muestran que una de las estrategias del hospedero para luchar contra ciertos agentes virales, es editar el genoma de ciertos virus, y en particular de retrovirus. La enzima responsable de este fenómeno es APOBEC3; dos proteínas de esta familia, hA3G y hA3F, han sido implicadas en la inducción de hipermutaciones, lo que ocasionalmente conduce a la inhibición de la replicación de algunos retrovirus como VIH-1, virus de la inmunodeficiencia simiana (SIV), virus de la anemia infecciosa equina (EIAV), virus de la leucemia murina (MLV) y virus Foamy (21, 26, 27, 28, 29). También se ha visto involucrada en la inhibición de algunos transposones (22, 30) y otros virus como Hepatitis B (HBV) (24, 31, 32) y Parvovirus (AAV) (30) (Véase Tabla 1).
Proteínas APOBEC 3 y Retrotransposones
Los humanos y algunos mamíferos integran en su genoma un gran número de retrovirus endogénos, conocidos como retrotransposones, debido posiblemente a infecciones ocurridas hace millones de años en células germinales (33). Los retrotransposones son secuencias repetitivas capaces de movilizarse de un sitio a otro del genoma durante la recombinación genética, integrándose en lugares prácticamente aleatorios. Se dividen en dos familias: los que contienen LTR y los que no contienen LTR (34).
Retrotransposones LTR
Los retrotransposones LTR codifican únicamente para Gag, Pol y producen algunas partículas virales que se ensamblan en el citoplasma de la célula. La transcriptasa reversa es funcional y el producto es translocado al núcleo donde es integrado nuevamente en el genoma celular. Estos retrotransposones incluyen una partícula A intracisternal (IAP) que conserva una región defectuosa de env, además de familia de proteínas Ty1 (34).
En el 2005, dos grupos de investigadores se preguntaron si APOBEC3 sería capaz de inhibir los retrotransposones por medio del sitio Ty1, y efectivamente descubrieron que la expresión de hA3G y hA3F en levaduras bloqueaba la replicación de LTR retrotransposon Ty1, gracias a varias mutaciones de GA (35, 36). Los autores demostraron que Ty1 Gag interactúa con hA3G y hA3F, empaquetándolos dentro de las partículas tipo virus. Schumacher et al (36), plantearon que Vif de VIH-1 requiere de un factor celular para inhibir hA3G, ya que al cotransfectar las mismas levaduras con hA3G y Vif, la inhibición de los retrotransposones no fue bloqueada; estos autores proponen como cofactor a E3 ubiquitin ligasa, la cual es reclutada por Vif en células humanas e induce la degradación de hA3G (36.
Retrotransposones no LTR
El más prevalente es LINE 1 (del ingles Long Interspersed Element 1) o L1. Esta familia comprende el 17% del genoma humano (34). Cabe anotar que la diferencia de LINE 1 con los demás retrotransposones y retrovirus endogénos es que la transcripción reversa ocurre en el interior del núcleo.
L1 es conocido por ser la causa de varias enfermedades genéticas, como la hemofilia A y hemofilia B (37), además de cáncer de colón, lo que sugiere que la transposición no sólo se presenta en células germinales, sino en células somáticas. Recientemente Kinomoto et al (22) demostraron que la transposición de L1 es inhibida por las proteínas hA3A, hA3B, hA3G, aunque las proteínas restantes mostraban también un bloqueo parcial dirigido a L1, lo que indica que todas las proteínas de la familia APOBEC3, actúan de forma diferente para bloquear la transposición del elemento L1 (22).
Respuesta anti-VIH-1, mediada por APOBEC3G (hA3G)
Las deaminasas de citosina deaminan este nucleótido por uracilo en DNA/RNA foráneos, actuando como un sistema de defensa innato de las células. En ese sentido se ha descrito que la deaminación del cDNA determina la respuesta innata a infecciones por retrovirus (36).
En el 2003 se describió por primera vez que hA3G (también llamada CEM15) estaba implicada en la resistencia antiviral. Los autores demostraron que hA3G ejerce una actividad antiviral durante la transcripción reversa, al inducir pequeñas mutaciones (fenómeno llamado hipermutación) en el cDNA naciente (39). Estas mutaciones registran una transición de Guanosina a adeniosina en el DNA de sentido positivo, lo que indica una edición de citidinas en la cadena en sentido negativo (40). La reacción de deaminación de citidinas inducida por APOBEC la convierte en uracilo, gracias a la adición de agua y a la eliminación de un grupo amino (Véase Figura 2)
APOBEC3G ingresa al virión durante el ensamblaje, y cuando el virus portador de hA3G infecta una nueva célula y la TR da inicio a su actividad, hA3G tiene la oportunidad de deaminar el cDNA naciente, generando así las hipermutaciones (41), lo que produce una progenie viral con la proteína viral Vif mutada (VIH∆Vif), consecuencia de la gran cantidad de mutaciones G A, en el gen que codifica por esa proteína.
A, en el gen que codifica por esa proteína.
estudios han demostrado que el empaquetamiento de hA3G se produce gracias a la interacción con la proteína estructural Gag (42). Sin embargo, otros estudios plantean que la integración de hA3G/Gag puede deberse a la interacción dependiente de RNA, ya que ambas proteínas presentan dominios de unión a RNA (43). Esto sugiere que aún hay controversia sobre la manera como hA3G pasa a hacer parte del virus; además se desconoce si en este proceso intervienen, o no, otros factores celulares.
La resistencia antiviral mediada por hA3G, puede ser consecuencia de uno o más de los siguientes mecanismos (40):
1. La infección puede llegar hasta la fase de provirus, pero las secuencias (genoma) estarían genéticamente comprometidas, hasta el punto que no sería capaz de producir una progenie infecciosa.
2. El nuevo cDNA (editado) podría ser reconocido y corregido por enzimas de reparación de DNA, creando sitios que luego son reconocidos, clivados y por ultimo, degradados.
3. hA3G puede bloquear la formación de transcritos reversos, mediante un mecanismo aún desconocido.
4. Posiblemente la enzima induce la degradación de los transcritos reversos del VIH-1, a través de un proceso independiente de la edición, en el que endonucleasas celulares pueden estar implicadas.
Sin embargo, la edición del cDNA parece no ser la única actividad antiviral de hA3G. Dos estudios recientes han demostrado que hA3G y hA3F pueden inhibir la replicación del virus por un mecanismo posiblemente no enzimático. Al observar el metabolismo del genoma viral expuesto a hA3G, se encontró que el cDNA exhibe defectos en el apareamiento con el cebador tRNA, alteraciones en la transferencia e integración del cDNA viral y, una reducción en la eficiencia del complejo de preintegración (PIC), lo que conlleva a la disminución de la infectividad viral; además se ha descrito una reducción en la tasa de síntesis del cDNA (44). Igualmente se ha demostrado que hA3G y hA3F interfieren con la formación del provirus, interactuando con la nucleocápside y la integrasa, lo cual altera la transcripción reversa y la integración del cDNA (45).
Ciertos estudios in vivo muestran una controversia sobre la actividad antiviral de hA3G. Por ejemplo, un estudio con 3000 individuos infectados por el HIV-1 demostró que existía un polimorfismo (R186H) en la hA3G, el cual fue asociado con una rápida progresión a SIDA (46). Sin embargo un año después, (47) y colaboradores, no encontraron el mismo resultado trabajando con una población de 327 pacientes VIH-1. A pesar de estos resultados, existe evidencia (48) que muestra que en células T CD4+ de pacientes infectados, la hA3G se encuentra activa en células T CD4+ inactivas (no infectadas); los autores sugieren que esto puede estar relacionado con la baja tasa de replicación del VIH en este tipo de células. Por otro lado, en las células CD4+ activadas (en las cuales se da una alta replicación del virus) hA3G se encuentra en estado inactivo (48). Igualmente se ha demostrado que en los pacientes VIH-1 (+) con una mayor transcripción (mRNA) del gen de la hA3G, presentan menor carga viral; esto sugire que posiblemente existe una correlación entre el contenido de la proteína y la multiplicación del virus. Adicionalmente se ha sugerido que los individuos conocidos como progresores lentos (individuos que han estado en contacto con el VIH-1, pero no son infectados) presentan un aumento en los niveles de hA3G (49). En efecto, en un reciente estudio con 30 individuos expuestos seronegativos, se demostró que la alta expresión de hA3G se podía corelacionar con la resistencia a la infección por el virus (50). Estos estudios son el punto de partida en la búsqueda de nuevas alternativas de lucha contra el VIH-1, y la hA3G se convierte en una nueva opión.
El conjunto de los diferentes reportes muestra que APOBEC3 tiene la capacidad de inhibir la replicación del virus, tanto por mecanismos dependientes como independientes de la edición de citidinas; pero hasta la fecha no se conoce muy bien el mecanismo utilizado por dicha enzima para alterar la producción de partículas virales de manera independiente de la edición. Igualmente se desconoce en detalle la estrategia utilizada por el virus para evadir este nuevo mecanismo de resistencia innata. Sin embargo, desde el momento en que se describió que APOBEC tenía una actividad anti-VIH-1, se han escrito una serie de artículos que muestran la importancia de dicha proteína, no sólo para la comunidad científica, sino también para las casas farmacéuticas; como resultado, como se discutirá mas adelante, ha permitido avanzar en la comprensión del mecanismo que involucra la enzima, así como también en la estrategia utilizada por el virus para escapar a la activivad antiviral de la enzima.
No sólo las APOBEC presentes en células humanas pueden inhibir la replicación de VIH-1; se han reportado estudios donde se evalúa la actividad antiviral de hA3G y hA3F, codificadas por el genoma de varios primates no humanos, o la proteína producida por algunas especies de ratas (rA1) o ratones (mA3), entre otros, las cuales muestran altos niveles de inhibición viral (40).
Proteína viral Vif
El factor de infectividad viral (Vif), es una fosfoproteína básica codificada por el genoma de muchos Lentivirus de primates y no primates. Vif fue descrita por primera vez en células infectadas por el VIH-1; se ha demostrado que es fundamental para la infección, ya que juega un papel muy importante en la replicación del genoma, y en consecuencia, es primordial en la patogénesis viral, ya que virus con Vif defectuosa (conocidos como VIHΔVif) producen viriones no infecciosos (51). Inclusive se ha sugerido que Vif es importante en el estado latente para suprimir la respuesta innata de los linfocitos T.
Las células que comúnmente son blancos del VIH-1 presentan fenotipos permisivos y no- permisivos a virus VIH∆Vif. Vif es requerida para la replicación del virus en células CD4+ como linfocitos T, macrófagos derivados de monocitos y en las líneas celulares CEM (ATCC CCL-199, células T linfoblastoides) y H9 (HTB-176, linfocitos T cutáneos) (52). A las células que replican genomas VIH∆Vif, se las conoce como "células no permisivas"; por el contrario, las células que no necesiten de Vif para una correcta replicación se conocen como "células permisivas" (53). Los viriones VIH∆Vif producto de células permisivas, son capaces de infectar células no permisivas, pero las progenies virales producidas en las células no permisivas son incapaces de producir infección (54). Esto sugiere que Vif es fundamental para producir partículas infecciosas.
Con base en la anterior información, se han sugerido dos hipótesis: las células permisivas presentan un factor capaz de proveer la misma función de Vif (complementación), o las células no-permisivas contienen un factor que previene la replicación de VIH∆Vif (inhibición). Con base en estas hipótesis, dos grupos demostraron que existía un factor celular capaz de inhibir la replicación de viriones en células infectadas con VIH∆Vif (41, 55). Estos trabajos potenciaron una serie de artículos que describen la importancia de APOBEC3 en la inhibición de la replicación del VIH-1.
Teniendo en cuenta que en ausencia de Vif se producían partículas virales no infecciosas, Sheehy et al. (41) decidieron trabajar con células que no expresaban el gen CEM15. Al realizar una expresión transitoria o estable de dicho gen, encontraron que la sola expresión de este gen inducía la producción de partículas no infecciosas, pero en su ausencia se restablecía el fenotipo infeccioso (41). Es decir, la actividad antiviral celular era potenciada por el producto de CEM15. Luego se describió que el producto del gen CEM15, era la proteína APOBE3G (56).
Neutralización de APOBEC3G por Vif.
Es evidente que el VIH-1 presenta un mecanismo que le permite evadir la respuesta antiviral celular mediada por hA3G y, con base en los resultados descritos anteriormente, se muestra que Vif, además de participar en el proceso de infección, en la replicación y en la patogénesis, tiene como función adicional, neutralizar la función de hA3, permitiendo que el virus pueda seguir su ciclo replicativo sin interrupción. Para llegar a esta conclusión, primero se realizaron diferentes análisis que permitieron determinar que una pequeña cantidad de hA3G era incorporada en partículas virales (57). A continuación, en presencia de Vif, se detecto una gran disminución en la cantidad de hA3G endógena, lo que indicó que Vif promueve la degradación de hA3G (58); esta degradación podía ser corregida si se trataban las células con inhibidores de proteasa, y se concluyó que Vif promueve la ubiquitinación de hA3G y su degradación proteosomal (37).
Subsecuentes análisis sugieren que Vif actúa como una proteína adaptadora, conectando hA3G en un complejo E3 ubiquitin ligasa, el cual está compuesto por las proteínas celulares Cullin 5, ElonginB y ElonginC (40, 59). La interacción de este complejo se da por una secuencia altamente conservada en Vif, llamada SOCS (del ingles supresor of cytokine signalling). Este mecanismo es similar al usado por las proteínas llamadas F-box, las cuales actúan como puente entre el complejo, la proteína a ser ubiquitinada y degradada por el proteosoma. Un aspecto interesante, recientemente descrito, es que la función de Vif es dependiente de cinc, ya que la quelación del cinc inhibe la función de Vif, lo que conlleva a que el virus sea más sensible a la acción antiviral de hA3G (60). Los autores demuestran que la quelación del cinc no tiene ningún efecto en el ensamble del complejo Cul5-SOCS3 E3, por lo cual ellos sugieren que el ensamble de E3 dependiente de cinc es un mecanismo exclusivo de VIH-1.
Si bien más de 20 años de estudio sobre la relación VIH-1/célula hospedera, han permitido desarrollar y almacenar un gran conocimiento que ha permitido controlar la replicación del virus y mas bienestar para los pacientes VIH/SIDA, los nuevos conocimientos y en especial los obtenidos con la proteína APOBEC, abren una nueva puerta, y posiblemente nuevas perspectivas para el tratamiento de los individuos VIH+. Es evidente que aún existe un gran camino por recorrer, pero la identificación de una proteína con un gran potencial antiviral es ya un gran avance.
CONCLUSIONES
El estudio de las interacciones proteína-proteína y proteína-RNA, va en aumento. Se han publicado trabajos en los cuales se trata de establecer una integración del conocimiento con miras a entender las diferentes vías que utilizan los virus para cumplir su función y a la vez alterar la biología celular. Son este tipo de estudios los que nos van a permitir disponer de nuevas herramientas, o alternativas, para luchar contra este tipo de infecciones. Los nuevos avances y prometedores resultados obtenidos en este campo reorientan el panorama y posibilitan el uso de nuevas estrategias con fines profilácticos o terapéuticos, basados en la modulación de la expresión de estos genes o en la inhibición/potenciación de estos sistemas.
Desde el descubrimiento del sistema APOBEC se han realizado diversos estudios con el fin de conocer sus funciones naturales y la capacidad que tiene como defensa innata intracelular contra algunos virus como el HIV-1. Después de estos estudios, es inevitable pensar en este sistema como una posible alternativa terapéutica. Actualmente se deben proyectar estudios que busquen inhibidores del proteosoma o bloqueadores de la ubiquitin ligasa E3, con el fin de facilitar a hA3G su función de hipermutar el genoma viral, al punto de convertirlo en un virus defectuoso/no infeccioso, pero que, a la vez, no sea tóxico para la misma célula.
Esto muestra cómo la mejor comprensión de los procesos moleculares implicados tanto en la infección como en la respuesta antiviral, son fundamentales para la prevención o para el desarrollo de nuevas estrategias terapéuticas.
REFERENCIAS BIBLIOGRÁFICAS
1. Chinen J., Terapia genética para la infección por VIH. Rev Med Hered. 1997; 8: 72-77. [ Links ]
2. Freed EO. HIV-1 Replication. Somatic Cell and Molecular Genetics, 2002. Vol. 26, pag: 1-6 [ Links ]
3. Forero, J.E., Urcuqui, S. Principales factores en el transporte núcleo-citoplasma: papel de rev en el proceso del transporte de transcritos de VIH-1. Coloma Med. 2004; 35: 215-223. [ Links ]
4. De Clercq, E., Antiviral drugs in current clinical use. J Clin Virol, 2004. 30(2): p. 115-33. [ Links ]
5. Urdaneta, A.M. and Vivas, M., Avances en la terapia antirretroviral: inhibidores de entrada. Infectio 2006; 10(4): 266-272. [ Links ]
6. Giguere, J.F. and M.J. Tremblay, Statin compounds reduce human immunodeficiency virus type 1 replication by preventing the interaction between virion-associated host intercellular adhesion molecule 1 and its natural cell surface ligand LFA-1. J Virol, 2004. 78(21): p. 12062-5. [ Links ]
7. Rullas, J., et al., Prostratin induces HIV activation and downregulates HIV receptors in peripheral blood lymphocytes. Antivir Ther, 2004. 9(4): p. 545-54. [ Links ]
8. Ichiyama, K., et al., A duodenally absorbable CXC chemokine receptor 4 antagonist, KRH-1636, exhibits a potent and selective anti-HIV-1 activity. Proc Natl Acad Sci U S A, 2003. 100(7): p. 4185-90. [ Links ]
9. Billich, A., et al., Mode of action of SDZ NIM 811, a nonimmunosuppressive cyclosporin A analog with activity against human immunodeficiency virus (HIV) type 1: interference with HIV protein-cyclophilin A interactions. J Virol, 1995. 69(4): p. 2451-61. [ Links ]
10. Vallur, A.C., et al., AID in antibody perfection. Cell. Mol. Life Sci, 2007. 64. pag, 555-565 [ Links ]
11. Takaori-Kondo, A., APOBEC family proteins: novel antiviral innate immunity. Int J Hematol, 2006. 83(3): p. 213-6. [ Links ]
12. Bishop, K.N., et al., APOBEC-mediated editing of viral RNA. Science, 2004. 305(5684): p.5. [ Links ]
13. Lau, P.P., et al., A DnaJ protein, apobec-1-binding protein-2, modulates apolipoprotein B mRNA editing. J Biol Chem, 2001. 276(49): p. 46445-52. [ Links ]
14. Muramatsu M, Kinoshita K, Fagarasan S, Yamada S, Shinkai Y, Honjo T. Class switch recombination and hypermutation require activation-induced cytidine deaminase (AID), a potential RNA editing enzyme. Cell 2000;102: p.553-63 [ Links ]
15. Revy P, Muto T, Levy Y, Geissmann F, Plebani A, Sanal O, Catalan N, Forveille M, Dufourcq-Labelouse R, Gennery A, Tezcan I, Ersoy F, Kayserili H, Ugazio AG, Brousse N, Muramatsu M, Notarangelo LD, Kinoshita K, Honjo T, Fischer A, Durandy A. Activation-induced cytidine deaminase (AID) deficiency causes the autosomal recessive form of the Hyper-IgM syndrome (HIGM2). Cell 2000;102: p.565-75. [ Links ]
16. Arakawa, H., J. Hauschild, and J.M. Buerstedde, Requirement of the activation-induced deaminase (AID) gene for immunoglobulin gene conversion. Science, 2002. 295(5558): p. 1301-6. [ Links ]
17. Prochnow, C., et al., The APOBEC-2 crystal structure and functional implications for the deaminase AID. Nature, 2007. 445(7126): p. 447-51. [ Links ]
18. Mikl, M.C., et al., Mice deficient in APOBEC2 and APOBEC3. Mol Cell Biol, 2005. 25(16): p. 7270-7. [ Links ]
19. Rogozin, I.B., et al., APOBEC4, a new member of the AID/APOBEC family of polynucleotide (deoxy) cytidine deaminases predicted by computational analysis. Cell Cycle, 2005. 4(9): p. 1281-5. [ Links ]
20. Cullen, B.R., Role and mechanism of action of the APOBEC3 family of antiretroviral resistance factors. J Virol, 2006. 80(3): p. 1067-76. [ Links ]
21. Bogerd, H.P., et al., The intrinsic antiretroviral factor APOBEC3B contains two enzymatically active cytidine deaminase domains. Virology, 2007. Aug 1; 364 (2):486-93. [ Links ]
22. Kinomoto, M., et al., All APOBEC3 family proteins differentially inhibit LINE-1 retrotransposition. Nucleic Acids Res, 2007; 35 (9):2955-64. [ Links ]
23. Chen, K., et al., Alpha interferon potently enhances the anti-human immunodeficiency virus tipe I activity of APOBEC3G in resting -primary CD4 T cell. J. Virol 2006. 80(15): p: 7645-7657. [ Links ]
24. Tanaka Y., et al. Anti-viral protein APOBEC3G is induced by interferon-alpha stimulation in human hepatocytes.Biochem Biophys Res Commun. 2006 Mar 10; 341 (2):314-9. [ Links ]
25. Stopak, K.S,. et al. Distinct patterns of cytokine regulation of APOBEC3G expresión and activity in primary lymphocytes, macrophages and dendritic cell. J. Biol. Chem. 282 (6), 3539-3546. [ Links ]
26. Mariani, R., et al., Species-specific exclusion of APOBEC3G from HIV-1 virions by Vif. Cell, 2003. 114(1): p. 21-31. [ Links ]
27. Liu, B., et al., Influence of primate lentiviral Vif and proteasome inhibitors on human immunodeficiency virus type 1 virion packaging of APOBEC3G. J Virol, 2004. 78 (4): p. 2072-81. [ Links ]
28. Schrofelbauer, B., D. Chen, and N.R. Landau, A single amino acid of APOBEC3G controls its species-specific interaction with virion infectivity factor (Vif). Proc Natl Acad Sci U S A, 2004. 101(11): p. 3927-32. [ Links ]
29. Russell, R.A., et al., Foamy virus Bet proteins function as novel inhibitors of the APOBEC3 family of innate antiretroviral defense factors. J Virol, 2005. 79(14): p. 8724-3. [ Links ]
30. Chen, H,. et al,. APOBEC3A is a potent inhibitor of adeno-associated virus and retrotransposons. Curr. Biol. 2006. 16, 480-485. [ Links ]
31. Turelli, P., et al., Inhibition of hepatitis B virus replication by APOBEC3G. Science, 2004. 303(5665): p. 1829. [ Links ]
32. Noguchi, C, et al,. G to A hypermutation of hepatitis B virus. Hepatology. 2005. 41, 626-633. [ Links ]
33. Seifarth, W., et al., Comprehensive analysis of human endogenous retrovirus transcriptional activity in human tissues with a retrovirus-specific microarray. J Virol, 2005. 79(1): p. 341-52. [ Links ]
34. Kazazian, H.H., Jr., Mobile elements: drivers of genome evolution. Science, 2004. 303(5664): p. 1626-32. [ Links ]
35. Dutko, J.A., et al., Inhibition of a yeast LTR retrotransposon by human APOBEC3 cytidine deaminases. Curr Biol, 2005. 15(7): p. 661-6. [ Links ]
36. Schumacher, A.J., D.V. Nissley, and R.S. Harris, APOBEC3G hypermutates genomic DNA and inhibits Ty1 retrotransposition in yeast. Proc Natl Acad Sci U S A, 2005. 102(28): p. 9854-9. [ Links ]
37. Li, X., et al., Frequency of recent retrotransposition events in the human factor IX gene. Hum Mutat, 2001. 17(6): p. 511-9. [ Links ]
38. Harris, R.S., et al., DNA deamination mediates innate immunity to retroviral infection. Cell, 2003. 113(6): p. 803-9. [ Links ]
39. Mangeat, B., et al., Broad antiretroviral defence by human APOBEC3G through lethal editing of nascent reverse transcripts. Nature, 2003. 424(6944): p. 99-103. [ Links ]
40. Holmes, R.K,. APOBEC-mediated viral restriction: not simply editin? Trend. 2007. Vol 32 No 3. [ Links ]
41. Sheehy, A.M., et al., Isolation of a human gene that inhibits HIV-1 infection and is suppressed by the viral Vif protein. Nature, 2002. 418(6898): p. 646-50. [ Links ]
42. Alce, T.M. and W. Popik, APOBEC3G is incorporated into virus-like particles by a direct interaction with HIV-1 Gag nucleocapsid protein. J Biol Chem, 2004. 279(33): p. 34083-6. [ Links ]
43. Svarovskaia, E.S., et al., Human apolipoprotein B mRNA-editing enzyme-catalytic polypeptide-like 3G (APOBEC3G) is incorporated into HIV-1 virions through interactions with viral and nonviral RNAs. J Biol Chem, 2004. 279(34): p. 35822-8. [ Links ]
44. Mbisa, J.L,. et al,. CDNAs produced in the presence of APOBEC3G exhibit defects in plus-strand DNA transfer and integration. J. Virol, 2007. 81(13), 7099-7110. [ Links ]
45. Luo, K.,. et al,. Cytidine deaminases APOBEC3G and APOBEC3F interact with HIV-1 integrase and inhibit proviral DNA formation. J. Virol. 2007. 81(13), 7238-7248. [ Links ]
46. P. An, G. Bleiber, P. Duggal et al. APOBEC3G genetic variants and their influence on the progression to AIDS. J Virol 78 (2004) 11070-11076. [ Links ]
47. H. Do, A. Vasilescu, G. Dipo et al, Exhaustive genotyping of the CEM15 gene and absence of association with AIDS progressión in a french cohort. J. Infect Dis 191 (2005) 159-163. [ Links ]
48. Ya-Lin Chiu, V. Soros, J. Kreisberg et al Cellular APOBEC3G restricts HIV-1 infection in resting CD4+ T cells. Nature 435 (5 de may 2005). [ Links ]
49. Xia Jin, A. Brooks, H. Chen et al. APOBEC3G mRNA levels associated inversely with Human Immunodeficiency Virus Viremia. J. Virol 79 (2005) 11513-11516. [ Links ]
50. M. Biasin, L. Piacentini, S. Caputo, et al. APOBEC3G: A posible role in the resístanse to HIB of HIV- exposed seronegative individuals. J. Infer Dis. 195 (2007) 960-964. [ Links ]
51. Oberste, M.S. and M.A. Gonda, Conservation of amino-acid sequence motifs in lentivirus Vif proteins. Virus Genes, 1992. 6(1): p. 95-102. [ Links ]
52. Von Schwedler, U., et al., Vif is crucial for human immunodeficiency virus type 1 proviral DNA synthesis in infected cells. J Virol, 1993. 67(8): p. 4945-55. [ Links ]
53. Sakai, H., et al., Cell-dependent requirement of human immunodeficiency virus type 1 Vif protein for maturation of virus particles. J Virol, 1993. 67(3): p. 1663-6. [ Links ]
54. Sova, P. and D.J. Volsky, Efficiency of viral DNA synthesis during infection of permissive and nonpermissive cells with vif-negative human immunodeficiency virus type 1. J Virol, 1993. 67(10): p. 6322-6. [ Links ]
55. Madani, N. and D. Kabat, An endogenous inhibitor of human immunodeficiency virus in human lymphocytes is overcome by the viral Vif protein. J Virol, 1998. 72(12): p. 10251-5. [ Links ]
56. Jarmuz, A., et al., An anthropoid-specific locus of orphan C to U RNA-editing enzymes on chromosome 22. Genomics, 2002. 79(3): p. 285-96. [ Links ]
57. Stopak, K., et al., HIV-1 Vif blocks the antiviral activity of APOBEC3G by impairing both its translation and intracellular stability. Mol Cell, 2003. 12(3): p. 591-601. [ Links ]
58. Kao, S., et al., The human immunodeficiency virus type 1 Vif protein reduces intracellular expression and inhibits packaging of APOBEC3G (CEM15), a cellular inhibitor of virus infectivity. J Virol, 2003. 77(21): p. 11398-407. [ Links ]
59. Yu, X., et al., Induction of APOBEC3G ubiquitination and degradation by an HIV-1 Vif-Cul5-SCF complex. Science, 2003. 302(5647): p. 1056-60. [ Links ]
60. Xiao, Z et al., Zinc chelation inhibits VIH Vif activity and lierates antiviral function of the cytidine deaminase APOBEC. Faseb J, 2007. 21 (1): p.217-22 [ Links ]
61. Franca, Raffaella, et al., APOBEC deaminases as cellular antiviral factors: a novel matural host defense mechanism. ;ed Sci Mont, 2006; 132 (5): RA92-98 [ Links ]
AGRADECIMIENTOS
A Wildeman Zapata, Mario Eduardo Archila, por facilitarnos la figura 1 y por las modificaciones realizadas en la misma; a Colciencias por la financiación de este trabajo a través del proyecto código No. 111534319145.
Recibido: Agosto 6 de 2007; Aceptado: Noviembre 28 de 2007
* Autor a quien se debe dirigir la correspondecia: cpatigno@virologia.udea.edu.co