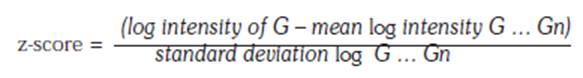Introducción
La obesidad es una condición de salud compleja que, generalmente, se asocia con desórdenes metabólicos como la diabetes y el daño cardiovascular, entre otros; se caracteriza por la acumulación de grasa corporal, producida por un desbalance entre las calorías ingeridas y las eliminadas 1,2. Estudios epidemiológicos han mostrado de manera consistente que ciertas dietas y estilos de vida acentúan el riesgo de obesidad. En este sentido, existe evidencia que el consumo continuo de bebidas azucaradas 3-5, alimentos fritos 6, baja actividad física o sedentarismo 7, interactúan con ciertas variantes génicas en la asociación con la obesidad.
Desde mediados del siglo pasado, la obesidad y el sobrepeso se han convertido en un gran estigma en la mayoría de los países del mundo. Por su parte, la Organización Mundial de la Salud (OMS) la considera una enfermedad dependiendo del lugar del mundo donde se presenta 8. Reportes previos documentan que la obesidad en el mundo se ha duplicado desde 1980. Así pues, en 2014 se estimaba que más de 1,9 millones de adultos mayores de dieciocho años tenían sobrepeso y, de estos, 600 millones eran obesos. Más preocupante todavía es que aproximadamente 41 millones de niños menores de cinco años eran obesos principalmente en países desarrollados 9. Ahora bien, tanto el manejo y como tratamiento de la obesidad son difíciles debido a su naturaleza multifactorial, pues depende de factores genéticos, epigenéticos, ambientales y, también importante, de aspectos socioeconómicos 10-14.
La complejidad de la obesidad se refleja en que el aprendizaje y los hábitos generan estimulación en diferentes áreas del cerebro, cuya respuesta promueve un incremento de la captación de energía y el consecuente aumento de la masa corporal. En estos procesos, el hipocampo y la amígdala están involucrados en el control de señales internas de hambre, memoria y los procesos inhibitorios relacionados con la ingesta alimentaria 15. Diferentes líneas de evidencia sugieren la existencia de una correlación entre la función del sistema dopaminérgico con marcadores de obesidad durante su progresión 16. Se postula que tanto procesos de neuroplasticidad como diferentes endofenotipos individuales estarían jugando un papel importante en el sobrepeso moderado y en la obesidad severa 17.
El tejido adiposo juega un papel importante en muchas de las condiciones patológicas asociadas con la obesidad, actuando como un órgano endocrino integral en la regulación de la homeóstasis energética, la sensibilidad a la insulina y la tolerancia a la glucosa 18,19. Adicionalmente al adipocito, el tejido adiposo blanco contiene pre-adipocitos, células endoteliales, fibroblastos y leucocitos 20. En la condición de obesidad, el número de leucocitos del tejido adiposo se incrementa dramáticamente, promoviendo un estado crónico de un bajo grado de inflamación 21. En este estado, los leucocitos activados, macrófagos y células T secretan interleukina-1 (IL-1), interleukina-6 (IL-6), y factor de necrosis tumoral α (TNFa), que son citosinas inflamatorias 22,23.
El acelerado desarrollo de las ciencias ómicas en el último decenio ha contribuido a un mayor conocimiento de enfermedades complejas como la obesidad. Los análisis de expresión global de genes a partir de experimentos de micromatrices de ADN y de análisis poblacionales de genomas han permitido comenzar a reconocer aspectos dinámicos de la regulación de la expresión de tejido específica de genes y las complejas redes de interacción en diferentes patologías 24-27. En este contexto, el estudio de transcriptomas ha hecho posible abordar la relación entre la expresión global de genes en el tejido adiposo con la obesidad y sus patologías asociadas. Durante la restricción calórica, la adipogénesis y la síntesis de ácidos grasos insaturados se asocia con cambios metabólicos en esta condición 28. Se ha demostrado la asociación de la actividad de la stearoil-CoA desaturasa 1 y los índices de saturación de ácidos grasos en tejido adiposo con la resistencia a insulina en la obesidad 29.
Varios estudios en torno a la obesidad han enfocado sus esfuerzos en encontrar biomarcadores asociados con esta enfermedad que sean útiles en la identificación temprana de individuos susceptibles y puedan agregar valor al riesgo atribuible de desarrollar patologías asociadas 30-32.
Este estudio tuvo como objetivo la analizar la complejidad de expresión de genes asociados con obesidad en el tejido adiposo blanco de personas obesas. El punto de partida de los análisis consignados en este trabajo fueron los valores de expresión global de genes consignados en diferentes experimentos de micromatrices de ADN de genes asociados con diferentes procesos metabólicos y fisiológicos, característicos de la obesidad. Para ello, se realizó una búsqueda bibliográfica en PubMed, en la cual se seleccionaron 37 genes asociados a obesidad, con íold change mayor a 1,5. A partir del cálculo de valores z-score obtenidos de estas micromatrices de ADN, se evaluó la complejidad de la expresión de estos genes. Se construyó una red de interacción génica y se aplicaron pruebas de estadística multivariada, que incluyeron un Análisis de Agrupación y uno Análisis de Componentes Principales. En conjunto, los resultados mostraron que existe una sobreexpresión diferencial de ciertos genes en el tejido adiposo de personas obesas.
Materiales y métodos
En primer lugar, se seleccionaron 39 artículos pertinentes al tema de investigación, publicados entre 2010 y 2016, luego de una búsqueda en la base de datos bibliográficos PubMed del Centro Nacional para la Información Biotecnológica (NCBI). La búsqueda se llevó a cabo usando los siguientes descriptores: obesidad, tejido adiposo, expresión génica, micromatrices de ADN, log>1,5 y valor p<0,05. De los artículos seleccionados, se obtuvo información genómica y funcional correspondiente a los siguientes 37 genes que se asociaron con obesidad y algunas de sus patologías asociadas: ANGPTL4, ADIPOQ, ACACB, ELOVL6, LPL, VEGFA, TNMD, MMP9, FTO, SCD, FASN, NEGR1, TMEM18, MAF, MTCH2, LEP, C9orf116, IL6, PPARGC1A, SOD2, IRAK3, IPO8, KDM2B, ITIH5, KCNMA1, MEDAG, ADCY3, MC4R, BDNF, SH2B1, SULT1A2, HMGCR, NPC1, IGF1, NOS3, NRF1 y KEAP1. Todos ellos tuvieron valores fold change mayores de 1,5 y valores P menores de 0,05.
Las descripciones y características generales de los genes incluidos en este estudio se obtuvieron del Genome Browser de la Universidad de California en Santa Cruz (UCSC). Estas incluyeron el locus cromosómico, el número de exones, el tamaño del gen en pares de bases y su respectivo ARNm en nucleótidos y el total de genes y de repeticiones en el cromosoma correspondiente. Asimismo, se calculó un índice de complejidad de cada gen, aplicando la relación entre el tamaño del ARNm y el tamaño de su gen codificante (Suplemento 1).
A partir de la información correspondiente de las diferentes bases de datos de acceso libre del Genome Browser y del Map Viewer del NCBI se realizó un paseo cro-mosómico en las zonas 100 Kpb upstream y downstream para cada uno de los genes incluidos en este estudio, calculando el número de secuencias repetidas Alu, islas CpG y genes.
Para analizar la expresión de los genes seleccionados y asociados con obesidad, se tomaron los datos de un trabajo depositado por Oñate y colaboradores 25, en la base de datos de acceso libre Gene Expression Omnibus del NCBI. El experimento de micromatrices se ADN, base de este estudio, está registrado bajo el número de plataforma GPL6244, serie GSE48964 que incluye muestras de pacientes con obesidad mórbida (códigos GSM1187673, GSM1187674 y GSM1187675) y muestras control de personas no obesas (códigos GSM1187676, GSM1187677, GSM1187678). De acuerdo con lo reportado en 2013 por Oñate y su equipo, el ARN total de las muestras, se extrajo de cultivos de células troncales derivadas de tejido adiposo blanco subcutáneo de personas obesas que se habían sometido a intervenciones bariátricas.
Los valores log>2,0 consignados en la plataforma GPL6244, serie GSE48964, para cada uno genes asociados a obesidad incluidos en este trabajo, se normalizaron por medio de la transformación zscore de acuerdo con la siguiente ecuación.
Los valores z-score calculados para cada gen se emplearon en la construcción de una red de interacción de genes, por medio de la plataforma Cytoscape 3.2, que es de acceso libre (sitio web: www.cytoscape.org/down-load.php) (33). Los valores de los diferentes parámetros topológicos de la red obtenida se extrajeron por medio de la función Network Analyzer. La información acerca de las interacciones físicas de mayor peso entre los genes evaluados asociados con obesidad se extrajo por medio de la interfase Web GeneMANIA. Esta información se complementó con la contenida en la base de datos Biological General Repository for Interaction Datasets (Bio-GRID) y en la plataforma Protein Interaction Network Analysis platform PINA2. Los valores p estadísticamente significativos obtenidos, luego de aplicar la corrección por técnica de ajuste secuencial de Bonferroni para las primeras 20 categorías ontológicas (GO, por sus siglas en inglés), fueron extraídos utilizando el plugin Biological Networks Gene Ontology tool (BiNGO) de la plataforma Cytoscape 3.2.
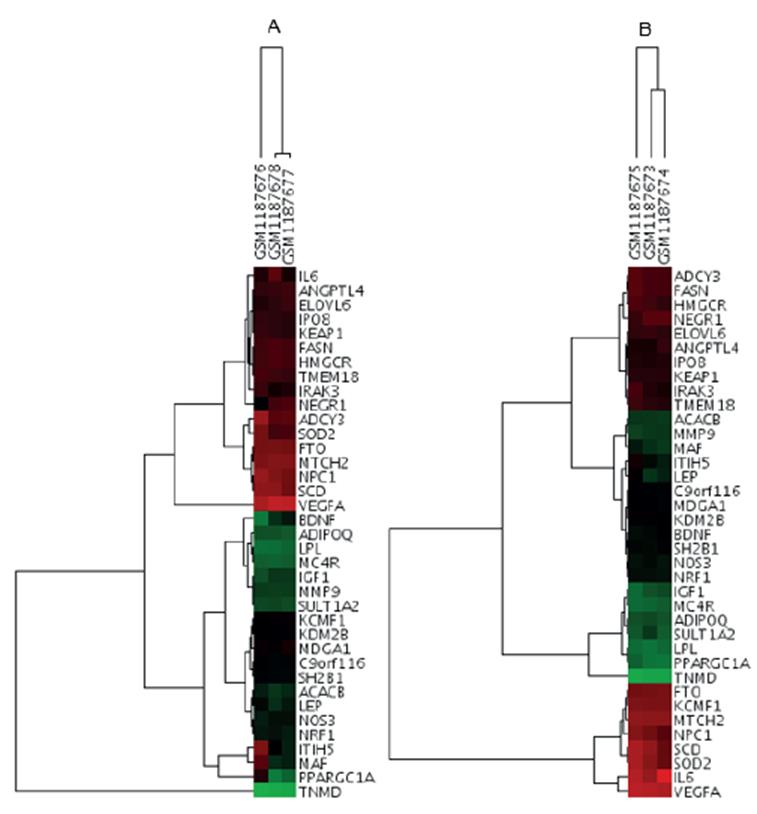
Con el fin de jerarquizar la expresión de cada uno de los genes en los dos grupos evaluados, se efectuó la evaluación de su significancia estadística mediante pruebas de estadística multivariable incluyendo un Análisis de Componentes Principales (ACP), llevado a cabo con el programa SPSS 20.0. Para visualizar las agrupaciones diferenciales de aquellos genes con mayor o menor expresión en tejido adiposo de personas obesas y del grupo control, se efectuó un análisis de agrupación obteniendo mapas de calor Heat-Map, disponible en la misma plataforma Cytoscape 3.2.
Resultados
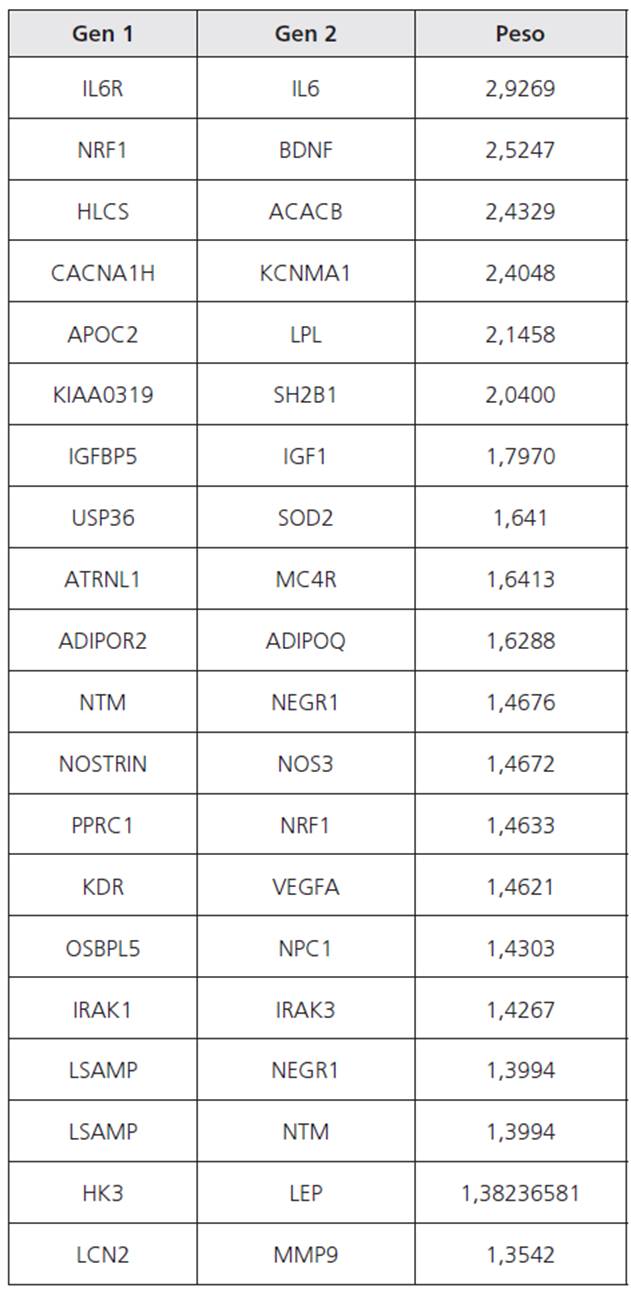
En la red construida se observaron dos nodos principales, con cantidad significativamente alta de interacciones: el más grande tiene como centro la proteína NRF1 (figura 1). Además, se destacaron por su elevada expresión los genes que codifican para VEGFA (z-score 2,02), FTO (z-score 1,31), NEGR1 (z-score 0,88), MTCH2 (z-score 1,57), IL6 (z-score 2,15) y NPC1 (z-score 1,25). De los valores de interacción obtenidos de la red de genes asociados con obesidad, la proteína que codifica el gen factor respiratorio nuclear 1 (NRF1) fue la que obtuvo mayor número de interacciones, en total 908; seguida por el canal activado por potasio de la subfamilia M alfa (1KCNMA1), con 140 interacciones; y, en tercer lugar, el ácido grado (FASN) con 127 interacciones (figura 2). Se determinó que los pares de interacciones físicas con mayor peso ocurrieron en los genes asociados con obesidad interleukina-6 (IL6) y su receptor IL6R (peso de 2,9269) y entre el factor nuclear respiratorio 1 (NRF1) y el Factor Neurotrópico Derivado del Cerebro BDNF (peso de 2,5247). Los datos se presentan en la tabla 1. Se agrega que se encontraron otras reacciones de importancia como entre NOSTRIN y NOS3 y LEP con HK3.
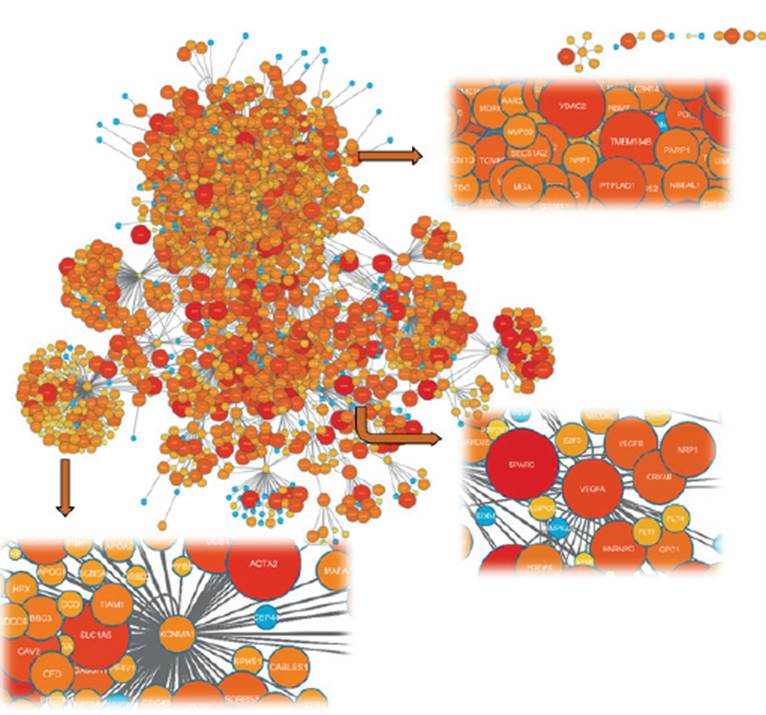
Figura 1 Red de interacción y expresión de proteínas codificadas por genes asociados con obesidad, obtenida por medio del software Cytoscape 3.2. La expresión de los genes proviene de células troncales del tejido adiposo blanco de personas obesas, tomadas del estudio de Oñate y colaboradores (25). Se resaltan los nodos KCNMA1, NRF1 y VEGFA. El color rojo indica mayor expresión y el amarillo, menor.
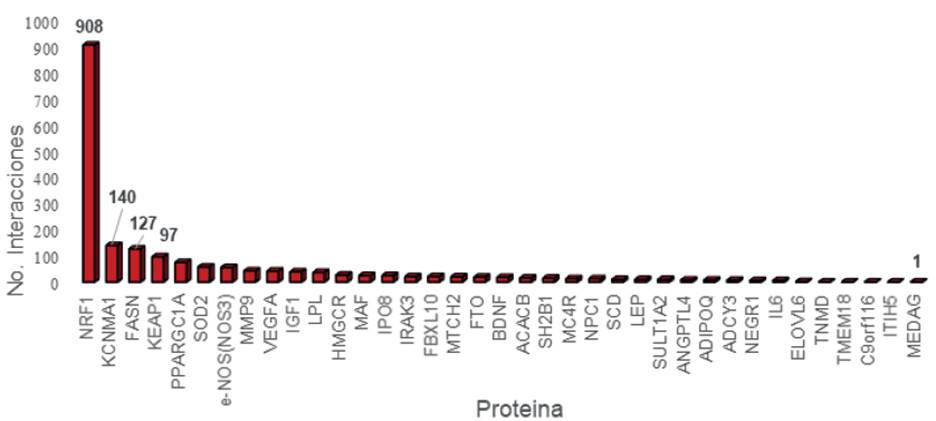
Figura 2 Número de interacciones proteicas encontradas de las proteínas asociadas con obesidad evaluadas en este estudio, obtenidas a partir de las bases de datos de BlOGrid y PINA2. La proteína NRF1 presentó la mayor cantidad de interacciones (90S).
Tabla 1 Valores de peso de los pares de interacciones físicas de proteínas codificadas por genes asociados con obesidad, empleando la interfase GeneMANÍA.
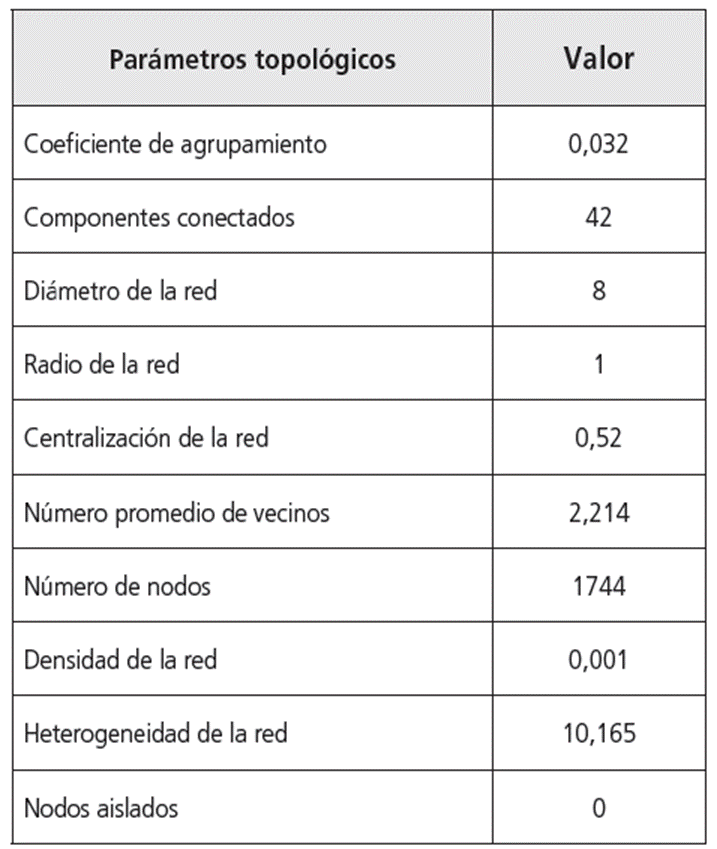
Los valores de los parámetros topológicos de la red mostraron una topología compleja que se aglutina: el número total de nodos fue de 1744 y el número promedio de vecinos de cada nodo de 2 214. El valor de centralización de la red fue significantemente alto: 0,52, lo que indica que la red, en su mayoría, está centrada en uno de los nodos. En este caso, el gen nodal es NRF1, como se muestra en la tabla 2.
Tabla 2 Datos topológicos de la red construida con Cytoscape 3.2, tomando como base los valores de expresión de 37 genes asociados con obesidad de células troncales de tejido adiposo blanco de individuos con obesidad mórbida.
En cuanto a las categorías ontológicas (GO) extraídas de la red se destacaron procesos biológicos muy relacionados con eventos patológicas ligados a la obesidad. Entre estos cabe resaltar 1) el proceso metabólico de lipoproteínas, 2) el ciclo de los ácidos tricarboxílicos, 3) la activación de las MAP-quinasas, 4) la cascada JNK y 5) la acumulación de células T, como se muestra en la tabla 3. El número de dinucleótidos CpG en la región de 10Kpb de la zona upstream fue mucho más alto, en comparación no solo con la zona 10 Kpb downstream sino también con el resto de las zonas. El número de secuencias Alu y de genes fue muy similar en todos los genes analizados durante el paseo cromosómico realizado (figura 3).
Tabla 3 Las veinte principales categorías ontológicas extraídas de la red de obesidad y sus respectivos valores de p obtenidos con la corrección por técnica de ajuste secuencial de Bonferroni. Los valores de p se obtuvieron ejecutando la aplicación con el plugin BiNGO de Cytoscape 3.2.
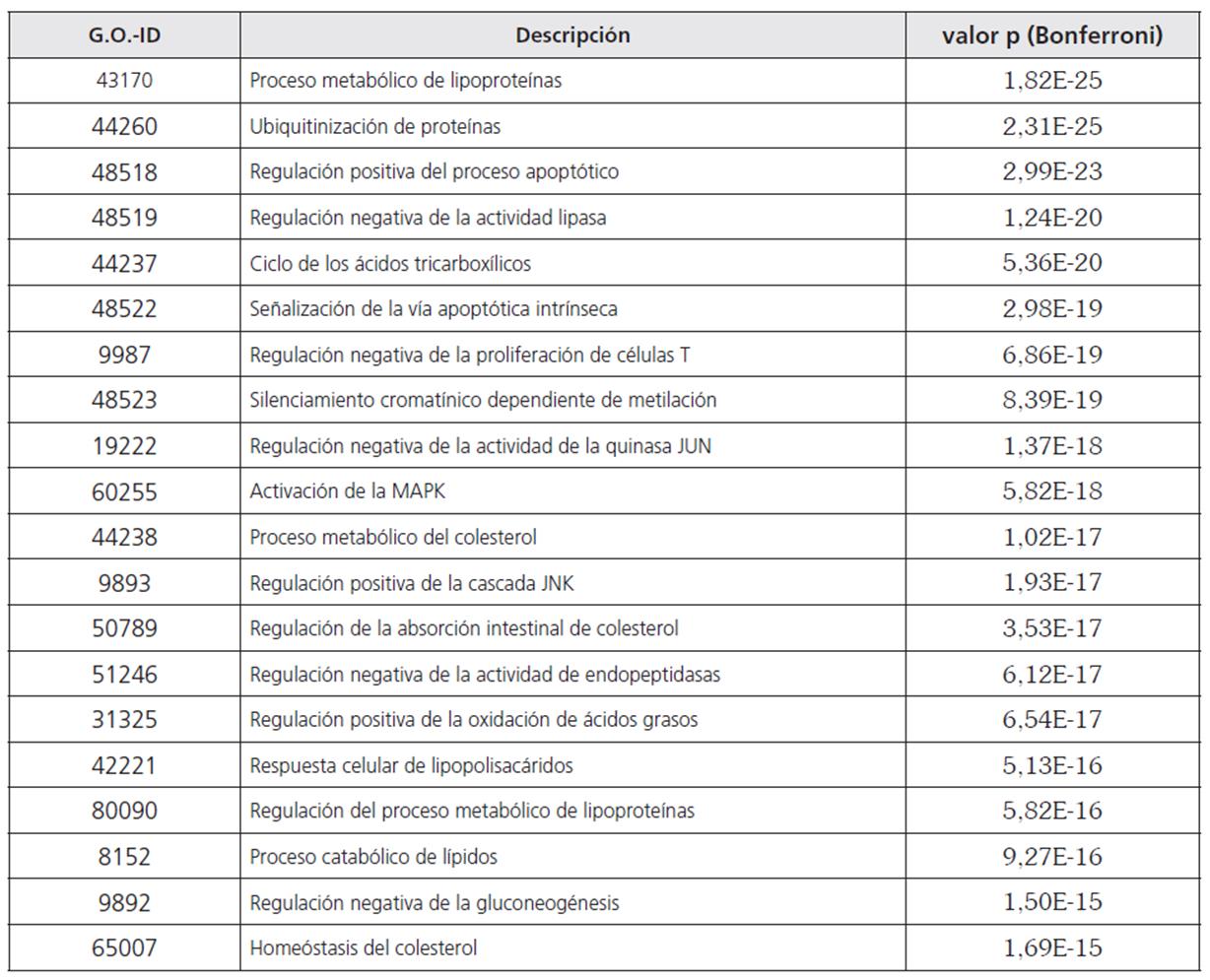
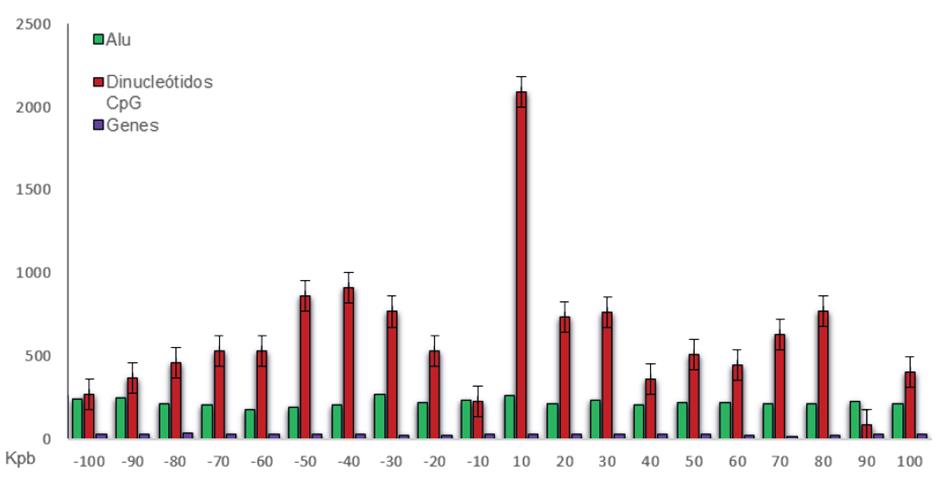
Figura 3 Paseo cromosómico realizado en una ventana de 100 Kpb, rodeando cada uno de los 37 genes evaluados, tanto corriente arriba como corriente abajo. Se tomaron en cuenta secuencias repetidas Alu, islas CpG y genes, con pasos de 10 Kpb utilizando información del Genome Browser de la UCSC y del Map Viewer del NCBl.
El ACP permitió reducir a 2 componentes los 37 genes evaluados en ambos grupos. Se pudo establecer estadísticamente que la contribución de la varianza de los genes asociados con obesidad en los dos componentes fue diferente entre personas obesas y el grupo de control. En los primeros, el componente 1 incluyó 21 genes, de los cuales ACACB, HMGR, IRAK3, ITH5, KDM2B, KEAP1, MAF, NFR1 y C9orf116 fueron los que tuvieron mayores valores de contribución a la varianza. Lo anterior, en contraste con el componente 2, que incluyó 11 genes de los cuales IL6, IPO8, LEP, ADCY3, AGPTL4, BDNF, FASN, FTO y KCMF1 presentaron mayores valores de contribución a la varianza total del componente. El gráfico biplot mostró que existen agrupaciones diferenciales de coexpresión entre personas obesas y las del grupo de control. En personas obesas se observó un grupo conformado por LEP, KCMF1, ITH55, FASN, MAF e IRAK3; diferente al del grupo de control, que incluyó a los genes FTO, SCD, ADPQ, SHB e IPO8 (figura 4).
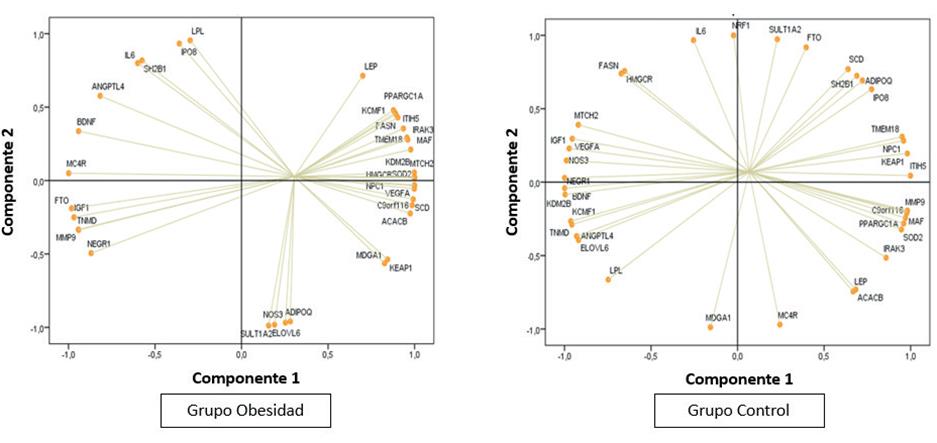
Figura 4 Gráficos Biplot del análisis de componentes principales para los grupos de obesidad y control, obtenidos con el programa SPSS v22.0.
En el Heat Map (mapa de calor), el análisis de agrupación reveló agrupaciones en dos diferentes bloques. El gen IL6 se encontró expresado diferencialmente, con mayor expresión en personas obesas, en comparación con el grupo de control (figura 5). Es también de gran importancia resaltar los genes VEGFA, SOD2, SCD, KCNMA1 y FTO, los cuales están altamente expresados en personas obesas. En general estos genes están involucrados en los procesos de angiogénesis, control del índice de masa corporal (IMC) y metabolismo.
Discusión
La evidencia obtenida de los análisis desarrollados en este trabajo permite asociar los genes IL6, NFR1, VE-GFA, y FTO con la fisiopatología de la obesidad. Un resultado importante y que confirma hallazgos bioquímicos previos fue la sobrexpresión del gen de IL6 en las células troncales, provenientes de individuos obesos, incluidos en el análisis. Este hallazgo permite su postulación como biomarcador potencial para un diagnóstico clínico proteómico, debido a que su expresión fue estadísticamente significativa en las células troncales de tejido adiposo blanco de personas obesas. La mayor abundancia de secuencias CpG y Alu, de 20-40 Kpb de la región del genoma humano en la cual se localizan estos genes, sugiere indirectamente la existencia de un posible mecanismo de regulación epigenética por metilación de las CpG localizadas en los promotores de genes asociados en personas obesas. También se sugiere que el mecanismo de regulación de los genes evaluados podría ser por metilación de las islas CpG ubicadas en sus promotores. Previamente, se han reportado potenciales interacciones entre el ambiente y mecanismos epigenéticos que regulan la expresión de genes asociados con el incremento del IMC y la obesidad.
También se ha determinado una regulación epigenética en el gen FTO que codifica una ADN desmetilasa 34 y en el gen MC4R. Dicha regulación reduce la metilación a causa de la acción a largo plazo de una dieta alta en grasas 35. Este efecto, en conjunto con la acetilación histónica mediada por el receptor gamma de proliferación de peroxisomas -PPARy 36- sobre los genes POMC (37) y LEP 38, remarcan la importancia de la metilación del ADN en los promotores de estos genes que se caracterizan por una riqueza de islas CpG, los cuales son potenciales blancos de metilación mC5".
Es importante mencionar las proteínas que resultaron con mayor número de interacciones físicas están involucradas en diferentes procesos y vías metabólicas, dife-rencialmente reguladas. Una de ellas es el NRF1, proteína implicada en el metabolismo de lípidos en el que su sobreexpresión se relaciona con la imposibilidad de ganar peso 39. De otra parte, el FASN, una enzima que participa de procesos de lipogénesis y cuya expresión en tejido adiposo está relacionada con la acumulación de grasa visceral, incremento de la IL6, leptina y RBP4 circulatoria, lo que sugiere un rol central de las rutas lipogénicas en la relación entre las consecuencias del exceso de energía y el desarrollo de obesidad y diabetes tipo 2 40.
Otra interacción importante fue la de VEGFA con su receptor VEFGR2 (KDR). Previamente se ha demostrado que, durante la expansión de grasa en personas obesas, la vascularización del tejido adiposo es insuficiente para mantener la normoxia tisular, lo que genera una hipoxia local que puede conllevar a una expresión alterada de adipocinas, reclutamiento de macrófagos proinflamato-rios y resistencia a la insulina, una de las patologías asociados con la obesidad 41.
El presente análisis confirmó la importancia del gen que codifica para el IL6 y su interacción con el ILR6. Además, se determinó que el gen IL6 tuvo una mayor expresión en tejido adiposo de personas obesas, en comparación con el tejido de las del grupo de control. Lo anterior lo hace un potencial biomarcador de obesidad, que podría estudiarse a futuro en personas con obesidad, incluyendo un rango más amplio de edades que permita determinar si el IL6 se sobreexpresa incluso en la niñez. En el estudio de Sindhu y sus colaboradores 42, se demostró por medio de técnicas inmunohistoquímicas, RT-PCR y microscopía confocal, que la obesidad es un modulador positivo de la expresión de los genes IL6 e ILR6 en tejido adiposo, lo cual podría ser un mecanismo que contribuya a la inducción de inflamación metabólica. Esta interleukina es secretada principalmente por adipocitos, especialmente del tejido adiposo visceral, de dos o tres veces más IL-6, en comparación con el tejido adiposo subcutáneo 43. En la obesidad, parece que el estado inflamatorio se origina a partir de estas células, que son activadas por macrófagos M1, los cuales, a su vez, producen citocinas proinflamatorias como TNF-α e IL-6, lo que produce una conexión entre obesidad, inflamación y resistencia a la insulina 44-45.
Se ha encontrado relación del gen FTO con la predisposición a la diabetes y se ha demostrado que modula el IMC 46 y el apetito 47. Además, los estudios realizados por Melhorn y sus colaboradores 48 y Cyrus y sus colaboradores 49 reportaron que variantes en el gen FTO aumentan el riesgo de obesidad debido a un mal procesamiento del sistema nervioso, en cuanto a la saciedad, lo que aumenta la ingesta de comida. Se ha reportado que niveles bajos de FTO en ratones conducen, entre otros efectos, a una reducción significativa del tejido adiposo y masa corporal baja. Por otro lado, el gen NEGR1 codifica para una proteína miembro de la superfamilia de inmunoglobulinas sobreexpresada en el hipotálamo, donde parece que modula el número de sinapsis en neuronas. En este sentido, nuestros resultados aportan una evidencia fuerte de que el gen NEGRI sería buen candidato funcional en la obesidad, especialmente, si se tiene en cuenta el origen neurológico de esta enfermedad 50.
En nuestro estudio se determinó que las categorías GO más importantes se asociaron con procesos metabólicos alterados en la obesidad. Es así como el proceso metabólico de lipoproteínas es esencial para la correcta metabolización de grasas, colesterol y glúcidos ingeridos 51, cualquier alteración en este proceso podría, junto con otros factores, conllevar a la mala asimilación y posterior acumulación de estos en el cuerpo y al desarrollo de sobrepeso. Otra categoría GO importante fue los procesos que ocurren en el ciclo de los ácidos tricarboxílicos, conocido como embudo del metabolismo, en el que se catabolizan, entre otros, ácidos grasos para la correspondiente producción de energía para la célula 52, además de algunos intermediarios que se convierten en la base de moléculas como la glucosa, ácidos grasos y aminoácidos no esenciales. De otra parte, la activación de las MAP-quinasas y la cascada JNK puede darse por el exceso de especies reactivas de oxígeno (ROS), generadas, a su vez, por la elevada acumulación de lípidos 53,54.
Comprender las complejas interacciones entre la alimentación y la salud es de gran importancia para promover el desarrollo de intervenciones nutricionales más eficientes. que promuevan el bienestar y que puedan reducir los factores de riesgo para enfermedades crónicas no trasmisibles asociadas con la obesidad y que promueven una la alteración de la homeóstasis metabólica. Los resultados obtenidos validan la importancia de la aplicación de herramientas bioinformáticas para poder encontrar evidencias sobre la expresión diferencial tejido específica y las interacciones funcionales que ocurren entre genes, las cuales contribuyen a un estado metabólico normal. La simulación computacional de resultados de experimentos de micromatrices de ADN, consignados en distintas bases de datos y el empleo de plataformas ómicas que se emplearon en este trabajo, abren un nuevo camino para abordar la obesidad como un evento de múltiples causas que genera alteraciones importantes en la expresión de genes claves en el metabolismo energético y cuyo manejo debe incluir nuevos enfoques desde las ciencias ómicas y la bioinformática.