Introducción
En los humanos, el cromosoma 2 es el segundo más grande, después del cromosoma 1 que representa, aproximadamente, el 8,0 % del material genético. El cromosoma 2 posee una longitud de 242 193 529 pb, un total de 1301 genes codificantes ,1578 genes no codificantes y 1075 pseudogenes 1. Se caracteriza por ser un cromosoma autosómico y, por tanto, no influye en la determinación del sexo los individuos.
El mapeo del cromosoma 2 ha permitido reconocer que su alteración, tanto duplicación como deleción, puede conducir a una serie de enfermedades asociadas 2; mientras tanto, el análisis genético de dichas enfermedades puede tener un valor pronóstico importante, así como la realización adecuada del consejo genético. Las enfermedades asociadas al cromosoma 2 pueden dividirse con base en su localización, es decir, localizadas en el brazo corto o en el brazo largo, haciendo referencia a los locus donde se encuentran.
Una de las enfermedades asociadas al cromosoma 2 es el síndrome 2p25, un trastorno raro y clínicamente heterogéneo, que se manifiesta por material genético adicional en porciones de tamaño variable del brazo corto de este cromosoma. Las características comunes de esta patología incluyen retraso del desarrollo, características dismórficas craneofaciales (incluido hipertelorismo y frente prominente) y malformaciones cardiovasculares. La duplicación 2p25 se había informado hasta 2016 solo en diez pacientes, como una aberración aislada de la región terminal de 2p 3.
El síndrome de microdeleción 2q37 es también una enfermedad asociada al cromosoma 2, que puede afectar muchos órganos del cuerpo. Esta enfermedad es causada por una deleción del material genético de la región específica q37 en el brazo largo (q) de este cromosoma. La deleción ocurre cerca del final del cromosoma (deleción terminal) en el lugar designado 2q37 4. Hasta 2013, se habían reportado cerca de 60 casos de deleción o monosomía 2q37, resultantes de translocaciones desequilibradas. La variabilidad significativa en la presentación clínica es evidente, pero casi todos los pacientes tienen algún grado de retraso mental y dismorfia facial. Las anomalías congénitas del corazón, con respecto a este síndrome, están presentes en aproximadamente el 20 % de los pacientes. La mayoría de las personas con este síndrome tienen una deleción de novo, lo que significa que no es heredado y que sus padres tienen cromosomas normales 5. Esta deleción cromosómica ocurre como un acontecimiento al azar, durante la formación de las células reproductoras o durante el desarrollo fetal temprano. Aproximadamente en 5 % de los casos publicados, los pacientes heredan la deleción de un padre portador de una translocación equilibrada.
La presencia de malformaciones congénitas múltiples y su gravedad son los factores más importantes para el pronóstico y para determinar la esperanza de vida de los pacientes. Los reportes de pacientes adultos con estos síndromes son pocos; por lo que se hace necesario conocer más sobre la historia natural de la enfermedad para suministrar la atención integral transdisciplinaria adecuada, para los pacientes afectados por estos defectos genéticos y sus familias 6.
Esta es la primera vez que se describe un reordenamiento cromosómico con una duplicación (2p25.3) y una microdeleción (2q37.3.) en un mismo individuo. De allí la importancia de su caracterización fenotípica, descripción sindrómica y análisis genómico bioinformático, para poder instaurar tratamientos dirigidos que impacten en la morbilidad y mortalidad de los pacientes y para poder realizar la consejería genética.
Metodología
Se determinaron las características fenotípicas de interés de una paciente, mediante las cuales se pudiera llegar a un diagnóstico preciso. Posteriormente, se hizo la cuantificación de actividad enzimática para (β-galactosidasa, galactosa-6-sulfato sulfatasa y arilsulfatasa B, por métodos fluorométricos, además de realizar la cuantificación de hormonas TSH, T4L, TGO-AST y TGP-ALT en plasma.
Se llevó a cabo un estudio molecular de hibridación genómica comparativa (HGC), basado en matrices que incluyen 180 000 oligonucleótidos o sondas, diseñadas con la secuencia complementaria a las 180 000 mutaciones más frecuentes del genoma, con el objetivo de indagar los 1714 genes que presentan mayor compromiso con el desarrollo de enfermedades genéticas, 700 micro-RNA y todo el genoma mitocondrial. Esta técnica permite detectar la pérdida de un número variable de copias (deleción), ganancia de números variables de copias (duplicación) o establecer el número normal de copias.
Finalmente, se realizó un estudio genómico bioinformático utilizando las herramientas Clinvar, Genecards, Online Mendelian Inheritance in Man (OMIM) y Human Gene Mutation Database (HGMD) con el fin de identificar las funciones descritas de los genes involucrados y se evaluaron las redes de expresión genómicas entre los genes afectados, utilizando el software STITCH 5 (http://stitch.embl.de/).
Resultados
En la evaluación realizada a una paciente de sexo femenino, de 18 años, se encontró una talla de 145 cm (desviación estándar [DE]: 21,21 cm), 36 kg de peso (DE: 12 Kg) y 17, 12 kg/m2 de índice de masa corporal (IMC), lo que indica delgadez, según la resolución colombiana 2465 de 2016.
El análisis de exploración física indicó presencia de facies dismórfica, con cara abotagada y cuello corto; además se reportó retraso psicomotor moderado y autismo, microadenoma hipofisiario con engrosamiento fusiforme del infundíbulo de aproximadamente 4,7x2,5 mm, que corresponde a neurohipófisis ectópica, observada en la resonancia nuclear de base de cráneo. Asimismo, se evidenció agenesia renal unilateral izquierda, aracnodactilia en manos y pies, además de la ausencia de ovario izquierdo, uterodesviación izquierda y afecciones por cardiopatías.
Al realizar la cuantificación por métodos fluo-rométricos de actividad enzimática para (β-galactosidasa, galactosa-6-sulfato sulfatasa y arilsulfatasa B a partir de leucocitos, se observó normalidad en los resultados enzimáticos, pues se encontraban dentro los valores de referencias normales. Asimismo, se cuantificaron las hormonas TSH, T4L, TGO-AST y TGP-ALT plasmáticas, lo cual también arrojó resultados normales.
El estudio molecular de deleciones y duplicaciones por la técnica de hibridación genómica comparativa (HGC) determinó una duplicación patogénica en la región cromosómica 2p25.3p24.3, con coordenadas genómicas Chr2:17019_16364856 de 16,34 Mb, relacionada con la afección de 36 genes (Tabla 1). Adicionalmente se identificó una deleción patogénica en la citobanda 2q37.3, coordenadas genómicas Chr2: 238043371_243040276 de 4,99 Mb; deleción relacionada con la afección de 36 genes (Tabla 2).
Tabla 1 Genes afectados por la duplicación 2p25.3p24.3
| Localización cromosómica | Tipo de CNV | Clasificación CNV | Genes afectados |
|---|---|---|---|
| 2p25.3p24.3 | Duplicación | Patogénica | ACP1, TMEM18, TPO, PXDN, MYT1L, TSSC1, TRAPPC12, ADI1, COLEC11, ALLC, SOX11, CMPK2, RSAD2, ID2, KIDINS220, MBOAT2, ITGB1BPT, ADAM17, GRHL1, K1F11, RRM2, HPCAL1, ODCI, NOL10, PDIA6, KCNFL1, ROCK2, GREB1, NTSR2, LPIN1, FAM84A, NBA5, DDX1, MYCNUT, MYCNOS, MYCN, GACAT |
Tabla 2 Genes afectados por la deleción 2q37.3
| Localización cromosómica | Tipo de CNV | Clasificación CNV | Genes afectados |
|---|---|---|---|
| 2q37.3 | Deleción | Patogénica | COL6A3, MLPH, RAB17, LRRFIP1, RAMP1, SCLY, FAM132B, HES6, PER2, TRAF3IP1, ASB1, TWIST2, HDAC4, NDUFAX0, GPC1, MIR149, RNPEPL1, CAPN10, GPR35, AQP12A, KIF1A, AGXT, SNED1, MTERD4, PASK, PPP1R7, HDLBP, SEPT2, STK25, THAP4, DTYMK, ING5, 02HGDH, GAL3ST, NEU4, PDCD1 |
Esta es la primera vez que se reporta la aparición de estas dos variantes en un mismo individuo, en el número de copias simultáneamente, por lo que se describieron las alteraciones por separado para llegar a un cuadro clínico que pudiera explicar el fenotipo de la paciente y su etiología molecular.
La identificación de las funciones descritas de los genes involucrados en la duplicación 2p25.3p24.3 (Tabla 3) y la deleción en 2q37.3 (Tabla 4) mostró la afección de genes implicados en la regulación de procesos celulares, como el crecimiento celular, la senescencia, la diferenciación, la apoptosis, la angiogénesis y la transformación neoplásica, así como múltiples procesos asociados a factores de crecimiento y afecciones en los patrones circadianos, que resultan afectados por estas duplicaciones-deleciones.
Tabla 3 Funciones descritas para genes afectados por la duplicación 2p25.3p24.3
| Gen | Número OMIN | Nombre del gen | Localización cromosómica | Función |
|---|---|---|---|---|
| ACP1 | *171500 | Acid phosphatase 1 | 2p25.3 | Actúa sobre proteínas fosforiladas en tirosina, arilfosfatos de bajo peso molecular y acilfosfatos, naturales y sintéticos. |
| TMEM18 | *613220 | Transmembrane protein 18 | 2p25.3 | Afecta los niveles de energía a través de la señalización de insulina y glucagón. |
| TPO | *606765 | Thyroid peroxidase | 2p25.3 | Yodación de los residuos de tirosina, para generar las hormonas tiroideas, tiroxina y triyodotironina. |
| PXDN | *605158 | Peroxidasin | 2p25.3 | Formación de la matriz extracelular y puede funcionar en la respuesta fibrogénica fisiológica y patológica en el riñón fibrótico. |
| MYT1L | *613084 | Myelin transcription factor 1-like | 2p25.3 | Funcionan en el sistema nervioso central de los mamíferos en desarrollo. |
| TSSC1 | *608998 | Earp complex and garp complex interacting protein 1 | 2p25.3 | Supresión de tumores. |
| TRAPPC12 | *614139 | Trafficking protein particle complex 12 | 2p25.3 | Ensamblaje del cinetocoro y estabilidad; controla el reclutamiento de CENPE para los cinetocoros. |
| ADI1 | *613400 | Acireductone dioxygenase 1 | 2p25.3 | Regula el procesamiento de ARNM en el núcleo. |
| COLEC11 | *612502 | Collectin 11 | 2p25.3 | Migración de las células de la cresta neural. |
| ALLC | *612396 | Allantoicase | 2p25.3 | Soporte en la degradación del ácido úrico. |
| SOX11 | *600898 | Sry-box 11 | 2p25.3 | Actores de transcripción involucrados en la regulación del desarrollo embrionario y en la determinación del destino celular. |
| CMPK2 | *611787 | Cytidine/uridine monophosphate kinase 2 | 2p25.3 | Participa en la síntesis de DUTP y DCTP en las mitocondrias. |
| RSAD2 | *607810 | Radical S-adenosyl methionine domaincontaining protein 2 | 2p25.2 | Promueve la producción dependiente de tlr7 y tlr9 de la producción de IFN-β en células plasmocitoides dendríticas (pdc) al facilitar la ubiquitinación ligada a Lys-63 de irak1. Juega un papel en la activación y diferenciación de las células T cd4. |
| ID2 | *600386 | Ιnhibitor of DNA binding-1 | 2p25.1 | Implicado en la regulación de una variedad de procesos celulares, incluido el crecimiento celular, la senescencia, la diferenciación, la apoptosis, la angiogénesis y la transformación neoplásica. |
| KIDINS220 | *615759 | Kinase D-interacting substrate | 2p25.1 | Desempeña un papel importante en la regulación de la transducción de señales postsinápticas a través de la localización mediada por sintrofina de las tirosina quinasas receptoras, como la EPHA4. |
| MBOAT2 | *611949 | Membrane-bound O-acyltransferase domain-containing protein 2 | 2p25.1 | Cataliza también la acilación del ácido lisofosfatídico en ácido fosfatídico (actividad lpaat). |
| ADAM17 | *603639 | A disintegrin and metalloproteinase domain 17 | 2p25.1 | Funciona en el procesamiento de numerosos sustratos, incluidas las proteínas de adhesión celular, los receptores de citoquinas y factores de crecimiento y los ligandos del receptor del factor de crecimiento epidérmico. |
| GRHL1 | *609786 | Grainyhead like transcription factor 1 | 2p25.1 | Factor de transcripción durante el desarrollo. |
| RRM2 | *180390 | Ribonucleotide reductase regulatory subunit M2 | 2p25.1 | Proporciona los precursores necesarios para la síntesis de ADN. Cataliza la biosíntesis de desoxirribonucleótidos a partir de los ribonucleótidos correspondientes. |
| HPCAL1 | *600207 | Hippocalcin like 1 | 2p25.1 | Participar en la regulación dependiente de calcio de la fosforilación de rodopsina. |
| NOL10 | *616197 | Nucleolar protein | 2p25.1 | Ensamblaje de partículas ribosómicas. |
| PDIA6 | *611099 | Protein disulfide isomerase family A member 6 | 2p25.1 | Chaperona que inhibe la agregación de proteínas mal plegadas. Regula negativamente la respuesta de la proteína desplegada. |
| ROCK2 | *604002 | Rho associated coiledcoil containing protein kinase 2 | 2p25.1 | Regula la citocinesis, la contracción del músculo liso, la formación de fibras de estrés de actina y las adherencias focales. |
| GREB1 | *611736 | Growth regulating estrogen receptor binding 1 | 2p25.1 | Proliferación celular estimulada por estrógenos. Actúa como un regulador del crecimiento del cáncer dependiente de hormonas en los cánceres de mama y próstata. |
| NTSR2 | *605538 | Neurotensin receptor 2 | 2p25.1 | Se asocia con proteínas G que activan un sistema de segundo mensajero de fosfatidilinositol y calcio. |
| LPIN1 | *605518 | Lipin 1 | 2p25.1 | Control del metabolismo de los ácidos grasos en diferentes niveles. Actúa como una enzima fosfatasa dependiente de magnesio que cataliza la conversión de ácido fosfatídico a diacilglicerol. |
| FAM84A | *611234 | Family with sequence similarity 84 member A | 2p24.3 | Desempeña un papel en la migración celular. |
| DDX1 | *601257 | DEAD-box helicase 1 | 2p24.3 | Este gen codifica una proteína de DEAD-box de función desconocida. |
| MYCNUT | *615968 | MYCN upstream transcript | 2p24.3 | Control de neuroblastomas. |
| MYCNOS | *605374 | MYCN opposite strand | 2p24.3 | Regula la estabilidad de mycn en células de neuroblastoma mediante la inhibición de la fosforilación de mycn mediada por GSK3B. |
| MYCN | *164840 | MYCN proto-oncogene, BHLH transcription factor | 2p24.3 | Regularmente regula la transcripción de MYCNOS en células de neuroblastoma. |
Tabla 4 Funciones descritas para genes afectados por la deleción en 2q37.3
| Gen | Número OMIN | Nombre del gen | Localización cromosómica | Función |
|---|---|---|---|---|
| COL6A3 | *120220 | Collagen type vi alpha 3 chain | 2q37.3 | Colágeno tipo vi, alfa 3 (300 kDa), microfibrilar, supuestamente gen diana c-Myc regulado por disminución. |
| MLPH | *606526 | Melanophilin | 2q37.3 | Implicada en el transporte de melanosomas. Sirve como enlace entre rab27A unido a melanosoma y la proteína motora myo5A. |
| RAB17 | *602206 | Ras-associated protein | 2q37.3 | Controla el tráfico de membrana a través de endosomas apical de reciclaje. |
| LRRFIP1 | *603256 | LRR binding flii interacting protein 1 | 2q37.3 | Regula la expresión de TNF, EGFR y PDGFA. Puede controlar la proliferación de las células musculares lisas. |
| RAMP1 | *605153 | Receptor activity modifying protein 1 | 2q37.3 | Actúa como un receptor para el péptido relacionado con el gen de la calcitonina. |
| SCLY | *611056 | Selenocysteine lyase | 2q37.3 | Cataliza la descomposición de L-selenocisteína a L-alanina y selenio elemental. |
| FAM132B | *615099 | Erythroferrone | 2q37.3 | Hormona reguladora del hierro que actúa como un regulador eritroide. |
| HES6 | *610331 | Hes family BHLH transcription factor 6 | 2q37.3 | Promueve la diferenciación celular. |
| PER2 | *603426 | Period circadian regulator 2 | 2q37.3 | Se expresa en un patrón circadiano en el núcleo supraquiasmático. |
| TRAF3IP1 | *607380 | traf3 interacting protein 1 | 2q37.3 | Participa en la supresión de la fosforilación de stat6 inducida por il-13, la actividad transcripcional y la unión al adn. Recluta traf3 y disc1 a los microtúbulos. Participa en el desarrollo renal y en la morfogénesis epitelial. |
| ASB1 | *605758 | Ankyrin repeat and SOCS box containing 1 | 2q37.3 | degradación proteasomal de las proteínas diana. |
| TWIST2 | *607556 | Twist family bhlh transcription factor 2 | 2q37.3 | Participa en el almacenamiento de glucógeno posnatal y el metabolismo energético. |
| HDAC4 | *605314 | Histone deacetylase 4 | 2q37.3 | Desempeña un papel importante en la regulación transcripcional, la progresión del ciclo celular y los eventos de desarrollo. |
| GPC1 | *600395 | Glypican 1 | 2q37.3 | Participa en la mielinización de células de Schwann. |
| MIR149 | *615209 | Micro-RNA 149 | 2q37.3 | Supresor de tumores. |
| RNPEPL1 | *605287 | Arginyl aminopeptidase like 1 | 2q37.3 | Liberación de aminoácidos N-terminales, preferentemente metionina, a partir de péptidos y arilamidas. |
| CAPN10 | *605286 | Calpain 10 | 2q37.3 | Captación de glucosa estimulada por la insulina. |
| GPR35 | *602646 | G protein-coupled receptor 35 | 2q37.3 | Actúa como un receptor para el ácido kinurénico. |
| AQP12A | *609789 | Aquaporin 12A | 2q37.3 | Facilitan el transporte de agua y pequeños solutos neutros a través de las membranas celulares. |
| KIF1A | *601255 | Kinesin family member 1ª | 2q37.3 | Motor para el transporte axonal anterógrado de precursores de vesículas sinápticas. |
| AGXT | *604285 | Alanine-glyoxylate and serine- pyruvate aminotransferase | 2q37.3 | Capacidad de dimerizar. |
| SNED1 | *616634 | Sushi, nidogen and EGF like domains 1 | 2q37.3 | Interactúa con la somatostatina, la espermidina sintasa y TMEM132C. |
| PASK | *607505 | PAS domain containing serine/threonine kinase | 2q37.3 | Sensor involucrado en la homeostasis de la energía: regula la síntesis de la glucógeno sintasa mediante la mediación de la fosforilación de GYS1. |
| PPP1R7 | *602877 | Protein Phosphatase 1 Regulatory Subunit 7 | 2q37.3 | Subunidad reguladora de la proteína fosfatasa 1. |
| HDLBP | *142695 | High density lipoprotein binding protein | 2q37.3 | Metabolismo de los esteroles celulares. Puede funcionar para proteger a las células de la acumulación excesiva de colesterol. |
| SEPT2 | *601506 | Septin 2 | 2q37.3 | Requerido para la organización normal del citoesqueleto de actina. Desempeña un papel en la biogénesis del epitelio al mantener los microtúbulos poliglutamilados, con lo cual facilita el transporte eficiente de vesículas e impide la unión de MAP4 a la tubulina. |
| STK25 | *602255 | Serine/threonine kinase 25 | 2q37.3 | Regula los eventos de transporte de proteínas, la adhesión celular y los complejos de polaridad importantes para la migración celular. |
| THAP | *612533 | THAP domain containing 4 | 2q37.3 | Actividad de homodimerización de proteínas. |
| DTYMK | *188345 | Deoxythymidylate kinase | 2q37.3 | Actividad de homodimerización de proteínas. |
| ING5 | *608525 | Inhibitor of growth family member 5 | 2q37.3 | Actividad de acetiltransferasa específica de histona H4. |
| NEU4 | *608527 | Neuraminidase 4 | 2q37.3 | Catabolismo lisosomal de glicoconjugados sialilados. |
| PDCD1 | *600244 | Programmed cell DEATH 1 | 2q37.3 | Receptor inhibitorio de la superficie celular, involucrado en la regulación de la función de las células T durante la inmunidad y la tolerancia. |
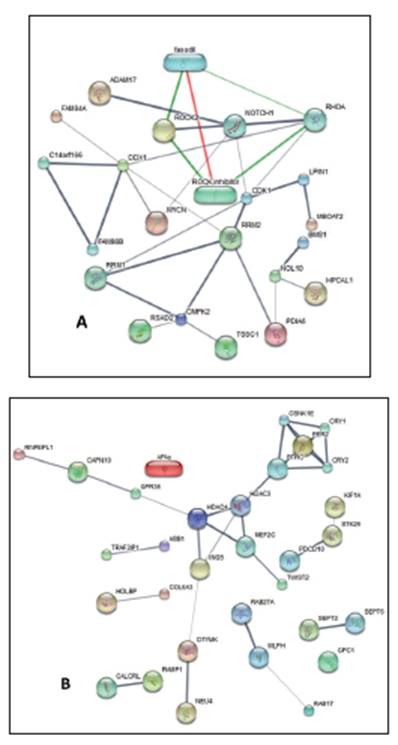
Las redes de expresión génicas, usadas para determinar las interrelaciones metabólicas de las proteínas expresadas con otras moléculas pequeñas, mostró procesos biológicos afectados por la duplicación en la citobanda 2p25.3p24.3 (Figura 1a) como la morfogénesis de lámina epitelial, el empalme de ARNt, a través de escisión endonucleolítica y procesos biosintéticos desoxirribunucleótidos. Además, las funciones moleculares como la reducción en la actividad del difosfato ribonucleó-tido también se vieron alteradas.
De otro lado, la red de interacción entre los genes afectados por la deleción en la 2q37.3 (Figura 1b) permitió observar afecciones relacionadas con procesos biológicos, como la regulación circadiana de los genes de expresión, regulación del transporte de L-glutamato, muerte celular programada, mediada por peróxido de hidrógeno. Funciones moleculares como la actividad fotoliasa del ADN y actividad en los receptores de calcitoninas.
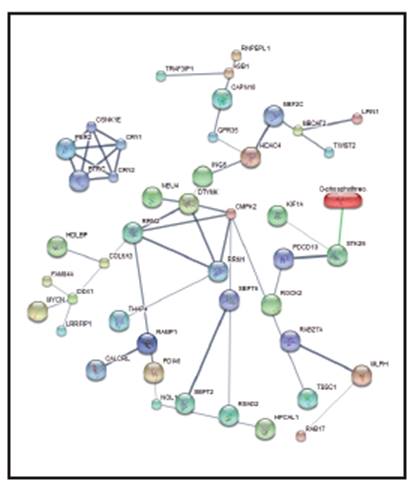
Finalmente, la red de interacción que relacionaba los genes afectados tanto por duplicación como por deleción (Figura 2), mostró una interacción asociada entre los dos tipos de mutaciones e implican ambos tipos de genes en una sola ruta de afecciones. Se observaron alteraciones en procesos biológicos tales como rutas de desoxirribonucleótidos, regulación del ritmo circadiano y regulación del transporte de L-glutamato. En cuanto a las funciones moleculares, se observaron alteraciones en la actividad fotoliasa del ADN, actividad quinasa timidilato, actividad en los receptores calcitoninos. Las rutas que se vieron más afectadas por las interacciones génicas fueron aquellas relacionadas con el ritmo circadiano.
Discusión
El síndrome de duplicación de la citobanda 2p25.3 se ha asociado con diferentes fenotipos del neurodesarrollo, como retardo en el desarrollo psicomotor y del lenguaje, discapacidad intelectual y autismo; además de rasgos faciales dismórficos 7. Estudios realizados por Buizer-Voskamp y sus colaboradores en 2011 sugirieron que las modificaciones en el locus 2p25.3 dan lugar a diferentes fenotipos del neurodesarrollo 8.
En 2008, Vrijenhoek y su equipo fueron los primeros en informar de dos pacientes con esquizofrenia y la presencia de microduplicaciones que interrumpieron el gen MYTIL, en la citobanda 2p25.3 9. Posteriormente, informaron de dos pacientes más con esquizofrenia de inicio en la niñez (COS) que también presentaban microduplicaciones que afectaban los genes MYTIL y PXDN 10. Asimismo, Lee y sus colaboradores, en 2012, reportaron dos pacientes con COS que portaban las microduplicaciones que afectaban los mismos genes evaluados, ello indicaba una fuerte evidencia de asociación entre microduplicaciones de tamaño variable que involucran genes como MYTIL y la esquizofrenia 11.
Adicionalmente, en 2016, investigaciones realizadas por Sperry y su equipo reportaron el caso de una paciente de 15 años con características clínicas del síndrome de Charge y una ganancia de novo de 6,5 Mb de material genómico en 2p25.3. La región duplicada contenía 24 genes, incluido el gen del factor de transcripción temprano y ampliamente expresado SOX11.
Actualmente, en bases de datos de CNV poblacionales como Database of Genomics Variants (DGV), no se encuentra reporte de duplicaciones de tamaño similar. Sin embargo, en bases de datos que incluyen variantes patogénicas como Decipher, se han reportado duplicaciones de tamaño similar a la identificada en este paciente, clasificadas como patogénicas y relacionadas con retardo en el desarrollo psicomotor.
De otro lado, la deleción encontrada en la cito-banda 2q37.3 se asoció con el síndrome de microdeleción 2q37, también conocido como síndrome de braquidactilia, asociado con anomalías cognitivas y de comportamiento, incluyendo trastornos del sueño.
Los afectados de esta deleción desarrollan talla baja, hipotonía, braquidactilia tipo E, dismorfias faciales como frente prominente con cejas arqueadas, ojos hundidos, puente nasal deprimido, labio superior fino y, más raramente, defectos cardiacos, convulsiones e incluso tumor de Wilms 5. También se ha reportado que, aproximadamente, un tercio de los afectados con síndrome de microdeleción 2q37 tiene características similares al autismo o autismo, que muestran una variación significativa entre los individuos. No obstante, ningún fenotipo conductual parece ser específico del síndrome de microdeleción 2q37 12.
La deleción encontrada en la paciente evaluada hace parte del grupo de deleciones pequeñas ocurridas en dicha región cromosómica; lo anterior, debido a que la deleción telomérica más grande informada en la región cromosómica 2q37 es de aproximadamente 10 Mb, mientras que la más pequeña es con frecuencia de alrededor de 3 a 4 Mb 13.
Es muy probable que la eliminación de los genes en esta región cromosómica sea el defecto genético que se sabe que está asociado con síndrome de microdeleción 2q37. Actualmente, se ha propuesto a la mutación de HDAC4 (histona deacetilasa 4) como la causante de la mayoría de las características del síndrome de microdeleción 2q37 14. Estas mutaciones de HDAC4 se han asociado fuertemente con trastornos del sueño que incluyen múltiples despertares durante la noche en la infancia y episodios prolongados de sueño ininterrumpido en la edad adulta con ausencia total de sueño.
Especíicamente, este gen se encontró en el análisis de redes de interacción junto a 37 genes asociados tanto a la duplicación como aquellos reportados para la microdeleción. Se ha reportado que muchas personas afectadas sin microdeleciones tuvieron una mutación inactivante de HDAC4, un gen en la región eliminada 2q37, lo que llevó a la propuesta de que la mutación de este gen puede ser causante de las características sindrómicas de la microdeleción 2q37 15.
HDAC4 es un gen en 2q37.3, cuya localización genómica se designa como 239,969,864-240,323,348, fundamental para el desarrollo adecuado de los huesos y de los cartílagos, así como para el desarrollo adecuado del corazón 16. También actúa en la supervivencia de las células nerviosas y juega un papel importante en el desarrollo de trastornos de la conducta, las crisis y la discapacidad intelectual. Probablemente, un desorden en los cromosomas que interieren con la expresión de este gen, aunque no lo elimina por completo, pueden causar leves síntomas del síndrome de la deleción 2q37. Sin embargo, los factores que interfieren con el hdac4 aún no han sido completamente explicados.
Desconcertantemente, algunas personas pueden no tener el HDAC4 (y otros genes candidatos), pero no tienen ninguno o sólo algunos de los síntomas esperados 14,16.
Por otro lado, estudios realizados en 2017 por Tomita, en los cuales evaluaron los niveles de me-tilación del ADN en regiones promotoras asociadas con nueve genes circadianos (PER1, PER2, PER3, cryptochrome 1 y 2, CLOCK, BMALI), revelaron que la mayor frecuencia de metilación de la isla CpG del promotor asociado con el gen circadiano está presente en pacientes con retrasos psicomotores y, específicamente, aquellos afectados por la deleción 2q37 17.
Los resultados obtenidos en nuestro estudio mostraron una alteración en uno de los genes asociados con afecciones en el sueño (gen PER2), el cual se expresa en un patrón circadiano en el núcleo supraquiasmático, lo que corresponde con los hallazgos encontrados en la literatura del tema.
Finalmente, en 2018, estudios realizados por Correa y sus colaboradores reportaron un paciente masculino de 16 años con evidencia de retraso en el desarrollo, discapacidad intelectual grave y una dismorfia facial, por lo cual el paciente depende de la atención de sus padres 18. Este paciente presenta, dos alteraciones cromosómicas terminales: una duplicación de 14.7 Mb de un segmento del brazo corto del cromosoma 2 (2p25.3p24.3) y una deleción de 4 Mb en el brazo largo del cromosoma 4 (4q35.1q35.2). La presencia de dos modificaciones en el número de copias del terminal sugirió una translocación desequilibrada, que fue validada por fish en metafases.
Conclusión
El uso de herramientas diagnósticas, como la hibridación genómica comparativa, se ha posicionado como la tecnología de elección para analizar la cantidad y estructura de los cromosomas, comparándolo con uno de referencia, ya que tiene mayor rendimiento diagnóstico que el cariotipo convencional y mayor sensibilidad y especificidad. Esto ha permitido identificar un número creciente de síndromes asociados con microduplicación o microdeleción. Por lo que podría llegar a ser la prueba de primera categoría para el análisis cromosómico en los próximos años.
El estudio molecular evidenció la duplicación patogénica en 2p25.3p24.3 y la deleción patogénica en 2q37.3, ambas anteriormente reportadas por separado. El fenotipo de la persona está en consonancia con los de la literatura existente para los síndromes de duplicación y deleción; sin embargo, esta es la primera vez que se reporta este tipo particular de reordenamiento cromosómico.
Las redes de interacción de genes afectados que otorgan el fenotipo de la paciente son importantes, ya que permiten determinar, en conjunto, las características sindrómicas causantes de la patología, además de brindar un acercamiento oportuno a la presentación clínica y molecular determinada en este estudio.
A pesar de tratarse de un ordenamiento no reportado, existe una correlación del diagnóstico clínico de la paciente con la duplicación/deleción encontrada en el estudio molecular, pues ambas, al considerarse patogénicas, llevan al paciente a sufrir las graves consecuencias de la enfermedad.
El conocimiento del genotipo, el endotipo y la expresión fenotípica de los pacientes permite acercarse a una medicina personalizada, basada en el diagnóstico precoz, tratamiento oportuno y reporte de nuevos casos y variantes genéticas, a fin de contribuir a la medicina de precisión que necesitamos para proveer un cuidado de calidad a los pacientes, su familia y la sociedad.