1. INTRODUCCIÓN
Las fisuras orofaciales son el defecto craneofacial más común en nacimientos vivos las cuales ocasionan complicaciones que afectan la alimentación, el habla y el desarrollo psicosocial [1, 2]. Dentro de las fisuras orofaciales se encuentran las fisuras de labio (FL), las fisuras labio y palatinas (FLP) y las fisuras palatinas (FP). Se cree que las FL y FLP son etiológicamente distintas de las FP, por tal motivo se denominan colectivamente fisuras labio y/o palatinas (FL/P) [3, 4]. Las FL/P presentan una prevalencia de aproximadamente 1 cada 1.000 nacimientos vivos, sin embargo, varía según la posición geográfica y la etnia, donde es mayor en asiáticos (1:500), seguido de europeos (1:1.000) y africanos (1:2.500) [1, 2, 5]. Asimismo, la frecuencia de dicha anomalía difiere según el sexo donde hay 2 hombres por 1 mujer entre los pacientes con FL/P y 2 mujeres por 1 hombre entre los pacientes con FP [6, 7].
El 70% de las FL/P son de tipo no-sindrómico, haciendo referencia a que se encuentran aisladas y no están acompañadas de anomalías físicas o cognitivas adicionales, el porcentaje restante son FL/P sindrómicas que están acompañadas de anormalidades congénitas adicionales[8-10]. Las FL/P no-sindrómicas son de origen multifactorial, parcialmente heredables e involucran un complejo de factores genéticos y ambientales[2, 5]. Más de 40 genes incluyendo IRF6, VAX1, MSX1, FOXE1, MYH9, MAFB, ABCA4, BMP4, FGFR2, TGFA, TGFB3, MTHFR, GSTT1, PDGFC, FGF8, PVRL1, SUMO1 y CRISPLD2 se han asociado a la presencia de FL/P no sindrómicas [2, 3, 11-14]
Con el desarrollo de las nuevas tecnologías de secuenciación del genoma humano, se han identificado variantes polimórficas que pueden estar asociados con la presencia de las FL/P no sindrómicas, lo que ha contribuido a comprender la etiología multifactorial de este fenotipo. Teniendo en cuenta que las FL/P se han asociado con la presencia de múltiples polimorfismos en diferentes poblaciones, en esta revisión se describen las variantes polimórficas comúnmente asociadas a dicho fenotipo y su papel en su etiología.
2. MATERIALES Y MÉTODOS
Se realizó una búsqueda sistemática de la literatura siguiendo los ítems de propuestos por "systematic reviews and meta-analyses (PRISMA)", los cuales incluyen la identificación, selección e inclusión de la literatura revisada. Se realizó una búsqueda en PubMed y en el National Center for Biotechnology Information (NCBI) (http://www.ncbi.nlm.nih.gov/omim/) utilizando los siguientes descriptores: "polymorphisms'", "cleft lip'", "cleft palate'" y "cleft lip and palate". Los artículos seleccionados fueron publicados hasta el 2018. Los criterios de inclusión fueron los siguientes: a) tipo de estudio experimental; (b) estudios de casos-controles; (c) FL/P no sindrómicas.
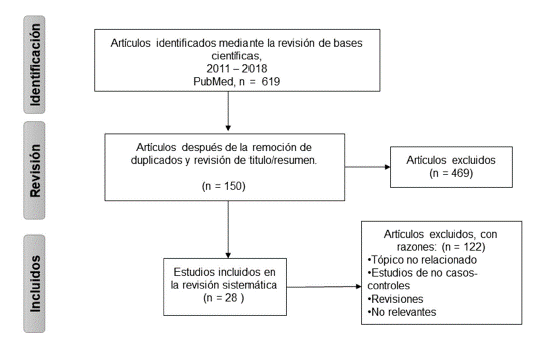
Para esta revisión se identificaron un total 619 artículos en la búsqueda en la base de datos PubMed y fueron evaluados mediante la revisión de títulos y resumen, posterior a la remoción de artículos duplicados y revisión de estudios, 150 artículos fueron sometidos a revisión de texto completo. Acorde con los criterios de inclusión y exclusión, se incluyeron en la síntesis los datos de un total de 28 artículos científicos (Figura suplementaria 1). Los resultados son expuestos en diferentes secciones y se mencionarán los genes de mayor a menor frecuencia de variables polimórficas.
3. RESULTADOS
3.1 Patobiología y genética de las FL/P no sindrómicas
La embriogénesis del labio y paladar se inicia con la formación de una depresión ectodérmica denominada estomodeo compuesta por la cavidad bucal y nasal sin diferenciación, entre la 8-12 semanas se lleva a cabo la palatogénesis donde se origina el paladar el cual divide el estomodeo dando lugar a la cavidad bucal y la nariz por separado. En las etapas iniciales de la palatogénesis los procesos palatinos aparecen como protrusiones provenientes de las células mesenquimatosas derivadas de la cresta neural en las paredes laterales de la cavidad del estomodeo y crecen alrededor de la lengua verticalmente mediante la proliferación de las células mesenquimales derivadas de la cresta neural. La formación del labio se da a partir de la fusión de los procesos medial y lateral nasal, y posteriormente la fusión de éstos con el proceso maxilar, mediante dichas fusiones los procesos se contactan, se adhieren y se produce una lámina epitelial que posteriormente desaparece mediante procesos de muerte celular, cualquier falla en los procesos de formación del labio y paladar conducirá a la formación de FL/P [15-18].
Las fisuras orofaciales no sindrómicas incluyen las FL/P, las cuales poseen una etiología multifactorial con un fuerte componente genético [19, 20], donde el desarrollo temprano de las estructuras involucradas en los procesos de palatogenésis y formación de labio, está mediada por las interacciones epitelio-mesenquimáticas lo cual depende de una amplia gama de moléculas de señalizaciones entre los cuales se incluyen fibroblast growth factors (FGFs), bone morphogenetic proteins (BMPs), y transforming growth factors (TGFs), de igual manera, están involucrados varios factores de desarrollo tales como msh homeobox (MSX), paired box (PAX),e interferon regulatory factor (IRF) [21, 22].
La vía de señalización FGF (fibroblast growth factor), juega un papel esencial en el desarrollo craneofacial, mediante la migración de las células de la cresta neural y regulación de las interacciones mesenquimales epiteliales durante la fusión de los procesos que originan las estructuras labio y palatinas [13, 21], al igual que la anterior vía, la señalización dada por la proteína bone morphogenetic protein (BMP), está involucrada en la inducción, formación y migración de las células de la cresta neural craneal que dan lugar a la mayoría de las estructuras craneofaciales, además, está involucrada en procesos de formación de los primordios faciales [22]. En referencia a los factores TGF (transforming growth factors), tienen un rol fundamental en procesos de palatogénesis, dado que se encuentran involucrados en procesos de fusión palatina, transformación epitelio-mesenquimática, proliferación celular e inductor de muerte celular programa necesarios para la formación craneofacial [22]. El factor de desarrollo MSX, se encuentra involucrado en las interacciones entre células epiteliales y mesenquimales que se dan durante la organogénesis, de igual manera, los genes que hacen parte de los factores de transcripción PAX juegan un papel importante durante el desarrollo fetal, especialmente En el desarrollo de la cresta neural que origina posteriormente las estructuras palatinas [18, 23], en referencia a los factores de transcripción IRF se ven involucrados en los procesos de regulación transcripcional en la morfogénesis de los labios, cuya expresión es detectable en el ectodermo orofacial temprano y luego se restringe a la capa peridérmica más externa antes de la fusión de labios y paladar [24, 25]. Otros genes se han visto involucrados en los procesos relacionados con la formación de estructuras labio/palatinas, como en los procesos de palatogénesis y odontogénesis: los genes MSX1, PAX9, morfogénesis: VaAX1, FGF1, uniones de tejidos: CHD1, procesos de proliferación, diferenciación y diferenciación celular: MSX1, FGF1, WNT3 y organogénesis: GREM1.
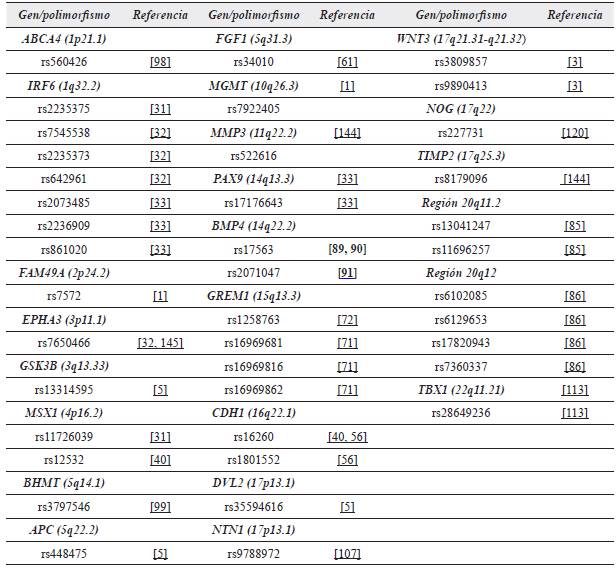
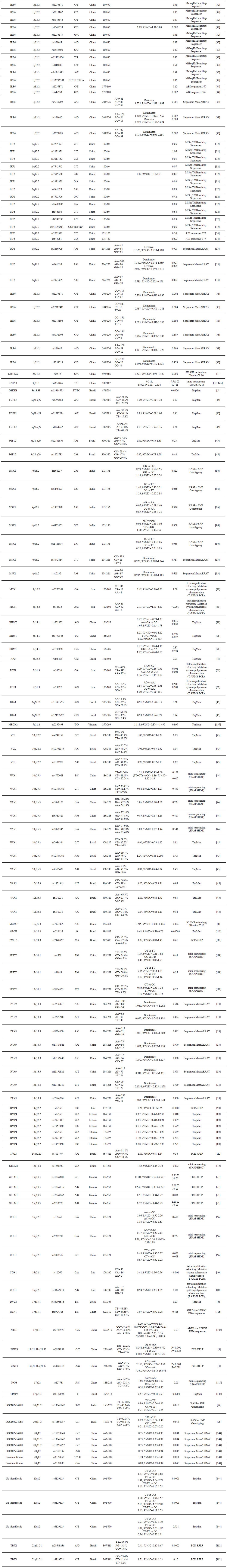
A continuación, se describen cada una de las variantes genéticas que se han asociado con la presencia de FL/P. En la Tabla suplementaria 1 se describen en detalle las variantes polimórficas estudiadas en la susceptibilidad del fenotipo de FL/P, especificando gen, ubicación cromosómica, SNP (del inglés, polimorfismo de nucleótido simple) con su "rs", variante, población analizada, número de muestra analizada, frecuencia genotípica, odds ratio, valor de p, y método de genotipificación utilizado en las diferentes investigaciones.
4. VARIANTES POLIMORFICAS ASOCIADAS CON LA PRESENCIA DE FL/P
Los principales cromosomas que albergan variantes polimórficas asociadas a las FL/P son los cromosoma 17 (DVL2-17p13.1, NTN1-17p13.1, WNT3-17q21.31-q21.32, NOG-17q22, TIMP2-17q25.3), el cromosoma 1 (PAX7-1p36.13, ABCA4-1p21.1, IRF6-1q32.2) , el cromosoma 3 (EPHA3-3p11.1, GSK3B-3q13.33, FGF12-3q28-q29), el cromosoma 5 (BHMT-5q14.1, APC-5q22.2, FGF1-5q31.3), el cromosoma 10 (VCL-10q22.2, VAX1-10q25.3, MGMT-10q26.3) y el cromosoma 14 (PAX9-14q13.3, BMP4-14q22.2, JAG2-14q32.33), y los cromosomas cuyos genes no han sido estudiados en la búsqueda de polimorfismos asociados a la presencia de FL/P son el cromosoma 8, 9, 12, 18, 19, 21, X e Y (Figura 1). A continuación se mencionarán cada uno de los genes cuyas variantes polimórficas se han asociado al fenotipo de FL/P de acuerdo a la frecuencia de polimorfismos reportados.
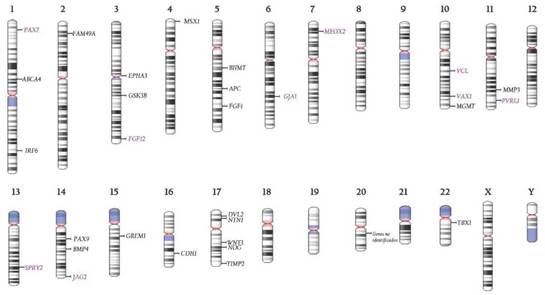
Figura 1 Genes cuyos polimorfismos han sido estudiados en la asociación con FL/P. Los genes resaltados en negro son los genes cuyos polimorfismos han sido asociados significativamente con las FL/P no sindrómicas y, los genes resaltados en morado son genes cuyas variantes genéticas, aunque han sido estudiadas no han sido asociadas significativamente con las FL/P no sindrómicas. Ideograma tomado de "The National Center for Biotechnology Information" (https://www.ncbi.nlm.nih.gov/genome/tools/gdp).
IRF6
El gen IRF6 (interferon regulatory factor 6) se localiza en el cromosoma 1 en la región q32.2, molecularmente se localiza entre las pares de bases 209.785.617 a 209.806.175 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13) [26]. Este gen lo conforman 9 exones, 20.354 pares de bases y se expresa normalmente en células epiteliales [27]. IRF6 hace parte de una familia de nueve factores de transcripción que comparten un dominio de unión a ADN muy conservado y un dominio de unión a proteína menos conservado, en lo referente a IRF6 codifica un factor de transcripción que regula la expresión de los interferones y otros genes objetivo que son críticos para la función inmune y la curación de heridas, así como el desarrollo craneofacial mediante el control de la proliferación y diferenciación celular durante el desarrollo embrionario [28-30].
Se ha estudiado la asociación de 20 polimorfismos del gen IRF6 y la presencia de fisuras orofaciales de tipo no sindrómico. En un estudio realizado por Gurramkonda, et al. [31], en 176 pacientes con FL/P no sindrómicas y 173 controles provenientes de India se reportó que el polimorfismo rs2235375 ubicado en el intrón 6 juega un rol importante en el riesgo de desarrollar las fisuras orofaciales no sindrómicas (p= 0.028). Mijiti, et al. [32] estudiaron la asociación de 12 polimorfismos (rs7545538, rs2235377, rs2235371, rs7545542, rs861019, rs7552506, rs12403006, rs846808, rs34743335, rs151296501, rs2013162, rs2235373) del gen IRF6 en un grupo de 100 pacientes con fisura labio y/o palatina y 60 controles de origen chino, de los 12 polimorfismos se identificó que el SNP rs7545538 mostraba una diferencia significativa en la frecuencia alélica entre los pacientes con fisura y los pacientes control donde los pacientes portadores del alelo G estaban un riesgo superior presentar dicha la anormalidad (p=0.007).
En otro estudio realizado en 204 pacientes con fisuras orofaciales no sindrómicas y 226 controles procedentes de china, se analizaron 9 polimorfismos (rs2236909, rs861020, rs2073485, rs2235371, rs17317411, rs2013196, rs7552506, rs861019, rs3753518) ubicados en la región 1q32.2, y se encontraron diferencias significativas entre el grupo control y el grupo con fisuras en los polimorfismos rs2073485 (p=0,002), rs2235371 (p=0,002), rs2236909 (p=0.001) y rs861020 (p=0.007), donde los otros SNPs no mostraron asociación significativa con el fenotipo de fisuras orofaciales no sindrómicas [33]. Por otro lado, en un estudio desarrollado por Shi, et al. [34] en el cual se estudiaron 2 polimorfismos (rs2235371, rs642961) para el gen IRF6 con el fin de establecer su asociación con las FL/P en una muestra de 175 individuos portadores de dicho fenotipo y 160 casos control, únicamente el SNP rs642961 mostró diferencias significativas entre los dos grupos (rs2235371 - p=0.20, rs642961 - p=0.002). Lo anterior sugiere que los polimorfismos presentes en el gen IFR6 dado su rol en los procesos de desarrollo y expresión en tejidos faciales, pueden tener un efecto en la etiología de las fisuras orofaciales no sindrómicas.
MSX1
El gen MSX1 (muscle segment homeobox 1 ) se localiza en el cromosoma 4 en la región p16.2, molecularmente se ubica entre las pares de pases 4.859.665 a 4.863.936 175 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13) [35]. Este gen está conformado por 2 exones y 4.271 pares de bases [36]. La familia MSX (muscle segment homeobox) está compuesta por dos genes MSX1 y MSX2, que se expresan parcialmente sobreexpuestos en la región craneofacial en el desarrollo embrionario. El gen MSX1 origina un represor transcripcional el cual es crucial en los procesos de palatogénesis y odontogénesis debido al rol que ocupa en la proliferación, diferenciación y muerte celular [37-39].
Cerca de 8 polimorfismos del gen MSX1 han sido estudiados con el fin de encontrar asociaciones con la presencia de las fisuras orofaciales no sindrómicas. En un estudio realizado por Gurramkonda, et al. [31] en el cual se buscó identificar la asociación entre 5 polimorfismos (rs868257, rs6446693, rs1907998, rs6832405, rs11726039) y la presencia de FL/P en una población proveniente de India, únicamente se observó diferencias significativas entre el grupo control (176) y el grupo de pacientes portadores del fenotipo (173) del SNP rs11726039 (p=0.038), indicando que el polimorfismo rs11726039 se asocia con la presencia de las fisuras orofaciales. Por otro lado, se estudió la asociación de los polimorfismos del gen MSX1 rs3775261 y rs12532 en una población de 200 individuos (100 individuos con fisuras orofaciales no sindrómicas/100 controles) provenientes de Irán, y se evidenció que el polimorfismo rs12532 es un factor de riesgo de susceptibilidad para el fenotipo de FL/P (p<0,001) [40], sin embargo, en un estudio desarrollado por Song, et al. [33] en el cual se analizó la asociación de los polimorfismos rs12532 y rs1042484 en una población proveniente China (204 individuos con FL/P y 226 controles) no se evidenció asociación significativa entre dichos polimorfismos del gen MSX1 y las fisuras orofaciales (rs12532-p=0.663 y rs1042484-p=0.587).
VAX1
El gen VAX1 (ventral anterior homeobox 1) está compuesto por 3 exones y 4.767 bases, localizándose en la región cromosómica 10q25.3 entre las pares de bases 117.128.520 a 117.138.270 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13). El gen VAX1 pertenece a una familia de genes que están involucrados en la regulación del desarrollo corporal y morfogénesis. La proteína codificada puede jugar un papel importante en el desarrollo del cerebro y el sistema visual [41-43]. En un estudio de caso-control en China (186 portadores del fenotipo de fisuras orofaciales no sindrómicas y 223 controles) se determinó la asociación de 5 SNPs ubicados en el gen VAX1 (rs4752028, rs10787760, rs7078160, rs6585429, rs1871345), donde únicamente el genotipo CC (modelo recesivo) del polimorfismo rs4752028 mostró asociación significativa con el fenotipo de FL/P no sindrómicas con un incremento de riesgo ((TT+CT) vs CC, odds ratio: 1.89, 95%IC= 1.12-3.19 p=0.017) [44] , de igual manera, en un estudio realizado a una población proveniente de Brasil compuesta por 300 pacientes con FL/P no sindrómicas y 385 casos controles no relacionados, en el cual se estudió la asociación de los polimorfismos rs10787760, rs6585429, rs1871345, rs7086344, rs751231 y rs751233 del gen VAX1 con la presencia de fisuras orofaciales no sindrómicas, aunque los SNPs rs7086344, rs6585429 y rs751233 demostraban tener un efecto proyectivo contra dicho fenotipo (odd ratio <1) ningún polimorfismo reveló diferencias significativas entre la asolación con fisuras orales (rs10787760-p=0.40, rs6585429-p=0.43, rs1871345-p=0.96, rs7086344-p=0.12, rs751231-p=0.83 y rs751233-p=0.58) [45].
PAX9
En la región cromosómica 14q13.3 se ubica el gen PAX9 (paired box 9) entre los pares de bases 36.657.568 a 36.677.807 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarcando cerca de 20.237 bases y 5 exones [46, 47]. El gen PAX9 hace parte de la familia "paired box (PAX)" de genes de trascripción que juega un rol importante en los mecanismos de ontogenia, puntualmente el gen PAX9 codifica factores de transcripción que se expresan en el mesénquima en todas las etapas de la odontogénesis, generando productos proteicos que son responsables de la comunicación cruzada entre los tejidos epiteliales y mesenquimales, y modulan la expresión de otros genes reguladores del desarrollo cruciales, comoMSX1 y BMP4 [48-51]. La asociación entre la presencia de las fisuras orofaciales y los SNPs ubicados en el gen PAX9 rs2236007, rs2295218, rs8004560, rs17104928, rs17176643, rs11156926, rs10131337, y rs7144276 fue estudiada por Song, et al. [33] en una población China compuesta por 204 pacientes con FL/P no sindrómicas y 226 casos controles no relacionados. Se encontró que de los 8 SNPs estudiados, únicamente el polimorfismo rs17176643 mostró diferencias significativas en la frecuencia alélica entre el grupo de pacientes con fisuras y el grupo de pacientes control (p=0.030), indicando que dicho polimorfismo presenta un factor de riesgo en la susceptibilidad de presentar fisuras orofaciales.
CDH1
El gen CDH1 (cadherin 1) se localiza en el cromosoma 16 en el brazo largo en la posición 22.1 (16q22.1) abarcando las pares de bases desde la 68.737.292 hasta 68.835.537 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), contiene 16 exones que se distribuyen a lo largo de 98.324 pares de bases que conforman al gen [52, 53]. CDH1 es miembro de la familia "cadherin" que codifica glicoproteínas transmembrana y juega un rol esencial en la adhesión célula-célula y en las uniones adherentes entre tejidos [54, 55]Tamara</ author><author>Frampton, Adam</author></ authors></contributors><titles><title>Gene of the month: E-cadherin (CDH1. En una investigación desarrollada por Rafighdoost, et al. [40] cuyo objetivo era determinar la asociación entre los polimorfismos del gen CDH1 y el fenotipo de FL/P de tipo no sindrómico, se estudiaron 100 individuos con fisuras no sindrómicas y 100 casos controles no relacionados provenientes de Irán a los cuales se les realizó la identificación de los polimorfismos rs16260 y rs11642413, encontrando únicamente diferencias significativas entre los grupos del polimorfismo rs16260 (p<0,001) , concluyendo que este SNP está asociado con el incremento del riesgo de presentar fisuras orofaciales, especialmente con las FL o FLP. Sin embargo, en un estudio con población de origen chino (331 pacientes con FL/P y 271 controles no relacionados) no se encontró asociación significativa entre el polimorfismo rs16260 (p=0.670) y la presencia de las fisuras orofaciales, de igual manera, en el mismo estudio se realizó la asociación entre los polimorfismos rs9929218 y rs1801552 del gen CDH1 y se encontró que el SNP rs1801552 con el genotipo TT se relacionaba significativamente con la disminución en la susceptibilidad de presentar FL/P en comparación con el genotipo CC (p=0.002) [56].
4.1 Familia FGF
FGF1
El gen FGF1 (fibroblast growth factor 1) se localiza en la región cromosómica 5q31.3 entre las pares de bases 142.592.179 y 142.698.070 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarcando cerca de 105.892 pares de bases y 4 exones. FGF1 es un factor de crecimiento teniendo un papel importante de la proliferación, migración y diferenciación celular en procesos biológicos como el desarrollo embrionario, la morfogénesis, la angiogénesis y la inflamación [57-60]. En una población proveniente de Irán (100 pacientes con FL/P y 100 pacientes controles no emparentados) se realizó la asociación entre los polimorfismos rs34010 y rs13317, y la susceptibilidad de presentar fisuras orofaciales, respecto al polimorfismo rs13317 no se encontró diferencia significativa entre los grupos y por ende no se encontró asociación entre el SNP y el fenotipo (p=0.588), por otro lado, se encontraron diferencias significativas entre la distribución del SNP rs34010 entre el grupo control y el grupo de pacientes con fisuras orofaciales donde, el genotipo CA y el genotipo CA + AA presentaban una mayor frecuencia en el grupo control comparado con el grupo de pacientes con FL/P constituyendo un factor protector contra la presencia de fisuras orofaciales (p=0.001) [61].
FGF12
El gen FGF12 (fibroblast growth factor 12) hace parte de la familia factores de crecimiento de fibroblasto (fibroblast growth factors) la cual está compuesta por cerca de 22 genes que juegan un papel importante en el crecimiento celular, la morfogénesis, la embriogénesis, la angiogénesis y la reparación de heridas, especialmente los productos del gen FGF12 interactúan con los canales neuronales de sodio y contienen compuestos que pueden actuar como señal de localización nuclear, sin embargo, la función específica de este gen aún no se ha determinado [62-64]. El gen FGF12 se localiza en el cromosoma 3 en la posición q28 a 29, entre las pares de bases 192.139.390 a 192.727.541 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13) y lo conforman 5 exones y 266.541 pares de bases [65, 66]. En una investigación realizada por de Aquino, et al. [45] se estudió la asociación de 5 polimorfismos en el gen FGF12 (rs6790664, rs11717284, rs1464942, rs12106855 y rs1875735) con la presencia de FL/P de tipo no sindrómico en una población proveniente de Brasil (300 pacientes con fisuras orofaciales no sindrómicas y 385 pacientes controles no relacionados), donde ninguno de los SNPs mostró diferencias significativas entre los grupos (rs6790664-p=0.50, rs11717284-p=0.36, rs1464942-p=0.74, rs12106855-p=0.23 y rs1875735-p=0.44).
GREM1
El gen GREM1 (gremlin 1, DAN family BMP antagonist) se ubica en el cromosoma 15q13.3 entre las pares de bases 32.718.004 a 32.745.106 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13) abarcando cerca de 27.134 pares de bases y 2 exones. Codifica un miembro de la familia antagonista BMP (bone morphogenic protein) y desempeña un rol importante en la regulación de la organogénesis, diferenciación de tejidos y diferenciación de patrones corporales [67-70]. En un estudio realizado en una población proveniente de Polonia (334 pacientes con fisuras orofaciales no sindrómicas y 955 controles) donde se realizó el tamizaje de 4 polimorfismos del gen GREM1 (rs16969681, rs16969816, rs16969862 y rs1258763) con el fin de determinar asociaciones con el fenotipo de fisuras orofaciales, se determinó que todos los polimorfismos mostraban diferencias significativas entre los grupos control y afectados (rs16969681-p=2.37 x 10-05, rs16969816-p=2.60 X 10-05, rs16969862-p=0.001 y rs1258763-p=1.10 X 1005), donde al presentar dichos polimorfismos se presentaba un menor riesgo de presentar fisuras orofaciales y, principalmente se encontró que al portar genotipo T del SNP rs16969681 se presenta 2,6 menor riesgo de presentar la anormalidad, concluyendo por tal motivo que las variaciones genéticas influencian los niveles de expresión del gen GREM1 contribuyendo a la etiología de las FL/P no sindrómicas [71]. Sin embargo, en un estudio realizado por Wang, et al. [72] en una población proveniente china (331 pacientes con fisuras orofaciales no sindrómicas y 271 pacientes control) se encontró que la presencia del polimorfismo rs1258763 juega un papel importante en el riesgo de desarrollar fisuras de tipo no sindrómico (p=0.022).
WNT3
Las vías de señalización de los genes pertenecientes a la familia WNT (wingless-type MMTV integration site family), juegan un papel fundamental en la embriogénesis craneofacial dado que controlan la actividad de otros genes y regulan las interacciones entre células durante el desarrollo embrionario [6, 73]. El gen WNT3 (Wnt family member 3) es el gen candidato más ampliamente estudiado para las FL/P no sindrómicas, el gen se localiza en la región 17q21.31-q21.32 entre las pares de bases 46.762.506 a 46.818.692 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38. p13) abarcando cerca de 56.255 pares de bases y 5 exones. WNT3 se ha visto involucrada en procesos de desarrollo embrionario, dando lugar a las extremidades, determinando la diferenciación del eje anterior y posterior y, se ha visto que se encuentra involucrado en procesos de formación facial [6, 74-76]. En un estudio realizado por Lu, et al. [3] en una población China compuesta por 236 pacientes con fisuras orofaciales y 400 pacientes control no emparentados, se encontró una asociación significativa del polimorfismo rs3809857 (p=0.001) del gen WNT3 y el riesgo de desarrollar FL/P donde los individuos portadores del alelo T mostraban 2 veces menor riesgo de presentar fisuras orofaciales indicando que el alelo G constituía un factor de riesgo y el alelo T un factor protector contra la susceptibilidad de presentar FL/P. En el mismo estudio, se realizó la asociación entre el polimorfismo rs9890413 del gen WNT3 y las fisuras orofaciales (p=0.008), encontrando que los individuos que portaban el alelo G presentaban un riego 2,37 mayor de presentar FL/P.
APC
El gen APC (WNT signaling pathway regulator) se localiza molecularmente entre las pares de bases 112.707.498 a 112.846.239 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13) en el cromosoma 5 en la región q22.2 y abarca cerca de 108.352 pares de bases y 16 exones. APC codifica para una proteína con múltiples funciones de unión siendo regulador clave en la vía de señalización WNT y está involucrada en procesos de crecimiento, migración y adhesión celular [77-79]. Se describió la asociación entre el polimorfismo rs448475 del gen APC en una población compuesta por 975 pacientes con fisuras orofaciales y 504 pacientes controles no relacionados provenientes de Brasil, encontrando asociación significativa con el fenotipo de fisuras orofaciales (p=0.01) [5].
GSK3
El gen GSK3 (glycogen synthase kinase 3 beta) se localiza en la región cromosómica 3q13.33 desde las pares de bases 119.826.686 hasta 120.093.666 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38. p13), abarcando aproximadamente 266.981 pares de bases y 12 exones, éste gen integra la vía de señalización WNT y desempeña un rol importante en la homeostasis de las células epiteliales [73, 80, 81]. En un estudio realizado en una población compuesta por 975 pacientes con fisuras orofaciales y 504 pacientes controles no relacionados provenientes de Brasil se encontró asociación significativa entre la presencia del SNP rs13314595 del gen GSK3 y las fisuras orofaciales (p=0.0006) [5].
DVL2
Asimismo, el gen DVL2 (dishevelled segment polarity protein 2) está involucrado en la vía de señalización WNT y en la regulación de procesos biológicos desde la especificación del destino celular hasta el comportamiento celular, se localiza en la región cromosómica 17p13.1 entre las pares de bases 7.225.341 a 7.234.545 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13) abarcando cerca de 9.205 pares de bases y 15 exones [82-84]. rs35594616 del gen DVL2, en una población compuesta por 975 pacientes con fisuras orofaciales y 504 pacientes controles no relacionados provenientes de Brasil, encontrando una asociación con el fenotipo de fisuras orofaciales, esto debido a que dichos genes tienen importantes efectos en la dinámica de la vía de señalización WNT responsable de procesos relacionados con el desarrollo embrionario y diferenciación celular (p=0.03) [5].
BMP4
En la región 14q22.2 se localiza el gen BMP4 (bone morphogeneticprotein 4) entre las pares de bases 53.949.736 a 53.956.891 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13) abarcando un total de 7.076 bases que contienen 4 exones. BMP4 es miembro de la familia "transforming growth factor-beta (TGF-f})", la cual juega un papel importante en desarrollo dental, diferenciación celular y formación ósea, especialmente BMP4 durante el desarrollo dental juega un rol importante en las interacciones epitelio-mesenquimatosas [85-88]. Se ha estudiado la relación de 3 polimorfismos del gen BMP4 (rs17563, rs2071047 y rs1957860J y la presencia de fisuras orofaciales. En una investigación realizada por Kempa, et al. [89] donde se buscaba encontrar la relación entre la presencia de los polimorfismos: rs17563, rs2071047 y rs1957860, y la presencia de FL/P, en dos poblaciones diferentes de Europa (Letonia: 164 pacientes con fisuras orofaciales y 190 pacientes control; Lituania: 127 pacientes con fisuras orofaciales y 127 pacientes de control), se encontró que en la población proveniente de Lituania ningún polimorfismo presente asociación significativa con el fenotipo de FL/P (rs17563-p=0.569, rs2071047-p=0.224 y rs1957860-p=0.271) , sin embargo, en la población de Letonia los SNPs rs17563 (p=0.018) y rs2071047 (p=0.009) mostraron una asociación significativa con las FL y las FLP, disminuyendo la susceptibilidad de ser portadores del fenotipo. Lo anterior concuerda con Rafighdoost, et al. [90], quienes en un estudio realizado en una población iraní (132 pacientes con fisuras y 156 controles) encuentran asociación significativa entre el fenotipo y la presencia del polimorfismo rs17563 (p<0,001), donde la presencia de éste disminuye significativamente el riesgo de presentar FL/P.
ABCA4
En el cromosoma 1, en el brazo corto en la región p21.1 se localiza el gen ABCA4 (ATP binding cassette subfamily A member 4) entre las pares de bases 93.992.834 a 94.121.148, abarcando cerca de 128.296 bases que contienen 50 exones. El gen ABCA4 es exclusivamente expresado en fotorreceptores, conos y bastones, cuya función es el transporte de varias moléculas a través de membranas extra a e intracelulares [91-95]. Se ha estudiado la asociación de dos polimorfismos ubicados en el gen ABCA4: rs560426 y rs481931, y la presencia de fisuras orofaciales no sindrómicas en poblaciones provenientes de India y China. En una investigación realizada por [Babu Gurramkonda, et al. [96] en el cual se incluyeron 173 pacientes con FL/P y 176 casos controles no relacionados provenientes de India, no se encontró asociación significativa entre la presencia de dichos fenotipos con los polimorfismos rs560426 (p=0.720) y rs481931 (p=0.387), sin embargo, en una población proveniente de China se estudiaron a 344 pacientes con FL/P y a 324 casos controles no relacionados, y aunque el polimorfismo rs481931 no mostró diferencias significativas entre los dos grupos (p=0.95) se evidenció que el polimorfismo rs560426 mostraba asociación significativa con el fenotipo de fisuras orofaciales no sindrómicas pudiendo ser un factor de riego para desarrollar el fenotipo (p=0.0041) [97].
BHMT
En el cromosoma 5, en la posición 14.1 del brazo largo (5q14.1) se localiza el gen BHMT (betaine-homocysteine S-methyltransferase) entre las pares de bases 79.111.809 a 79.132.288 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarcando cerca de 20.512 bases y 8 exones. Codifica para una enzima esencial que cataliza la reacción de re-metilación de la homocisteína a la metionina, y participa en la conservación de la metionina y la desintoxicación de la homocisteína y, es responsable de hasta el 50% de la capacidad de metilación de homocisteína del hígado [98-101]. En un estudio llevado a cabo por Hu, et al. [98] que buscaba determinar la asociación entre los polimorfismos rs651852, rs3797546 y rs3733890 del gen BHMT y la presencia de FL/P en una población de origen chino compuesta por 166 pacientes con fisuras orofaciales no sindrómicas y 285 pacientes control, se encontró una asociación significativa entre el genotipo CC del polimorfismo rs3797546 y las fisuras orofaciales (p=0.020), sin embargo no se detectó asociación alélica significativa entre los polimorfismos y la presencia de las FL/P (p=0.199).
NTN1
El gen NTN1 (netrin 1) se localiza en el cromosoma 17 en la región p13.1 entre las pares de bases 9.021.510 a 9.244.000 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), conteniendo cerca de 222.459 bases y 7 exones. Hace parte de la familia de las Netrin, la cual origina un grupo de proteínas extracelulares que regulan la migración y la supervivencia celular durante el desarrollo y edad adulta, puntualmente NTN1 participa en la migración celular durante la embriogénesis e invasión de las células mesenquimales, siendo un paso crítico en la fusión palatina [102-105]. Se estudió la relación entre los polimorfismos rs9904526 y rs9788972 del gen NTN1 en una población China conformada por 602 pacientes con fisuras orofaciales y 510 pacientes controles no relacionados, concluyendo que el SNP rs9788972 (p=0.07) se asociaba significativamente con el incremento del riesgo de presentar fisuras orofaciales y, que la distribución del genotipo AG del polimorfismo rs9788972 difiere significativamente entre los grupos caso-control (p=0.006), mientras que el genotipo AG+AA del SNP rs9788972 incrementa la susceptibilidad de presentar fisuras orofaciales (p=0.014) [106].
TBX1
El gen TBX1 (T-box 1) se localiza en la región cromosómica 22q11.2 entre las pares 19.756.703 a 19.783.593 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38. p13), abarcando cerca de 10.632 pares de bases y 9 exones. Pertenece a una amplia familia de factores de transcripción, los cuales comparten un dominio conocido como T-box y juegan un papel importante en la formación de órganos y tejidos, La expresión TBX1 varia espacial y temporalmente entre los tejidos y se ha observado que está involucrado en procesos de relacionados con la regulación de la proliferación y diferenciación del epitelio del primordio palatino, jugando un rol importante en los procesos de fusión palatina. TBX1 es el principal gen candidato para el síndrome de deleción 22q11.2 el cual se caracteriza por hipoplasia del timo, anormalidades cardiovasculares, insuficiencia velofaríngea, esquizofrenia defectos craneofaciales entre los cuales se destacan las FL/P [107-111]. En un estudio realizado por Paranaiba, et al. [112], se investigó la asociación entre la presencia de los polimorfismos rs28649236 y rs4819522 del gen TBX1 y la susceptibilidad de presentar hendiduras orofaciales en una población preveniente de Brasil (367 casos con fisuras orofaciales y 413 controles no emparentados), encontrando respecto al SNP rs28649236 (p=0.0002) una diferencia significativa en la distribución alélica del entre los grupos y una asociación significativa entre la presencia del alelo G y la ocurrencia de las FL/P y, aunque no se encontraban diferencias significativas entre grupos respecto al SNP rs4819522 (p=0.10) se evidencia una mayor frecuencia de los genotipos CC vs CT+TT entre los grupos por lo anterior concluyen que existe una relación significativa entre los polimorfismos del gen TBX1 y la presencia de FLP.
PAX7
En la región 1p36.13 entre las pares de bases 18.630.846 a 18.748.866 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38. p13) se localiza el gen PAX7 (paired box 7), el cual abarca cerca de 117.346 bases y 9 exones. Pertenece a la familia Paired Box la cual tiene un rol importante durante el desarrollo fetal y crecimiento del cáncer, sin embargo, aunque se ha comprobado que el factor de transcripción para el que codifica PAX7 se expresa en células satelitales de estado quiescente a proliferativo y se especula que está involucrado en la supresión de tumores, su función específica del gen aún no ha sido esclarecida [70, 113-115]. En un estudio realizado por Guo, et al. [106] cuyo objetivo era encontrar la asociación entre 4 polimorfismos (rs742071, rs6659735, rs766325 y rs4920520) del gen PAX7 en una población proveniente de China compuesta por 602 pacientes con fisuras orofaciales y 510 pacientes controles, se encontró que ningún polimorfismo presentaba asociación significativa con la presencia de las fisuras orofaciales concluyendo que los polimorfismos del gen PAX7 (rs742071-p=0.792, rs6659735-p=0.852, rs766325- p=0.699 y rs4920520-p=0.782) no tienen efecto en el riesgo de presentar dicho fenotipo.
SPRY2
El gen SPRY2 (sprouty RTK signaling antagonist 2) se ubica en el cromosoma número 13 en la posición q13.1, entre las pares de bases 80.335.976 a 80.341.126 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38. p13), abarcando cerca de 4.976 bases y 2 exones. Codifica una proteína que posee un dominio rico en cisteína esencial para la actividad inhibitoria de las proteínas de señalización del receptor tirosina quinasa jugando un papel importante en las funciones regulatorias en los procesos biológicas, además puede actuar en el desarrollo del paladar hendido dado que estimula la vía de los factores de crecimiento de fibroblastos (fibroblast growth factor-FGF) [116-118]. En un estudio desarrollado en una población china conformada por 188 pacientes con fisuras orofaciales no sindrómicas y 228 casos controles, se investigó la asociación entre los polimorfismos rs4728, rs11911 y rs9574565 del gen SPRY2 y la presencia de las FL/P no sindrómicas, sin embargo, ninguno de éstos polimorfismos mostró una asociación significativa con el fenotipo estudiado (rs4728-p=0.44, rs11911-p=0.35 y rs9574565-p=0.72) [119].
VCL
En la posición q22.2 del cromosoma 10 entre las pares de bases 73.998.114 a 74.121.363 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarcando cerca de 122.047 pares de bases y 22 exones, se localiza el gen VCL (vinculin). Este gen codifica para una proteína citoplasmática de unión a actina que reside en complejos de adhesión asociados a la membrana para formar uniones entre las células adyacentes y con la matriz extracelular [120-122]. En una población proveniente de Brasil conformada por 300 pacientes con fisuras orofaciales y 385 casos controles, de Aquino, et al. [45] realizó un estudio con el fin de asociar los polimorfismos rs4746172, rs10762573 y rs2131960 a dicho fenotipo, sin embargo, ninguno de los SNPs mostró asociación significativa con las FL/P (rs4746172-p=0.83, rs10762573-p=0.94 y rs2131960-p=0.62)
GJA1
El gen GJA1 (Gap Junction protein alpha 1) también llamado CX43 se localiza en la región cromosómica 6q22.31 entre las pares de bases 121.435.646 a 121.449.727 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarca cerca de 14.151 y contiene 2 exones. GJA1 hace parte de la familia "connexin" y codifica para la proteína connexin 43 (Cx43) la cual forma canales transmembrana de unión gap que facilitan la comunicación entre citoplasma de dos células adyacentes permitiendo el intercambio intercelular de iones y metabolitos, asimismo contribuye a la migración normal celular y a la invasión de células tumorales [123-127]. En un estudio realizado en una población compuesta por 300 pacientes con FL/P y 385 pacientes control de Brasil, no se encontró asociación entre los polimorfismos rs11961755 (p=0.68) y rs12197797 (p=0.94) del gen CX43 y el fenotipo de FL/P no sindrómicas [45].
JAG2
En el cromosoma 14 en la posición q32.33 se localiza el gen JAG2 (jagged canonical Notch ligand 2) entra las pares de bases 105.140.981 a 105.168.824 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarcando cerca de 27.844 bases y 26 exones. JAG2 hace parte de la vida de señalización NOTCH la cual está implicada en variedad de procesos incluyendo destino celular, regulación, proliferación diferenciación y muerte celular, siendo esencial en los procesos involucrados en el desarrollo embrionario, especialmente JAG2 es un receptor en la vía de señalización y se encuentra ampliamente involucrado en el mantenimiento y expansión de las células madre [128-132]. Con el fin de determinar la asociación entre el fenotipo de fisuras orofaciales no sindrómicas y el polimorfismo rs1057744, Paranaiba, et al. [112] identificaron la presencia de éste en una población proveniente de Brasil conformada por 367 pacientes con fisuras no sindrómicas y 413 controles no relacionados, concluyendo que aunque la frecuencia del genotipo GA del polimorfismo es mayor que los genotipos GG y AA, no se encuentra una diferencia significativa entre los grupos de estudio (p=0.36).
MEOX2
En la región cromosómica 7p21.2 entre las pares de bases 15.611.212 a 15.686.812 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarcando aproximadamente 75.601 con 3 exones se localiza el genMEOX2 (mesenchyme homeobox 2, también conocido como GAX, Growth Arrest-specific homeoboX). Junto con MEOX1 (mesenchyme homeobox 1) componen la fa milia MEOX (Mesenchyme homeobox), y codifica un factor de transcripción que contiene un homeodominio que se expresa tanto en músculo liso vascular como en las células endoteliales vasculares, tiene función específica de regulador del crecimiento mediante la inhibición de la proliferación celular [133-136]. En un estudio realizado por Tran, et al. [137] no se encontró relación significativa entre el polimorfismo rs2237493 y la presencia de hendiduras orofaciales en una población de Vietman (277 pacientes con FL/P y 293 casos controles no relacionados), indicando que no existen diferencias significativas de la distribución alélica y genotípica entre el grupo control y el grupo afectado (p=0.995).
PVRL1
PVRL1 (encoding poliovirus receptor-like 1, también conocido como NECTIN1 gene nectin cell adhesion molecule 1) se localiza en la región cromosómica 11q23.3 desde las pares de bases 119.638.098 a 119.729.200 (Homo sapiens Updated Annotation Release 109.20190607, GRCh38.p13), abarcando cerca de 68.093 pares de bases y 6 exones. Es miembro de la familia de las proteínas adhesivas de superficie celular y su función principal se relaciona con la formación de uniones entre células epiteliales. Juega un papel importante en el desarrollo del paladar dado que para que éste se origine se debe unir y fusionar el epitelio palatino, proceso que se encuentra regulado por la expresión de PVRL1 [138-141]. Se estudió la asociación entre el fenotipo de FL/P y la presencia del polimorfismo rs7940667 en un población proveniente de Brasil (367 pacientes con fisuras orofaciales y 413 casos controles no relacionados), encontrando que el genotipo CC del SNP rs7940667 es más frecuente en el grupo control en comparación con el grupo con fisuras orofaciales, sin embargo, no se encontró asociación significativa entre el riesgo de presentar la anormalidad y la presencia de dicho polimorfismo (p=0.61) [112].
EPHA3
Se encontró en un estudio realizado por Yan, et al. [142] asociación significativa entre el rs7650466 del gen EPHA3 localizado en la región cromosómica 3p11.1 y la presencia de las FL/P en una población de China compuesta por 180 individuos con fisuras orofaciales no sindrómicas y 167 pacientes control no relacionados, donde los individuos que portaban el alelo C presentaban una reducción del riesgo del 80% de presentar la anormalidad orofacial en comparación con los individuos portadores del alelo T (p=9.763 X 10-11).
FAM49A
En un estudio realizado en 1062 pacientes provenientes de China (596 pacientes con fisuras orofaciales y 466 pacientes control) se encontró que la frecuencia alélica entre los grupos era significativamente diferente confirmando la asociación significativa entre el polimorfismo rs7572 del gen FAM49A localizado en 2p24.2 y la presencia de las FL/P no sindrómicas (p=0.006). Los pacientes portadores del alelo G mostraban un incremento del riesgo de presentar la anormalidad en comparación con los pacientes portadores del alelo A [1].
MGMT
Chen, et al. [1] estudiaron la asociación del polimorfismo rs7922405 del gen MGMT ubicado en la región cromosómica 10q26.3 y la presencia de fisuras orofaciales no sindrómicas en una población compuesta por 596 pacientes con FL/P y 466 pacientes control no relacionados provenientes de China, encontrando asociación significativa entre el polimorfismo y el fenotipo estudiado, donde los individuos portadores del alelo A mostraban un incremento del riesgo de desarrollar fisuras orofaciales no sindrómicas a comparación de los portadores del alelo G (p=0.024) .
MMP3
Se encontró una asociación significativa entre la presencia del polimorfismo rs522616 (p=0.0003) ubicado en el gen MMP3 localizado en la región cromosómica 11q22.2 y la presencia de fisuras orofaciales de tipo no sindrómico en una población proveniente de Brasil compuesta por 494 individuos con FL/P y 413 individuos control, donde los individuos con al menos una copia del alelo A mostraban asociación con la presencia de hendiduras orofaciales, especialmente con las FLP y FP [143].
TIMP2
En un estudio realizado a una probación originaria de Brasil (494 pacientes con fisuras orofaciales y 413 casos control no relacionados) se encontró asociación significativa entre el SNP rs8179096 del gen TIMP2 ubicado en la posición cromosómica 17q25.3 y la presencia de FL/P, donde los individuos que eran portadores de 1 o 2 copias del alelo C presentaban un mayor riesgo de presentar fisuras orofaciales a comparación de los individuos con el alelo T. Concluyendo que el gen TIMP2 se encuentra involucrado como un gen de susceptibilidad en las hendiduras orofaciales (p=0.0004) [143].
NOG
Song, et al. [119] realizaron un estudio en una población proveniente de China (416 individuos: 188 pacientes con fisuras orofaciales y 288 controles sin parentesco) el cual pretendía determinar la asociación del polimorfismo rs227731 del gen NOG ubicado en 17q22 con la presencia de las fisuras orofaciales, encontrando una diferencia significativa entre los grupos donde, el genotipo rs227731-CC era menos frecuente en pacientes con hendiduras orofaciales asociándose con la disminución en la susceptibilidad de presentar FL/P no sindrómicas en comparación con el genotipo AA (p=0.03).
4.3 Genes no identificados
En dos estudios realizados en poblaciones provenientes de China e India, se realizó el estudio de asociación de 6 polimorfismos ubicados en la región 20q12 (rs17820943, rs13041247, rs11696257, rs7360337, rs6129653 y rs6102085) y el fenotipo de fisuras orofaciales no sindrómicas. En el estudio desarrollado en la población proveniente de India, se involucraron 173 pacientes con fisuras orofaciales y 176 casos controles a los cuales se les realizó estudio de asociación con los polimorfismos rs13041247 y rs11696257, se encontró evidencia de la asociación significativa entre dichos polimorfismos y la presencia de las hendiduras orofaciales (rs13041247-p=0.013 y rs11696257-p=0.013) [96]. Lo anterior concuerda con un estudio realizado por Yin, et al. [143], quienes identifican una asociación significativa entre los polimorfismos rs17820943 (p=0.001), rs13041247(p=0.004), rs11696257 (p=0.002), rs7360337 (p=0.006), rs6129653 (p=0.011) y rs6102085 (p=0.045) y, el fenotipo de fisuras orofaciales en una población proveniente de China la cual incluyó 602 pacientes con hendiduras orofaciales no sindrómicas y 590 pacientes control.
DISCUSIÓN
Con el desarrollo de tecnologías y su aplicación a la genética médica, han permitido identificar variantes polimórficas relacionadas con la presencia de las fisuras orofaciales. Los principales genes en los cuales se han identificado variantes polimórficas relacionadas con las FL/P son IRF6, MSX1, VAX1, PAX9, CHD1, FGF1, GREM1 y WNT3, los cuales pueden contribuir a entender la etiología de las fisuras orofaciales.
Los genes mencionados anteriormente, cuyos polimorfismos se han visto mayormente asociados a la presencia de FL/P es debido a sus funciones genéticas, como el desarrollo craneofacial (IRF6), procesos de palatogénesis y odontogénesis (MSX1, PAX9), morfogénesis VAX1, FGF1), uniones de tejidos (CHD1), procesos de proliferación, diferenciación y diferenciación celular (MSX1, FGF1, WNT3) y organogénesis (GREM1), todos se encuentran involucradas en procesos necesarios para el desarrollo embrionario y procesos de formación de las estructuras labio y/o palatinas. Pese a que se han encontrado variaciones polimórficas en dicho gen, cabe resaltar que se encuentran algunas diferencias según la población de estudio y que no se han replicado en todas las poblaciones existentes, por lo tanto, es aconsejable realizar estudios en diferentes poblaciones para establecer con certeza variantes polimórficas relacionadas con la etiología de las fisuras orofaciales (Tabla 1).
Dentro de la literatura revisada se identificaron otros genes menos frecuentes, como APC, GSK3, DVL2, BMP4, ABCA4, BHMT, NTN1, TBX1, EPHA3, FAM49A, MGMT, MMP3, TIMP2y NOG, y aunque algunos estudios reportan la asociación entre las FL/P y las variantes polimórficas de dichos genes, su función y la relación con sus procesos biológicos aun no es bien conocida. Por otro lado, aunque se estudiaron las variantes polimórficas de los genes PAX7, SPRY2, VLC, GJA1, JAG2, MEOX2 y PVRL1, no se reportó en ningún estudio revisado su asociación con la susceptibilidad de presentar FL/P (Tabla 1.).
Por último, es importante continuar con el desarrollo de éste tipo de estudio dado que no solo nos permite identificar polimorfismos en diferentes poblaciones, sino también elucidar la función biológica de cada gen lo que permitirá comprender la asociación con el fenotipo de fisuras orofaciales.
CONCLUSIÓN
Las variantes polimórficas ubicadas en los genes IFR6, MSX1, VAX1, PAX9, CHD1, FGF1, GREM1 y WNT3 se han relacionado con la presencia de FL/P, los SNPs ubicados en los genes APC, GSK3, DVL2, BMP4, ABCA4, BHMT, NTN1, TBX1, EPHA3, FAM49A, MGMT, MMP3, TIMP2 y NOG aunque se ha reportado su asociación con la presencia de las fisuras orofaciales aún no es clara su relación con dicho fenotipo.
Es importante resaltar que la presencia de algunos polimorfismos en una población puede estar relacionada con su composición étnica, debido a esto, es necesario realizar investigaciones futuras que abarquen poblaciones específicas con el fin de poder comprender la etiología de las FL/P de tipo no sindrómico.