Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268On-line version ISSN 1909-0544
Rev.MVZ Cordoba vol.13 no.3 Córdoba Sept./Dec. 2008
ORIGINAL
DETECTION OF Brucella abortus IN BLOOD AND MILK OF DAIRY CATTLE BY PCR METHOD
Xiomara Mosquera C,1* Ing Biotec, Carmen Bernal V,1 Bact, Carlos Muskus L,1 P.hD, Jesús Berdugo G,2 MV.
1 Universidad de Antioquia. Sede de Investigación Universitaria-SIU. Programa de Estudio y Control de Enfermedades Tropicales- PECET. Calle 62 # 52-59. Medellín, Antioquia.
2 Universidad de Antioquia.Sede de Investigación Universitaria- SIU. Grupo de Reproducción. Calle 62 # 52-59. Medellín,Colombia.
*Correspondencia: xiomara@udea.edu.co
Recibido: Agosto 21 de 2008; Aceptado: Diciembre 19 de 2008
RESUMEN
Objetivo. Evaluar el uso de la Reacción en Cadena de la Polimerasa (PCR) para la detección de Brucella abortus en muestras de sangre y leche de vacunos. Materiales y métodos. Este estudio de tipo descriptivo fue realizado durante los años 2004 y 2005. Se analizaron 136 animales de tres fincas localizadas en el municipio de Durania, Norte de Santander, Colombia. Se evaluó la presencia de anticuerpos en la leche mediante la prueba del anillo (PAL). Se amplificó el fragmento de 223pb del gen BCSP31. Se emplearon los cebadores B4 y B5 de la región interna de la secuencia del gen BCSP31 (GenBank, número M20404). Resultados. En aquellos animales positivos se obtuvo una muestra de sangre y leche para el análisis por PCR, la sangre no fue analizada por serología. Se evaluaron diferentes métodos de extracción de ADN. Se encontró que un 13.2% (18/136) de las muestras de leche fueron positivas a la PAL. Se analizaron 33 muestras de leche negativas por PAL de las cuales el 30.3% (10/33) resultaron positivas por PCR. Al analizar las muestras de sangre de los animales positivos por PAL el 94.1% (16/17) fueron positivas por PCR, mientras que el 47% (8/17) de las muestras de leche positivas por PAL, fueron positivas por PCR. Conclusiones. Se demostró la amplificación de un fragmento de ADN de Brucella abortus en muestras de sangre y leche de vacunos. Los resultados preliminares demostraron que es posible usar PCR como prueba diagnóstica de brucelosis en Colombia.
Palabras clave: Brucella abortus, PCR, leche, sangre, bovinos.
ABSTRACT
Objective. To evaluate the use of Polymerase Chain Reaction in the detection of Brucella abortus in cattle blood and milk samples. Materials and methods. Between 2004 and 2005 a descriptive study was carried out. One hundred and thirty six females from three different herds located in Durania, Norte de Santander, Colombia were used. The antibodies to Brucella were detected using ring test (RT). Primers B4 and B5 of internal region of gen BCSP31 (GenBank, number access M20404) were used. From those RT positive animals, blood and milk samples were obtained and along with 33 negative milk samples were evaluated by PCR. Blood samples were not analyzed for antibody detection. Different DNA extraction protocols were evaluated and a Brucella abortus gene was amplified using specific primers. Results. The 13.2% of milk samples were positive to RT (18/136), 30.3% (10/33) of negative samples for RT and 94.1% (16/17) of positive samples were both positive by PCR. It was demonstrated that PCR is an useful tool to detect Brucella abortus DNA, either in milk and blood samples. Conclusions. It was determined by PCR a DNA fragment of Brucella abortus in blood and milk of cattle. The preliminary results showed that it is possible to perform PCR as diagnostic test of brucellosis in Colombia.
Key words: Brucella abortus, PCR, milk, blood, bovines.
INTRODUCCIÓN
La brucelosis es una enfermedad que produce un impacto económico negativo en explotaciones de ganado vacuno debido a los abortos, la disminución en la producción de leche, carne y problemas de fertilidad. Se calcula que las pérdidas económicas del sector pecuario en el continente Americano ascienden a $270 millones de dólares; que se distribuyen de la siguiente manera: producción de crías (47%), producción de leche (41%) y costos de reposición (12%). En Colombia, el Instituto Colombiano Agropecuario, ICA, ha estimado pérdidas aproximadas a los $46 millones de dólares (1).
Los métodos tradicionales o comúnmente empleados en el diagnóstico de brucelosis bovina son: la demostración directa de la bacteria mediante el cultivo, considerado el estándar de oro. Sin embargo, requiere laboratorios con alto grado de bioseguridad, y las pruebas indirectas (serología) pueden dar resultados falsos positivos debido a la reacción cruzada con otras bacterias gramnegativas (2) y falsos negativos por un nivel bajo de anticuerpos. Debido a esto, es necesario buscar y aplicar métodos alternativos que contribuyan al mejoramiento del diagnóstico de la enfermedad y que se eviten los problemas relacionados con la serología. La Reacción en Cadena de la Polimerasa (PCR), ha sido empleada en varios estudios para detectar la presencia de Brucella spp, en muestras de sangre, leche y otros materiales contaminados. El hecho de amplificar secuencias específicas de ADN bacteriano permite plantear que sea utilizada como un método rápido y confiable en el diagnóstico de brucelosis (3-5). El objetivo del presente trabajo fue evaluar la PCR para la detección de Brucella abortus mediante la amplificación de una secuencia genómica a partir de ADN aislado de muestras de sangre y leche de ganado vacuno.
MATERIALES Y MÉTODOS
Muestras. Se recolectaron 136 muestras de leche y 17 muestras de sangre de vacunos pertenecientes a tres fincas con antecedentes de brucelosis ubicadas en el corregimiento Hato Viejo, municipio de Durania, Norte de Santander. Los animales con cruce Pardo Suizo-Cebú, eran mayores de tres años y al momento de la toma de la muestra no presentaban síntomas clínicos de la enfermedad. Las muestras de sangre y leche se almacenaron a -20 y -80°C respectivamente hasta su uso para la PCR, y una alícuota de cada muestra de leche se almacenó a 4 °C para la detección de anticuerpos por la Prueba del Anillo en Leche (PAL).
Detección de anticuerpos en leche. La detección de anticuerpos anti-Brucella fue llevada a cabo individualmente en las instalaciones del Instituto Colombiano Agropecuario (ICA), seccional Norte de Santander, utilizando la PAL. Para este procedimiento, a 1 ml de leche se le adicionaron 30 µl de antígeno coloreado de Brucella y se incubaron a 37°C durante 1 h. La formación de un anillo azul solo en la superficie del tubo se consideró como reacción positiva. El color azul disperso en todo el tubo, se consideró como reacción negativa. A los animales con la prueba serológica positiva, se les tomó una muestra de sangre para el diagnostico molecular por PCR. Las muestras de sangre no fueron evaluadas por serología.
Extracción de ADN. Para la obtención de ADN a partir de sangre bovina se emplearon dos métodos, un estuche comercial (Promega WI, USA) siguiendo las instrucciones del fabricante y un protocolo basado en la utilización de fosfato de sodio (6) empleado en el Programa de Estudio y Control de Enfermedades Tropicales (PECET), Universidad de Antioquia, para diagnóstico de malaria, con algunas modificaciones. Brevemente, a 500 µl de sangre se le adicionaron 1 ml de agua ultra pura estéril, se mezclaron y se centrifugaron tres veces durante 2 min., a 2000 x g. El precipitado fue resuspendido en 50 µl de agua ultra pura estéril y seguido de la adición de 500 µl de solución fría de fosfato de sodio (NaH2PO4, pH 8), se mezcló por vortex y se centrifugo a 3000 x g. El sobrenadante se descartó y este paso se repitió dos veces. Posteriormente, se adicionaron 50 µl de solución de Chelex al 5%, se mezcló por vortex, se hirvió durante 10 minutos, y se centrifugó a 3000 x g durante 10 minutos. El ADN, contenido en el sobrenadante, se almacenó a -20°C en un tubo estéril hasta el momento de su utilización.
Para la obtención de ADN a partir de leche, se evaluaron tres métodos: dos empleando estuches comerciales (Promega WI, USA y Fermentas Glen Burnie, Maryland, USA), siguiendo las indicaciones del fabricante y un protocolo descrito por Romero y López (7). Un paso de centrifugación, previo a la extracción (8) fue incluido en este último protocolo. En este procedimiento se utilizó el precipitado y la fase grasa, obtenidos de la centrifugación de 1 ml de leche a 6000 x g por 10 min. Estos fueron disueltos en 500 µl de agua ultrapura estéril. Para la extracción, también se utilizaron 500 µl de la muestra de leche completa. En ambos casos se adicionaron 100 µl de Buffer NET (50 mM de NaCl, 125 mM de EDTA, 50 mM de Tris HCl pH 7.6) y 100 µl de solución de SDS al 24%. Estas muestras se incubaron a 80°C por 10 min., seguidas de enfriamiento en hielo. Se adicionaron 325 µg/ml de proteinasa K (Fermentas Glen Burnie, Maryland, USA), y se incubaron durante 90 min a 50°C. El ADN se extrajo empleando Fenol: cloroformo: alcohol isoamilico (25:24:1), se resuspendió en TE y se almacenó a -20°C hasta su utilización. La extracción del ADN de Brucella abortus se realizó a partir de 200 µl de la cepa vacunal equivalentes a 2x109 UFC (Vacuna Cepa 19 resuspendida en 2 ml de agua equivalentes a 20x109 UFC/ml de Brucella abortus viva liofilizada VECOL), utilizando un estuche comercial (Fermentas Glen Burnie, Maryland, USA).
La cuantificación del ADN total (ADN vacuno + ADN bacteriano) como reflejo de la eficacia y calidad del ADN obtenido por los métodos de extracción empleados, se llevó a cabo por espectrofotometría, a 260 y 280 nm.
Evaluación de la amplificación a partir del ADN obtenido de la leche. Para optimizar las condiciones de amplificación del ADN obtenido de la leche, se evaluaron diferentes concentraciones de EDTA: 0.5, 1, 2.5, 5 y 10 mM. A 1 ml de leche estéril se le adicionaron 20 µl de la cepa vacunal equivalentes a 2x108 UFC. A esta mezcla se le adicionaron 500 µl de la solución de EDTA a diferentes concentraciones. Los ensayos se realizaron por duplicado. La obtención del ADN se realizó empleando un estuche comercial (Promega WI, USA), utilizando el precipitado y el sobrenadante, luego de centrifugar la leche a 6000 x g por 10 min. El EDTA fue utilizado como un posible quelante del calcio presente en la leche, ya que hipotéticamente se planteó que el exceso de iones podría afectar la acción de la Taq polimerasa durante la PCR.
Nivel de detección del ADN de Brucella abortus en sangre y leche por PCR. Para establecer el nivel de detección de la técnica en sangre, se inocularon 4 µl de la vacuna, (4 x 107 UFC), en 36 µl de una muestra de sangre de un animal previamente negativo por PAL y PCR. A partir de esta, se hicieron diluciones seriadas en base diez y la extracción se llevó a cabo con el protocolo reportado por Foley et al en 1992 (6).
Para establecer el nivel de detección de la técnica en la leche, se inocularon 100 µl de la vacuna (1 x 109 UFC), en 900 µl de leche pasteurizada. Similarmente, se hicieron 9 diluciones seriadas en base diez, a partir de la dilución inicial con una concentración de 1x108 UFC. La extracción de ADN se llevó a cabo empleando los protocolos reportados anteriormente por Romero y López (7) y la centrifugación previa reportada por Sreevatsan et al (8). Cada ensayo fue realizado por duplicado.
Amplificación del fragmento de 223pb del gen BCSP31 de Brucella abortus. Se emplearon los cebadores B4 (5'-TGGCTCGGTTGCCAATATCAA-3') y B5 (5'-CGCGCTTGCCTTTCAGGTCTG-3') los cuales reconocen una región interna de la secuencia del gen BCSP31 (GenBank, número de acceso M20404), de Brucella abortus, amplificando un fragmento de 223 pb. Las condiciones de la PCR empleadas, con algunas modificaciones, fueron descritas previamente en la literatura (9). La mezcla de reacción contenía 0.7 U de Taq polimerasa (Fermentas Glen Burnie, Maryland, USA), 2.5 µl de buffer 10X, 1.5 mM de MgCl2, 0.2 mM de cada dNTP, 0.5 µM de cada cebador y 5 µl de ADN sin diluir y diluido 1:5 y 1:10 en un volumen final de reacción de 25 ml. El control positivo consistió en ADN aislado de las bacterias de la vacuna Cepa 19. La amplificación se llevó a cabo en un termociclador (Perkin Elmer 9700, Applied Biosystems Foster city, California, USA). Los productos amplificados se visualizaron en gel de agarosa al 1.5%, teñido con 0,5 µg/ml de bromuro de etidio empleando el sistema GelDoc y el Software Quantity one (Bio-Rad Hercules, California, USA).
RESULTADOS
Detección de anticuerpos en leche. De las 136 muestras de leche analizadas por PAL, el 13.23% (18/136), dieron la prueba positiva (Tabla 1).
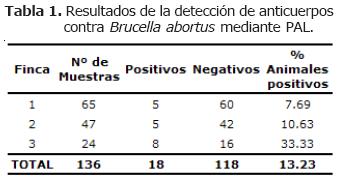
La finca 1 presentó el 7.69% (5/65) de casos positivos, la finca 2 el 10.6% (5/47) y la finca 3, el 33.3% (8/24) de los casos.
Evaluación de los métodos de extracción. Todos los métodos empleados para extraer y purificar ADN total de sangre y leche produjeron ADN en buena cantidad y calidad, de acuerdo a los resultados obtenidos por electroforesis y espectrofotometría. Sin embargo, análisis preliminares mostraron mejores resultados para la detección de Brucella por PCR cuando el ADN se obtuvo por los métodos a base de fosfatos (6) y Fenol:clorofomo (7) a partir de sangre y leche, respectivamente (Datos no mostrados). De aquí en adelante todas las muestras se procesaron por estos dos métodos.
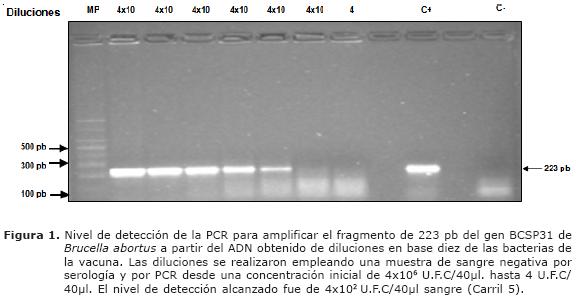
Nivel de detección del fragmento de 223 pb del ADN de Brucella abortus por PCR. Para evaluar el nivel de detección de la técnica por los métodos seleccionados, se corrieron PCRs empleando ADN aislado de sangre inoculada con diferentes diluciones de la cepa bacteriana. Se encontró que la PCR era positiva en sangre que fue inoculada hasta con 4 x 102 UFC, en geles coloreados con bromuro de etidio (Figura 1).
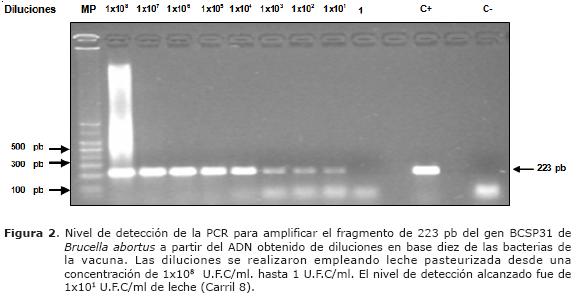
Similar análisis a partir de muestras de leche pasteurizada y contaminada experimentalmente, mostró un nivel de detección de hasta 10 UFC (Figura 2).
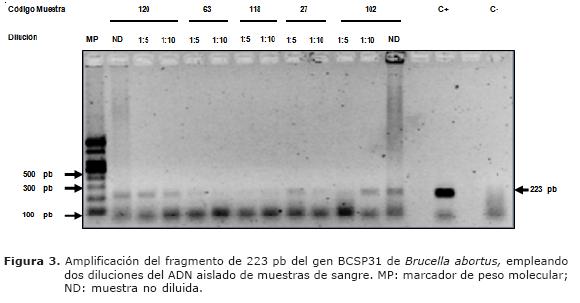
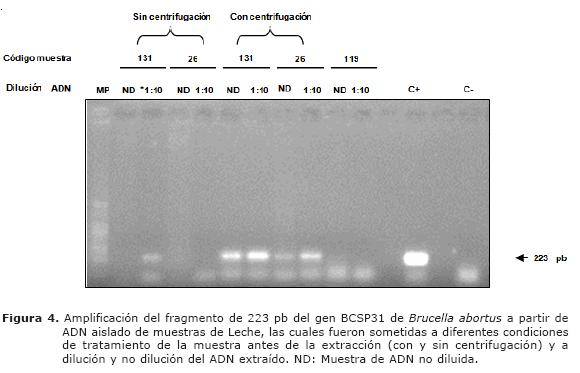
Detección directa de Brucella abortus. Para la detección de Brucella en las muestras de bovinos se empleo ADN diluido 1:5 y 1:10 y en algunas muestras se empleó además ADN sin diluir. Se detectó ADN de Brucella abortus en 16 muestras de sangre de los vacunos que tenían un resultado positivo a la PAL, independientemente de la dilución de ADN empleado para la PCR (Figura 3). También se observó amplificación en 8 muestras de leche de animales positivos por PAL y en 10 muestras de leche de animales negativos por PAL, a partir de ADN sin diluir y/o diluido 1:10 cuando la muestra fue sometida a un paso adicional de centrifugación (Figura 4).
Evaluación de la amplificación a partir del ADN obtenido de la leche. Al variar algunas de las condiciones de extracción del ADN a partir de leche, interesantemente se observó un incremento en la positividad cuando el ADN era extraído a partir del precipitado, independiente de la concentración de EDTA empleado y se observó amplificación con el ADN purificado a partir tanto del precipitado como del sobrenadante de las muestras, si no se incluía EDTA en la extracción (Datos no mostrados).
Correlación de la detección de anticuerpos y del ADN de Brucella abortus. De acuerdo a los resultados obtenidos, el 94.1% (16/17) de las muestras de sangre provenientes de animales positivos a la PAL, fueron también positivos por PCR y el 5.8% (1/17) resultó negativo por PCR (Tabla 2).
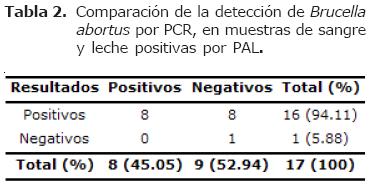
En este caso, 16 resultados positivos por serología coincidieron con los resultados de la PCR.
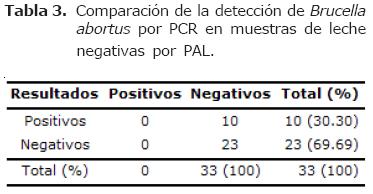
Al evaluar 33 muestras de leche que habían dado la prueba de PAL negativa, se encontró que el 30.3% (10/33), dieron la PCR positiva (Tabla 3).
DISCUSIÓN
La Prueba del Anillo en Leche (PAL), es una técnica empleada rutinariamente en el tamizaje y monitoreo de la Brucelosis en ganado lechero (10). Aunque su sensibilidad es confiable (11), su especificidad ha sido cuestionada (12). En las fincas incluidas en el estudio se encontraron animales positivos a la PAL. Sin embargo, los resultados de animales positivos a la PAL fueron más altos en la finca 3 con respecto a las otras dos fincas (Tabla 1).
En este trabajo, se evaluaron 17 muestras de sangre de animales positivos por PAL, de las cuales el 94.1% resultaron igualmente positivas por PCR (Tabla 2).
Interesantemente, al evaluar 33 muestras de leche de animales con prueba de PAL negativa, el 30.3%, resultaron positivos por PCR (Tabla 3). Estos resultados concuerdan con los hallazgos obtenidos por otros investigadores como Hamdy y Amin (3), quienes obtuvieron PCR positivas en muestras previamente positivas por el cultivo y/o por la PAL. Leal-Klevezas et al (13) obtuvieron PCR positivas en muestras que habían sido previamente negativas por la prueba Rosa de Bengala y el cultivo. En Colombia, Rivera et al (1), reportaron reacciones inespecíficas de la PAL, al obtener resultados falsos negativos que fueron confirmados como positivos utilizando la PCR.
En el caso del resultado positivo por la PAL el cual fue negativo por PCR, y asumiendo la mayor sensibilidad de la PCR, permite sugerir que posiblemente podría tratarse de una reacción cruzada con anticuerpos generados por infecciones contra otras bacterias gramnegativas presentes en el animal y que fueron detectados por la PAL. Se han descrito reacciones cruzadas con bacterias gramnegativas tales como Escherichia coli O 157:H7 y Yersinia enterocolitica 0:9 (2).
La detección del ADN de Brucella abortus en muestras de animales negativos por la PAL, indica que la bacteria está presente en el organismo del animal, pero que los niveles de anticuerpos al momento de la toma de la muestra no era lo suficientemente altos para ser detectados por la PAL. En el trabajo realizado por Leal-Klevezas et al (13) encontraron casos de animales clínicamente sanos, negativos a las pruebas serológicas y positivos por PCR en los cuales el ADN de la bacteria fue amplificado a partir de muestras tomadas en los primeros 10 días post-infección, mientras que la serología mostró resultados positivos solo después de 18 días postinfección. Basados en las anteriores evidencias experimentales es posible proponer que la PCR podría detectar el microorganismo durante la etapa temprana de la enfermedad, cuando los animales no presentan aun síntomas clínicos. Estos resultados falsos negativos favorecen la permanencia latente de la brucelosis en las fincas, ya que los animales pueden continuar transmitiendo la infección a los animales sanos, además de que también ponen en riesgo la salud de las personas.
La extracción del ADN es considerada una de las etapas críticas en la detección no solo de Brucella en la leche o en la sangre sino para cualquier microorganismo que pueda ser detectado en estos tipos de muestras, ya que parte del éxito de la detección por amplificación está relacionado con la calidad y cantidad del ADN obtenido. En este trabajo se evaluaron diferentes métodos de extracción de ADN a partir de sangre y leche, con el fin de establecer cual era el más eficiente en cada fluido.
La extracción de ADN total de sangre y leche, con algunos estuches comerciales fue adecuada, sin embargo, los resultados de las amplificaciones no fueron reproducibles. En contraste, con el método de extracción de ADN de sangre, empleando fosfato de sodio (6), se obtuvieron resultados más consistentes y reproducibles los cuales se observaron en la amplificación. Por lo tanto, este fue el método escogido para obtener el ADN de todas las muestras de sangre. La extracción de ADN total de leche, con soluciones compuestas por EDTA, Tris-HCl, NaCl, SDS, un tratamiento enzimático y la utilización de la solución de Fenol-Cloroformo- Alcohol isoamilico, permitió la obtención de ADN, que fue posible amplificar bajo las condiciones de la PCR y además los resultados fueron reproducibles. Estos coinciden con la experiencia reportada por Romero y López (7), quienes evaluaron diferentes componentes y condiciones para la extracción a partir de leche con el fin de mejorar la detección directa de Brucella. Además, la mayor parte de los trabajos reportados basados en la detección de la bacteria en la leche, realizan la extracción del ADN utilizando Fenol (1,14,15) con el fin de eliminar la mayor cantidad de sustancias contaminantes que pueden interferir con la amplificación.
Al determinar las mejores condiciones para la amplificación del ADN de la leche, se encontró que todas las muestras amplificaron cuando el ADN era extraído a partir del precipitado, independiente de la concentración de EDTA empleado. En contraste, en las muestras de leche a la cual no se le adicionó EDTA, se observó amplificación en el ADN purificado a partir tanto del precipitado como del sobrenadante (Datos no mostrados). Debido a estos resultados, las muestras de leche de los animales se procesaron sin la adición de EDTA ya que según lo observado, el calcio presente en la leche no afectó la amplificación.
Se conoce que en los animales infectados, las bacterias pueden localizarse en la glándula mamaria (16). Por lo tanto, el microorganismo puede ser excretado en la leche la cual en muchos casos es consumida sin un adecuado proceso de pasteurización, teniendo como consecuencia el aumento del número de casos de esta zoonosis en los humanos. Otra de las características de Brucella spp., es su afinidad por la grasa de la leche. Según lo reportado por Rijpens et al (15), la utilización de la fase grasa para el cultivo es un método clásico aplicado a la prueba bacteriológica para la detección de brucelosis. En el presente trabajo, al realizar las amplificaciones del fragmento de 223 pb del gen BCSP31 de Brucella abortus a partir del ADN obtenido del precipitado, más la grasa de la leche que fueron sometidas a un paso de centrifugación adicional, previo a la extracción, se observaron amplificaciones del fragmento en las muestras de ADN sin diluir y una banda más intensa al utilizar una dilución de las mismas (Figura 4).
En contraste, en las muestras que no fueron centrifugadas previamente, no se observaron amplificaciones. Sin embargo, en la dilución de una de estas muestras se observó el fragmento, pero la señal fue débil al compararla con las anteriores (Figura 4).
Esto permite sugerir que el paso extra de centrifugación incrementa el chance o la posibilidad de detectar la bacteria ya que según los resultados obtenidos, muchas de las muestras sometidas al paso de centrifugación previo adicional y que dieron la prueba positiva hubieran podido ser consideradas negativas. Por lo tanto y basado en estos resultados, se sugiere la centrifugación adicional previa de las muestras, la utilización de diluciones del ADN y el protocolo de extracción recomendado en la literatura (7,8) para el diagnostico de la brucelosis por PCR. La amplificación del fragmento de 223 pb a mayores diluciones y no amplificación en las muestras poco diluidas o sin diluir podría ser el resultado de contaminantes presentes en las muestras, que por el efecto de dilución se pierden o se disminuyen permitiendo la amplificación (Figura 4).
Adicionalmente, el ADN de los bovinos presente quizás en mayor cantidad que el ADN bacteriano en algunos animales podría tener efecto negativo sobre la eficiencia de la PCR.
En este trabajo, los cebadores B4 y B5 utilizados en la PCR, amplificaron el fragmento del tamaño esperado, en la vacuna, la sangre y la leche. Según lo reportado por Baily et al (9), estos cebadores produjeron los mejores resultados luego de la evaluación de diferentes secuencias. Estos investigadores también realizaron ensayos con diferentes muestras de ADN de bacterias gramnegativas relacionadas con el género Brucella spp., y no se presentaron amplificaciones inespecíficas confirmando la especificidad de los cebadores.
Un 50% de las muestras que resultaron positivas por PCR en sangre (Tabla 2), resultaron negativas por PCR en la leche. Una probable explicación es que la bacteria no estaba siendo excretada por la leche en el momento de la toma de muestras.
El nivel de detección de la PCR a partir del ADN obtenido de la leche contaminada experimentalmente se considera aceptable, llegando a detectar el equivalente a 10 UFC/ ml. (Figura 4). Este nivel de detección coincide con el reportado por Leal-Kevezas et al (13). Por otra parte, Romero y Lopez-Goñi (7) reportaron un nivel entre 5 - 50 UFC/ml de leche y Hamdy y Amin (3) reportaron un nivel de 100 UFC/ml en leche. A pesar de detectar el fragmento de ADN de Brucella abortus aislado de muestras de sangre, este no se consideró lo suficientemente bueno, lo que sugiere evaluar otro método de extracción con el fin de mejorar aun mas el nivel de detección en sangre o evaluar otros cebadores o condiciones de PCR (Figura 1).
Los resultados en general sugieren que la PCR podría ser una prueba de diagnóstico confiable para el diagnostico de Brucella ya que los resultados positivos de la PCR coincidieron en un alto porcentaje con los de la PAL y se encontraron casos positivos por PCR en muestras negativas inicialmente por PAL. Sin embargo, se necesita ampliar este estudio, empleando un mayor número de muestras, animales de diferentes regiones con diferentes prevalencias de brucelosis y empleo de ELISA competitiva para la evaluación de anticuerpos en sangre.
Agradecimientos
Programa de Estudio y Control de Enfermedades Tropicales - PECET. Universidad de Antioquia. Grupo de Biotecnología. Universidad de Antioquia. Dr. Javier Esteban López, Miriam Suárez Galán, auxiliar técnico de epidemiología y Dra. Ángela Sánchez Pérez; funcionarios del Instituto Colombiano Agropecuario (ICA) Seccional Norte de Santander, por su colaboración en el desarrollo de la etapa inicial del trabajo. Al Dr. Francisco Javier Pabón, funcionario de la Secretaria de Agricultura de Antioquia.
REFERENCIAS
1. Rivera D, Rueda O, Calderon C, Mariño O, Gall D, Nielsen K. Comparative evaluation of the indirect enzyme-linked immunosorbant assay in milk for the detection of cattle infected with Brucella abortus, in herds located in the province of Cundinamarca, Colombia. Rev Sci Tech 2003; 22:1065-1075. [ Links ]
2. Nielsen K, Smith P, Widdison J, Gall D, Kelly L, Kelly W, Nicoletti P. Serological relationship between cattle exposed to Brucella abortus, Yersinia enterocolitica O.9 and Escherichia coli O157: H Vet Microbiol 2004; 100:25-30. [ Links ]
3. Hamdy E, Amin S. Detection of Brucella species in the milk of infected Cattle, Sheep, goats and camels by PCR. Vet J 2002; 163: 299-305. [ Links ]
4. Lavaroni O, Aguirre N, Manzini V, Lugaresi C, Torioni de Echaide S. Evaluación de la reacción en cadena de la polimerasa para el diagnóstico de la brucelosis en un rebaño lechero infectado con Brucella spp. Rev Argent Microbiol 2004; 36:101-106. [ Links ]
5. Renteria T, Organes H, Licea A, Medina G, Nielsen K, Montaño M, Moreno J, Pujol L. Evaluation of the Polimerase Chain Reaction test (PCR), for the diagnosis of bovine brucellosis. Tec Pec Mex 2005; 43: 117-126. [ Links ]
6. Foley M, Ranford-Cartwrigth L, Babiker H. Rapid and simple method for isolating malaria DNA from fingerprick samples of blood. Mol Biochem Parasitol 1992; 53: 241-244. [ Links ]
7. Romero C, López-Goñi I. Improved Method for purification of Bacterial DNA from Bovine Milk for Detection of Brucella spp. by PCR. Appl environ Microbiol 1999; 65:3735-3737. [ Links ]
8. Sreevatsan S, Bookout J, Ringpis F, Perumaalla V, Ficht T, Adams G, Hagius S, Elzer P, Bricker B, Kumar G, Rajasekhar M, Isloor S, Barathur R. A Multiplex Approach to Molecular Detection of Brucella abortus and/or Mycobacterium bovis Infection in Cattle. J Clin Microbiol 2000; 38:2602. [ Links ]
9. Baily G, Krahn J, Drasar B, Stoker N. Detection of Brucella melitensis and Brucella abortus by DNA amplification. J Trop Med Hyg 1992; 95:271-275. [ Links ]
10. Alton Jones, Angus R, y Verger J. Techniques for the Brucellosis laboratory. Institute National de la Recherche Agronomique 1988. [ Links ]
11. Huber J, Nicoletti P. Comparison of the results of Card, Rivanol, Complement-fixation and Mil Ring Test with the isolation rate of Brucella abortus from cattle. Am J Vet Res 1986; 47:1529-1531. [ Links ]
12. Rolfe C, Sykes E. Monitoring of dairy herds for Brucella abortus infection when prevalence is low. Aust Vet J 1987; 4:97-100. [ Links ]
13. Leal-Klevezas D, Martínez-Vázquez I, García-Cantú J, López-Merino A, Martínez-Soriano J. Use of Polymerase Chain reaction to detect Brucella abortus biovar 1 in intected goats. Vet Microbiol 2000; 75:91-97. [ Links ]
14. Leal-Klevezas D, Martínez-Vázquez I, López-Merino A, Martínez-Soriano J. Single-Step PCR for Detection of Brucella spp. From Blood and Milk of Infected Animals. J Clin Microbiol 1995; 33:3087-3090. [ Links ]
15. Rijpens N, Jannes G, Van Asbroeck M, Rossau R, Herman L. Direct Detection of Brucella spp. in Raw Milk by PCR and Reverse Hybridization with 16s-23s rRNA Spacer Probes. Appl Environ Microbiol 1996; 62:1683-1688. [ Links ]
16. Cordes D, Carter M. Persistency of Brucella abortus infection in six herds of cattle under brucellosis eradication. N Z Vet J 1979; 27:255-259. [ Links ]














