Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268On-line version ISSN 1909-0544
Rev.MVZ Cordoba vol.15 no.1 Córdoba Jan./Apr. 2010
ORIGINAL
Determinación y cuantificación de bacterias acidolácticas por PCR en tiempo real
Identification and quantification of lactic acid bacteria by real-time PCR
Vienvilay Phandanouvong L,1 M.Sc., Liliana Betancourt L,2 M.Sc., Fernando Rodriguez V,1* Ph.D.
1Corpoica, Centro de Biotecnología y Bioindustria, Laboratorio de Microbiología Molecular, Tibaitatá, Km 14 vía Mosquera, Colombia.
2Universidad de La Salle, Facultad de Ciencias Agropecuarias, Cra. 7 # 172-85, Bogotá, Colombia.
*Correspondencia: frodriguez@corpoica.org.co
Recibido: Febrero 11 de 2009; Aceptado: Octubre 20 de 2009
RESUMEN
Objetivo. Establecer un ensayo de PCR-TR para determinar el tamaño poblacional y la composición de bacterias totales y de ácido lácticas, en particular las pertenecientes a los géneros Lactobacillus y Bifidobacterium. Materiales y métodos. La especificidad de los iniciadores fue verificada utilizando la técnica de PCR convencional. Diluciones de 101 a 10-4 ng/µl de ADN fueron preparadas a partir de cada cultivo microbiano y utilizadas en las curvas de calibración. La temperatura de disociación y la eficiencia de cada reacción de PCR-TR se determinaron con el software del iQCycler (Bio Rad®), versión 3.1. Resultados. Los juegos de iniciadores utilizados resultaron específicos para cada grupo microbiano, sin detectar reacción cruzada. Las eficiencias de las reacciones de la PCR-TR para bacterias totales, acidolácticas, Lactobacillus y Bifidobacterium fueron 104.4%, 98.1%, 113.3% y 103.3%, respectivamente. Conclusiones. Al obtener reacciones específicas y eficiencias cercanas al 100%, es posible cuantificar las poblaciones bacterianas totales, acidolácticas, Lactobacillus y Bifidobacterium con una alta especificidad. Por lo tanto, la técnica de PCR-TR puede utilizarse para monitorear cambios poblacionales bacterianos en ambientes como el tracto gastrointestinal de pollos de engorde, donde las bacterias acidolácticas, Lactobacillus y Bifidobacterium son habitantes comunes.
Palabras clave: PCR-TR, especificidad, eficiencia, bacterias acidolácticas, Lactobacillus, Bifidobacterium.
ABSTRACT
Objetive. To establish a PCR-TR assay to assess the population size and composition of total bacteria, total lactic acid bacteria, and particularly bacteria of the genus Lactobacillus and Bifidobacterium in samples of the gastrointestinal tract of chickens. Materials and methods. The specificity of primers was verified using conventional PCR technique. Dilutions, 101 to 10-4 ng/µl, of bacterial DNA prepared from each microbial culture were used in the calibration curves. Melting temperature and efficiency of each RT-PCR reaction were determined using iQCycler software 3.1 (BioRad®). Results. The primers sets used were found to be specific for each bacterial group with no detectable cross reaction. Efficiencies of RT-PCR reactions for total bacteria, lactic acid bacteria, Lactobacillus and Bifidobacterium were 104.4%, 98.1%, 113.3% and 103.3%, respectively. Conclusions. As specific RT-PCR reactions were obtained and efficiencies of RT-PCR reactions were about 100%, the total bacteria, lactic acid bacteria, Lactobacillus and Bifidobacterium could be quantified with high specificity. Therefore, based on the results found in this study, the RT-PCR technique can be used to monitoring bacterial population shifts in environments such as the gastrointestinal tract of broiler chickens, where lactic acid bacteria, Lactobacillus and Bifidobacterium are common habitants.
Key words: RT-PCR, specificity, efficiency, lactic acid bacteria, Lactobacillus, Bifidobacterium.
INTRODUCCIÓN
La técnica de PCR en tiempo real (PCR-TR) permite visualizar de forma inmediata cada ciclo de amplificación, a través de la aparición de una señal de fluorescencia, que permite determinar la concentración de ADN presente en una muestra (1). La cantidad de ADN de un microorganismo, al igual que la concentración de un gen en una muestra, se evalúan mediante la determinación de un valor Ct (Ciclo umbral) en cada ciclo de amplificación. Este valor Ct es el ciclo de la PCR en el cual la fluorescencia es detectada y se correlaciona con la concentración del producto de ADN amplificado (2). Así, el valor Ct es inversamente proporcional a la cantidad de ADN presente en una muestra, lo que significa que una alta concentración de ADN tendrá un valor Ct menor que se expresa y visualiza tempranamente (por una señal de fluorescencia) (3).
En la PCR-TR, los fluorocromos como el SYBR Green o sondas marcadas, son adicionados a la reacción de PCR para producir las señales de fluorescencia (4,5). El SYBR Green produce estas señales fluorescentes cuando se une al ADN de doble cadena (ADNds), con lo cual si se obtienen productos inespecíficos, estos generan fluorescencia junto con la del amplicón deseado (5). Con el fin de obtener valores de Ct producto de amplificaciones específicas, se determina la temperatura de disociación (Tm) de los amplificados. En ésta temperatura la mitad del ADN amplificado se encuentra denaturado, donde fragmentos largos tienen una Tm mayor a la de los fragmentos cortos, y también puede aumentar si el fragmento tiene mayor contenido de guanina y citosina (6). Cuando una reacción de PCR es inespecífica se obtienen varias temperaturas, por lo tanto un valor de Ct es correcto cuando se reporta una sola temperatura de disociación en cada reacción de PCR-TR (5).
La eficiencia de una reacción de PCR-TR se determina con una curva de calibración, realizada con diluciones seriadas de una concentración de ADN conocida y sus valores de Ct (1,3). Dicha eficiencia debe ser aproximadamente 2 o cercana al 100%, si estos valores no se obtienen, se deben mejorar las condiciones de corrida de la reacción (2).
La cuantificación de ADN por medio de PCR-TR puede ser absoluta o relativa (1,7). En la cuantificación absoluta, la curva de calibración es utilizada para determinar la concentración de ADN en una muestra, puesto que dicha concentración se infiere según el valor de Ct encontrado (7,8).
En la cuantificación relativa, la concentración de ADN de un gen o de un microorganismo de interés se halla en relación con un gen o grupo microbiano de referencia, por ejemplo cuando se estudian muestras de contenido gastrointestinal el grupo de referencia es el de las bacterias totales, que es considerado el grupo o población más abundante (9). En este tipo de cuantificación se usa la curva de calibración como control de calidad. Entonces la concentración de ADN en una muestra se determina calculando un valor de DCt, el cual corresponde a la diferencia entre los valores de Ct del grupo de interés y el de referencia (10). Cuando se desea evaluar las concentraciones de ADN entre tratamientos se halla el DDCt, y asumiendo que las reacciones de PCR-TR tienen la misma eficiencia se calcula el índice 2-DD Ct que indica la diferencia entre tratamientos (1,2,10).
Para identificar bacterias del tracto gastrointestinal (TGI) se ha utilizado el gen del ARNr 16S, puesto que se han encontrado en el secuencias blanco que permiten diferenciar entre una especie bacteriana de otra (11,12). Con la técnica de PCR-TR se han amplificado y cuantificado dichas secuencias, para determinar la presencia y abundancia de las bacterias, aunque se encuentren en baja proporción (13,14). Por ejemplo en muestras fecales de humanos se han identificado los grupos Clostridium coccoides, Clostridium leptum, Bacteroides fragilis, Bifidobacterium y el cluster Atopobium como los más abundantes (11).
En este estudio, luego de seleccionar y validar cuatro juegos de iniciadores para detectar grupos bacterianos del TGI de pollos de engorde, la técnica de PCR-TR fue estandarizada para evaluar la abundancia de cuatro grupos de microorganismos: bacterias totales (grupo de referencia), bacterias acidolácticas, el género Lactobacillus y el género Bifidobacterium.
MATERIALES Y MÉTODOS
Microorganismos de referencia. En total cinco cepas bacterianas fueron empleadas, tres cepas de enterobacterias: Escherichia coli ATCC 25922, Salmonella thyphimurium ATCC 14028, Salmonella enteritidis ATCC 13922, y dos cepas de bacterias acidolácticas: Lactobacillus acidophillus ATCC 4356 y Bifidobacterium breve ATCC 15700. Las cepas fueron reactivadas en caldo nutritivo estándar I, incubándose a 37°C durante 24 horas. Luego 10 µl de este cultivo fue inoculado en medios específicos: las enterobacterias en caldo Muller-Hinton y las bacterias acidolácticas en caldo MRS (Man, Rogosa y Sharpe).
Extracción y dilución de ADN. El ADN se obtuvo a partir de las cepas de referencia utilizando el kit de Extracción Microbial DNA Isolation (Mo Bio®). La extracción de ADN de las bacterias se realizó después de 8-10 horas de su incubación a 37°C en caldo Muller-Hilton, y después de 36 horas a 37°C para las bacterias acidolácticas en caldo MRS.
El ADN extraído fue cuantificado con un nano-espectrofótometro, ND-100 (NanoDrop®, Wilmington, Delaware, Estados Unidos), a una longitud de onda de 260 nm. La pureza del ADN se determinó estableciendo la relación de absorbancia 260/280 entre un rango de 1.8 - 2.0. Las muestras de ADN fueron ajustadas a una concentración de 10 ng/µl. Para realizar las curvas de calibración de la PCR-TR, se realizaron diluciones seriadas (1/10) hasta alcanzar una concentración de 10-4 ng/µl. Entonces la curva de calibración se realizó con seis diluciones así: 101, 100, 10-1, 10-2, 10-3 y 10-4 ng/µl.
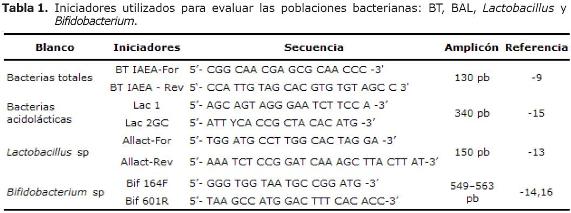
Validación de iniciadores para la detección de grupos bacterianos mediante la técnica de PCR convencional. Las cuatro parejas de iniciadores que fueron seleccionadas para evaluar poblaciones bacterianas comunes del tracto gastrointestinal (TGI) de pollos de engorde se presentan en la tabla 1. Una pareja de iniciadores se utilizó para detectar el grupo bacterias totales (BT), otra para el grupo bacterias acidolácticas (BAL), una pareja género-específica para evaluar Lactobacillus y otra para el género Bifidobacterium.
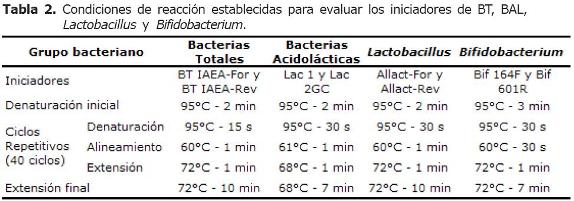
La especificidad de los iniciadores se verificó con la técnica de PCR convencional empleando un termociclador PTC-100 (MJ Research®, Inc. Minnesota, Estados Unidos). Los iniciadores se evaluaron en un rango de temperatura de alineamiento de 58 a 62°C, estableciendo las condiciones de reacción adecuadas (Tabla 2).
La mezcla de reacción de PCR se realizó en un volumen de 24 µl y contenía: 2.5 µl de buffer 10X, 0.5 µl de BSA 20X, 1 µl de MgCl2 50 mM, 0.5 µl de dNTPs mix 10 mM, 0.4 µl de Taq polimerasa (5 U/µl), 0.5 µl de cada iniciador en una concentración de 10 µM y 18.1 µl de agua ultrapura libre de nucleasas. Luego 1 µl del ADN extraído, ajustado a una concentración de 10 ng/µl, se adicionó a la mezcla para obtener una reacción final de PCR de 25 µl.
Electroforesis. Los productos de amplificación de ADN por PCR convencional se visualizaron utilizando la técnica de electroforésis, en geles de agarosa al 2.0% teñidos con SYBR Safe (Invitrogen®). Acorde al tamaño de los amplicones esperados (130-500 pb), se empleó un marcador de peso molecular de 25 a 500 pb (BioFermentas®). El corrido electroforético se realizó a 100 V durante 20 min, utilizando TAE 1X (Tris-acetato EDTA) como buffer de corrida. Los geles se visualizaron con luz ultravioleta y se fotografiaron con una cámara Polaroid.
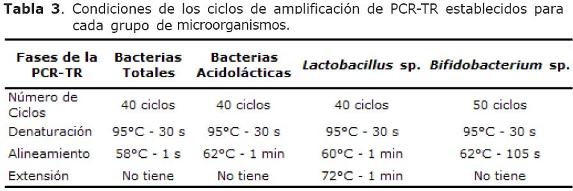
Establecimiento de la técnica de PCR-TR. Los iniciadores utilizados en las reacciones de PCR-TR fueron aquellos seleccionados y evaluados mediante la técnica de PCR convencional (Tabla 1). Para estandarizar la técnica de PCR-TR se utilizó el equipo iQ Cycler (Bio Rad®, Hercules, California, Estados Unidos), adicionando SYBR Green como fluorocromo a la reacción de PCR.
En una reacción de 25 µl de PCR-TR se adicionó: 5 µl de ADN de la muestra a una mezcla de 20 µl que contenía 8.4 µl de Platinum SYBR Green Supermix (Invitrogen®), 0.5 µl de ROX, 0.75 µl de cada iniciador en una concentración de 10 µM y 9.6 µl de agua ultrapura libre de nucleasas.
Para establecer las condiciones de reacción de la PCR-TR, los iniciadores se evaluaron en un rango de temperatura de alineamiento de 55°C a 63°C. En la tabla 3 se presentan las condiciones de reacción de cada pareja de iniciadores para evaluar grupos bacterianos del TGI de pollos de engorde. La temperatura de disociación (Tm) de los fragmentos amplificados se determinó mediante ciclos repetitivos de aumento y disminución de temperatura de 95°C por 2 min y luego 80 repeticiones de 60°C por 15 s y 95°C por 15 s.
Curvas de calibración. Para cada pareja de iniciadores utilizado se realizó una curva de calibración con seis concentraciones de ADN conocidas: 101, 100, 10-1, 10-2, 10-3 y 10-4 ng/µl, y tres réplicas por dilución. En la curva de calibración de los iniciadores para el grupo bacterias totales se utilizó el ADN extraído de E. coli ATCC 25922, para las curvas de los iniciadores de los grupos bacterias acidolácticas y Lactobacillus sp. se empleó el ADN obtenido de L. acidophillus ATCC 4356, y para la curva de calibración de los iniciadores del grupo género-específico Bifidobacterium se utilizó el ADN de B. breve ATCC 15700.
La eficiencia de la reacción de PCR-TR se determinó utilizando el software del iQCycler (Bio Rad®), versión 3.1. Una curva de calibración apropiada fue definida en aquellas relaciones de cantidad de ADN y valor Ct, en las cuales la eficiencia obtenida fue cercana al 100%.
RESULTADOS
Concentración y pureza del ADN obtenido a partir de cultivos bacterianos. La mayor concentración de ADN obtenida fue de 36.34 ng/µl, en el cultivo de B. breve ATCC 15700, seguida por L. acidophillus ATCC 4356, S. thyphimurium ATCC 14028, E. coli ATCC 25922 y S. enteritidis ATCC 13922, en los cuales se obtuvo 34.04 ng/µl, 18.05 ng/µl, 17.44 ng/µl y 13.96 ng/µl, respectivamente.
Todas las extracciones de ADN obtenidas presentaron una relación de absorbancia A260/A280 de 1.7 - 2.1, indicando que el ADN no contenía proteínas que pudieran interferir con las reacciones de amplificación de PCR (17). La concentración de ADN obtenida en este estudio a partir de los cultivos microbianos (> 10 ng/µl) fue adecuada para realizar diluciones seriadas (1/10) que se utilizaron en las curvas de calibración de la PCR-TR.
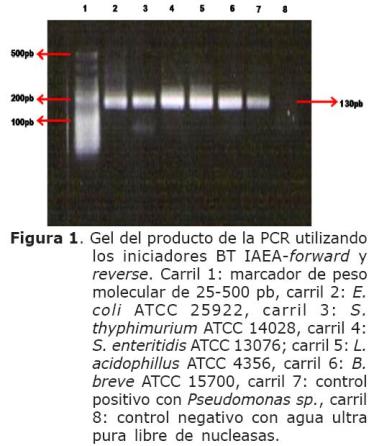
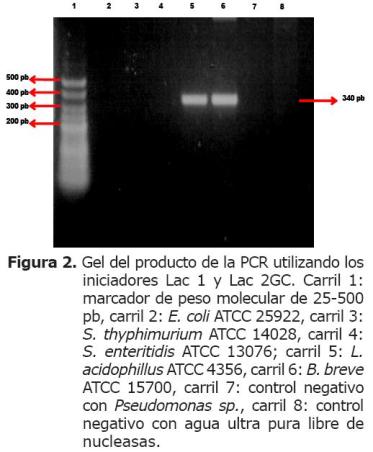
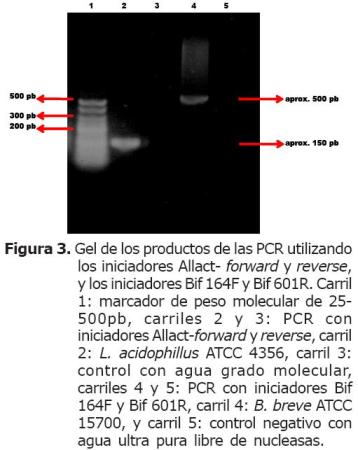
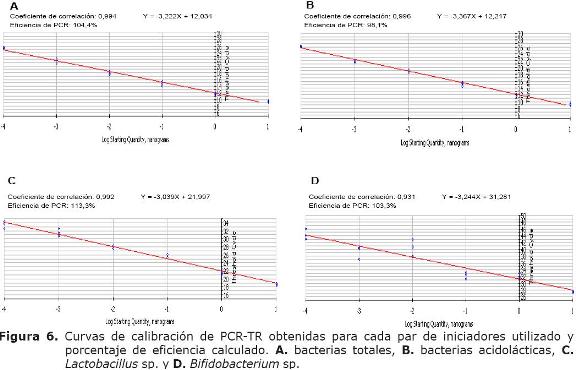
Validación de iniciadores para la detección de bacterias totales, bacterias acidolácticas, y los géneros Lactobacillus y Bifidobacterium, mediante la técnica de PCR. Las cuatro parejas de iniciadores evaluadas (Tabla 1) fueron específicas para los grupos bacterianos estudiados, siguiendo las condiciones de PCR establecidas para cada grupo (Tabla 2). El tamaño de los fragmentos amplificados para los grupos bacterianos: bacterias totales (BT), bacterias acidolácticas (BAL), Lactobacillus y Bifidobacterium fue de aproximadamente 130, 340, 150 y 500 pb, respectivamente (Figuras 1, 2 y 3).
Estandarización de la técnica PCR-TR para monitorear poblaciones de bacterias totales, bacterias acidolácticas, y los géneros Lactobacillus y Bifidobacterium. Las condiciones de corrida para cada par de iniciadores con la técnica de PCR-TR fueron similares a las establecidas con la PCR convencional, cambiando principalmente la temperatura de alineamiento (Tabla 3). En el programa de PCR-TR establecido para el género Lactobacillus se adicionó al ciclo de corrida una fase de extensión y en el programa para el género de Bifidobacterium se aumento el tiempo de alineamiento de 30 s, a 1 min y 45 s.
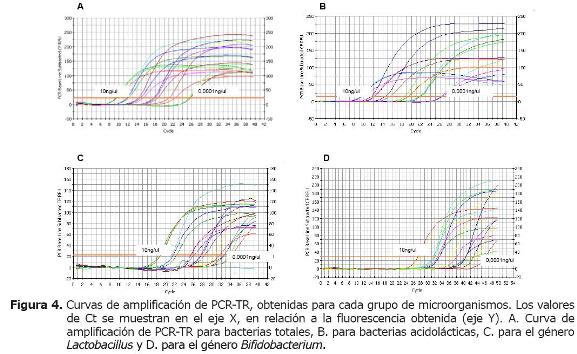
En la figura 4 se presentan las curvas de amplificación obtenidas con la utilización de cada par de iniciadores para los diferentes grupos de microorganismos, encontrándose concordancia entre el valor Ct y la concentración de ADN, de manera que con concentraciones altas de ADN se obtuvieron los menores valores de Ct.
Los programas de PCR-TR establecidos para cuantificar los grupos microbianos estudiados, requirieron 40 ciclos de amplificación para detectar la menor cantidad de ADN (0.0001 ng/µl), a excepción del establecido para detectar y cuantificar el género Bifidobacterium (50 ciclos). Los valores de Ct obtenidos con la mayor concentración de ADN (10 ng/µl) correspondieron a ciclos tempranos (ciclos 8 a 18), en relación con el valor de Ct para el género Bifidobacterium, el cual fue tardío (ciclo 27) (Figura 4).
Entonces con 50 ciclos de amplificación en el programa para detectar Bifidobacterium, se logró determinar que bajo las condiciones estandarizadas en este estudio, se podrían detectar cantidades bajas de Bifidobacterium sp. en una muestra de contenido intestinal de pollo de engorde (Ct = 46 con la menor concentración de ADN, 0.0001 ng/µl).
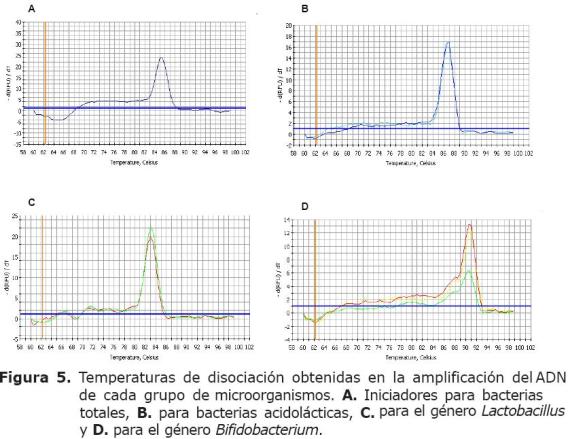
Todas las reacciones de PCR-TR obtenidas con cada par de iniciadores fueron específicas, esto es, que cada una de ellas presentó gráficamente un único pico (o valor) en la temperatura de disociación (Figura 5). La mayor temperatura (93°C) se obtuvo en la reacción de PCR-TR establecida para el género Bifidobacterium, debido a que los iniciadores empleados en esta reacción amplifican el fragmento más largo, de 549 a 563 pb (14,16).
En general las reacciones establecidas de PCR-TR presentaron eficiencias similares, para bacterias acidolácticas se obtuvo la menor eficiencia (98.1%) y la mayor para el género Lactobacillus (113.3%). Las eficiencias de las reacciones para bacterias totales y para el género Bifidobacterium fueron de 104.4% y 103.3%, respectivamente. En la figura 6 se presentan las curvas de calibración para cada par de iniciadores, con la ecuación de la recta, el coeficiente de correlación y el porcentaje de eficiencia.
DISCUSIÓN
Los iniciadores BT IAEA forward y reverse amplificaron todos los microorganismos de referencia utilizados, incluyendo el control positivo Pseudomonas sp (Figura 1). Numerosos estudios verifican que los iniciadores BT IAEA forward y reverse son apropiados para identificar bacterias en muestras intestinales (9,18). En este estudio se obtuvo amplificación del ADN proveniente de 5 géneros bacterianos diferentes: Escherichia, Salmonella, Lactobacillus, Bifidobacterium y Pseudomonas utilizando estos iniciadores seleccionados para evaluar el grupo BT.
El grupo BAL se identificó específicamente con los iniciadores Lac 1 y Lac 2GC, puesto que con las muestras que contenían ADN de Lactobacillus y Bifidobacterium se obtuvo un fragmento de 340 pb aproximadamente, mientras que las muestras con ADN de Escherichia, Salmonella y Pseudomonas no presentaron ningún fragmento de amplificación (Figura 2). Con los iniciadores Allact forward y reverse, y Bif 164F y 601R fue posible diferenciar entre dos géneros de BAL, Lactobacillus y Bifidobacterium, puesto que se identificaron dos amplicones de tamaños diferentes de 150 y 500 pb aproximadamente (Figura 3). El tamaño de estos amplicones concuerda con lo reportado previamente por Lan et al (15), Bartosch et al (14,16), y Haarman y Knol (13).
Con la técnica de PCR convencional se logró seleccionar y validar juegos de iniciadores para identificar específicamente cuatro grupos microbianos: bacterias totales, bacterias acidolácticas, Lactobacillus y Bifidobacterium en muestras de contenido gastrointestinal de pollos de engorde.
Cuando se utiliza SYBR Green en una PCR-TR no siempre se realiza una fase de extensión, pues la fluorescencia comienza a producirse cuando los iniciadores se están alineando, registrándose una fluorescencia adecuada para cuantificar el ADN presente en la muestra (4). En el programa de corrida para detectar el género Bifidobacterium fue necesario aumentar el tiempo de alineamiento a 1 min y 45 s, puesto que con menor tiempo los valores de Ct obtenidos no corresponden con la concentración de ADN. Entonces es probable que estos iniciadores no se alineen rápidamente y así no se reconozcan todas las secuencias blanco presentes en una muestra, como no sucede con los iniciadores de bacterias acidolácticas que tienen la misma temperatura de alineamiento, donde esta fase solo dura 1 min. Para detectar el ADN del género Lactobacillus se incluyó en el programa de corrida una fase de extensión, donde se registra la fluorescencia, en lugar de la fase de alineamiento, puesto que la fluorescencia producida durante la extensión fue adecuada para hallar la relación entre la concentración de ADN y el valor Ct.
Además de verificar la especificidad de los iniciadores por PCR convencional, en los ensayos de PCR-TR donde se utiliza SYBR Green como fluorocromo, es necesario determinar la temperatura de disociación (Tm) de los fragmentos amplificados, de tal modo que al obtenerse una sola temperatura se confirme la amplificación de un único fragmento (5). Con los iniciadores seleccionados, bajo las condiciones de reacción establecidas para la PCR-TR en este estudio, fue posible amplificar un único fragmento para cada grupo bacteriano evaluado, demostrando así que la fluorescencia registrada en cada reacción de PCR-TR corresponde a la amplificación de este único fragmento. Entonces como las reacciones de PCR-TR no fueron inespecíficas, los valores de Ct obtenidos para cada grupo bacteriano no fueron sobrestimados (10).
La obtención de eficiencias cercanas al 100% y similares entre las reacciones de PCR-TR estandarizadas, indican que las condiciones de corrida aquí establecidas, son adecuadas para obtener valores de cuantificación confiables (Cts) y que se pueden realizar comparaciones entre grupos bacterianos y tratamientos. Por lo tanto es posible calcular los índices 2-ΔCt y 2-ΔΔCt, los cuales se fundamentan en eficiencias cercanas a 2 (ó 100%) y similares entre tratamientos (1,10).
De acuerdo con la especificidad de los iniciadores seleccionados y las eficiencias obtenidas, la técnica de PCR-TR establecida en este estudio permite sugerir su aplicación a futuros estudios de ecología microbiana en el tractogastrointestinal de pollos de engorde, y el seguimiento específico de los grupos bacterianos: bacterias totales, bacterias acidolácticas, y los géneros Lactobacillus y Bifidobacterium.
Agradecimientos
Este estudio fue financiado por la Vicerrectoría de Investigaciones de la Universidad de La Salle y la Corporación Colombiana de Investigación Agropecuaria (CORPOICA). Los autores agradecen al Laboratorio de Microbiología del Instituto Nacional de Salud (INS) por facilitar la utilización de las cepas de referencia: Escherichia coli ATCC 25922 y Salmonella thyphymurium ATCC 14028.
REFERENCIAS
1. Pfaffl MW. A new mathematical model for relative quantification in real time RT-PCR. Nucleic Acids Res 2001; 29(9): 2003-2007. [ Links ]
2. Kubista M, Andrade JM, Bengtsson M, Forootan A, Jonak J, Lind K et al. The real-time polymerase chain reaction. Mol Aspects Med 2006; 27:95-125. [ Links ]
3. Larionov A, Krause A, Miller W. A standard curve based method for relative real time PCR data processing. BMC Bioinformatics 2005; 6:62-78. [ Links ]
4. Viljoen GJ, Nel LH, Crowther JR. Molecular diagnostic PCR handbook. Dordrecht, Holanda: Editorial Springer; 2005. [ Links ]
5. Granja CB, Vidal OM, Parra G, Salazar M. Hyperthermia reduces viral load of white spot syndrome virus in Penaeus vannamei. Dis Aquat Org 2006; 68:175-180. [ Links ]
6. Bustin SA, Mueller R. Real-time reverse transcription PCR (qRT-PCR) and its potential use in clinical diagnosis. Clin Sci 2005; 109:365-379. [ Links ]
7. Rutledge RG, Côté C. Mathematics of quantitative kinetic PCR and the application of standard curves. Nucleic Acids Res 2003; 31(16):e93-e99. [ Links ]
8. Mocellin S, Rossi CR, Pilati P., Nitti D, Marincola FM. Quantitative real-time PCR: a powerful ally in cancer research. Trends Mol Med 2003; 9(5):189-195. [ Links ]
9. Makkar HPS, McSweeney CS. Methods in gut microbial ecology for rumiants. Dordrecht, Holanda: Editorial Springer; 2005. [ Links ]
10. Dorak MT. Real-time PCR. New York, EUA: Editorial Taylor & Francis Group; 2006. [ Links ]
11. Matsuki T, Watanabe K, Fujimoto J, Takada T, Tanaka R. Use of 16S rDNA gene-targeted group-specific primers for real-time PCR analysis of predominant bacteria in human feces. Appl Environm Microbiol 2004; 70(12):7220-7228. [ Links ]
12. Wang RF, Cao WW, Cerniglia CE. PCR detection and quantification of predominant anaerobic bacteria in human and animal fecal samples. Appl Environm Microbiol 1996; 62(4):1242-1247. [ Links ]
13. Haarmann M, Knol J. Quantitative real-time PCR analysis of fecal Lactobacillus species in infants receiving a prebiotic infant formula. Appl Environm Microbiol 2006; 72:2359-2365. [ Links ]
14. Bartosch S, Fite A, Macfarlane JT, McMurdo MET. Characterization of bacterial communities in feces from healthy elderly volunteers and hospitalized elderly patients by using real-time PCR and effects of antibiotic treatment on the fecal Microbiota. Appl Environm Microbiol 2004; 70(6):3575-3581. [ Links ]
15. Lan Y, Xun S, Tamminga S, Williams BA, Verstegen MW, Erdit G. Real-Time PCR detection of lactic acid bacteria in cecal contents of Eimeria tenella-infected broilers fed soybean oligosaccharides and soluble soybean polysaccharides. Poult Sci 2004; 83:1696-1702. [ Links ]
16. Bartosch S, Woodmansey EJ, Paterson JCM, McMurdo MET, Macfarlane GT. Microbiological effects of consuming a symbiotic containing Bifidobacterium bifidum, Bifidobacterium lactis and oligofructose in elderly persons, determined by real-time polymerase chain reaction and counting of viable bacteria. Clin Infect Dis 2005; 40(1):28-37. [ Links ]
17. Thakuria D, Schimdt O, Siurtain MM, Egan D, Doohan FM. Importance of DNA quality in comparative soil microbial community structure analysis. Soil Biol Biochem 2008; 40:1390-1403. [ Links ]
18. Tajima K, Aminov RI, Nagamine T, Matsui H, Nakamura M, Benno Y. Diet-dependent shifts in the bacterial population of the rumen revealed with real-time PCR. Appl Environ Microbiol 2001; 67:2766-2774. [ Links ]