Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268On-line version ISSN 1909-0544
Rev.MVZ Cordoba vol.16 no.2 Córdoba May/Aug. 2011
ORIGINAL
Presencia de Spiroplasma penaei en plancton, bentos y fauna acompañante en fincas camaroneras de Colombia
Presence of Spiroplasma penaei in plankton, benthos and fauna in shrimps farms of Colombia
José Altamiranda M,1 Acuicultor, Marcela Salazar V,1 M.D, Boris Briñez R,1,2* M.Sc.
1Centro de Investigaciones para la Acuacultura de Colombia (Ceniacua), Cra 9 C No. 114-60, Bogotá, Colombia.
2Universidad Estatal de Campinas - UNICAMP, Departmento de Genética, Evolución y Bioagentes. Instituto de Biologia, Brazil.
*Correspondencia: borisbrinez@hotmail.com.
Recibido: Junio de 2010; Aceptado: Febrero de 2011.
Resumen
Objetivo. Determinar la presencia de Spiroplasma penaei en el plancton, bentos y fauna acompañante en 2 fincas comerciales de camarones. Materiales y métodos. Fueron colectadas 200 muestras para identificación de lesiones características de Spiroplasma, a través de la técnica de histología, mientras que para la técnica de PCR se tomaron 326 muestras de plancton, bentos y fauna acompañante. Las muestras colectadas fueron preservadas en tubos estériles con etanol al 95% para análisis de PCR y en solución Davidson para análisis histológicos. Resultados. En los muestreos realizados en las dos fincas camaroneras fue detectada la presencia de Spiroplasma en una muestra de un representante de los dípteros (mosca de agua) a través de la técnica de PCR en tiempo real, el cual arrojo una Tm=84, similar a la del control positivo de Spiroplasma utilizado. Esta muestra fue secuenciada y comparada con secuencias de bacterias almacenadas en el banco de datos GenBank usando el algoritmo BLAST, encontrando 100% de homología con un fragmento de los genes ribosomales 16S de la bacteria Spiroplasma penaei. Conclusiones. La mosca de agua es portadora de Spiroplasma penaei, sin embargo no se puede afirmar que este organismo sea el transmisor de la infección, por lo que se recomienda realizar ensayos de tipo experimental con moscas de agua infectadas con Spiroplasma penaei, en camarones libres de patógenos, para evaluar si en realidad es el vector de infección.
Palabras clave: Bentos, PCR, Penaeus vannamei, pláncton, Spiroplasma. (Fuente: AIMS).
Abstract
Objective. To determine the presence of Spiroplasma penaei on plankton, benhtos and fauna samples, in two commercial shrimp farms. Material and methods. A total of 526 samples of plankton, benthos and fauna were collected; 200 samples were used for histological examination to identify the typical lesions of Spiroplasma, and 326 samples were used for PCR. The samples collected were keep in sterile tubes and preserved in Davidson solution for histological analyses and in 95% ethanol solution for PCR. Results. The presence of Spiroplasma was detected in a water fly through the technique of real time PCR with a melting temperature Tm=84, similar to the positive control used. This sample was sequenced and compared with sequences of bacteria stored in the GenBank database using the BLAST algorithm, founding 100% homology with a fragment of the 16S ribosomal genes of the bacterium Spiroplasma penaei. Conclusions. The water fly can carry the Spiroplasma penaei, however more experimental assays are required, using water flies infected with Spiroplasma penaei and pathogen-free shrimp to prove this findings.
Key words: Benhtos, PCR, Penaeus vannamei, plankton, Spiroplasma. (Source: AIMS).
Introducción
En el comercio internacional el producto acuícola de mayor importancia es el camarón marino. Aproximadamente el 26% del producto total proviene de las especies de Peneidos criados en estanques (1), siendo el predominante el Penaeus (litopenaeus) vannamei, por lo que la industria camaronera colombiana es generadora de importantes divisas para el país a través de las exportaciones de camarones cultivados (2).
El cultivo de estos animales en estanques a altas densidades conlleva a situaciones de enfermedad por confinamiento (3). Por lo general las enfermedades más severas del camarón son causadas por virus y bacterias, ocasionando de este modo grandes pérdidas económicas a los cultivadores (4).
La causa de las enfermedades bacterianas está relacionada con el tipo de manejo que se aplique y a las condiciones medioambientales presentes. En sistemas semi-intensivo e intensivo, el incremento de la densidad de cultivo, las heces y el recambio inapropiado de agua, hacen susceptibles a los peneidos para que sean atacados por bacterias (5).
Dentro del grupo de las bacterias que afectan el monocultivo de camarón encontramos al Spiroplasma. La mayoría de las especies de Spiroplasma son naturalmente patógenos de plantas y artrópodos, principalmente insectos (6-7), la cual se ha aislado de la hemolinfa de los mismos (6).
La detección de Spiroplasma en crustáceos es relativamente nueva. El primer reporte de este patógeno fue hecho por Wang et al (7-8) quienes demostraron la presencia de este patógeno en el cangrejo Eriocheir sinensis, el cual causa la enfermedad de "tremor" (7). Wang y Gu (9) encontraron que esta enfermedad se presenta en temperaturas relativamente altas (28°C) y las principales sintomatologías son debilitamiento, anorexia y muerte. En el mismo año Zbinden y Cambon-Bonavita (10) reportaron Spiroplasma en el camarón Rimicaris exoculata. Mortalidades atípicas y severas aparecieron en una finca camaronera colombiana localizada en la costa Atlántica. Durante los meses siguientes estanques adicionales experimentaron altas mortalidades, donde la sobrevivencia fue de 40%. El diagnóstico presuntivo para las mortalidades sufridas fue atribuido a una infección bacteriana severa. Este fue confirmado a través de la hibridación in situ y bioensayos. La infección fue causada por una nueva especie del género Spiroplasma, denominada S. Penaei. Esta especie de Spiroplasma es considerada como el agente causal del "síndrome del muerto parado", el cual ocasiona sintomatologías como: tracto digestivo vacío, nado errático y producción de gas en el cefalotórax, lo que origina una flotación vertical anormal del animal (11). El último reporte acerca de este género fue realizado por Wang et al (12), catalogándolo como causante de patologías en la langosta de agua dulce Procambarus clarkii, el cual produjo severas mortalidades estimadas entre 92 y 96% en el sudeste de China. De este mismo modo Nunan et al (13) reportaron mortalidades en cultivo de camarón Penaeus (litopenaeus) vannamei en piscinas que presentaban valores de salinidad bajos y altas temperaturas
Spiroplasma es una bacteria gram positiva que por estar asociada con artrópodos, principalmente insectos (6), es probable que estos animales sean los transmisores de esta enfermedad en camarones teniendo en cuenta que la dieta de estos es omnívora (14-15).
Debido a que en la columna de agua y sobre la superficie de las piscinas camaroneras se encuentra además de plancton y bentos, una gran variedad de insectos, es posible que estos estén actuando como vectores de infección, por lo que el objetivo de este trabajo fue la identificación de los vectores de Spiroplasma.
Materiales y métodos
Área de estudio. El estudio fue realizado en dos fincas camaroneras ubicadas en el municipio de San Antero, en el norte del departamento de Córdoba, Colombia, a 5 metros sobre el nivel del mar y cuyas coordenadas geográficas son 09° 23' de latitud Norte y 75° 46' de longitud oeste de Greenwich con temperatura promedio de 28°C.
Toma de muestras. Fueron muestreadas piscinas que presentaron antecedentes de Spiroplasma, y dos piscinas control en cada finca. Se tomaron muestras de plancton, bentos y fauna acompañante cada 15 días durante dos ciclos de cultivo (240 días). Las muestras colectadas fueron colocadas en tubos eppendorf estériles con etanol al 95% para análisis de PCR y se fijaron camarones con solución Davidson para histopatología. También se tomaron muestras de plancton y fauna acompañante del manglar y del canal de entrada que surte de agua a las piscinas.
Identificación de las muestras. Las muestras de plancton fueron montadas al microscopio con el fin de identificar los organismos hasta el nivel taxonómico más bajo posible. Las muestras de bentos, fauna acompañante e insectos, fueron observadas individualmente al estereoscopio para su identificación. Parte de las muestras fueron enviadas al laboratorio de biología molecular, para la extracción del ADN y respectiva amplificación con los iniciadores específicos para Spiroplasma.
Extracción y amplificación de ADN. El ADN extraido y la PCR fueron realizados conforme a lo propuesto por Nunan et al (11). El fragmento fue amplificado por el par de primers CSF: 5' TAG CCG AAC TGAGAG GTT GA 3' y CSR 5' GAT AAC GCT TGC CACCTA TG 3' con 520 pares de bases aproximadamente. El volumen de la reacción de PCR fue de 25 ul, conteniendo Tris-KCl (20 mM Tris-HCl pH 8.4 com 50 mM KCl), 2.0 mM MgCl2, 2.5 µM de cada primer, 0.1 mM de cada dNTP, 2.5 U de Taq DNA polimerasa, 15 ng de ADN e água Milli-Q para completar el volumen a 25 µL el mix fue colocado en termociclador y el DNA desnaturado a 94°C por 5 minutos. La amplificación fue en 35 ciclos a 94°C por 30 segundos, 55°C por 30 segundos e 72°C por 30 segundos, seguido de una extensión final de 72°C por 4 minutos.
El ADN de las muestras que presentaron fragmentos amplificados de tamaño similar al control positivo de Spiroplasma fueron amplificadas por PCR en tiempo real en un termociclador Opticon (MJ Research, Hatboro, USA). Como fluorómetro se empleó Sybr Green el cual se une a la hebra de ADN de doble cadena y emite fluorescencia a una longitud de onda de 530nm. Fueron realizados análisis de las curvas de disociación para verificar la especificidad del producto amplificado.
Secuenciamento. Las muestras que mostraron curvas de disociación similares al del control positivo del Spiroplasma fueron secuenciadas en un secuenciador automático MegaBace (Amersham) de acuerdo con las instrucciones del fabricante, utilizando el primer CSF: 5' TAG CCG AAC TGAGAG GTT GA 3'. Las secuencias obtenidas fueron alineadas utilizando el programa CLUSTAL W (16) y editadas manualmente con el programa BIOEDIT (17). Estas secuencias fueron comparadas con las bases de datos públicas, usando la herramienta Blast2-NCBI (18).
Histología. Los camarones y poliquetos respectivamente fijados en solución Davidson, fueron remitidos al laboratorio de histología de CENIACUA en Cartagena, en donde fueron realizados micropreparados coloreados con eosina y hematoxilina según el protocolo estandarizado por CENIACUA.
Resultados
Reacción en cadena de la polimerasa. Un total de 326 muestras fueron analizadas por la reacción en cadena de la polimerasa (PCR). Fue detectada la presencia de Spiroplasma en: 4 muestras de músculo de camarón, 1 muestra de plancton y en 10 de insectos (mosca de agua, Hesperocorixa, Uca, Callinectes sapidus, Orthóptera sp1 y Lepidóptera sp2). Los camarones que fueron positivos por PCR también presentaron las lesiones típicas de eSpiroplasma por histopatologia (Figura 1).
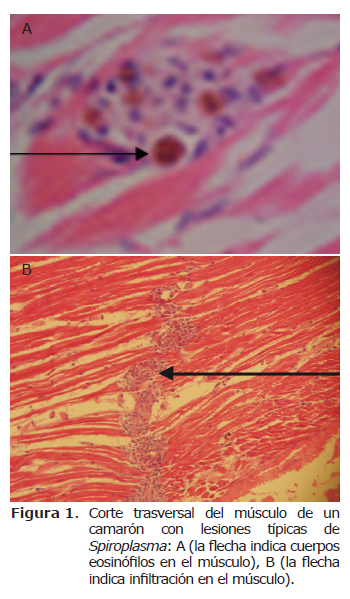
Las muestras de plancton, bentos, fauna acompañante e insectos de los muestreos realizados durante los dos ciclos de cultivo, usando iniciadores específicos de 16S de rRNA de Spiroplasma mostró la presencia de la bacteria en las muestras de Hesperocorixa, mosca de agua perteneciente al orden Díptera, representantes del orden Lepidóptera (sp2) y representantes del orden Orthóptera (sp1). Resultados no detectables fueron encontrados en las muestras de Upogebia y Uca.
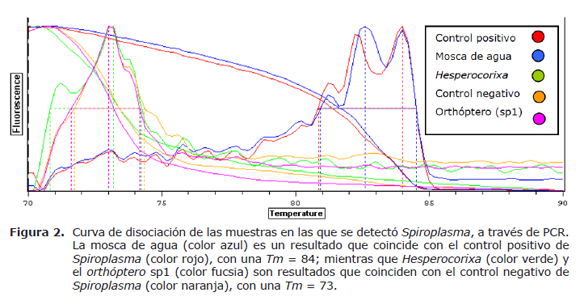
PCR en tiempo real. Los resultados positivos por PCR fueron procesados a través de PCR en tiempo real. El organismo en el que se observó presencia de Spiroplasma por PCR en tiempo real fue la mosca de agua con una Tm = 84, similar a la del control positivo de Spiroplasma utilizado (Figura 2). Esta muestra fue secuenciada para verificar la presencia de esta especie de bacteria.
Secuenciación - Blast. En este estudio sólo se procesaron las muestras de la mosca de agua puesto que fue la única que resultó positiva a través de PCR en tiempo real. Esta muestra fue comparada con secuencias de bacterias pertenecientes al GenBank usando el algoritmo BLAST, encontrando 100% de homología con un fragmento de los genes ribosomales 16S de la bacteria Spiroplasma penaei.
Discusión
Las piscinas comenzaron a presentar mortalidades en la época de lluvia, momento en el cual la salinidad bajó considerablemente en el primer ciclo de 27.6 ± 0.86 UPS hasta 16 y 17 UPS en todas las piscinas, además para este periodo la temperatura en las horas de la tarde no bajó de los 30.8°C, mientras que en el segundo periodo la salinidad llegó a bajar hasta 11 UPS y la temperatura no bajó de 31.7°C en las horas de la tarde, lo que coincide con lo reportado por Wang y Gu (9), quienes encontraron mortalidades hasta del 90% en cultivo del cangrejo de agua dulce Eriocheir sinensi, con temperaturas relativamente altas, al igual que lo reportado por Wang et al (12) quienes detectaron la presencia de Spiroplasma en la langosta de agua dulce Procambarus clarkii, en periodo de verano, donde las temperaturas son elevadas. De este mismo modo Nunan et al (13) reportaron mortalidades en cultivo de camarón Penaeus (litopenaeus) vannamei en piscinas que presentaban valores de salinidad bajos y altas temperaturas.
En las fincas muestreadas los valores de temperatura variaron entre 28.9 ± 1.36°C y 35.1 ± 0.94°C, observándose altos valores en los meses de junio y julio, mientras que los valores de salinidad variaron entre 21.0 ± 1.73 UPS y 29.5 ± 0.50 UPS. Los valores más bajos se observaron en el mes de junio y julio. En el mes de julio se presentaron mortalidades ocasionadas por Spiroplasma, lo que confirma que esta bacteria se encuentra presente en aguas de baja salinidad con elevadas temperaturas.
En los muestreos realizados se detectó la presencia de Spiroplasma en el (4.6%) de las muestras procesadas, dentro de estos sobresalieron los insectos pertenecientes al orden Orthóptera, Lepidóptera, espécies del género Hesperocorixa y un representante de los Dípteros (mosca de agua). Hesperocorixa fue positivo por PCR en todas las piscinas muestreadas, al igual que las moscas de agua. En todas las piscinas muestreadas se detectó Spiroplasma por PCR en las muestras de músculo de camarón LitoPenaeus vannamei.
CENIACUA en muestreos realizados en años anteriores detectó Spiroplasma en muestras de callianassa; sin embargo en el presente estudio en ningún momento se encontraron callianassas sintomáticas y no se detectó la presencia de la bacteria por las técnicas de histología y PCR, por lo que se descarta la posibilidad de que las callianassas sean vectores de infección.
Aunque se piensa que el manglar de algún modo influye con la incidencia de la bacteria, dado que las primeras piscinas en presentar síntomas de la enfermedad son las cercanas a este, no se pudo detectar la presencia de Spiroplasma al igual que en el canal reservorio; sin embargo no se descarta la posibilidad de que la bacteria está asociada al manglar.
La PCR de bacterias utilizando iniciadores de genes 16S pueden presentar coamplificación con otros géneros como Micoplasma hominis y Micoplasma mycoides que poseen homología con la secuencia de Spiroplasma en algunos pares de bases Gasparich et al (19). Además, debido a la elevada sensibilidad del método, el riesgo de contaminación es considerable, por lo que existe la posibilidad de obtener resultados falsos positivos (20). Por esto es necesario procesar los resultados positivos a través de PCR en tiempo real, debido a que por medio de esta técnica además de cuantificar la cantidad de ADN amplificado, se puede evaluar la especificidad al calcular la fluorescencia versus temperatura en una curva de disociación. Estas curvas dan lugar a la temperatura de disociación (Tm) que depende sobre todo de la composición de las bases y de la longitud de la secuencia amplificada, pudiéndose diferenciar la secuencia blanco de otras secuencias amplificadas como dímeros de iniciadores que se disocian a menores temperaturas (21-22).
El organismo en el que se observó presencia de Spiroplasma por PCR en tiempo real fue la mosca de agua. A pesar que Hesperocorixa y el representante del orden orthóptera (sp1) arrojaron resultados positivos por PCR, no se pudo comprobar que estos eran portadores de la bacteria por PCR en tiempo real. Otro aspecto a tomar en cuenta es el hecho de que los primers están diseñados para secuencias del género Spiroplasma a partir de una región 16S, por lo que no se puede asegurar que el Spiroplasma detectado en la mosca de agua, sea el mismo que afecta a los camarones de cultivo. Por tal razón fue necesario secuenciar esta muestra para verificar la presencia de esta especie de bacteria.
Al comparar esta muestra con secuencias de bacterias pertenecientes al GenBank usando el algoritmo BLAST, se encontró 100% de homología con un fragmento de los genes ribosomales 16S de la bacteria Spiroplasma penae, acceso gb|AY771927.1.
A través de este método se comprobó que la mosca de agua es portadora de S. penaei; sin embargo no se puede afirmar que este organismo sea el vector de la infección, puesto que para comprobarlo se sugiere realizar ensayos experimentales con moscas de agua infectadas con Spiroplasma penaei en camarones libres de patógenos, y así determinar si son transmisores de la enfermedad.
El género Spiroplasma se ha logrado aislar de varias especies de plantas y de insectos pertenecientes a los órdenes Díptera, Himenóptera, Coleóptera, Lepidóptera, Homóptera y Hemíptera (6-23-24). Fletcher et al (23) reportaron a los insectos Circulifer tenellus, Circulifer hematoceps y Circulifer opacipennis como vectores de infección de S. citri en árboles cítricos; mientras que Tsai (25) encontró que S. kunkelli, patógeno del maíz es transmitido a través del género Dalbulus. En general los insectos son infectados con Spiroplasma a través del polen de la flor, causando de este modo grandes pérdidas económicas a más de 300 especies de plantas agrícolamente importantes (26).
Como conclusión, se plantea la probabilidad de que la mosca de agua sea el vector de infección de S. penaei en camarones, teniendo en cuenta que el camarón es omnívoro (14-15) y que la mayoría de los vectores de Spiroplasma son los insectos (6-23-24). Como los organismos involucrados con Spiroplasma fueron los insectos, se hace recomendable evaluar las muestras pertenecientes a este grupo con bioensayos ex situ. Además de tomar ciertas medidas de bioseguridad en las fincas tales como limpiar la maleza que se encuentra en el terraplén y alrededor de las piscinas, para destruir el hábitat de organismos indeseables en el ciclo de cultivo de camarón.
Referencias
1. FAO (Food & Agriculture Organization of the United Nations)(2002). The State of World Fisheries and Aquaculture (SOFIA). Rome: FAO. [ Links ]
2. Kautsky N, Ronnback P, Tedengren M, Troell M. Ecosystem perspectives on management of disease in shrimp pond farming. Aquaculture 2000; 191:145-161. [ Links ]
3. Rosemberry B. World shrimp farming. Annual Report Shrimp News International. San Diego: World Aquaculture Society; 2001. [ Links ]
4. Kautsky N, Ronnback P, Tedengren M, Troell M. Ecosystem perspectives on management of disease in shrimp pond farming. Aquaculture 2000; 191:145-161. [ Links ]
5. Aguirre GG and Valle AF. Infectious disease in shrimp species with aquaculture potential. Recent Res Dev Microbiol 2000; 4:333-348. [ Links ]
6. Martin GG, Rubin N, Swanson E. Vibrio parahaemolyticus and V. harveyi cause detachment of the epithelium from the midgut trunk of the penaeid shrimp Sicyonia ingenti. Dis Aquat Organ 2004; 60: 21-29. [ Links ]
7. Yokomi R, Mello AFS, Saponari M and Fletcher J. Polymerase chain reaction-based detection of Spiroplasma citri associated with citrus stubborn disease. Plant Dis 2008; 92:253-260. [ Links ]
8. Wang W, Rong L, Gu W, Du K, Chen J. Study on experimental infections of Spiroplasma from the Chinese mitten crab in crayfish, mice and embryonated chikens. Res Microbiol 2003; 154: 677-680. [ Links ]
9. Wang W, Chen J, Du K, Xu Z. Morphology of Spiroplasmas in the Chinese mitten crab Eriocheir sinensis associated with tremor disease. Res Microbiol 2004; 155:630-635. [ Links ]
10. Wang W, Gu ZF. Rickettsia-like organism associated with tremor disease and mortality of the Chinese mitten crab Eriocheir sinensis. Dis Aquat Org 2002; 48: 149-153. [ Links ]
11. Zbinden M, Cambon-Bonavita MA. Occurrence of Deferribacterales and Entomoplasmatales in the deep-sea alvinocarid shrimp Rimicaris exoculata gut. FEMS Microbiol Ecol 2003; 46: 23-30. [ Links ]
12. Nunan LM, Pantoja CR, Salazar M, Aranguren F, Lightner DV. Characterization and molecular methods for detection of a novel Spiroplasma pathogenic to Penaeus vannamei. Dis Aquat Organ 2004; 62: 255-264. [ Links ]
13. Wang W, Gu W, Ding Z, Ren Y, Chen J, Hou Y. A novel Spiroplasma pathogen causing systemic infection in the crayfish Procambarus clarkii (Crustacea: Decapod), in China. FEMS Microbiol Lett 2005; 249: 131-137. [ Links ]
14. Nunan LM, Lightner DV, Oduori MA, Gasparich GE. Spiroplasma penaei sp. nov., associated with mortalities in Penaeus vannamei, Pacific white shrimp. Int J Syst Evol Microbiol 2005; 55: 2317-2322. [ Links ]
15. Delgado JG, Poveda CM, Cahu C. Digestive enzyme activity and food ingesta in juvenile shrimp Litopenaeus vannamei (Boone, 1931) as a function of body weight. Aquac Res 2003; 34:1403-1411. [ Links ]
16. Otoshi CA, Montgomery AD, Look AM and Moss SM. Effects of diet and water source on the nursery production of pacific white shrimp Litopenaeus vannamei. J World Aquac Soc 2001; 32: 243-249. [ Links ]
17. Chena R, Sugawara H, Koike T, Lopez R, Gibson TJ, Higgins DG and Thompson JD. Multiple sequence alignment with the Clustal series of programs. Nucleic Acids Res 2003; 31: 3497-3500. [ Links ]
18. Hall TA. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symp Ser 1999; 41: 95-98. [ Links ]
19. Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol 1990; 215: 403-10. [ Links ]
20. Gasparich GE, Whitcomb RF, Dodge D, French FE, Glass, J, Williamson DL. The genus Spiroplasma and its nonhelical descendants: phylogenetic classification, correlation with phenotype and roots of the Mycoplasma micoydes clade. Int J Syst Evol Microbiol 2004; 54: 893-918. [ Links ]
21. Costa J. Real time PCR. Enferm Infecc Microbiol Clin 2004; 22: 299-305. [ Links ]
22. Erali M and Wittwer C. High resolution melting analysis for gene scanning. Methods 2010; 50:250-261. [ Links ]
23. Philip B and Wittwer C. Homogeneous amplification and variant detection by fluorescent hybridization probes. Clin Chem 2000; 46:147-148. [ Links ]
24. Fletcher J, Melcher V, Wayadende A. The Phytopathogenic Spiroplasmas. In: Dworkin M, Falkow S, Rosenberg E, Schleifer K, Stackebrandt E. The Prokaryotes. New York. Springer; 2006. p 905-947. [ Links ]
25. Barros TS, Davis RE, Resende RO. Design of polymerase chain reaction for specific detection of corn stunt Spiroplasma. Plant Diseases 2001; 85: 475-480. [ Links ]
26. Seemüller E, Garnier M and Schneider B. Mycoplasmas of plants and insects. In: Razin S and Herrmann R. Molecular biology and pathogenicity of mycoplasmas. New York: Kluwer Academic/Plenum Publishers; 2002. p 91-115. [ Links ]
27. Summers CG and Stapleton JJ. Management of corn leafhopper (Homoptera: Cicadellidae) and corn stunt disease in sweet corn using reflective mulch. J Econ Entomol 2002; 95:325-330. [ Links ]














