Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268
Rev.MVZ Cordoba vol.18 no.1 Córdoba Jan./Apr. 2013
ORIGINAL
Estructura y diversidad genética en vacas Holstein de Antioquia usando un polimorfismo del gen bGH
Structure and genetic diversity of a population Holstein cows of Antioquia department, using a polymorphism of bGH gene
Juan Rincon F,1* M.Sc, Albeiro Lopez H,1,2 Ph.D, Julian Echeverri Z,1,3 Ph.D.
1Grupo BIOGEM. Universidad Nacional de Colombia, Facultad de Ciencias Agropecuarias. Calle 59 A N° 63-20, Medellín, Colombia.
2Universidad Nacional de Colombia, Facultad de Ciencias Agropecuarias. Medellín, Colombia
3Universidad Nacional de Colombia, Facultad de Ciencias Agropecuarias. Medellín, Colombia.
*Correspondencia: jcrincon@unal.edu.co
Recibido: Mayo de 2011; Aceptado: Junio de 2012.
RESUMEN
Objetivo. Determinar las frecuencias alélicas y genotípicas del polimorfismo del intrón 3 del gen bGH y estimar algunos parámetros de estructura poblacional en ganado Holstein. Materiales y métodos. El estudio se realizó con 1366 vacas Holstein en 120 hatos de 11 municipios del departamento de Antioquia. Se extrajo DNA por el método de Salting out y la genotipificación se realizó usando la técnica de PCR-RFLPs. La diversidad genética se determinó mediante la comparación de las heterocigosidades, El equilibrio de Hardy-Weinberg (HW) y la diferenciación genética entre las poblaciones se realizó usando el software Arlequín 2.0 Las frecuencias alélicas y genotípicas se evaluaron mediante el paquete estadístico SAS®. Resultados. Las frecuencias genotípicas encontradas fueron 0.764 (+/+), 0.223 (+/-) y 0.013 (-/-) y las frecuencias alélicas 0.876 (+) y 0.124 (-). No se encontraron desviaciones del Equilibrio de Hardy Weinberg en ninguna de las subpoblaciones. La diversidad genética determinada mediante la comparación de las heterocigosidades fue relativamente baja entre poblaciones pero al interior de estas no. El valor de FST de toda la población fue de 0.0068 y significativo (p<0.05), algunos FST pareados también lo fueron, tomando valores desde 0.0 a 0.13. Los estadísticos FIT y FIS no fueron significativos. Conclusiones. El gen bGH es un candidato interesante para evaluar características de importancia económica ya que no parece haber sido sometido a selección directa, presenta una variabilidad media en las poblaciones, observándose diferenciación genética significativa entre distintos municipios, producto de los diferentes sistemas de producción y acceso a las biotecnologías.
Palabras clave:Ganado lechero, genética de poblaciones, marcadores moleculares, polimorfismo (Fuente: Agrovoc).
ABSTRACT
Objective. To determine the allele frequencies and genotypic polymorphism of the intron 3 of gene bGH and estimate structural parameters in Holstein cattle populations. Materials and methods. The study was conducted with 1366 Holstein cows belonging to 120 herds in 11 municipalities of the department of Antioquia. DNA was extracted by the Salting out method and genotyping was carried out using PCR-RFLP. Genetic diversity was determined by comparing the heterozygosities, Hardy-Weinberg (HW) and genetic differentiation between populations was performed using the Arlequin software 3.0. The allelic and genotyping frequencies were assessed using the SAS statistical software. Results. The genotype frequencies found were 0.764 and 0.013 (+/-) 0.223 (+/+), (-/-) and allele frequencies were 0.876 (+) and 0124 (-). There was no unbalance for Hardy Weinberg in the subpopulations. The genetic diversity determined by comparison of the heterozygosity was low among populations but within them it was not. The FST value of the entire population was 0.0068 and significant (p<0.05), FST also matched some were values ranging from 0.0 to 0.13. The statistical FIT and FIS were not significant. Conclusions. The gene bGH is an interesting candidate to economically evaluate important traits because it does not seem to have been subject to direct selection, has a mean variability in populations, showing significant genetic differentiation between several municipalities, resulting from the different production systems and access to biotechnologies.
Key words: Dairy cattle, molecular markers, polymorphism, population genetics (Source: Agrovoc).
INTRODUCCIÓN
El ganado Holstein en Antioquia ha sufrido cambios genéticos diversos en diferentes poblaciones, debido principalmente a la selección indirecta y a las diferencias en el tipo de producción y uso de tecnologías reproductivas como la inseminación artificial, por tanto es necesario determinar el efecto generado por dichos factores sobre la estructura genética poblacional. En la actualidad se conocen un amplio número de marcadores genéticos que pueden ser usados para evaluar la diferenciación poblacional (1). Entre estos marcadores se encuentran algunos relacionados con parámetros productivos en ganado de leche (2), como es el caso del polimorfismo en un solo nucleótido o SNP que son marcadores polimórficos que pueden ser reconocidos mediante varias técnicas (3), entre las cuales se encuentra la PCR (reacción en cadena de la polimerasa) asociada a RFLP (polimorfismo en la longitud de los fragmentos de restricción) (4). Uno de estos marcadores es un SNP del gen de la Hormona del crecimiento Bovina (bGH) que permite la amplificación de un segmento que contiene un sitio polimórfico que puede ser reconocido por la endonucleasa MspI, generando 3 genotipos diferentes (+/+,+/- y -/-) (4).
El gen de bGH contiene 1800 pb, con 5 exones y 4 intrones (5), localizados en el cromosoma 19 que produce una proteína compuesta de 190 a 191 aminoácidos con un peso molecular aproximado de 22 KDa (6) que contienen Ala o Phe en el extremo N-terminal debido a un procesamiento alternativo de los precursores de bGH (7,8). Actualmente se conocen múltiples polimorfismos al interior del gen o en regiones reguladoras (9). Una de estas variantes reportadas corresponde a un sitio que puede ser reconocido por la endonucleasa de restricción MspI (10), el cual fue localizado como un polimorfismo en el intrón 3 en la posición 1547, que se debe a la transición de Timina, T a Citosina, C (11).
Algunos parámetros poblacionales estimados usando el gen de bGH, permiten calcular las frecuencias genotípicas, alélicas y el flujo de genes entre las poblaciones para determinar su grado de diversidad y diferenciación (1). La determinación del equilibrio de Hardy-Weinberg (HW), la estimación de las frecuencias alélicas y genotípicas, posibilitan no solo determinar el comportamiento poblacional, sino que de acuerdo a las frecuencias de los alelos en la población permiten determinar la importancia de dicho polimorfismo en un programa de mejoramiento genético, ya que aquellos alelos que se encuentran casi fijados en una población tendrían diferente respuesta a la selección que aquellos alelos que se encuentran en proporciones intermedias.
Los parámetros de estructura poblacional describen el grado de reducción de heterocigosidad en una población, con base en el equilibrio de Hardy-Weinberg, lo cual faculta la comparación entre grupos a diferentes escalas jerárquicas y determinar el grado de diferenciación entre ellos (12), de tal forma que posibilitan valorar el modo de acción y la intensidad con que se debe actuar para aumentar la presencia de un alelo específico dentro de un programa de selección asistida por marcadores moleculares.
El objetivo de la presente investigación fue determinar los parámetros de estructura poblacional, las frecuencias alélicas y genotípicas del gen de bGH en vacas Holstein del departamento de Antioquia, como aporte para la implementación de programas de selección asistida por marcadores moleculares (MAS).
MATERIALES Y MÉTODOS
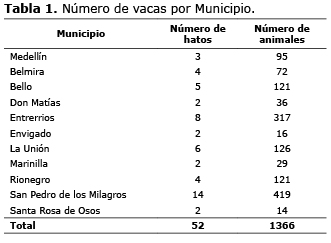
Sitio de estudio. La presente investigación se llevó a cabo en el trópico alto de la región norte y oriente del departamento de Antioquia, e incluyó 1366 vacas de la raza Holstein, localizadas en 120 hatos de lechería especializada, pertenecientes a 11 municipios (subpoblaciones) del departamento. Los municipios evaluados fueron San Pedro de los Milagros (Altura: 2.475 m.s.n.m; T°: 14°C), Belmira (Altura: 2.550 m.s.n.m; T°: 14°C), Entrerrios (Altura: 2.300 m.s.n.m; T°: 16°C), La Unión (Altura: 2.500 m.s.n.m; T°: 13°C), Envigado (2000-2300 m.s.n.m; T°: 16°C), Bello (Zonas entre los 2000 a 2400 m.s.n.m; T°: 16°C), Medellín (Zonas entre los 2000 a 2500 m.s.n.m; T°:15°C), Santa Rosa de Osos (2581 m.s.n.m; T°: 14°C), Don Matías (2200 m.s.n.m; T°: 16°C), Marinilla (2150 m.s.n.m; T°: 17°C) y Rionegro (2150 m.s.n.m; T°: 17°C) (Tabla 1).
Extracción del DNA. Para la toma de las muestras de sangre se utilizó BD vacutainer de 5ml con EDTA como anticoagulante, con agujas número 18 (BD VacutainerTM). Los tubos fueron homogenizados por inversión, rotulados para su identificación y refrigerados con hielo durante su transporte hasta el laboratorio de Biología Celular y Molecular de la Universidad Nacional de Colombia sede Medellín, para realizar la extracción del DNA.
A partir de estas muestras se realizó el protocolo de extracción de DNA basado en el método de “salting out” modificado (13) y el DNA se almacenó en Buffer TE 1X (tris-EDTA) a 4°C hasta el momento del análisis.
La cantidad y calidad del ADN extraído se evaluó en gel de agarosa al 0.8% teñido con bromuro de etidio, y se semicuantificó con el transiluminador (Biometra®). La pureza del DNA genómico se determinó mediante un análisis de absorbancia en dos longitudes de onda. Sólo el ADN genómico con una pureza entre 1.8-2.0 se consideró para los estudios a realizar.
Amplificación de la región polimórfica del gen bGH. Con base en las secuencias del gen bGH y a los reportes de Dybus (14), se sintetizaron 2 oligonucleotidos de 22 pares de bases que permitieron amplificar el fragmento de 329 pb que presenta el sitio de restricción para la endonucleasa MspI. Los cebadores sintetizados fueron:
F 5`- CCCACGGGCAAGAATGAGGC-3`
R 5`- TGAGGAACTGCAGGGGCCCA-3`
Se realizó una amplificación por PCR individual para cada región específica en un volumen final de 25 µL, que contenía 30-60 ng de DNA genómico, 2.5 µL de buffer PCR 10X (1.0 -1.5 mM de MgCl2, 50 mM de KCl, 10 mM de Tris-HCl, pH de 8.3), 2 mM de MgCl2, 0.2 µM de cebadores; 0.4 mM de cada dNTP y 0.5-1 unidad de taq polimerasa (Bioline®).
Las condiciones de la reacción en cadena de la polimerasa (PCR) Fueron: 1. Desnaturalización a 95°C por 6 min, 2. Desnaturalización a 95°C por min, 3. Temperatura de alineamiento (anneling) de 60°C por 30 seg, 4. Extensión a 72°C por 40 seg, 5. Se repitió desde el paso 2 durante 39 ciclos para finalizar con una extensión por 5 min a 72°C. La PCR se llevó a cabo en un termociclador (Biometra®).
Los productos de PCR fueron resueltos mediante electroforesis en gel de agarosa al 2% (Amresco®), teñido con bromuro de etidio (Promega®). En cada pozo se sirvieron 5 µl del producto de PCR diluidos en 2 µl de buffer carga 1X (fermentas®) y se utilizaron 2 µl de marcador de peso molecular (low range Fermentas ®). Los geles fueron visualizados mediante un fotodocumentador (BiometraTM®) de geles para tomar evidencia fotográfica. En todos los casos en la PCR se utilizó un control negativo sin DNA y un control positivo que poseía un DNA previamente amplificado con éxito.
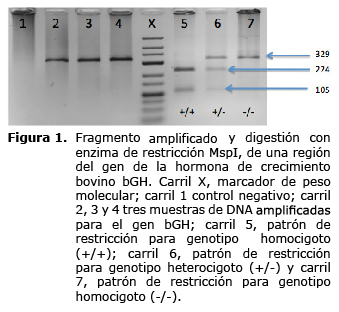
Digestión con enzimas de restricción (PCR-RFLP). Una vez se comprobó la amplificación del fragmento de 329 pb, los productos de PCR fueron almacenados y posteriormente sometidos a digestión con la endonucleasa de restricción MspI, la cual cortó el fragmento en presencia de un sitio especifico, generando un patrón de bandas que permiten clasificar los alelos como (+) cuando presentaron corte y como (-) cuando no presentaron corte (14), de tal forma que los individuos de las poblaciones tuvieron genotipos (+/+), (+/-) y (-/-) de acuerdo al patrón de bandas generado. El alelo (-) no presentó sitio de corte, por tanto su fragmento fue de 329 pb, el alelo (+) generó 2 fragmentos de 224 y 105 pb (Figura 1).
Para la digestión se utilizaron 2 µl de buffer Tango 10X para obtener una concentración final de 1X y un volumen de reacción de 20 µl, se adicionó 5 unidades de enzima, se completó con agua ultrapura hasta 15 µl y se utilizaron 5 µl del producto de PCR obtenido. Una vez realizada la mezcla esta se sometió a digestión durante 3 horas a 37°C. La enzima en esta reacción se utilizó en exceso para evitar la formación de falsos heterocigotos, debido a digestiones parciales del producto de PCR.
En todas las digestiones se incluyó un control positivo que presentaba el sitio de restricción en ambos alelos, y de esta manera se procuraba evitar la generación de falsos positivos o negativos.
Los productos de la digestión fueron resueltos por electrofóresis en gel de agarosa al 2.5% (Amresco®) en buffer TBE 1X (EDTA 0.05M, Tris base 0.089 M y Ácido bórico 0.089 M), teñido con Bromuro de Etidio (Promega®). En cada pozo se sirvieron 15 µl del producto de la digestión diluidos en 5 µl Buffer de carga 1X (Fermentas®) y se utilizaron 2 µL de un marcador de peso molecular de muy bajo rango (Low Range Fermentas®). Los geles fueron visualizados bajo luz ultravioleta en un equipo de fotodocumentación de geles (BiometraTM®) con el cual se obtuvo una fotografía digital.
Frecuencias alélicas y genotípicas. La frecuencia de los diferentes alelos se realizó determinando la proporción de cada forma del gen entre el número de copias totales de la población en estudio. Se identificaron los homocigotos (dos copias del mismo alelo) y los heterocigotos (una copia de cada alelo) y se calculó la frecuencia F de cada alelo contando los homocigotos y añadiendo la mitad de los heterocigotos, con el método descrito por Hartl, (15). La frecuencia genotípica se calculó como la sumatoria de cada genotipo sobre el total de la población (15). Ambas frecuencias se determinaron usando el programa SAS® (16).
Diversidad genética. La diversidad genética de la población fue determinada mediante la comparación de la Heterocigosidad observada (Ho) y la Heterocigosidad esperada (He). Este análisis se llevó a cabo usando el software Arlequín v 3.0 (17), Que utiliza el método de la cadena de Markov (18).
Estructura genética. El equilibrio de Hardy-Weinberg se calculó con base en las frecuencias alélicas y genotípicas esperadas y observadas mediante el uso del software Arlequín v 3.0 (17). Los parámetros de estructura poblacional, se calcularon utilizando el método Propuesto por Wright que parte el coeficiente de endogamia de una población subdividida (FIT), entre el componente debido a apareamientos no aleatorios dentro de una población (FIS) y las subdivisiones entre poblaciones FST (19) teniendo en cuenta dos niveles jerárquicos que incluyen a Antioquia como la población total y los municipios actuando como subpoblaciones. Teniendo en cuenta lo anterior se determinó el parámetro FIT= HT-Hi/HT, que corresponde a la endogamia total, el parámetro FIS= HS-HI/HS que mide la subdivisión intrapoblacional y el FST=FIT-FIS/1-FIS que mide la subdivisión poblacional (19); donde HT se refiere a la heterocigosidad esperada en la población total, HI es la heterocigosidad promedio observada en un grupo de poblaciones y HS es la heterocigosidad promedio esperada de cada población. Los cálculos fueron realizados con el uso del software Arlequín v 3.0 (17), utilizando para ello el análisis de varianza molecular (AMOVA) que permite analizar la variación entre y dentro de poblaciones, con su significancia estadística, bajo la hipótesis nula “los alelos o genotipos tienen la misma distribución en todas las poblaciones”, pero sin requerir equilibrio de Hardy Weinberg.
El flujo génico expresado como el numero de migrantes (Nm) por generación, se calculó con base a la siguiente expresión derivada de los estadísticos de Wright (19).
RESULTADOS
Determinación de las frecuencias alélicas y genotípicas. Los fragmentos del gen bGH de 329 pb fueron amplificados exitosamente en 1366 muestras. La digestión del fragmento de DNA amplificado fue digerido correctamente con la enzima de restricción MspI. Las muestras fueron catalogadas como (+/+, +/- y -/-) de acuerdo a los tamaños de los fragmentos generados (figura. 1).
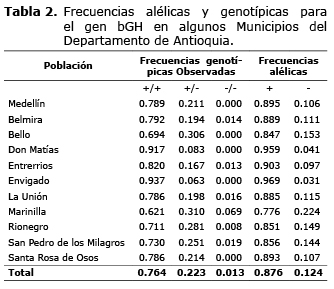
Las frecuencias de los alelos + y - en la población total fueron 0.876 y 0.124 respectivamente. La frecuencia de los genotipos +/+, +/- y -/- fueron 0.764, 0.223 y 0.013 respectivamente. Las frecuencias alélicas y genotípicas por municipios y la total se presentan en la tabla 2.
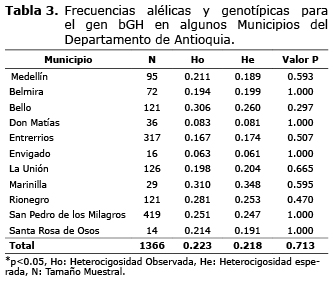
Diversidad genética. La Heterocigosidad observada y esperada tuvo una variación de 0.063 a 0.310 y de 0.061 a 0.348, respectivamente, lo que sugiere que hay una variación genética considerable entre las poblaciones, pero, al interior de las mismas no se percibe evidencia de que estas hayan estado sometidas a alguna fuerza genética como selección, mutación, u otra (Tabla 3).
En ninguno de los municipios evaluados se encontraron evidencias suficientes para suponer desvíos significativos (p>0.05) en el equilibrio de Hardy Weinberg, por lo tanto no hay evidencias para suponer cambios en las frecuencias genotípicas de una generación a otra (Tabla 3).
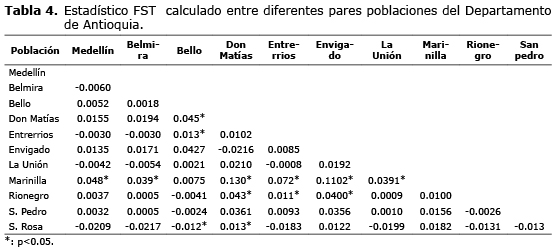
Estructura y diferenciación genética de las poblaciones. El estadístico FST para la población total fue 0.0068 con un valor de significancia p=0.0107, indicando que algunas de las poblaciones se han diferenciado, aunque la magnitud de la diferenciación genética es baja según los rangos propuestos por Wright (19).
Los FST pareados por municipios, estuvieron entre 0.00 y 0.130, con algunos valores significativos (Tabla 4); indicando diferenciación baja ó media en algunos casos, y ausencia de significancia en la diferenciación de algunas poblaciones pareadas. Los valores de FST pareados entre las poblaciones Marinilla-Envigado (0.11) y Marinilla-Don Matías (0.13) presentaron una diferenciación Media y significativa según los rangos propuestos por Wright (19).
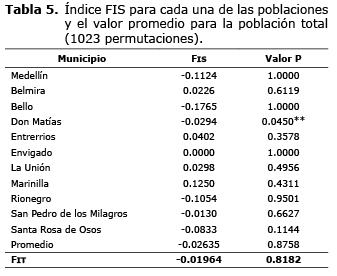
El valor de FIS promedio obtenido fue de -0.02527, pero no fue significativo (p>0.05), del mismo modo los valores de FIS por municipio carecieron de significancia (p>0.05), por lo tanto no es posible asumir tendencias a la endogamia, ni a la exogamia en la mayoría de los municipios analizados, a excepción del Municipio de Don Matías que fue el único que presentó un FIS significativo, con un valor de -0.0294, indicando una pequeña tendencia a la exogamia en esta población (Tabla 5).
El estadístico FIT obtenido fue de -0.0207, pero no fue significativo (p>0.05) y por tanto no se tienen argumentos suficientes para suponer una tendencia a la exogamia en la población total (tabla 5).
Flujo génico. El valor medio de flujo de genes representado como Nm (Número de migrantes) fue de 36, un valor muy alto considerando que la tasa efectiva de migración de más de un inmigrante por generación, es suficiente para evitar una diferenciación por efectos de deriva génica. La baja diferenciación genética (FST) y la alta tasa efectiva de migración (Nm) observada en estas poblaciones puede estar relacionada directamente con la utilización de inseminación artificial y la comercialización de vacas entre los diferentes municipios.
DISCUSIÓN
Las poblaciones evaluadas en el departamento de Antioquia no presentaron desviaciones significativas (p>0.05) del equilibrio de Hardy-Weinberg para el gen de bGH, lo que parece indicar que dichas poblaciones no han sido sometidas a un proceso fuerte de selección sobre este gen recientemente, de tal manera que no se ha perturbado el equilibrio de una generación a la siguiente. Los resultados obtenidos concuerdan con los encontrados por Gorbani et al (20) en el 2009 al evaluar 183 toros iraníes, pero difieren de los encontrados por Mohammadabadi et al (21) en 2010 al usar el gen bGH en toros Iraníes, pero con la enzima de restricción AluI. En Colombia Echeverri et al (22) en el 2010 no encontraron diferencias significativas entre las frecuencias alélicas ni genotípicas entre fincas, al evaluar 165 vacas Holstein en diferentes municipios del Departamento de Antioquia (Colombia). Usme en el 2009 reportó equilibrio HW en el departamento de Antioquia con el gen de kappa-caseina (23).
La heterocigosidad puede ser utilizada como un indicador de diversidad genética y en el presente trabajo tomó valores de medios a bajos (Tabla 3) de acuerdo a la población. Resultados similares se han encontrado en diferentes estudios (14, 20 y 22) con heterocigosidades mayores para este polimorfismo (MspI) solo reportadas en Bos indicus (20). Con respecto a la heterocigosidad observada y esperada no se encontraron diferencias significativas (p>0.05) en ninguno de los municipios, ni en la población total (Tabla 3), por lo tanto no se puede asumir que haya déficit o exceso de heterocigotos. La mayor heterocigosidad encontrada se presentó en Marinilla (h=0.310), lo que indica que dicha población presenta una diversidad genética importante posiblemente porque en esta población no se han realizado esfuerzos importantes en la selección de animales. Los municipios de Don Matías y Envigado presentaron valores de heterocigosidad muy bajos (0.083 y 0.063) indicando una muy baja variabilidad genética, posiblemente por selección indirecta, en dichas poblaciones de importancia en la producción de leche. En el caso de Don Matías, el estadístico FIS fue negativo mostrando una pequeña tendencia a la exogamia que debería corresponder a un aumento en la diversidad sobre dicha población, pero que es tan pequeño que puede no haber sido suficientemente fuerte para mostrar un cambio en la heterocigosidad.
Los resultados del presente estudio mostraron que el alelo MspI (+) fue más frecuente que el (-) (0.876 vs 0.124) en la población total, y se comportó de manera similar en las diferentes subpoblaciones variando de 0.776 a 0.969 para el alelo (+), y de 0.031 a 0.224 para el alelo (-), lo que parece indicar una variabilidad media entre municipios. En Colombia Echeverri et al (22) encontraron frecuencias alélicas de 0.85 para el alelo MspI+ y 0.15 para el MspI- en el departamento de Antioquia, valores que se encuentran entre los rangos encontrados en este trabajo. Datos similares han sido reportados no solo en la raza Holstein sino en la mayoría de razas europeas que difieren en mucho a las frecuencias encontradas en los Bos indicus (20).
Las frecuencias genotípicas mostraron que la mayoría de los animales (76.4%) fueron homocigotos (+/+), el 22.3% fueron heterocigotos y solo un pequeño porcentaje (1.3%) fueron homocigotos (-/-). Estos hallazgos fueron similares a los reportados previamente en ganado Holstein lechero (20) en los cuales se encuentra una muy alta proporción del genotipo (+/+) con una muy baja frecuencia del genotipo homocigótico (-/-), lo que parece indicar una tendencia a la fijación del alelo (+), posiblemente por la selección lenta e indirecta sobre el gen que posibilita la prevalencia de una forma alélica, pero que no es lo suficientemente fuerte y directa para ser reconocida en una prueba de Hardy Weinberg, ya que las variaciones alélicas para el alelo (+) que se encuentra en mayores proporciones no llega a ser significativa. En los municipios de Don Matías, Bello, Medellín y Envigado no se encontró el genotipo (-/-), y las frecuencias alélicas muestran una prevalencia del alelo (+) llegando incluso a valores de 96.9% lo que indica que el alelo se encuentra casi fijado (Tabla 2).
Las diferencias en las frecuencias encontradas también hablan de la selección indirecta que se ha generado en los diferentes municipios, principalmente por el apareamiento dirigido.
Los valores obtenidos de FIS y FIT no fueron significativos posiblemente por la alta variabilidad encontrada dentro de las subpoblaciones y dentro de la población total, por lo tanto, no es posible inferir alguna conclusión sobre tendencias a la endogamia y a la exogamia en ninguno de los niveles.
La población total presentó una pequeña diferenciación (FST=0.0068) significativa (p<0.05) para el gen de bGH-MspI, es decir, que por lo menos hay dos poblaciones que se encuentran bien diferenciadas entre sí, lo cual significa que Antioquia no se comporta como una única población. Los valores de FST pareados permitieron determinar de que forma se presenta la estructuración entre municipios (Tabla 4). De acuerdo a lo anterior se pudo determinar que Marinilla presenta las mayores diferencias entre poblaciones con 6 diferencias significativas, además su valor de FST pareado fue el mayor (0.13) al compararse con el municipio de Don Matías, mostrando una diferenciación moderada entre estos (19). Tal diferenciación en Marinilla se pudo haber generado por el aislamiento que presenta esta población con respecto a las demás y por la falta de programas de selección. Las demás poblaciones presentaron diferencias muy bajas y solo unas pocas fueron significativas, por tal motivo el valor de flujo genético fue muy alto (Nm=36), teniendo en cuenta que solo es necesario un inmigrante por generación para evitar la diferenciación por deriva genética.
Los programas de inseminación, en los que es posible integrar semen de toros de todo el mundo, derriban las barreras geográficas y aumentan el flujo genético, pero en algunos casos los sistemas de producción no permiten que todas las poblaciones tengan acceso a estos programas y se pueden generar barreras de flujo entre poblaciones y por lo tanto una diferenciación genética.
Los resultados obtenidos en el presente trabajo postulan al gen de bGH-MspI como un candidato interesante para la evaluación de características de importancia económica en Colombia, ya que este no ha sido sometido a procesos de selección de manera directa, presenta variabilidades medias en las frecuencias alélicas de algunas poblaciones y es de gran importancia por su papel en la lactancia, incluso puede generar resultados satisfactorios si se determina el genotipo superior para estas condiciones, con respecto a alguna característica de importancia económica.
Teniendo en cuenta lo anterior, se debe tener en cuenta que para el desarrollo de un programa de selección asistido por marcadores moleculares, es pertinente realizar la evaluación a nivel local sobre el genotipo más deseado por su relación con parámetros productivos, debido a que los reportes son ambiguos (9,14,24) y pueden ser variables de acuerdo al lugar donde son evaluados y a la raza de los animales (24). Además, en Colombia no se han reportado asociaciones significativas entre el alelo MspI posiblemente porque la única investigación llevada a cabo por Echeverri et al (22), no poseía un número de animales que permita lograr la significancia deseada.
Agradecimientos
A los propietarios de las fincas en cada municipio por permitir el desarrollo de este estudio, Al Laboratorio de Biología Molecular y Celular de la Universidad Nacional Sede Medellín.
REFERENCIAS
1. Lirón P, Ripoli V, De Luca C, Peral C and. Giovambattista G. Analysis of genetic diversity and population structure in Argentine and Bolivian Creole cattle using five loci related to milk production. Genet mol biol 2002; 25(4):413-419. [ Links ]
2. Bedoya G, Carvajal L, Bermudez N, Moreno F, Marquez M, Davies S et al. Estructura molecular y poblacional del Ganado criollo Colombiano (GCC). Rev Col Cienc Pec 2001; 14(2):109-120. [ Links ]
3. Sadeghi M, Moradi Shahr-e-Babak M, Rahimi G and Nejati A. Association Between Gene Polymorphism of Bovine Growth Hormone and Milk Traits in the Iranian Holstein Bulls. Asian J Anim Sci 2008; 2:1-6. [ Links ]
4. Zwierzchowski L, Krzyzewki J, Strzalkowskan N, Siadkowska E, and Ryniewicz Z. Effects of polymorphism of growth hormone (GH), Pit-1 and leptin (LEP) genes, cows age, lactation stage and somatic cell count on milk yield and composition of polish Black and White cows. Anim Sci Pap Rep 2002; 20:213-227. [ Links ]
5. Pawar R, Tajane K, Joshi C, Rank D and Bramkshtri B. Growth hormone gene polymorphism and its association with lactation yield in dairy cattle. Indian J Anim Sci 2007; 77(9):884-888. [ Links ]
6. Dybus A, Grzesiak W, Szatkowska I, Blaszczyk P. Association between the growth hormone combined genotypes and dairy traits in Polish Black-and-White cows. Anim Sci Pap Rep 2004; 22(2):185-194. [ Links ]
7. Zhou GL, Jin HG, Liu C, Guo SL, Zhu Q and Wu YH. Association of genetic polymorphism in GH gene with milk production traits in Beijing Holstein cows. J Biosci 2005; 30(5):595-598. [ Links ]
8. Zakizadeh S, Rahimi G, Mirae-Ashtiani S, Nejati-Javaremi A, MoradiShahrbabak M, Reinecke P, Reissmann M, Masoudi A, Amirinia C, Mirhadi S. Analysis of Bovine Growth Hormone Gene Polymorphisms in Three Iranian Native Breeds and Holstein Cattle by RFLP-PCR. Biotechnology 2006; 5(3):385-390. [ Links ]
9. Ge W, Davis M, Hines H. Association of single nucleotide polymorphisms in the growth hormone and growth hormone receptor genes with blood serum insulin like growth factor I concentration and growth traits in angus cattle. J Anim Sci 2003; 81(3):641-648. [ Links ]
10. Khatami S, Lazebny O, Maksimenko V, Sulimova G. Association of DNA polymorphisms of the growth hormone and Prolactin genes with productivity in Yaroslavl and Black and White cattle. Russian J Genet 2005; 41:167-173. [ Links ]
11. Sodhi M, Mukesh M, Prakash B, Mishra B, Sobti R. MspI Allelic Pattern of Bovine Growth Hormone Gene in Indian Zebu Cattle (Bos indicus) Breeds. Biochem Genet 2007; 45(1-2):145-153. [ Links ]
12. Hartl L, Clark G. Principles of population genetics. 4th Ed. Sunderland: Sinauer Associates Inc. Publishers; 2007. [ Links ]
13. Pashaei S, Azari M, Hasani S, Khanahmadi A, Rostamzadeh J. Genetic diversity in mazandaranian native cattle: a comparison with Holstein cattle, using ISSR marker. Pak J Biol Sci 2009; 12(9):717-721. [ Links ]
14. Dybus A. Association of Growth Hormone and Prolactin genes polymorphisms with milk production traits in Polish Black and White Cattle. Anim Sci 2002; 20:203-212. [ Links ]
15. Hartl L. A primer of populations Genetics. 3rd ed. Sunderland: Sinauer Associates Inc. Publishiers; 2000. [ Links ]
16. SAS Institute Inc. SAS/STAT User`s Guide: Version 9th edition [Programa para computador]. Cary (NC), USA: SAS Institute Inc; 2002. [ Links ]
17. Arlequin: An integrated software package for population genetics data analysis [programa de computador]. Version 3.0. Berna, Suiza: Evolutionary Bioinformatics Online 2005. [ Links ]
18. Hamilton M. Population Genetics. 1st Ed. London: Blackwell Publishing; 2009. [ Links ]
19. Provine W. The origins of theoretical population genetics. Chicago: University of Chicago Press; 2001. [ Links ]
20. Gorbani A, Vaez R, Bonyadi M and Amirinia C. A MspI PCR-RFLP within bovin growth hormone gene and its association with sperm quality traits in Iranian Holstein Bulls. Afr J Biotechnol 2009; 8(19):4811-4816. [ Links ]
21. Mohammadabadi R, Torabi A, Tahmourespoor M, Baghizadeh A, Esmailizadeh A and Mohammadi A. Analysis of bovine growth hormone gene polymorphism of local and Holstein cattle breeds in Kerman province of Iran using polymerase chain reaction restriction fragment length polymorphism (PCR-RFLP). Afr J Biotechnol 2010; 9(41):6848-6852. [ Links ]
22. Echeverri J, Vasquez N and Gallo Y. Polymorphism of the bovine somatrotropin and its association to the most important characteristics of dairy industry. Rev. Lasallista Investig (En linea) 2010 Febrero 24 (fecha de Acceso 15 de febrero de 2011); 7 (1): 58-65. URL disponible en: http://www.scielo.unal.edu.co/scielo.php?script=sci_arttext&pid=S1794-44492010000100008&lng=en&nrm=iso>.ISSN 1794-4449. [ Links ]
23. Usme J, Restrepo F y Trujillo-Bravo E. Kappa-Caseina Bovina y su asociación con el recuento de células somáticas en ganado Holstein. Actual Biol 2004; 26(80):17-22. [ Links ]
24. Mattos K, Lama S, Martinez M and Ferreira A. Association of bGH and Pit-1 gene variants with milk production traits in dairy Gyr bulls. Pesq Agropec Bras 2004; 39(2):147-150. [ Links ]