Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista MVZ Córdoba
Print version ISSN 0122-0268
Rev.MVZ Cordoba vol.18 no.1 Córdoba Jan./Apr. 2013
ORIGINAL
Estructura genética poblacional del gen lactoferrina bovino en vacas Holstein del departamento de Antioquia
Population genetic structure of bovine lactoferrine gen in holstein breed of Antioquia Department
Nancy Rodríguez C,1* M.Sc, Albeiro López H,1 Ph.D, Julián Echeverri Z,1 Ph.D.
1niversidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agrarias. Calle 59A N° 63-20, Medellin, Colombia.
*Correspondencia: nancyrodrigu@gmail.com
Recibido: Octubre de 2010; Aceptado: Diciembre de 2011.
RESUMEN
Objetivo. Estimar algunos parámetros de estructura poblacional en una población Holstein del departamento de Antioquia. Materiales y métodos. El estudio se realizó con 427 vacas de la raza Holstein pertenecientes a 5 municipios del departamento de Antioquia. La genotipificación se llevó a cabo usando la técnica de PCR-PFLPs. La Heterocigocidad observada (Ho) y Heterocigocidad esperada (He), la prueba de Hardy-Weinberg (HW) y la estructura y diferenciación genética entre las poblaciones se calculó mediante los parámetros F de Wright, evaluados mediante el software GENEPOP. Las frecuencias alélicas y genotípicas se evaluaron con el método descrito por Hartl. Resultados. Las frecuencias genotípicas encontradas fueron 0.61, 0.34 y 0.05 para los genotipos AA, AB y BB respectivamente y las frecuencias de los alelos fueron 0.78 y 0.22 para A y B, encontrándose la población en equilibrio de HW. La heterocigocidad fue media entre poblaciones (Ho=0.368). Los valores FIS, FST y FIT de la población total fueron -0.0717, 0.0099 y -0.0611. Conclusiones. No fue posible asumir endogamia, ni exogamia en los municipios analizados, exceptuando el municipio de San Pedro de los Milagros, en cuyo caso se percibe de manera más fuerte el efecto del mejoramiento genético y la disminución de la heterocigocidad.
Palabras clave: Análisis genético, caracterización, polimorfismo (Fuente: CAB).
ABSTRACT
Objective. Estimate some population structure parameters in a Holstein population of the Department of Antioquia. Materials and methods. Research involved 427 Holstein cows of five municipalities in the department of Antioquia. The cattle was genotyped using PCR-PFLP. Observed heterozygosity (Ho) and expected heterozygosity (He), the exact Hardy-Weinberg test (HW) and the structure and genetic differentiation among populations were calculated using F-Wright parameters which were assessed using the GENEPOP software. The allele and genotype frequencies were evaluated with the method described by Hartl. Results. The genotypic frequencies were 0.59, 0.37 y 0.04 to AA, AB y BB, respectively and allele frequencies were 0.78 and 0.22 of A and B. The population is in Hardy-Weinberg equilibrium. Heterozygosity was average among populations (Ho = 0.368). FIS, FST and FIT values of the total population were -0.0717, 0.0099 and -0.0611 but these were not significant. Conclusions. It was not possible to assume inbreeding or out breeding in the analyzed municipalities, except in the municipality of San Pedro de los Milagros, in which case the effect of genetic improvement and the reduction of heterozygosity was more strongly perceived.
Key words: Characterization, genetic analysis, polymorphism (Fuente: CAB).
INTRODUCCIÓN
La lactoferrina (LTF) bovina estimula o inhibe diversos componentes humorales y celulares de la inmunidad implicados en la prevención o resolución de infecciones y de la inflamación asociada a estas últimas.
La respuesta inmunitaria contra los agentes infecciosos involucra una interacción compleja entre diferentes tipos de células y sus productos, que culmina con la eliminación del agente infeccioso o la muerte del animal (1). La LTF se encuentra en diversas secreciones mucosas como la leche, las lágrimas y la saliva (2). También es un componente abundante de los neutrófilos y puede ser liberada al plasma sanguíneo por la acción de éstos (3). Las LTF humana y bovina, despliegan un amplio espectro antimicrobiano, actuando contra bacterias Gram positivas y Gram negativas y contra algunos virus y hongos (4). Inicialmente se pensó que esta actividad antimicrobiana se debía únicamente a su capacidad de secuestrar hierro (5).
El efecto bacteriostático de la LTF en su forma de apo LTF se ha atribuido a su habilidad de captar el ion Fe3+ y limitar así su utilización por bacterias patógenas, que lo requieren como factor esencial para su crecimiento y expresión de factores de virulencia (6); se ha demostrado que el efecto bacteriostático in vivo de la LTF no depende exclusivamente de sus propiedades quelantes del Fe3+, sino de su capacidad para estimular la absorción intestinal de carbohidratos y reducir su disponibilidad para el crecimiento bacteriano, ya que algunas bacterias patógenas no sólo son capaces de contrarrestar las propiedades quelantes de la LTF sino que además la aprovechan como fuente de hierro (7). Este último puede ser removido directamente de la LTF por receptores expresados en la superficie de ciertas bacterias patógenas Gram positivas y negativas (8). Se ha demostrado que, dependiendo de la dosis ensayada, la apo LTF inhibe el crecimiento de bacterias patógenas no invasoras como Escherichia coli enterotoxigénica (ETEC) y Helicobacter pylori (9); La LTF ejerce un efecto bactericida directo por su capacidad de unirse a porinas y al lípido A del lipopolisacarido (LPS), presentes en la membrana externa de bacterias gram negativas (7).
El gen de la lactoferrina bovina (bLTF) está localizado en el cromosoma 22 del genoma bovino (referencia NCBI: NC_007320.4), se extiende sobre 33.4 kb, contiene 17 exones y 16 intrones. Por la naturaleza inmunogenica de la proteína bLTF, el gen presenta una cantidad importante de polimorfismos entre los cuales se encuentran polimorfismos en el exón 4 y en el exón 11 que causan cambios importantes en la cadena de aminoácidos, en el exón 4, en la base 1155 A/G causando un cambio de isoleucina a valina, y el exón 11 en la base 1415 C/T causando un cambio de histidina a tirosina; se encontró que un polimorfismo en el intrón 6 (C/T), está ligado a los cambios en los exones 4 y 11, separados por 14.5 kb. Cuando el intrón 6 se encuentra en su forma no mutada (citocina), el exón 4 contiene una adenina y el exón 11 una citocina, cuando el intrón 6 se encuentra con su forma mutada (timina) el exón 4 se encuentra con una guanina y el exón 11 con una timina (10).
Las lecherías del departamento de Antioquia, aunque en su mayoría comparten características similares encuentran diferencias importantes en cuanto a tecnificación, la cual no ha tenido el mismo progreso en los diferentes municipios ya sea por cuestiones de topografía o accesibilidad al hato, lo que puede sugerir cambios génicos en las poblaciones de ganado Holstein. Conocer la estructura genética de las poblaciones locales de Holstein es de gran importancia, ya que una población para un locus determinado, en este caso el lugar del polimorfismo del intrón 6, podría producir cambios y por ende producir efectos genéticos importantes en los parámetros de la población, es decir, cambios en la frecuencia génica, éstas resultado usual del equilibrio entre selección, mutación, migración y azar (11). Lo que permitiría la interpretación de su evolución en la raza y la base para dilucidar sus efectos en las frecuencias génicas de las poblaciones de ganado Holstein.
MATERIALES Y MÉTODOS
Área y poblaciones de estudio. Se incluyeron 427 vacas de la raza Holstein, pertenecientes a nueve lecherías especializadas ubicadas en el departamento de Antioquia, el número de animales por disponibilidad en los municipios de San Pedro de los Milagros (Altura: 2.475 m.s.n.m; T°: 14°C), Belmira (Altura: 2.550 m.s.n.m; T°: 14°C), Entrerríos (Altura: 2.300 m.s.n.m; T°: 16°C), La Unión (Altura: 2.500 m.s.n.m; T°: 13°C) y Medellín, corregimiento Santa Elena (altura: 2.500m.s.n.m; T°: 14°C). Los municipios fueron tomados como subpoblaciones de Antioquia (Tabla 1), cada uno de estos presentan rasgos particulares en productividad y tecnificación. Además, de diferencias en los sistemas productivos; el inventario por hato no fue igual en todos los municipios, lo que derivó en un número desbalanceado de animales.
Toma de muestras. De cada uno de los individuos se colectó 5 ml de sangre periférica en tubos de vacío con EDTA como anticoagulante, utilizando agujas número 18 (BD Vacutainer® TM). Los tubos fueron homogenizados por inversión, rotulados para su identificación y refrigerados con hielo durante su transporte hasta el laboratorio de Biología Celular y Molecular de la Universidad Nacional de Colombia sede Medellín, en donde se realizó la extracción del ADN.
Extracción del ADN de células de sangre. Para la obtención del ADN de sangre periférica se utilizó el método de “salting out” modificado, descrito por Miller (12) y se almacenó a 4°C hasta el momento del análisis. La cantidad y calidad del ADN extraído se evaluó en gel de agarosa al 0.8% teñido con bromuro de etidio, y se cuantificó en un espectrofotómetro NanoDrop ND 2000. La pureza del ADN genómico se determinó mediante un análisis de absorbancia en dos longitudes de onda. Sólo el ADN genómico con una pureza ideal entre 1.8-2.0 se consideró para los estudios a realizar.
Amplificación y genotipaje del gen bLTF. El ADN aislado fue usado para la amplificación por PCR (reacción en cadena de la polimerasa) de un fragmento de 301 pb (pares de bases) con los siguientes cebadores: Directo: 5'-GCC TCA TGA CAA CTC CCA CAC-3' Reverso: 5'-CAG GTT GAC ACA TCG GTT GAC-3', según Seyfert y Kuhn (10). La PCR contenía 0.5 U de Taq polimerasa (Bioline®), 2.5 µl de Buffer para PCR 10x, 30ng/µl de DNA, 0.5 µl de MgCl2 a 50nM, 0.5 µl de dNTPs a 20nM y 0.5µl de cada primer y se aforó con agua desionizada y estéril para un volumen total de 25µl.
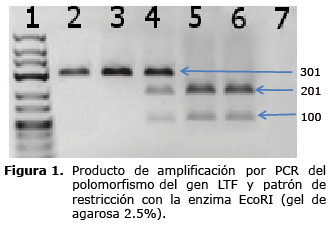
Se estandarizó la PCR de la siguiente manera: desnaturalización inicial a 95°C por 3 min, seguido de 28 ciclos a 95°C por 45s, 60°C por 30s, 72 por 20s, con una extensión final a 72°C por 3 min. El producto de la PCR se analizó por electroforesis en gel de agarosa al 2% teñido con bromuro de etidio. Luego se realizó la digestión con la enzima de restricción EcoRI de la siguiente manera: 5µl de reacción de PCR, 2µ de Buffer EcoRI (Fermentas®), 0.5µ EcoRI (Fermentas®) y 12.5 µl de agua desionizada y estéril, para un volumen total de 20 µl, se incubó por 3 horas a 37°C, el producto se observó por electroforesis en gel de agarosa al 2.5% y bromuro de etidio. El patrón de restricción esperado para el genotipo AA es un fragmento de 301pb, para el genotipo AB tres fragmentos (301pb, 201pb y 100pb) y para el genotipo BB dos fragmentos (201pb y 100pb).
Análisis Estadístico. Frecuencias alélicas y genotípicas. La frecuencia de los diferentes alelos se estimó determinando la proporción de cada forma del gen entre el número de copias totales de la población en estudio. Se identificaron los homocigotos (dos copias del mismo alelo) y los heterocigotos (una copia de cada alelo), y se calculó la frecuencia F de cada alelo contando los homocigotos y añadiendo la mitad de los heterocigotos, con el método descrito por Hartl (13).
Frecuencia total (p) del alelo 1 en la población es:
p= Fa/a +½ Fa/b
Frecuencia total (q) del alelo 2 en la población es:
q= Fb/b + ½Fa/b, donde;
F a/a = Homocigótico
F a/b = Heterocigótico
Diversidad genética. Se estimó comparando la proporción de los diferentes genotipos dentro de las poblaciones, mediante la Heterocigosidad observada (Ho) y Heterocigosidad esperada (He) en cada población, utilizando los algoritmos incluidos en el programa GENEPOP versión 4.0 (14). La significación estadística de estos análisis se obtuvo de acuerdo al método de cadena de Markov (15), utilizando el conjunto de programas GENEPOP versión 4.0 (14).
Estructura genética. Se determinó, la deficiencia o exceso de heterocigotos para el locus, en cada población y para la población total con la prueba exacta de Hardy-Weinberg (HW) con el programa GENEPOP versión 4.0 (14).
La estructura genética entre las poblaciones se calculó mediante los estadísticos F de Wright (11). Estos parten el coeficiente de endogamia de una población subdividida, calculando el parámetro FIT, que corresponde a la endogamia total, el parámetro FIS, que mide la subdivisión intrapoblacional y el FST, que mide la subdivisión poblacional; usando las siguientes formulas: para el parámetro FTI= HT-Hi/HT, para el parámetro FIS= HS-HI/HS y para el FST=FIT-FIS/1-FIS; donde HT se refiere a la heterocigosidad esperada en la población total, HI es la heterocigosidad promedio observada en un grupo de poblaciones y HS es la heterocigosidad promedio esperada de cada población. Estos cálculos se realizaron usando el conjunto de programas GENEPOP versión 4.0 (14), bajo la hipótesis nula “los alelos o genotipos tienen la misma distribución en todas las poblaciones”.
El flujo génico fue expresado como el número de migrantes (Nm) por generación, y se calculó con base a la siguiente expresión derivada de los estadísticos de Wright (11).
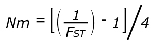
RESULTADOS
Determinación de las frecuencias alélicas y genotípicas del gen LTF. Se obtuvo amplificación del ADN de las 427 vacas de un fragmento de 301pb; el análisis de los fragmentos de restricción con la técnica RFLP usando la enzima EcoRI contenía fragmentos de 301pb (alelo A, sin sitio de restricción), 201 pb y 100 pb (alelo B) (Figura 1).
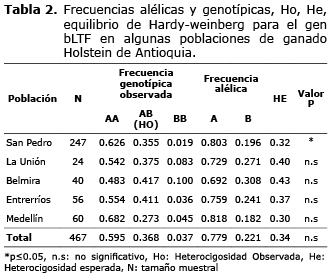
Las frecuencias de los alelos A y B fueron 0.78 y 0.22. Estos alelos forman tres genotipos AA, AB y BB, con frecuencias 0.59, 0.37 y 0.04 (Tabla 2). Es posible observar una presencia mayor de homocigotos BB en la subpoblación de Belmira y La Unión, comparada con las demás poblaciones y el promedio.
Diversidad genética. Solo la población de San Pedro de los Milagros, mostró desvíos significativos (p ≤ 0.05) para el análisis de equilibrio de HW, esta desviación se debió a un exceso de heterocigotos, lo que parece indicar una tendencia de apareamiento especifico en esta población. En ninguno de los otros municipios evaluados se encontraron evidencias suficientes para suponer desvíos significativos (p>0.05) (Tabla 2).
La Heterocigocidad observada y esperada tuvo una variación de 0.27 a 0.42 y de 0.30 a 0.43 respectivamente, indicando mediana variación entre las poblaciones considerable pero no significativa para toda la población (p>0.05).
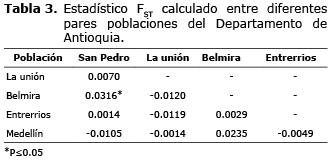
Estructura genética de las poblaciones. Los FST pareados por municipios, estuvieron entre -0.0049 y 0.0316, con diferencias significativas (p<0.05) para la población de San Pedro de los Milagros y Belmira (Tabla 3), encontrándose todos estos según los rangos propuestos por Wright (11), que define los valores de diferenciación genética (FST) en bajos (FST<0.05), moderados (FST de 0.05-0.15), altos (FST 0.15-0.25) y muy altos (FST>0.25), entre la categoría de los bajos. El estadístico FST para la población total presentó un valor de 0.0099 aunque este careció de significancia (p>0.05), por lo tanto, no es posible asumir tendencias a la endogamia, ni a la exogamia en los municipios analizados.
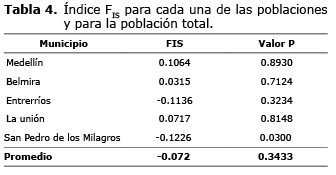
Los valores de FIS por municipio carecieron de significancia (p>0.05), excepto para el municipio de San Pedro de los Milagros (-0.12) indicando leve exceso de heterocigotos, lo cual parece indicar una tendencia a la exogamia en esta población. Dicho planteamiento es reforzado por el resultado planteado en la tabla 2, donde se observa una diferencia alta entre Ho y He, llevando a la perdida de equilibrio de HW.
El estadístico FIS obtenido fue de -0.0717 y no fue significativo (p>0.05), por tanto, no es posible afirmar que exista una tendencia a la endogamia (Tabla 4).
El estadístico FIT obtenido fue de -0.0611 resultando no significativo (p>0.05), por tanto, no es posible afirmar que exista una tendencia a la endogamia (Tabla 4).
Flujo génico. En este estudio el valor medio de flujo de genes Nm fue de 25, un valor muy alto considerando que la tasa efectiva de migración de más de un inmigrante por generación, es suficiente para evitar una diferenciación por efectos de deriva génica (11). La baja diferenciación genética (FST) y la alta tasa efectiva de migración (Nm) observada en estas poblaciones está ligada al apareamiento aleatorio entre individuos.
DISCUSIÓN
Todas las poblaciones estudiadas se encontraron en equilibrio de HW excepto San Pedro de los Milagros, para que se mantenga tal equilibrio se tienen que dar cinco condiciones, que exista una población grande para la reproducción, entrecruzamiento al azar, que no se dé ningún cambio alélico debido a mutaciones, que no se de migración y por último que no exista selección. Es posible que dado que la población de San Pedro de los Milagros es una de las más importantes para el sector lechero del departamento, se hayan concentrado allí muchos esfuerzos desde el punto de vista del mejoramiento genético aumentando los procesos de selección, para conseguir animales menos vulnerables a enfermedades infeccionas, ocasionando que en la actualidad el equilibrio para este gen se haya perdido, favoreciendo más alguna de las formas genotípicas.
Estos resultados, en general difieren de los encontrados por Wojdak-maksymiec et al (16), en una población de 124 vacas de la raza Holstein en Polonia, donde se encontraron frecuencias de 0.68, y 0.33 para los alelos A y B respectivamente del gen LTF, y para los genotipos AA, AB, y BB, frecuencias de 0.38, 0.60 y 0.20 respectivamente; para el cálculo de equilibrio de HW encontraron desviaciones significativas (p≤0.000252) con un exceso de heterocigóticos.
Igualmente en un trabajo realizado por Šrubarová y Dvorák (17) en una población de 94 vacas Holstein en República Checa, encontraron una frecuencia de 0.57 para el genotipo AA y 0.43 para el genotipo AB y no se encontró el genotipo BB, las frecuencias para el alelo A fueron de 0.79 y para el alelo B de 0.21, frecuencias muy similares a las encontradas en el presente estudio.
Rosero (18) encontró una población de 30 animales Holstein ubicados en el Valle del Cauca (Colombia) en equilibrio de HW, para los genes de Kappa caseína y β-Lactoalbúmina. Al igual que Usme-Ciro et al (19) en una población de 145 hembras Holstein de Antioquia (Colombia) para el gen de Kappa Caseína. No se encontraron reportes de equilibrio de HW para el gen LFT en Colombia.
Las diferencias en las frecuencias encontradas por otros autores denotan como en las poblaciones de Holstein se han trabajado apareamientos dirigidos (selección artificial), ya que el genotipo AA del gen LTF se ha asociado con un menor conteo de células somáticas, mientras que el genotipo AB se asocia con un mayor conteo (16); lo que podría explicar las frecuencias genotípicas de la población Checa.
La utilidad de una población en condiciones exogámicas, tal como es la tendencia de la población del departamento de Antioquia para el gen bLTF, reside aumentar genes portadores de los caracteres favorables sobre sus alelos, que aportan las expresiones menos deseables del mismo carácter. Por lo tanto, la exogamia mejora el comportamiento de los descendientes, pero disminuye la potencialidad genética de los individuos sobresalientes en los cuales inicialmente los genes favorables se presentan en forma de homocigotos. (20).
La diversidad genética medida en términos de Heterogocicidad observada (Ho) y Heterogocicidad esperada (He) indican entre media y baja variabilidad genética (Tabla 2). Resultados que no se alejan mucho de la variabilidad genética de otros genes tales como el gen Kappa-caseína, en un estudio realizado en el departamento de Nariño, con Ho de 0.30 y He de 0.32 (21); de igual manera Rosero (18) encontró para los genes de Kappa caseína, α-Lactoalbúmina, y β-Lactoalbúmina en ganado Holstein colombiano, un Ho de 0.567 y He de 0.457. En Colombia no se encontraron referencias para el gen LFT.
Como se observó en la tabla 3, los valores de FIS para las poblaciones de los municipios de la Unión, Belmira y Medellín indican una leve deficiencia de heterocigotos. El valor medio de FIS = -0.0717 muestra que la población estudiada tiende a la heterocigosis, condición que puede estar asociada a un apareamiento aleatorio dentro de poblaciones, lo que evidencia que no se han presentado en esta población cruzamientos dirigidos para el gen de LTF. Este índice es una medida indirecta de consanguinidad y concuerda con los resultados obtenidos mediante la prueba de HW (Tabla 2), donde se encuentra esta población en equilibrio. Pero algunos valores positivos en las poblaciones de Medellín (0.11), La Unión (0.07) y Belmira (0.03), indican la tendencia a la disminución de su heterocigocidad, a partir del coeficiente de endogamia entre los componentes de una población debido a apareamientos no aleatorios dentro de poblaciones.
El conocimiento poblacional del polimorfismo del gen LTF es de vital importancia para futuros trabajos en la asociación de este con enfermedades infecciosas como mastitis, problemas pódales y respiratorios, que aquejan y golpean el estado sanitario y la economía ganadera.
En conclusión, hasta este momento y con base en la población analizada se puede concluir que el gen de la lactoferrina bovina LTF no ha sido objeto de selección genética ni de otras fuerzas génicas que sugieran cambios en la estructura de las diferentes poblaciones, pese a que este gen se ha encontrado asociado a diferentes características, en las poblaciones estudiadas la selección indirecta que se ha llevado a cabo, no ha provocado cambios que impliquen desviaciones del equilibrio de HW.
Con respecto a la estructura de las diferentes poblaciones no se encontraron resultados significativos por lo tanto, no es posible asumir endogamia, ni exogamia en los municipios analizados para este gen. Exceptuando el Municipio de San Pedro de los Milagros, en cuyo caso se percibe de manera más fuerte el efecto del mejoramiento genético y la disminución de la heterocigocidad, para reforzar esta teoría se sugiere la realización de estudios con un tamaño muestral mayor al del presente trabajo.
Estos resultados son el primer reporte que caracteriza el gen lactoferrina bovino en ganado Holstein tropical colombiano. Los mismos, servirán de base para futuros estudios con este y otros marcadores moleculares, a fin de contribuir con la conservación y permanencia de alelos de importancia, su asociación con enfermedades de importancia económica y como posterior marcador de susceptibilidad o resistencia a las mismas; lo cual es de gran utilidad como criterio de selección.
Algunas poblaciones tienen una tendencia a disminuir la heterocigocidad, lo que incrementa el riesgo de erosión genética asociado a altos niveles de endogamia que se pueden presentar si los apareamientos consanguíneos persisten, por tal motivo en la labor pedagógica y de extensión se debe enfatizar que la evaluación del impacto de los procesos de mejoramiento genético a través de la selección, se logran en el mediano y largo plazo, debido al prolongado intervalo generacional de los bovinos.
REFERENCIAS
1. Hugot JP, Chamaillard M, Zouali H, Lesage S, Cezard JP, Belaiche J. Association of NOD2 leucine-rich repeat variants with susceptibility to Crohn's disease. Nature (London) 2001; 411: 599-603. [ Links ]
2. Wakabayashi H, Kurokawa M, Shin K, Teraguchi S, Tamura Y, Shiraki K. Oral lactoferrin prevents body weightloss and increases cytokine responses during herpes simplexvirus type 1 infection of mice. Biosci Biotechnol Biochem 2004; 68: 537-544. [ Links ]
3. Waleska DS, Carnelocce JL, Andre CO, Rafael BG. Conformational changes in bovine lactoferrin induced by slow or fast temperature. Increases Biol Chem 2008; 389: 1137-1142. [ Links ]
4. Shukla D, Spear PG. Herpesviruses and heparan sulfate:an intimate relationship in aid of viral entry. J Clin Invest 2001; 108:503-510. [ Links ]
5. Rong-Hua Y, Schryvers B. Bacterial lactoferrin receptors: insights from characterizing the Moraxella bovis receptors. Biochem Cell Biol 2002; 80: 81-90. [ Links ]
6. Orsi N. The antimicrobial activity of lactoferrin: current status and perspectives. Biometals 2004; 17: 189-196. [ Links ]
7. Van der Strate BW, Beljaars L, Molema G, Harmsen MC, Meijer DK. Antiviral activities of lactoferrin. Antiviral Res 2001; 52:225-239. [ Links ]
8. Drago S. La lactoferrina como modulador de la respuesta inmunitaria. Biochimie 2008; 33: 71-82. [ Links ]
9. Kutila T, Pyörälä S, Saloniemi H, Kaartinen L, Antibacterial Effect of Bovine Lactoferrin Against Udder Pathogens. Acta vet scand 2003; 44: 35-42. [ Links ]
10. Seyfert HM, Kuhn C. Characterization of a first bovine lactoferrin gene variant, based on an EcoRI polymorphism. Anim Genet 1994; 25: 54. [ Links ]
11. Wright S. Evolution and Genetics of Populations, The theory of gene frequencies. Vol. II. Chicago: University of Chicago Press; 1969. [ Links ]
12. Miller SA, Dykes DD, Polesky HF. A simple salting-out procedure for extracting DNA from human nucleated cells. Nucleic Acid Res 1988; 16:1215. [ Links ]
13. Hartl DL, and Clark AG. Principles of population genetics. 4th edition Sunderland (MA): Sinauer Associates Inc Publishers 2007. 5-138. [ Links ]
14. Raymond M, Rousset F. genepop'007: a complete re-implementation of the genepop software for Windows and Linux. Mol Ecol Resour 2008; 8:103-106. [ Links ]
15. Guo SW, Thompson EA. Performing the exact test of Hardy-Weinberg proportions for multiple alleles. Biometrics 1992; 48: 361-372. [ Links ]
16. Wojdak-maksymiec K, Kmiec M, Ziemak J. Associations between bovine lactoferrin genepolymorphism and somatic cell count in milk. Vet Med 2006; 51: 14-20. [ Links ]
17. Šrubarová P, Dvorák J. Association between Lactoferrin gene polymorphism and bovine mammary gland inflammation. MendelNet'09 Agro Page. (En linea). 2009. (fecha de acceso 25 de febrero de 2010). URL disponible en: http://web2.mendelu.cz/af_291_mendelnet/mendelnet09agro/files/articles/bz_srubarova.pdf.
18. Rosero AJ. Polimorfismo de los genes K-Caseina, ß-Lactoglobulina y A Lactoalbumina en razas bovinas criollas colombianas. [Tesis Magister en Ciencias Agrarias]. Colombia. Palmira: Universidad Universidad Nacional de Colombia, Facultad de Ciencias Agropecuarias; 2009.
19. Usme Ciro J, Trujillo Bravo ER. Kappa-caseína bovina y su asociación con el recuento de células somáticas en ganado Holstein Colombia. Actualidades Biológicas 2004; 26: 17-22. [ Links ]
20. Valle A. Cruzamiento de bovinos lecheros. FONAIAP divulga 2007; 57: 40-44. [ Links ]
21. Solarte-Portilla C E, Rosero C, Eraso JM, Zambrano G L, Cárdenas H, Burgos WO. Frecuencias alélicas del gen Kappa caseína en la raza Holstein del trópico alto de Nariño - Colombia. LRRD, 2009; 21:1-7. [ Links ]