Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista MVZ Córdoba
versão impressa ISSN 0122-0268versão On-line ISSN 1909-0544
Rev.MVZ Cordoba vol.24 no.1 Córdoba ene/abr. 2019
https://doi.org/10.21897/rmvz.1345
Artículo institucional
Polimorfismos de los genes calpaína y calpastatina en dos poblaciones de ovinos de pelo colombiano
3 Universidad de Sucre, Facultad de Ciencias Agropecuarias, Grupo de Investigación en Reproducción y Mejoramiento Genético Animal, Sincelejo, Colombia.
4 Universidad Nacional de Colombia, Facultad de Ciencias Agropecuarias, Grupo de Investigación en Recursos Zoogenéticos, Palmira, Colombia.
Objetivo.
El propósito del presente estudio fue caracterizar el polimorfismo genético tipo SNPs en los genes calpaína (CAPN) y calpastatina (CAST) en el ovino de pelo criollo colombiano (OPC).
Materiales y métodos.
300 individuos pertenecientes a dos subpoblaciones de OPC de los departamentos de Sucre (SC) y Valle del Cauca (VC) fueron genotipados por PCR-RFLP (MspI) para el locus CAST y por PCR-SSCP para el locus CAPN. Se calcularon las frecuencias genotípicas, alélicas, la heterocigocidad observada (Ho) y esperada (He), el índice de fijación (F), los desvíos del equilibrio de Hardy-Weinberg (EHW) y un análisis de varianza molecular para estimar los valores de FST, FIS y FIT.
Resultados.
En el locus CAST, el genotipo MM fue el más frecuente (83.9±1.1%), seguido por los otros genotipos (MN: 15.5±1.1; NN:6.0±0.0%) y la frecuencia alélica de M (91.7±0.4%) superó la del N (8.3±0.4%). Para el locus CAPN el genotipo heterocigoto (48.2±0.7%) fue el más frecuente; los otros genotipos presentaron frecuencias de TT:44.7±1.9 y CC:7.0±1.4%. El alelo T alcanzó una frecuencia de 68.8±1.5% (C:31.3±1.5%). Similares frecuencias alélicas y genotípicas se encontraron en las subpoblaciones. La He fue menor que la Ho en ambos loci, con valores negativos de F y desvíos de EHW solo en CAPN. Toda la variación encontrada fue debida a diferencias dentro de los individuos, con valores no significativos (p>0.05) de FST, FIS y FIT (0.002, -0.093 y -0.095, respectivamente).
Conclusiones.
Los loci estudiados tiene alta variabilidad, estos resultados pueden ser utilizados para futuros planes de selección asistida por genes para aumentar la productividad del OPC.
Palabras clave: Frecuencias alélicas; gen CAST; gen CAPN; recurso genético (Fuente: CAB)
Objetive.
Characterize the genetic polymorphism type SNPs in the calpain (CAPN) and calpastatin (CAST) genes of Colombian creole hair sheep (OPC).
Materials and methods.
In 300 individuals belonging to two OPC subpopulations from the departments of Sucre (SC) and Valle del Cauca (VC) were genotyped by PCR-RFLP (MspI) for the CAST locus and by PCR-SSCP for the CAPN locus. The allelic and genotypic frequencies, the observed (Ho) and expected heterozygosity (He), the fixation index (F) and the deviations from the Hardy-Weinberg equilibrium (HWE) were calculated and a molecular analysis of variance to estimate the values of FST, FIS and FIT.
Results.
In the CAST locus, the MM genotype was the most frequent (83.9±1.1%), followed by the other genotypes (MN: 15.5±1.1, NN: 6.0±0.0%) and the allelic frequency of M (91.7±0.4%) exceeded that of N (8.3±0.4%). For the CAPN locus the heterozygous genotype (48.2±0.7%) was the most frequent, the other genotypes presented frequencies TT:44.7 ± 1.9 and CC:7.0 ± 1.4%. The T allele reached a frequency of 68.8±1.5% (C: 31.3±1.5%). Similar percentages of allelic and genotypic frequencies were found in the subpopulations. The He was less than the Ho in both loci, with negative values of F and deviations of EHW only in CAPN. All the variation found was due to differences within the individuals, with non-significant values (p>0.05) of FST, FIS, and FIT (0.002, -0.093 and -0.095, respectively).
Conclusions.
The loci studied has high variability, these results can be used for future gene-assisted selection plans to increase OPC productivity.
Keywords: Allele frequency; CAST gene; CAPN gene; genetic resources; (Source: CAB)
INTRODUCCIÓN
La mayoría de los sistemas de producción ovina en Colombia, tienen como base genética la raza ovina criolla de pelo colombiana (OPC). El OPC presenta características adaptativas importantes al clima tropical, tales como la tolerancia al calor, ectoparásitos y la capacidad de consumir pasturas de escaso valor nutricional [1]. Sin embargo, se ha considerado a estos sistemas como una actividad secundaria de la ganadería que se realiza en sistemas tradicionales y/o familiares, con bajos requerimientos de insumos y en sistemas mixtos de producción con otras especies [2,3], lo que conlleva a que problemas en nutrición, reproducción, sanidad, progreso genético, bienestar animal y manejo empresarial; entre otros, sean comunes y se reflejen en una baja eficiencia productiva [1,4].
Los programas de mejoramiento genético en el OPC son escasos, se basan, en cruzamientos absorbentes con razas importadas para aprovechar el vigor híbrido [5]. Sin embargo, para aumentar la competitividad del sector, se requiere de la incorporación de nuevas tecnologías o prácticas de manejo que generen ventajas competitivas mediante sistemas de gestión de conocimiento que se adapten a las condiciones de la producción primaria de un país en desarrollo como Colombia [2, ,6]. Es así como las herramientas moleculares son utilizadas en los programas de mejora genética de animales a través de la selección asistida por marcadores moleculares. Esta práctica mejora la exactitud y aumenta el progreso genético a través de la identificación, mapeo y análisis del polimorfismo de los genes implicados en las principales vías metabólicas relacionadas con el crecimiento animal, la distribución de los nutrientes en los diferentes tejidos y calidad de productos [7]. La calpaína (CAPN) y la calpastatina (CAST) pueden ser considerados como genes candidatos con potencial de ser usados en programas de selección, pues polimorfismos de nucleótido simple (SNPs) se han asociado con mejora en la calidad de la carne [8].
El gen CAPN está asociado con la degradación de las proteínas miofibrilares post-morten, ocasionando el ablandamiento de la carne; se encuentra ubicado en el cromosoma siete y tiene 25 exones. El SPN g.24962426T>C ubicado en el exón diez ha sido el más estudiado [9]. Por otra parte, el gen CAST es el principal inhibidor de la acción de la calpaína ralentizando la velocidad y el grado de ablandamiento post-morten de la carne [8]. Está ubicado en el cromosoma 5 de la oveja, el sistema de las calpaínas está conformado por los genes de u-calpaína (CAPN1), m-calpaína (CAPN2) y calpaína 3 (CAPN3), en los cuales se han identificado numerosos SNPs correlacionados con la terneza de la carne en diferentes especies [9,10]. Además, las calpainas están involucradas en el crecimiento muscular en diferentes etapas de desarrollo y en la degradación de las proteínas musculares, lo que influye en la pérdida de masa muscular [7,8,9].
Los reportes de estudios de polimorfismos en los genes antes mencionados en ovinos de Colombia son limitados. Por lo tanto, el objetivo de este trabajo fue caracterizar el polimorfismo genético tipo SNPs en los genes calpaína y calpastatina en el ovino de pelo criollo colombiano.
MATERIALES Y MÉTODOS
Poblaciones, colecta de sangre y extracción de ADN. En la presente investigación se utilizaron 300 individuos pertenecientes a dos subpoblaciones de OPC ubicadas en los departamentos de Valle del Cauca (VC, n = 150) y Sucre (SC, n = 150). Las muestras de sangre se colectaron en tubos con anticoagulante (EDTA 7.2 mg) teniendo en cuenta para los procedimientos de recolección de muestras, manejo y conservación, las normas éticas, técnicas, científicas y administrativas para la investigación en animales contenidas en la Ley 84 (Congreso Nacional de Colombia, 1989). El ADN se extrajo usando el estuche comercial QIAamp® DNA Mini Kit de QIAGEN. Se evaluó la cantidad y calidad del ADN usando el NanoDrop 2000TM (Thermo Fisher Scientific).
Amplificación y genotipificación de los loci CAPN y CAST. Todas las reacciones de PCR fueron llevadas a cabo en un volumen final de 25 μl que contenían 20 ng de ADN, 250 nM de cada cebador y 1X del súper mix MangoMixTM (Bioline©). Los cebadores utilizados para el locus CAPN (CAPNS1, accesión: XM_027514355.1) fueron 5 '-AACATTCTCAACAAAGTGGTG-3 ' y 5'-ACATCCATTACAGCCACCAT-3' [11], para el locus CAST (CAST-A, accesión: KX_722534.1) 5'-TGGGGCCCAATGACGCCATCGATG-3' y 5'-GGTGGAGCAGCACTTCTGATCACC-3' [12]. Los perfiles térmicos constaron de una desnaturalización inicial a 95°C durante 5 minutos. Seguido por 35 ciclos de 95°C por 30s, 60°C por 60s y 72°C por 60s para CAPN y de 95°C por 60s, 58°C por 60s, 72°C por 60s para CAST; seguido por una extensión final de 10 minutos a 72°C. Las amplificaciones fueron realizadas en un termociclador MasterCycler Nexus Gradient de Eppendorf®. La visualización de los fragmentos amplificados se realizó por electroforesis en geles de agarosa al 1.2% a 80 V durante 45 minutos teñidos con GelRedTM (Biotium).
Los genotipos para el SNPs estudiado en el locus CAST se obtuvieron por PCR-RFLP con la endonucleasa de restricción MspI, en un volumen final de 15 μl que contenían en 5 μl del producto de PCR, 1 U de la enzima MspI y 1 X de tampón. La incubación se realizó a 37°C durante dos horas, seguido de 80°C durante 20 minutos. Los productos de digestión se visualizaron en geles de poliacrilamida al 9% (Acrilamida:Bis-acrilamida 37:1) a 150 V por 40 minutos y teñidos con GelRedTM (Biotium). Los genotipos fueron identificados según lo reportado por Azari et al [11] y Santos et al [12].
Para el gen CAPN los genotipos se obtuvieron por PCR-SSCP, en geles desnaturantes de poliacrilamida al 10% (100:1, Acrilamida:Bis-acrilamida) y 3.7% de glicerol. Cada muestra fue desnaturalizada con formamida (95% formamida, 0.05% de Xilene-Cianol, 0.05% de azul bromofenol, 20 mM de EDTA pH 8.0) y sometidos a 95°C por 5 minutos y luego enfriadas a 4°C. Los geles fueron corridos a 190 voltios durante 5.5 horas, en una cámara Multigel Biometra. La tinción se realizó con GelRedTM (Biotium). Los genotipos fueron identificados según lo reportado por Azari et al [11].
Análisis de datos. Se calcularon las frecuencias genotípicas, alélicas, la heterocigocidad observada (Ho) y esperada (He) y el índice de fijación (F) para cada subpoblación y en total. Se estimaron los desvíos del equilibrio de Hardy-Weinberg (EHW) y se realizó un análisis de varianza molecular para estimar los valores de FST, FIS y FIT. Se compararon las frecuencias genotípicas y alélicas encontradas entre locus y subpoblación utilizando el test de Fisher con una significancia del 5%. Todos los análisis fueron realizados con los programas Arlequín ver 3.5.2.2 (13) y GENALEX ver 6.5 [14].
RESULTADOS
Los dos SNPs evaluados fueron polimórficos. Las frecuencias alélicas y genotípicas no difirieron significativamente (p>0.05) entre subpoblaciones para ambos loci.
En el locus CAST, para toda la población en estudio, el genotipo MM fue el más frecuente, seguido por los otros dos genotipos (Tabla 1). El genotipo MM fue más frecuente en la subpoblación SC que en la subpoblación VC, aunque ocurrió lo contrario en el genotipo MN. Mientras que, el genotipo NN presentó igual frecuencia en ambas subpoblaciones (6%). Respecto a las frecuencias alélicas, el alelo M superó en frecuencia al alelo N (Tabla 1). El alelo M fue más frecuente en la subpoblación SC y el alelo N el más frecuente en la subpoblación VC.
Para el locus CAPN se encontró mayor frecuencia del genotipo heterocigoto, en toda la población (48.2±0.7%) como en las subpoblaciones SC (48.7%) y VC (47.6%). El genotipo CC presentó la menor frecuencia (Tabla 1). El alelo T del locus CAPN alcanzó una frecuencia de 68.8 ± 1.5% para toda la población, siendo más alta en la subpoblación VC, de otro lado, el alelo C presentó mayor frecuencia en la subpoblación SC (Tabla 1).
Tabla 1 Frecuencias genotípicas y alélicas en los loci CAST y CAPN en la raza OPC.
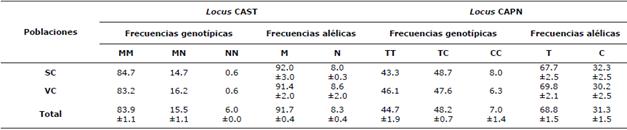
SC: Sucre. VC: Valle del Cauca.
En la tabla 2 se pueden apreciar los valores de diversidad genética en la población de OPC estudiada. En el locus CAST, los valores de Ho y He promedios fueron similares con un ligero exceso de heterocigotos no significativo; de otro lado, en el locus CAPN este exceso indujo a un valor negativo de F y desvíos significativos del EHW (p<0.05). Los valores de Ho y He para el locus CAST fueron más altos en la subpoblación VC y en el locus CAPN en la subpoblación SC. Solo el valor de F fue positivo en la subpoblación SC para el locus CAST (0.004) los demás valores fueron negativos. El análisis locus a locus indicó que todos se encontraban en EHW.
Tabla 2 Ho, He, F valores y desviaciones del HWE en los loci estudiados en la raza OPC.
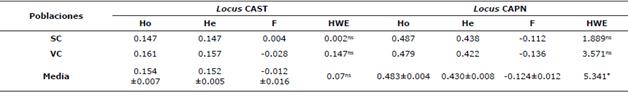
SC: Sucre. VC: Valle del Cauca. Ho: Heterocigosidad observada. He: Heterocigosidad esperada. F: índice de fijación. HWE: Equilibrio de Hardy-Weinberg (valor X2). *p<0.05.
El análisis de varianza molecular mostró que el 100% de la variación es debida a diferencias dentro de los individuos, con valores no significativos (p>0.05) de FST, FIS y FIT de 0.002, -0.093 y -0.095, respectivamente.
DISCUSIÓN
En el locus CAST (AF016006, AF016007 y AF016008.1) los fragmentos digeridos por la enzima MspI, se encontraron entre los exones 1C y 1D, donde ocurre una sustitución de A por G [15]. En ovejas Dalagh se encontraron los genotipos MM (36%), MN (38%) y NN (26%) con mayor frecuencia del alelo M (55%) [16], en ambas subpoblaciones estudiadas y para el OPC el genotipo MM y el alelo M presentaron mayor frecuencia (Tabla 1).
Lo anterior concuerda con las frecuencias encontradas por Santos et al [12] en tres de cuatro razas estudiadas en Brazil; entre ellas la Pantaneira (MM: 58%, MN: 23%, NN: 19%; M: 70%), la Sufflok (MM: 80%, MN: 16%, NN: 4%; M: 88%) y la Ile de France (MM: 82%, MN: 12%, NN: 6%; M: 88%) pero no con lo encontrado en la raza Bergamacia (MM: 28%, MN: 34%, NN: 38%; M: 45%). En ovinos Iraníes de raza Afshari se reportan los alelos M y N con frecuencias de 74% y 26%, respectivamente [17] y en las frecuencias genotípicas encontradas, el homocigoto NN presentó mayor frecuencia (10%) que la aquí mostrada; los otros genotipos tuvieron frecuencias de 57 y 33% para MM y NN, respectivamente.
Por otro lado, en la raza Zel las frecuencias alélicas fueron de 85.5% para M y de 14.5% para N [16], con frecuencia para los genotipos MM (75%) y NN (4%) menor a la encontrada en el OPC. En ovejas autóctonas de Pakistan Balkhi, Kajli y Beetal las frecuencias del alelo M fueron de 88, 86 y 100%, respectivamente; con el genotipo MM más frecuente (Balkhi: 76%, Kajli: 74%, Beetal: 100%) y ausencia del genotipo NN en las razas Balkhi y Beetal y con baja frecuencia (2%) en Kajli [18]. La frecuencia del alelo N en la raza Kurdi fue superior (12%) a la encontrada en los OPC con ausencia del genotipo NN y mayor frecuencia del MM (76%) [19]. Es de resaltar que el alelo M de este locus se ha asociado con altos pesos al nacimiento [15].
Para el locus CAST se ha comparado las técnicas PCR-RFLP y PCR-SSCP como alternativas de genotipaje. Se logró determinar que los alelos A y C obtenidos por SSCP podrían ser digeridos con la enzima MspI, también que los genotipos AA, AC y CC eran similares al genotipo MM determinado por RFLP, igualmente, el genotipo MN es similar con los genotipos AB y BC (por SSCP) y por último, el genotipo NN concuerda con el genotipo BB [11,16]. En otro locus del gen CAST (exones 10, 11 e intrón 10) en 80 animales de la raza OPC, las frecuencias alélicas encontradas fueron de 93.7% para A y de 6.3% para B, con frecuencias genotípicas de 88.75, 10 y 1.25% para AA, AB y BB respectivamente (20).
El valor de He en el locus CAST, fue menor al encontrado en las razas Dalagh [11] (49%) y Zel [16] (25%), la Pantaneira (42%), la Sufflok (21%) y la Ile de France (21%) y Bergamacia (50%) [12] Balkhi (21%) y Kajli (24%) [18] pero mayor al reportado en OPC (11%) [20] y el ovejas Kurdi (7.9%) [19]. De igual forma, en la presente investigación se encontró un exceso no significativo de heterocigotos, al igual que EHW. Lo anterior indica que no se ha realizado selección a favor de alguno de los alelos de este locus, o la ausencia de programas de mejoramiento genético asistido por herramientas moleculares. Por su parte, Molano [20] en el mismo grupo racial aquí estudiado, presentó un déficit de heterocigotos no significativo y ausencia de las proporciones teóricas del EHW. Resultados contrarios se presentan en ovejas Dalagh [11] y Zel [16] la Pantaneira, Ile de France y Bergamacia [12] donde los autores encontraron déficit de heterocigotos con desviaciones significativas de EHW (Tabla 2).
Para el locus CAPN (AF309634.1) se estudió una variación, ubicada entre los exones 5 y 6, que consiste en una transición sinónima de T/C en el nucleótido 44 del gen [11,16]. Variaciones en este locus ha sido asociado con características de crecimiento y calidad de canal en ovejas [21,22]. Contrario a lo aquí reportado, en tres razas de ovejas de Egipto (Barki, Rahmani y Ossimi) se presenta al genotipo CC y al alelo C como el más frecuente y con ausencia del genotipo TT en las razas Barki y Ossimi [22] (Tabla 1). En otro locus (CAPN316) del gen CAPN en la raza OPC, las frecuencias alélicas encontradas fueron A: 54.4% y B: 45.6%, con mayor frecuencia del genotipo heterocigoto (38.75%) que de los homocigotos (AA: 35% y BB: 26.25%) [20].
En ovejas Merino, se reportan tres alelos para este gen siendo el alelo A1 y el genotipo A1B1 los más frecuentes con valores de 46.7 y 41.9%, respectivamente [23]. Asimismo, en ovejas Baluchi se reportan tres patrones de bandas con frecuencias genotípicas de 8.2 (G1), 89 (G2) y 3% (G3) [11]. En ovinos Bandur reportan dos alelos con frecuencias 82 (A) y 18% (B), y dos genotipos AA (67%) y AB (33%) [24], de forma similar, en ovejas Zel solo se encontraron los genotipoas AA (69%) y AB (31%) con frecuencias alélicas de 84.5 y 15.5 para A y B, respectivamente [16] y en ovejas Kurdi [19] las frecuencias alélicas para A y B fueron de 96 y 4% con ausencia del genotipo BB y mayor frecuencia del genotipo AA.
Las investigaciones antes citadas, así como en el presente trabajo se utilizó la metodología de genotipaje PCR-SSCP para el locus CAPN, pese a la simplicidad de la técnica, en varios reportes, los patrones de movilidad de las bandas fueron diferentes. Algunas razones que podrían explicar por qué en las cadenas de ADN monocatenario puedan observarse más de dos bandas siendo la misma secuencia, se relacionan con la estabilidad molecular de la cadena que puede verse afectada por un exceso de cebadores, un manejo lento después de la desnaturalización que ocasione un emparejamiento o re-asociación biomolecular [25] y por las condiciones de electroforesis, donde, factores como la temperatura, tiempo de corrida, concentración del buffer, composición del gel, posición del SNP en el fragmento analizado al igual que el tamaño del fragmento, pueden influenciar la sensibilidad de la técnica [26].
La He encontrada en este estudio para el locus CAPN es superior a la reportada en las razas Bandur (29.5%) [24], Zel (26%) [16] y Kurdi (7%) [19], pero inferior a la informada por Molano [20] en OPC (49%). Por otro lado, en este locus se encontró un exceso no significativo de heterocigotos intra poblacional, pero al analizar toda la población en estudio (Tabla 2), este exceso fue significativo con desviaciones del EHW (p<0.05). Esto podría atribuirse a la falta de endogamia por la existencia de un flujo génico, así como el cruce rápido de hembras y machos migrantes con los individuos residentes en las subpoblaciones. Por el contrario, déficit de heterocigotos sin desvíos de EHW se presentan en OPC [20], y en la raza Zel este déficit ocacionó desvíos de EHW (p<0.05) [16]. Asimismo, en las razas Kurdi y Bandur el exceso de heterocigotos encontrado no causó desvíos significativos de EHW [19,24].
El análisis de varianza molecular mostró que la variación encontrada solo es debida a diferencias dentro de los individuos, lo que es confirmado por el bajo valor de FST encontrado (0.002, p=0.653), lo que indica una baja estructura poblacional con flujo de genes entre las subpoblaciones estudiadas [5], además, los valores de FIS y FIT negativos y no significativos (p>0.05) contrastan con lo reportado en OPC (FIS=0.183) [20], lo que podría explicarse por el origen diferente de los animales evaluados al igual que por diferencias en el manejo de los sistemas de producción respecto a sus sistemas o esquemas de cruzamiento.
Finalmente, los resultados del presente estudio mostraron que los loci estudiados tienen buena variabilidad que es necesario conservar. Adicionalmente, las frecuencias alélicas encontradas son considerablemente altas, por lo que, podrían usarse estos resultados en futuros planes de selección asistida por genes, en miras a aumentar la productividad de los sistemas ovinos que usan al OPC como base genética.
REFERENCIAS
1 Cuellar-Gamboa G, Jimenez-Robayo L, Grajels-Lombana H, Morales-Mendoza L, Leal-Gutierrez J, Sanchez-Isaza C. Factores que influencian la prolificidad en ovinos del centro agropecuario marengo, Colombia. Actas Iberoamericanas de Conservación Animal. 2015; 6(1):460-465. URL Available in: https://aicarevista.jimdo.com/app/download/11698017525/AICA2014_Trabajo011.pdf?t=1542558897. [ Links ]
2. Ospina Ó, Grajales H, C CM. Gestión del conocimiento: mayor producción y competitividad. Perspectivas para los sistemas de producción ovino-caprinos. Rev Med Vet. 2011; 22(2):95-113. DOI: https://doi.org/10.19052/mv.564. [ Links ]
3. Castellanos J, Rodriguez J, Toro W, Lenguas C. Agenda prospectiva de investigación y desarrollo tecnológico para la cadena productiva cárnica ovino - caprina en Colombia. Colombia: Ministerio de Agricultura y Desarrollo Rural; 2010. [Consultado: 2018 Julio 24]. Disponible en: https://sioc.minagricultura.gov.co/OvinoCaprina/Documentos/004%20-%20Documentos%20Competitividad%20Cadena/Agenda%20Prospectiva%20de%20Investigacion.pdf. [ Links ]
4. Simanca J, Vergara O, Bustamante M. Description of growth in sheep creole (Ovis aries) in two populations from Córdoba, Colombia. Rev MVZ Córdoba. 2017; 22(3):6310-6319. DOI: https://doi.org/10.21897/rmvz.1135. [ Links ]
5. Ocampo R, Martínez R, Rocha J, Cardona H. Genetic characterization of Colombian indigenous sheep. Rev Colomb Ciencias Pecu. 2017; 30(2):116- 125. DOI: https://doi.org/10.17533/udea.rccp.v30n2a03. [ Links ]
6. Pineda R, Montes D, Hernandez D. Association of the Polymorphisms FecXR, FecGH, and FecGI and Non-Genetic Factors that Affect the Prolificacy of Colombian Creole Sheep. Indian Journal of Science and Technology. 2018; 11(17). DOI: http://dx.doi.org/10.17485/ijst/2018/v11i17/122374. [ Links ]
7. Coria M, Carranza P, Palma G. Calpain System in meat tenderization: A molecular approach. Rev MVZ Córdoba. 2018; 23(1):6523-6536. DOI: 10.21897/rmvz.1247. DOI: https://doi.org/10.21897/rmvz.1247. [ Links ]
8. Georgieva S, Hristova D, Dimitrova I, Stancheva N, Bozhilova-Sakova M. Molecular analysis of ovine calpastatin (CAST) and myostatin (MSTN) genes in Synthetic Population Bulgarian Milk sheep using PCR-RFLP. J BioSci Biotechnol. 2015; 4(1):95-99. URL Available in: http://www.jbb.uni-plovdiv.bg/documents/27807/1014559/jbb_2015-4(1)-pages_95-99.pdf. [ Links ]
9. Bagatoli A, Gasparino E, Soares M, Amaral R, Macedo F, Voltolini D, et al. Expression of calpastatin and myostatin genes associated with lamb meat quality. Genet.Mol.Res. 2013; 12(4):6168-6175. DOI: http://dx.doi.org/10.4238/2013.December.4.3. [ Links ]
10. Knight MI, Daetwyler HD, Hayes BJ, Hayden MJ, Ball AJ, Pethick DW, et al. Discovery and trait association of single nucleotide polymorphisms from gene regions of influence on meat tenderness and long-chain omega-3 fatty acid content in Australian lamb. Animal Production Science. 2012; 52(7):591-600. DOI: https://doi.org/10.1071/AN11229. [ Links ]
11. Azari M, Dehnavi E, Yousefi S, Shahmohamadi L. Polymorphism of calpastatin, calpain and myostatin genes in native dalagh sheep in Iran. Slovak J Anim Sci. 2012; 45(1):1-6. URL Available in: http://www.cvzv.sk/slju/12_1/Azari-Dehnavi-SJAS-1-2012.pdf. [ Links ]
12. Santos C, Crispim A, Seno L, Vargas-Junior F, Grisolia B. Polymorphisms in the CAST gene of shepp from mato grosso do Sul, Brazil. Rev Colomb Cienc Animal - RECIA. 2016; 8(1):51-55. DOI: https://doi.org/10.24188/recia.v8.n1.2016.206. [ Links ]
13. Excoffier L, Lischer HEL. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Resour. 2010; 10(3):564-567. DOI: https://doi.org/10.1111/j.1755-0998.2010.02847x PMid:21565059. [ Links ]
14. Peakall R, Smouse PE. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research--an update. Bioinformatics. 2012; 28(19):2537-2539. DOI: https://doi.org/10.1093/bioinformatics/bts460 PMid:22820204. [ Links ]
15. Byun SO, Zhou H, Forrest RHJ, Frampton CM, Hickford JGH. Association of the ovine calpastatin gene with birth weight and growth rate to weaning. Anim Genet. 2008; 39(5):572-573. DOI: https://doi.org/10.1111/j.1365-2052.2008.01745.x PMid:18513277. [ Links ]
16. Dehnavi E, Azari M, Hasani S, Nassiry M, Mohajer M, Ahmadi A. Genetic Variability of Calpastatin and Calpain Genes in Iranian Zel Sheep Using PCR-RFLP and PCR-SSCP Methods. Iranian Journal of Biotechnology. 2012; 10(2):136-139. URL Available in: http://www.ijbiotech.com/article7186.html. [ Links ]
17. Nikmard M, Molaee V, Eskandarinasab M, Djadid D, Vajhi A. Calpastatin polymorphism in Afshari sheep and its possible correlation with growth and carcass traits. Journal of Applied Animal Research. 2012; 40(4):346-350. DOI: https://doi.org/10.1080/09712119.2012.692330. [ Links ]
18. Khan S, Riaz M, Ghaffar A, Khan M. Calpastatin (CAST) gene polymorphism and its association with average daily weight gain in Balkhi and Kajli sheep and Beetal goat breeds. Pakistan J Zool. 2012; 44(2):377-382. URL Available in: http://zsp.com.pk/pdf44/377-382%20_11_%20PJZ-607-11%20Corrected-Khan-et-al-PJZ.pdf. [ Links ]
19. Nassiry M, Shahroudi F, Tahmoorespur M, Javadmanesh A. Genetic variability amd population structure in Beta-lactoglobulin, Calpastatin and Calpain loci in Iranian Kurdi sheep. Pak J Biol Sci. 2007; 10(7):1062-1067. DOI: https://doi.org/10.3923/pjbs.2007.1062.1067 PMid:19070051. [ Links ]
20. Molano J. Identificación de polimorfismos de nucleótido simple en los genes Calpaína y Calpastatina en ovinos criollos colombianos. Universidad de Cundinamarca; 2016. [Consultado: 2018 Julio 24]. Disponible en: Disponible en: http://dspace.ucundinamarca.edu.co:8080/xmlui/bitstream/handle/123456789/422/IDENTIFICACI%C3%93N%20DE%20POLIMORFISMOS%20DE%20NUCLEOTIDO%20SIMPLE%20EN%20LOS%20GENES%20DE.pdf?sequence=1&isAllowed=y . [ Links ]
21. Fang Q, Forrest RH, Zhou H, Frampton CM, Hickford JGH. Variation in exon 10 of the ovine calpain 3 gene (CAPN3) and its association with meat yield in New Zealand Romney sheep. Meat Sci. 2013; 94(3):388-390. DOI: https://doi.org/10.1016/j.meatsci.2013.03.015 PMid:23567141. [ Links ]
22. Mahrous KF, Hassanane MS, Shafey HI, Abdel Mordy M, Rushdi HE. Association between single nucleotide polymorphism in ovine Calpain gene and growth performance in three Egyptian sheep breeds. Journal of Genetic Engineering and Biotechnology. 2016; 14(2):233-240. DOI: https://doi.org/10.1016/j.jgeb.2016.09.003. [ Links ]
23. Grochowska E, Borys B, Grzeskowiak E, Mroczkowski S. Effect of the calpain small subunit 1 gene (CAPNS1) polymorphism on meat quality traits in sheep. Small Ruminant Research. 2017; 150:15-21. DOI: https://doi.org/10.1016/j.smallrumres.2017.02.022. [ Links ]
24. Naveen K, Jayashankar M, Nagaraja R, Nagaraja C, Nadeem F, Satyanarayana K. Genetic polymorphism of ovine calpain gene in Bandur sheep. International Journal of Science, Environment and Technology. 2015; 4(3):804-812. URL Available in: http://www.ijset.net/journal/701.pdf. [ Links ]
25. Bahrami A, Behzadi S, Miraei-Ashtiani SR, Roh S-G, Katoh K. Genetic polymorphisms and protein structures in growth hormone, growth hormone receptor, ghrelin, insulin-like growth factor 1 and leptin in Mehraban sheep. Gene. 2013; 527(1):397-404. https://doi.org/10.1016/j.gene.2013.05.066 PMid:23747407. [ Links ]
26. Shojaei M, Abadi M, Fozi M, Dayani O, Khezri A, Akhondi M. Association of growth trait and Leptin gene polymorphism in Kermani sheep. Journal of Cell and Molecular Research. 2010; 2(1):67-73. URL Available in: https://jcmr.um.ac.ir/index.php/biology/article/download/3117/pdf. [ Links ]
Como citar (Vancouver) Montes VD, Lenis VC, Hernández HD Polimorfismos de los genes calpaína y calpastatina en dos poblaciones de ovinos de pelo colombiano. Rev MVZ Cordoba. 2019; 24(1):7113-7118. DOI: https://doi.org/10.21897/rmvz.1345
Recibido: Julio de 2018; Aprobado: Diciembre de 2018; Publicado: Enero de 2019











 texto em
texto em 


