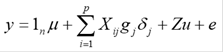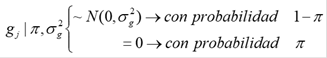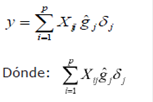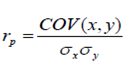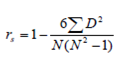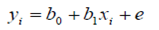INTRODUCCIÓN
La selección genómica (SG) es una metodología que incluye miles de marcadores moleculares, los cuales cubren todo el genoma uniformemente; de tal manera que cada QTL asociado a una característica en particular, esté en desequilibrio de ligamiento con al menos un marcador (1,2). Esta metodología combina la información de fenotipos (o pseudofenotipos: EBVs), el pedigrí y el genotipo de cada animal, para estimar valores de cría genómicos (GEBVs), los cuales son el punto central en los actuales esquemas de mejoramiento genético (3). Esto ha generado nuevas oportunidades para realizar una selección más eficiente en características que incluso presentan baja heredabilidad o que son difíciles de medir (4,5).
Sin embargo, el mayor obstáculo en la implementación de la SG, es el costo de genotipificación de animales, principalmente con chips de moderada y alta densidad (4). Para reducir los costos de genotipificación, una estrategia es usar chips de SNPs de baja densidad, los cuales pueden ser optimizados por procesos de imputación. Las estrategias de genotipificación para imputación generalmente involucran animales influyentes, principalmente machos genotipificados con chips de alta densidad (Illumina SNP50, Illumina BovineHD 800K), animales jóvenes (candidatos a selección) y vacas, usando chips de baja densidad (Illumina BovineLD) y en algunos casos individuos no genotipificados (6,7,8). La imputación de genotipos inicia con la reconstrucción de haplotipos portados por los individuos genotipificados con chips de alta densidad. Luego los genotipos de baja densidad son usados en conjunto con el pedigrí, el ligamiento familiar y la información de desequilibrio de ligamiento para determinar la combinación de haplotipos presentes en los individuos genotipificados con chips de baja densidad y deducir su genotipo a una densidad de SNPs mayor que la inicial (8).
Varios programas de imputación han sido creados como: fastPHASE (9), BEAGLE (10), IMPUTE2 (11), FINDHAP (12), FImpute (13), entre otros. Todos los programas usan diferentes métodos de reconstrucción de haplotipos y por lo tanto el tiempo de computación es diferente. FastPHASE, BEAGLE e IMPUTE2, son lentos ya que usan métodos bayesianos, los cuales pueden ser limitantes en la práctica cuando se usan grandes cantidades de datos. FINDHAP y FImpute usan métodos determinísticos y computacionalmente son muy rápidos y además las precisiones de imputación son comparables con fastPHASE, BEAGLE e IMPUTE2 (6,14). La imputación de genotipos en selección genómica, ha permitido estimar GEBVs en toros y vacas, con confiabilidades similares a las obtenidas, si los animales fueran genotipificados con chips de alta densidad (6,14). Esto ha facilitado la implementación de la SG en programas de mejoramiento genético, haciendo que esta metodología sea más confiable, más eficiente y menos costosa (5,15,16).
A pesar de que en Colombia existe una alta tecnificación en ganado lechero especializado, el mejoramiento genético ha presentado serias dificultades, debido a la limitada cantidad de registros productivos disponibles (17). Sin embargo, esto no ha sido un impedimento para iniciar con la implementación de metodologías que incluyen información genómica tanto en ganado de leche (18,19), en razas criollas (20), así como en ganado de carne (21). El objetivo de esta investigación fue determinar la precisión y el sesgo de valores genómicos directos (VGD) usando genotipos imputados a densidad media, en características productivas y reproductivas en ganado Holstein de Antioquia, Colombia.
MATERIALES Y MÉTODOS
Población. Esta investigación fue realizada con base en la información obtenida de 85 hatos lecheros para la raza Holstein, ubicados en 18 municipios del Departamento de Antioquia (Colombia) que cubre zonas de bosque muy húmedo montano bajo (bmh-mb), con una temperatura que oscila entre 12 y 18°C y un promedio anual de lluvias entre 2000 y 4000 mm. Normalmente se extienden en una faja altimétrica de 1800 a 2800 msnm. Las condiciones de manejo, alimentación y sanidad fueron variables en todos los hatos, así como su topografía y ubicación geográfica.
El número de registros utilizados para los análisis fue variable, de acuerdo con la característica dependiente analizada: 8772, 6624, 6595, 6426, 11562 y 11395 para producción de leche (PL), porcentaje de proteína (PRO), porcentaje de grasa (GRA), recuento de células somáticas (RCS), intervalo entre partos (IEP) y días abiertos (DA), respectivamente. El RCS fue transformado a puntaje de células somáticas (SCS) mediante la siguiente ecuación: SCS = [log2 (RCS/100000)] + 3, con el fin de mejorar la normalidad de los datos, como lo describe Ali y Shook (22). El número de animales en el pedigrí para la evaluación genética convencional fue de 9090 (que incluyó 721 padres y 944 madres) y para evaluación genómica fue de 144 (36 toros y 108 vacas).
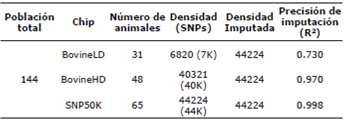
Genotipificación de animates con chips de alta densidad. Fueron genotipificados 144 animales con tres chips de Illumina (Illumina Inc, San Diego, CA): 31 con el chip Bovine LD, 65 con el chip BovineSNP50 v2 y 48 con el chip BovineHD. Después de aplicar el control de calidad: call rate > 0.90, alelos de menor frecuencia (MAF) > 0.02, equilibrio de Hardy Weinberg (HWE) p > 0.001 y descarte de marcadores con errores mendelianos, fueron obtenidos un total de 6716 marcadores comunes a los tres chips, cuya densidad fue denominada 6K. La edición de los genotipos fue realizada con los programas SAS versión 9.2 (SAS Institute Inc., Cary, E.E.U.U.) y PLINK v1.07 (CHGR., Massachusetts, E.E.U.U.)
Imputación de genotipos. Para obtener la base de datos de genotipos imputados, fueron seleccionados 6820 SNPs (panel 7K) del chip LD y 40321 SNPs (panel 40K) del chip HD, los cuales fueron comunes al chip SNP50v2. Los paneles 7K y 40K fueron imputados a 44224 SNPs (panel 44K_imputado). Los SNPs fueron definidos como genotipos discretos (0, 1 y 2), por lo cual se recodificaron como: BB=0 (homocigótico para el primer alelo), AB=1 (heterocigótico), AA=2 (homocigótico para el segundo alelo), B_=3 (alelo paterno conocido y alelo materno no conocido), _A=4 (Alelo paterno no conocido y alelo materno conocido) y 5 cuando ambos alelos son no conocidos. Los SNPs fueron ordenados por número de cromosoma y localización sobre el cromosoma, teniendo en cuenta el ensamble UMD3.1 del genoma bovino. El pedigrí de los 144 animales genotipificados que correspondió a 761 animales, fue incluido en el análisis, con el fin de mejorar la exactitud de la imputación.
La exactitud de la imputación fue determinada por comparación de los genotipos imputados (GI) con los genotipos verdaderos (GV) (genotipos obtenidos con el chip SNP50v2), mediante la correlación de Pearson (rGV;GI). El proceso de imputación fue llevado a cabo mediante el programa FINDHAP.f90 versión 4 (USDA., Beltsville, E.E.U.U.).
Estimación de los efectos de los marcadores y valores genómicos directos. La estimación de los efectos de los SNPs fue realizada empleando el método Bayes C (23). El modelo estadístico general fue descrito como:
Donde: y es el vector de valores fenotípicos corregidos por efectos fijos (que corresponde a los EBVs convencionales obtenidos mediante el método BLUP), para las características PL, SCS, PRO, GRA, DA e IEP; m corresponde a la media general, 1n es el vector de unos de longitud n, Xi;. es el vector columna que representa la covariable del marcador (SNP) en el locus j en el animal i. En cada jth marcador son tres posibles combinaciones de dos alelos (A o B), el homocigótico de un alelo (BB), el heterocigótico (AB) y el homocigótico del otro alelo (AA). Estas combinaciones de alelos (genotipos) son representados cuantitativamente como 0, 1 y 2 respectivamente (es decir; Xjj= 0, 1 o 2), g;. es el efecto aleatorio de sustitución alélica para el marcador j, el cual es condicional sobre o2g y se asume normalmente distribuido N~(0, tfg ) cuando 8j =1, pero gj = 0 cuando 5j=0. 8j es una variable aleatoria 0/1 que indica ausencia (con probabilidad n) o presencia (con probabilidad 1-n) del locus j en el modelo, u es el vector de efectos aleatorios poligénicos de longitud n (Z es la matriz de diseno asociada) y fue considerada como el ajuste de los genes no tenidos en cuenta por los efectos de los jth SNPs en g, u es asumido estar distribuido normalmente, u~N(0, Atfu ), donde A es la matriz de parentesco derivada del pedigrí de los animales genotipificados, e es el residual y también se asume estar distribuido normalmente, e~N(0, Id2,), donde I es una matriz identidad nxn.
De esta manera, el método bayes C asume una mezcla de dos distribuciones para los efectos de los SNPs. En la primera, (con probabilidad n) establece que muchos marcadores no tienen efecto ni varianza y para la segunda, (con probabilidad 1-n) existen marcadores con efecto y varianza, la cual es común para todos los SNPs (24).
Así, los efectos de los SNPs (gj) siguen una distribución a priori normal con media cero y varianza (Sy, mientras que las varianzas de los efectos de los marcadores (o2g) siguen una distribución a priori Chi cuadrada invertida escalada con parâmetros ng (grados de libertad) y s?g (parâmetro de escala). Por otra parte, n es tratada como una distribución a priori uniforme con media cero y varianza 1 (24), como se describe a continuación:
El VGD de los animales cuyo genotipo fue conocido, se determinó como:
Corresponde a la suma de todos los efectos de los SNPs estimados (gj). El programa utilizado para la estimación de los efectos de los SNPs y de los VGDs fue GS3 (INRA., Toulouse, Francia).
Validación cruzada. La precisión de la selección genómica fue determinada mediante validación cruzada, como lo describe Meuwissen et al. (25). La base de datos completa correspondiente a 144 animales con genotipo y fenotipo conocidos fue dividida aleatoriamente en S=12 grupos de igual tamano (S1-S12). En la primera etapa, S1 correspondió al grupo de validación, en el cual los animales presentaron genotipo pero no fenotipo ya que fue removido de la base de datos (fenotipo enmascarado=0), mientras que de S2 a S12 correspondieron al grupo de referencia (con fenotipo y genotipo), a partir del cual se estimaron los efectos de los SNPs, en S1 fueron estimados los VGDs de los 12 animales que conforman el grupo, usando los efectos de los SNPs estimados en los 132 animales remanentes (S2-S12). Esto se repitió para cada uno de los grupos, hasta llegar al grupo S12. Al final se estimaron los VGDs de todos los animales.
Precisión y sesgo de los VGD predichos. Los VGDs estimados mediante el método bayes C fueron comparados con los valores genéticos convencionales (EBV) mediante la correlación de Pearson, que fue considerada como la precisión de la selección genómica (26). La ecuación se representa como:
Donde, rp = coeficiente de correlación de Pearson, x=VGD para la característica i, y=EBV convencional para la característica i. También fue determinado el coeficiente de correlación por rangos de Spearman, el cual fue usado para determinar el grado de similitud entre las clasificaciones o ranking de los animales por sus valores genómicos (VGD o GEBV) y la clasificación de los mismos animales por el EBV convencional.
La ecuación empleada fue descrita como:
Donde:
D = diferencia entre los correspondientes valores genéticos de orden (x-y), x = VGD, y = EBV convencional,
N = número de parejas (x,y) y rs = coeficiente de correlación de Spearman. Un valor de correlación de 1 indica que la clasificación o ranking de los animales por sus valores genéticos (VGD y EBV convencional) es similar.
Finalmente, el coeficiente de regresión lineal de los EBVs convencionales sobre los VGDs (bVGD;EBV) fue determinado. El bVGD;EBV se consideró como el sesgo de la predicción de VGD. La regresión lineal fue definida como:
Donde yi = variable dependiente que corresponde EBV convencional para la característica i, xi = VGD para la característica i, b1 = coeficiente de regresión del EBV convencional sobre el VGD, b0 = intercepto y e = residual.
RESULTADOS
Análisis descriptivo para características productivas y reproductivas. Fueron determinados los promedios para las características productivas PL, PRO, GRA y SCS y para las características reproductivas IEP y DA (Tabla 1), que son resultados normales para la raza Holstein en condiciones del trópico alto colombiano. Por otra parte, las heredabilidades estimadas en la evaluación genética convencional para características productivas (GRA, PRO, PL y SCS) fueron moderadas con valores de 0.33, 0.41, 0.30 y 0.27 respectivamente y para las características reproductivas IEP y DA fueron bajas con valores de 0.11 en los dos casos.
Tabla 1 Análisis descriptivo para características productivas y reproductivas en ganado Holstein de Antioquia, Colombia.
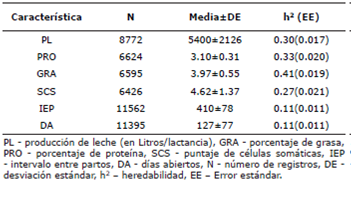
Característica N Media±DE h2 (EE)
PL - producción de leche (en Litros/lactancia), GRA - porcentaje de grasa, PRO - porcentaje de proteína, SCS - puntaje de células somáticas, IEP - intervalo entre partos, DA - días abiertos, N - número de registros, DE -desviación estândar, h2 - heredabilidad, EE - Error estândar.
Precisión de la imputación. La base de datos 7K conformada por 31 animales fue imputada a 44K, obteniendo una exactitud de 0.73. De igual manera, la base de datos 44K conformada por 48 animales fue imputada a 44K, logrando una exactitud de 0.97 (Tabla 2).
Análisis descriptivo para los efectos de los marcadores. Los efectos de los SNPs fueron estimados en todas las características incluídas en el anâlisis (PL, PRO, GRA, SCS, IEP y DA), usando genotipos de baja densidad (6K) y genotipos imputados (44K_imputado). Los efectos de los SNPs fueron mayores en todas las características cuando se usó la base de datos 6K, en comparación con los efectos de los SNPs estimados en la base de datos 44K_imputado (Tabla 3).
Tabla 3 Análisis estadístico descriptivo de los efectos de los SNPs para características productivas y reproductivas em ganado Holstein de Antioquia, Colombia
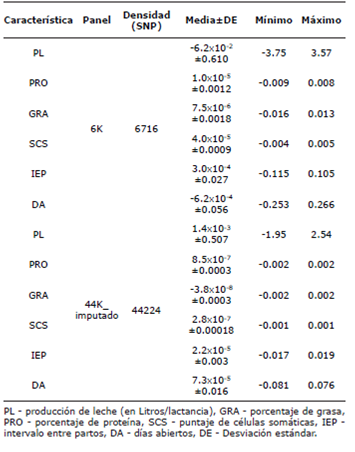
PL - producción de leche (en Litros/lactancia), GRA - porcentaje de grasa, PRO - porcentaje de proteína, SCS - puntaje de células somâticas, IEP -intervalo entre partos, DA - días abiertos, DE - Desviación estândar.
Precisión de los valores genómicos directos. Las precisiones genómicas (rpVGD.EBV) oscilaron entre 0.19 y 0.29, cuando se usó la base de datos de genotipos 6K, mientras que cuando se usó la base de datos 44K_ imputado, las precisiones genómicas fueron mayores y oscilaron entre 0.24 y 0.33 en todas las características evaluadas. Aunque las correlaciones obtenidas fueron bajas, tanto usando genotipos de baja densidad, como genotipos de alta densidad, puede evidenciarse un incremento de las precisiones cuando se usa una densidad mayor de SNPs y es mâs notorio en características de mayor heredabilidad (Tabla 4). De igual manera, las correlaciones de Spearman (rsVGD;EBV) oscilaron entre 0.16 y 0.33 cuando se usó la base de datos 6K, mientras que cuando se usaron datos imputados (44_imputado), las correlaciones fueron mayores, con valores que oscilaron entre 0.30 y 0.35, excepto en IEP que presentó un valor de 0.20 (Tabla 4).
Tabla 4 Precisión genómica de los valores genómicos directos y correlación de Spearman entre el VGD y el EBV.
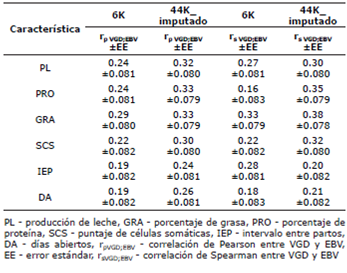
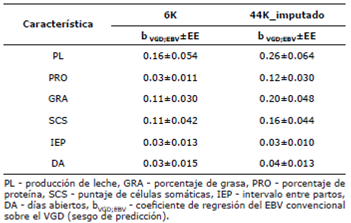
Los coeficientes de regresión (bVGI>EBV) obtenidos para PL, PRO, GRA, SCS, IEP y DA fueron bajos: 0.16, 0.03, 0.11 y 0.11 respectivamente, cuando se usó la base de datos 6K, mientras que cuando se usó la base de datos 44K_imputado, los coeficientes fueron mayores solo en PL, PRO, GRA y SCS con valores de: 0.26, 0.12, 0.20 y 0.16 respectivamente. Para IEP y DA no presentaron cambios significativos, con valores de 0.03 y 0.04 respectivamente (Tabla 5).
DISCUSIÓN
En la presente investigación, se determinó la precisión de imputación de genotipos, obteniendo valores de 0.97 cuando se imputó de 40K a 44K, mientras que la precisión fue menor (0.73), cuando se imputó genotipos de 6K a 44K. Khatkar et al (6), determinaron precisiones de imputación usando los programas IMPUTE2, Beagle y Fastphase con valores de 0.8983, 0.8595, y 0.7630, cuando imputaron genotipos de 3K a 50K y 0.9731, 0.9597 y 0.9626 cuando imputaron de 35K a 50K respectivamente para cada programa, usado una población de referencia de 136 animales; cuando la población de referencia fue de 27 animales, las precisiones de imputación fueron menores con valores de 0.8160, 0.6598 y 0.7409 cuando imputaron de 3K a 50K y 0.9229, 0.8543 y 0.9351 al imputar de 35K a 50K. En otra investigación, Chen et al (26), determinaron las precisiones de imputación para las densidades de SNPs: 6K, 3K, 1536SNP, 768SNP y 384SNP, cuando fueron imputadas a 50K, usando el programa FImpute2, obteniendo valores de 0.9723, 0.9367, 0.9120, 0.8285 y 0.7210 respectivamente para cada densidad. En esta investigación los autores incluyeron 2046 animales en la población de referencia (genotipos 50K). Estos resultados concuerdan con los resultados de la presente investigación y en todos los casos se sugiere que el número de animales en la población de referencia y la densidad de los marcadores son factores que afectan significativamente la precisión de imputación.
Por otra parte, el uso de paneles de SNPs de alta densidad puede incrementar la precisión de la selección genómica, mucho mâs que con paneles de baja densidad 6,27. Sin embargo, el costo de genotipificación de animales con chips de alta densidad es mucho mâs elevado, por lo cual, se hace necesario el uso de datos imputados, para mejorar la predicción de valores genómicos. En la presente investigación, se usaron genotipos de baja densidad (6K) y genotipos imputados (44K_imputado), en los cuales se determinó la precisión (rpVGD;EBV) y el sesgo de predicción (bVGD;EBV), para seis características de importancia económica (PL, PRO, GRA, SCS, IEP y DA). Las precisiones de predicción de VGD fueron en promedio 0.23 cuando se usó el panel 6K y 0.30 cuando se usó el panel 44K_imputado.
Khatkar et al 6, igualmente determinaron la precisión de los VGDs en una población de validación de 452 toros, usando los efectos de los SNPs de una población de referencia de 1753 animales. La precisión fue calculada usando los genotipos LD, 50K_imputado y 800K_ imputado. Las precisiones para PL (h2 =0.25) fueron: 0.481, 0.546 y 0.558 y para fertilidad (h2=0.04) fueron menores: 0.232, 0.246 y 0.256 respectivamente para cada densidad. Los resultados indican que la precisión fue mayor usando la densidad 800K_imputado, comparada con las precisiones obtenidas para 50K_imputado y LD. Por otra parte, Chen et al (26), determinaron mejores precisiones cuando usaron paneles con mayor densidad de SNPs para PL y PRO. Para PL las precisiones fueron: 0.64 (50K), 0.61 (3K), 0.58 (L1536), 0.52 (L768) y 0.42 (L384). Para PRO las precisiones fueron: 0.76 (50K), 0.72 (3K), 0.59 (L1536), 0.51 (L768) y 0.34 (L384). En la presente investigación, las precisiones obtenidas para PL fueron 0.24 y 0.32, para PRO 0.34 y 0.33, para IEP 0.19 y 0.24 y para DA 0.19 y 0.26 cuando se usaron los paneles 6K y 44K respectivamente. En la investigación de Chen et al 26, determinaron un incremento promedio en la precisión de 7 puntos cuando pasaron de 1536 a 6177 SNPs y de 7.8 puntos cuando pasaron de 1536 a 35790 SNPs, para cuatro características evaluadas (PL, PRO, GRA y SCS). En la presente investigación se determinó un incremento en la precisión de 6.8 puntos en promedio para las mismas características (PL, PRO, GRA y SCS), cuando se pasó del panel 6K al panel 44K_imputado, lo que indican que la densidad de los marcadores, juega un papel crucial en la precisión de la predicción genómica, tanto en características de alta como de baja heredabilidad, lo que muestra la importancia de usar chip de moderada o alta densidad y/o de realizar imputación de genotipos. La explicación a este aumento en la precisión de predicción de VGD se debe a que um panel con mayor número de SNPs permite cubrir una mayor región en el genoma, y por lo tanto, capturar un mayor número de QTLs involucrados en la expresión de las características cuantitativas.
En la actual investigación, las precisiones de predicción de los VGD para PRO y GRA fueron iguales con un valor de 0.33 para las dos características usando el panel 44K. De igual manera, Chen et al 26, determinaron las precisiones de predicción de VGD usando un panel de 50K, para PRO y GRA, obteniendo valores de: 0.75 y 0.76 respectivamente. En las dos investigaciones, se puede ver que PRO y GRA tienen una arquitectura genética similar, es decir que hay pocos QTLs con gran efecto, comparadas con otras características como SCS donde hay muchos QTLs con efecto pequeno. Ahora, la diferencia en las precisiones en las dos investigaciones estâ asociada al tamano de la población referencia. En la presente investigación fueron usados 132 animales, mientras que en la investigación de Chen et al 26 fueron usados 10309 animales. Ademâs, las metodologías usadas fueron diferentes, Bayes B en el primer caso y Bayes C en la actual investigación. En bayes B cada SNP tiene una varianza locus específica, mientras que en bayes C, la varianza es común para todos los SNPs 23.
Habier et al 28, determinaron la precisión de predicción de GEBVs en 113 toros jóvenes de raza Holstein, usando los efectos de 40764 SNPs estimados en una población de referencia de 4000 animales. Las precisiones obtenidas (rpGEBV;DEBV) para PL y SCS fueron 0.46 y 0.18 usando el método bayes A, 0.41 y 0.12 usando el método bayes B y 0.43 y 0.15 usando el método bayes Cpi. Para características de baja heredabilidad como SCS, muchos QTL con efectos pequenos no pueden ser detectados, ya que el desequilibrio de ligamiento entre marcadores y QTL puede ser muy bajo, por lo cual se requiere una mayor densidad de marcadores. Con un panel 50K, no ha sido posible aún capturar el 100% de la varianza genética en todas las características evaluadas en ganado lechero, el valor capturado está entre el 90% para PL a un 32% para características de fertilidad 28.
En otra investigación, Hayes et al 29, determinaron la confiabilidad (definida mediante la relación rGEBV;EBV/ rEBV;TEBV) de valores genómicos en animales jóvenes usando el método bayes A, para PRO y fertilidad, usando una población de referencia de 332 toros, a partir de los cuales se estimaron los efectos de 38259 SNPs. La confiabilidad para PRO fue de 0.36, mientras que para fertilidad fue de 0.14. Por su parte, Nicolazzi et al 30 determinaron la precisión de predicción de valores genómicos (rABV;DGV), usando los métodos Bayes A y Bayes LASSO-gamma para producción de proteína (PPRO) y GRA en una población de validación de 386 toros, usando los efectos de 39048 SNPs estimados en una población de referencia de 763 toros de raza Holstein-Friesian. Las precisiones de predicción obtenidas para PPRO fueron 0.52 y 0.48 respectivamente para cada método, mientras que para GRA los valores fueron 0.75 y 0.71 respectivamente. Los autores sugieren que los resultados de precisión de predicción son mejores para GRA, dado que esta característica es explicada en mâs de un 30% por mutaciones en los genes DGAT1 y GHR, indicando que GRA es una característica mâs heredable que PPRO. Colombani et al 31, también determinaron la precisión y el sesgo de los valores genómicos en las razas Holstein (2976 toros en la población de referencia) y Montbéliarde (950 toros en la población de referencia), usando el método bayes Cpi. Los SNPs evaluados fueron 38462 para Montbéliarde y 39738 para Holstein. Las precisiones para PL, GRA y tasa de concepción (TC) fueron: 0.57, 0.80 y 0.34 para raza Holstein y 0.44, 0.62 y 0.43 respectivamente para la raza Montbéliarde. Los coeficientes de regresión (sesgo de predicción) fueron: 0.73, 0.90 y 0.72 en Holstein y 0.74, 0.85 y 1.35 en la raza Montbéliarde.
En la actual investigación, las precisiones obtenidas para PRO y GRA fueron mayores (0.33 en los dos casos) comparadas con las obtenidas para DA e IEP que fueron 0.24 y 0.26 respectivamente. De igual manera, el coeficiente de regresión fue mayor para PRO y GRA (0.12 y 0.20) comparado con, IEP y DA que fueron 0.03 y 0.04 respectivamente. Estos coeficientes en todos los casos fueron menores de 1 (b<1) indicando que los VGD fueron sobreestimados con respecto a los EBVs convencionales, lo que sugiere una inflación de la varianza genética en todas las características. Hayes et al 29, hacen referencia a que la confiabilidad de los GEBVs para fertilidad fue baja comparada con PRO, considerando la alta diferencia de heredabilidad entre características evaluadas y sugieren que se requiere una mayor cantidad de registros para características reproductivas en experimentos iniciales para obtener GEBV con mayor confiabilidad y lograr que la selección genómica sea más eficiente.
Finalmente, debe considerase que en la presente investigación fueron estimados los efectos de los marcadores en una población de referencia pequena, por lo cual las precisiones y los coeficientes de regresión fueron bajos comparados con los resultados de las investigaciones de los autores mencionados anteriormente. Por lo tanto, es importante aclarar que esta investigación muestra resultados iniciales de evaluaciones genómicas para características de interés económico tanto de baja como de alta heredabilidad en el trópico alto colombiano. No obstante, es necesario incrementar el número de animales genotipificados y el número de SNPs para lograr consolidar una población referencia de la cual se pueda realizar evaluaciones genómicas con mayor precisión.











 text in
text in