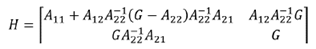INTRODUCCIÓN
Tradicionalmente, las evaluaciones genéticas están basadas en el análisis de información genealógica y fenotípica a través de los sistemas de ecuaciones de los modelos mixtos 1. Sin embargo, la disponibilidad de marcadores genéticos tipo SNP en las últimas décadas ha permitido a investigadores identificar genes de importancia productiva, medir la variabilidad genética y estimar valores genéticos genómicos. Esto ha sido recomendado especialmente para aquellas características con baja heredabilidad o difíciles de medir 2.
El peso a los ocho meses de edad (W8M), la edad al primer parto (AFC), y el primer intervalo entre partos (FCI) han sido usados como criterios de selección para incrementar la productividad y evaluar la eficiencia reproductiva en sistemas de producción bovina 2,3. La disminución de AFC y FCI reduce los costos de producción en las hembras de reemplazo y de terneros por año 4. Asimismo, la selección por un mayor crecimiento permite que los animales permanezcan menos tiempo en las praderas, lo cual disminuye el tiempo del ciclo de producción y aumenta la rentabilidad.
La estimación de parámetros genéticos con información genómica ha sido discrepante entre poblaciones de acuerdo con la estrategia de genotipado y las características evaluadas, especialmente para características reproductivas 5. No obstante, la inclusión de información genómica ha mostrado estimaciones más confiables, lo cual podría ser importante para características de baja heredabilidad 6. Entonces, el uso de la información genómica podría permitir a los criadores alcanzar un mayor progreso genético a través del incremento de la precisión y la reducción del intervalo generacional 7.
En Colombia, la raza Simmental es usada en sistemas de doble propósito y está actualmente distribuida a lo largo del territorio colombiano. Sin embargo, no hay estimaciones de parámetros genéticos, lo cual ha limitado el diseño e implementación de un programa de mejoramiento genético. Por lo tanto, el objetivo de este estudio fue estimar parámetros genéticos para W8M, AFC y FCI en ganado Simmental de Colombia utilizando modelo animal univariado y bivariados bajo el mejor predictor lineal insesgado convencional (BLUP) y genómico en una etapa (ssGBLUP).
MATERIALES Y MÉTODOS
La aprobación por parte de un comité de ética no fue necesaria en este estudio debido a que todos los registros utilizados en los análisis provenían de una base de datos ya existente y no involucraron experimentos o procedimientos con los animales.
Información de pedigrí y fenotípica. La información genealógica de la Asociación de Criadores de Ganado Simmental de Colombia (Asosimmental) fue usada en este estudio. El archivo de pedigrí incluyó 27986 animales nacidos desde 1975 hasta 2017 con 15 generaciones trazadas. El número de registros para W8M, AFC y FCI fue 481, 3063 y 1098, respectivamente. El archivo final de pedigrí para computar la matriz de parentesco tuvo 26376, 27037 y 26650 animales para W8M, AFC y FCI, respectivamente.
Genotipado y control de calidad. La población genotipada estuvo conformada por 718 animales usando el chip comercial GeenSeek Genomic Profiler-LD chip (GGP Bovine LD v4) con 30106 SNP. Para el control de calidad, los SNP con una ausencia de genotipado >10%, una frecuencia del alelo con menor frecuencia <0.05 y un valor p<0.001 para la prueba del equilibrio Hardy-Weinberg, fueron excluidos y los animales con una tasa de genotipado <90% también fueron excluidos usando PLINK 8. Un total de 661 animales y 22395 SNP permanecieron después del control de calidad, y fueron usados para estimar los parámetros genéticos. El número de animales con genotipos y fenotipos fueron 372 y 162 para AFC y FCI, respectivamente. Los animales con registros para W8M no tuvieron información genómica, pero estuvieron emparentados con la población genotipada.
Condiciones ecológicas y de manejo. En relación con las prácticas de producción de los ganaderos colombianos, las vacas en producción y los terneros son mantenidos en pastoreo todo el año. La dieta estuvo conformada por forraje, alimentos balanceados y suplementación mineral. La inseminación artificial es usada en lugar de la monta natural y no existen épocas fijas de reproducción, lo que significa que las vacas pueden ser inseminadas en cualquier mes del año.
Análisis genético. Los componentes de varianza fueron estimados usando modelo animal univariado (W8M, AFC, FCI) y bivariados (W8M x AFC and AFC x FCI). Los efectos fijos para W8M fueron grupo contemporáneo (sexo, año de nacimiento desde 2010 hasta 2015, y mes de nacimiento desde enero hasta diciembre), y la covariable edad. El W8M no incluyó efecto materno porque los animales evaluados fueron removidos de la madre y criados artificialmente. Los efectos fijos para AFC fue el año de nacimiento desde 1999 hasta 2014 y los efectos fijos para FCI fueron año de nacimiento desde 2003 hasta 2015, y el hato. El efecto de la región no fue incluido para todas las características porque no fue significativo o no estuvo disponible. Los efectos aleatorios incluidos en todos los modelos fueron el animal y el residual.
En notación matricial, el modelo animal usado es:
y= Xβ+Za+e,
Donde y es el vector de registros productivos; β es el vector de efectos fijos; a es el vector de soluciones para el efecto genético directo y aleatorio del animal; e es el vector de efectos residuales; X y Z, son las matrices de incidencia relacionando β y a con y. El modelo asumió que E[y] = Xβ;Var(a) = AGa;Var(e) = I ⨂R, donde A es la matriz de parentesco por pedigrí, ⨂es el producto directo de Kronecker, Ga es la matriz de (co)varianza de efectos genéticos aditivos directos, I es la matriz de identidad y R es la matriz de (co)varianza de efectos residuales.
Análisis genómico. Los componentes de varianza fueron estimados usado la metodología del ssGBLUP. Los mismos modelos univariados y bivariados descritos en la sección análisis genético fueron ejecutados, pero la matriz de parentesco por pedigrí (A) fue reemplazada por la matriz H, así:
donde, A11 es la matriz de parentesco por pedigrí de animales no genotipados, A22 es la matriz de parentesco por pedigrí de animales genotipados, A12 que es igual a A21 son matrices que contienen las relaciones de parentesco por pedigrí de los animales genotipados y no genotipados, G es la matriz de parentesco genómico. Por lo tanto, la matriz H incluye el parentesco basado en el pedigrí y en la información genómica 9.
Los componentes de varianza para el análisis genético y genómico fueron ejecutados usando el procedimiento del promedio de información por máxima verosimilitud restringida (AIREML) incluido en la familia de programas del BLUPF90 10.
RESULTADOS
Los promedios para W8M, AFC y FCI fueron 247±37 kg, 1080±269 d y 464±106 d, respectivamente. Las heredabilidades para W8M, AFC y FCI con el análisis genético y genómico son mostradas en la Tabla 1.
Tabla 1 Heredabilidades para el peso a los ocho meses de edad (W8M), edad al primer parto (AFC) y primer interval entre partos (FCI) usando análisis genético y genómico en ganado Simmental de Colombia.
| Carácter | Modelo | Heredabilidades | |
|---|---|---|---|
| Análisis genético | Análisis genómico | ||
| W8M | Univariado | 0.26±0.15 | 0.26±0.15 |
| Bivariado con AFC | 0.25±0.15 | 0.25±0.15 | |
| AFC | Univariado | 0.22±0.04 | 0.20±0.04 |
| Bivariado con W8M | 0.21±0.04 | 0.20±0.04 | |
| Bivariado con FCI | 0.21±0.04 | 0.20±0.04 | |
| FCI | Univariado | 0.04±0.05 | 0.07±0.06 |
| Bivariado con AFC | 0.05±0.05 | 0.08±0.06 | |
Todos los modelos usados para estimar la heredabilidad de AFC y que incluyeron información genómica mejoraron ligeramente (a partir del tercer decimal) la precisión de la heredabilidad. Por otro lado, no hubo incrementos en las precisiones cuando se incluyó información genómica para W8M y FCI, pero fueron ligeramente más altas con parentesco genómico (a partir del tercer decimal) que aquellas heredabilidades sin información genómica. Las correlaciones genéticas entre W8M y AFC fueron medias y negativas con valores de -0.34 y -0.27 con y sin parentesco genómico, respectivamente. Respecto a las correlaciones genéticas entre AFC y FCI, fueron positivas con valores de 0.23 y 0.25 con y sin parentesco genómico, respectivamente.
DISCUSIÓN
La media de W8M fue mayor a lo reportado en una población de ganado Brahman en Colombia, la cual presentó pesos inferiores (237±36 kg) a una edad mayor (270 días) 11. Los sistemas de producción de ganado Simmental en Colombia utilizan suplementos balanceados y forrajes de alta calidad nutricional, lo cual podría explicar parcialmente los pesos superiores encontrados en esta población. Sistemas de producción de doble propósito con la raza Pardo Suizo en México con similares condiciones ambientales y prácticas de manejo mostraron un peso a 240 días de 235±44 kg 12, valor muy similar al promedio reportado en este estudio.
El promedio de la AFC siempre fue más largo comparado con otros estudios reportados en razas taurinas. Por ejemplo, una población de ganado Holstein evaluada en Colombia presentó el valor más similar con un promedio de 962 días de edad al primer parto 13. Aunque los sistemas de producción de leche se han vuelto más intensivos en Colombia, el descarte de animales por AFC todavía no está implementado por los productores de ganado Simmental en Colombia. Eso podría contribuir a una AFC más larga. Además, el criterio para la primera inseminación en ganado Simmental ha sido el peso del animal, lo cual podría ser otro factor para incrementar la AFC si la ganancia media de peso desde el destete hasta la AFC es baja.
La media de FCI fue más alta comparada con una población de animales Holstein que tuvo 385 días FCI 14. Aunque las prácticas de manejo productivo y reproductivo de los sistemas de ganado Simmental son favorables e intensivas, los productores de ganado Simmental de Colombia mantienen animales con problemas reproductivos debido a la escasez de hembras. En el contexto anterior, los periodos de intervalos entre partos podrían ser más largos.
Las heredabilidades para W8M coincidieron con los valores reportados por otros autores con valores desde 0.11 hasta 0.35 15,16. Las heredabilidades directas estimadas para W8M fueron más altas que las reportadas por Guillen et al 15 en una población cebuína en condiciones tropicales, pero muy similar a lo reportado por Kebede y Komlosi 17 quienes estimaron una heredabilidad directa de 0.26 en otra población de ganado Simmental. La población cebuína consultada tenía una alta intensidad de selección para peso al destete (25%), lo cual podría explicar parcialmente una menor varianza genética debido a procesos de selección. En Colombia, los resultados de W8M indican que un importante mejoramiento para esa característica podría ser alcanzado mediante selección genética. Sin embargo, una mayor cantidad de registros fenotípicos y animales genotipados son aún necesarios para incrementar la precisión en las estimaciones, y consecuentemente el progreso genético.
Aunque las diferencias entre las heredabilidades para W8M entre modelos univariado y bivariado incluyendo o no información genómica fueron pequeñas (p<0.006), una menor heredabilidad estimada en modelos bivariado pudo ser debida a la presencia de pocos animales en la base de datos con información para todas las características 16. El incremento de la heredabilidad fue leve (0.0024) en modelos que incluyeron parentesco genómico. Esto fue coherente a lo reportado en otros estudios para características asociadas a crecimiento 2. Las leves diferencias encontradas podrían sugerir que la matriz de parentesco genómica y por pedigrí para los animales genotipados es similar, lo cual no alteraría en gran medida las estimaciones de los parámetros genéticos. Además, el hecho que no hubo animales con información genómica y fenotípica para W8M podría explicar los cambios leves de las estimaciones.
Heredabilidades para AFC han variado considerablemente entre poblaciones taurus e indicus, variando desde 0.08 hasta 0.47 18,19,20. Una población de ganado criollo en Colombia presentó un valor de 0.15 21 y otras poblaciones de ganado Holstein en Colombia y Brasil tuvieron valores más bajos, desde 0.13 hasta 0.19 13. Las diferencias en estimaciones podrían estar relacionadas con el tamaño de las bases de datos, los efectos incluidos en los modelos e incluso factores ambientales que no fueron registrados 22. Marques et al 18 encontraron una heredabilidad para AFC desde 0.16 hasta 0.18 y sugirieron que una disminución en esa característica podría ser hecha por selección genética. Por lo tanto, el progreso genético para AFC en ganado Simmental de Colombia podría ser posible.
Las diferencias en la heredabilidad para AFC de acuerdo con los modelos fueron diferentes a lo reportado por Buzanskas et al 23, quienes observaron mayores heredabilidades para AFC en modelos univariado versus modelos bivariado. La varianza genética más baja pudo ser debido al uso de los SNP, los cuales podrían identificar un grado de parentesco menor entre algunos animales, y con ello disminuir la proporción de la varianza genética conocida 9. Por otro lado, el efecto del parentesco genómico sobre los errores estándar coincidió con los resultados reportados por Haile-Mariam et al 24, quienes reportaron errores estándar más bajos para estimaciones con información genómica comparado con modelos que solamente incluyeron matriz de parentesco por pedigrí.
Las heredabilidades reportadas para FCI fueron menores comparadas con W8M y AFC. Las estimaciones de heredabilidades para FCI en ganado Simmental en Colombia fueron consistentes con las heredabilidades encontradas en otros estudios, las cuales variaron desde 0.01 a 0.011 para razas lecheras taurinas 3,14,19,25. Esas variaciones podrían ser dadas principalmente por diferencias en el ambiente, las cuales pueden estar relacionadas con los sistemas de alimentación y decisiones en los programas de monta estacional. Por otro lado, las razas taurinas han sido más seleccionadas por el desempeño reproductivo, lo cual reduce la varianza genética de las características en el tiempo. Aunque los productores todavía no han implementado un programa de mejora genética en Colombia, esa población de ganado proviene de poblaciones europeas y norte americanas intensivamente seleccionadas, lo cual podría explicar las bajas heredabilidades.
Las variaciones de las heredabilidades para FCI estuvieron en acuerdo con lo reportado por Haile y Price 26, donde la heredabilidad varió desde 0.03 hasta 0.06. Los incrementos observados de la heredabilidad en los modelos bivariado con y sin información genómica pueden ser debidos a la correlación genética positiva entre FCI y AFC, dirigiendo a una mayor explicación de la varianza genética aditiva. Igualmente, el uso simultáneo de modelos bivariado y parentesco genómico es favorable para las características con una menor cantidad de registros y generalmente permite explicar una mayor cantidad de la varianza genética 27.
Mayores relaciones entre la matriz de parentesco por pedigrí y genómico podría dirigir a estimaciones más precisas y a su vez podrían capturar una mayor varianza genética aditiva. La correlación entre la matriz genómica y por pedigrí fue alta (>0.8), lo cual indica una buena calidad de pedigrí. Esto podría explicar parcialmente las discrepancias en las estimaciones de la varianza genética aditiva para AFC y FCI al incluir información genómica. Sin embargo, el bajo número de animales genotipados en este estudio y la densidad del chip de genotipado tienen que ser considerado entre las diferencias de las estimaciones, lo cual podría afectar la varianza de las características evaluadas 24.
La correlación genética entre W8M y AFC fue igual a lo reportado por Chin-Collin et al 3, quienes reportaron un valor negativo de -0.34. Sin embargo, otros estudios han reportado valores cercanos a cero y ligeramente negativos de -0.02 12. Las heredabilidades y la correlación genética indican una oportunidad para estas características para ser incluidas en un programa de mejoramiento genético. Eso optimizaría el progreso genético porque animales con mayor W8M tendrían una AFC más corta. La mayor correlación genética incluyendo información genómica pudo estar asociada a una mayor relación de los efectos poligénicos de animales genotipados y no genotipados, lo cual contribuye con la construcción de la matriz de parentesco genómica y las diferencias encontradas 28.
La correlación genética entre AFC y FCI coincidió con lo reportado por Gutiérrez et al 22 en razas de carne. Sin embargo, Rocha et al (21) y Chin-Colli et al 3 reportaron correlaciones genéticas negativas entre estas características con valores de -0.43 y -0.26, respectivamente. Las diferencias respecto a esas poblaciones están posiblemente asociadas con diferentes criterios de selección que cambian la estructura genética a través del tiempo y la magnitud de las heredabilidades y las correlaciones genéticas. En Colombia, la disponibilidad y el uso simultáneo de toros tipo carne y leche que provienen de Europa y Norte América pueden estar afectando actualmente la estructura de la raza y las estimaciones de parámetros genéticos.
Aunque las heredabilidades y las correlaciones genéticas para AFC y FCI fueron bajas, su inclusión en programas de mejoramiento genético podría ser una estrategia confiable para incrementar la productividad debido al efecto económico 29. Adicionalmente, esos resultados en la población de ganado Simmental en Colombia sugieren que la selección de animales con un valor genético más alto para W8M y un valor genético más bajo para AFC representaría una mayor eficiencia reproductiva. Por otro lado, la inclusión de la AFC en esquemas de selección genética podría ser considerada para incrementar el progreso genético para características asociadas con fertilidad 30.
En conclusión, el uso de información genómica mediante la metodología del ssGBLUP podría optimizar el mejoramiento genético para las características evaluadas debido a los incrementos en las precisiones y las heredabilidades. Aunque las diferencias en los errores estándar y las heredabilidades entre las estimaciones con parentesco por pedigrí y genómico fueron muy pequeñas en esta población, estrategias como el genotipado de un mayor número de animales que estén más emparentados con la población no genotipada podría mejorar las estimaciones significativamente. Asimismo, una mayor recolección de información fenotípica es crucial para obtener un mayor beneficio del potencial ofrecido por la selección genómica en programas locales de mejoramiento genético