INTRODUCCIÓN
El parvovirus canino tipo 2 (PVC-2), es el agente causal más importante de muertes en cachorros alrededor del mundo, desde su aparición en 1978 1. Este virus se ha asociado a una enfermedad caracterizada por un síndrome que consiste en vómitos, diarrea hemorrágica severa, de aparición súbita 2.
El CPV-2 apareció como mutación de un virus similar al de la panleucopenia felina (FPLV), y logró adaptase a los caninos. A pesar de haber vacunas disponibles en el mercado, el parvovirus aún es el agente etiológico más importante de gastroenteritis virales en cachorros. La cepa original denominada parvovirus canino tipo 2 (CPV-2) desde que surgió, ha sufrido alteraciones genéticas, llevando a la aparición de nuevas variantes 3. En 1980 el CPV-2, mutó a la variante CPV-2A y en 1984 se describió el CPV-2B, asociada a una mutación, que resultó en un cambio del aminoácido asparagina por aspartato en la posición 426 del gen VP-2 4,5.
En el año 2000 se detectó en Italia por primera vez una tercera variante conocida como CPV-2C, con otra mutación en la misma posición, (glutamato). Esta nueva variante se ha diseminado rápidamente en la población canina en varias regiones 5. En Sudamérica, se han diagnosticado cepas CPV-2C más virulentas que sus antecesoras CPV-2A y CPV-2B (6-8)CPV-2b, and CPV-2c. En México los investigadores también refieren que la variante C es la predominante 9,10. En Centroamérica los estudios moleculares para identificar variantes son inexistentes. Por lo tanto, se hace necesario conocer las variantes de parvovirus canino-circulantes en Nicaragua y la relación filogenética con las cepas vacunales que actualmente se comercializan en la región.
El objetivo del presente trabajo fue identificar los genotipos de parvovirus canino-circulantes en cachorros en dos municipios de Nicaragua.
MATERIALES Y MÉTODOS
Recolección de muestras e información general. Se recolectaron muestras por hisopado rectal de 45 cachorros con y sin antecedentes de vacunación, los perros debían tener menos de seis meses de edad y presentar (n = 30) o no (n = 15) sintomatologia compatible con parvovirosis. Los datos sobre las diferentes variables fueron recolectados en el mismo momento de la toma de muestra, con ayuda de una ficha de colección de datos, que incluyó información general y sobre la enfermedad en el canino.
Extracción de ADN viral. Los hisopos con las muestras fueron colocados en viales de 1.5 ml, se agregaron 200 u.l de agua libre de nucleasas, se aplicó Vortex por 20 segundos, luego se utilizó el protocolo descrito por el fabricante (QIAamp DNA Mini Kit QIAGEn), con la diferencia de que, en el último paso, el ADN se eluyó en 200 u.l de agua libre de nucleasas. También se obtuvo ADN de las dos vacunas que son comercializadas en país; estas fueron identificadas como vacuna n°1 y vacuna n°2. Como control negativo para la extracción se utilizó agua libre de nucleasas.
Amplificación del gen VP2. Se utilizó PCR convencional con los cebadores Hfor/ Hrev (5'-CAGGTGATGAATTTGCTACA-3'/5'-CATTTGGATAAACTGGTGGT-3'), descritos previamente 5, que flanquean un segmento de « 630 pb del gen VP2 del parvovirus canino. El volumen final de reacción fueron 50 u.l, agregando 25 u.l de Máster Mix 2X (Promega, EE. UU), 12 u.l de agua libre de nucleasas, 4 u.l del cebador Hfor (1x103 nM), 4 u.l de cebador Hrev (1x103 nM) y 5u.l de muestra de ADN. La reacción de PCR se realizó con el Termociclador Applied Biosystem 2720, elevando la temperatura a 94°C durante 10 minutos, seguido de 40 ciclos (95°C por 50 segundos, 55°C por 1 minuto, 72°C por 1 minuto), una extensión final durante 7 minutos a 72°C. Se utilizó como control positivo el ADN extraído de las vacunas n°1 y n°2, como control negativo se utilizó la muestra de agua que fue procesada como una muestra en la extracción de ADN. Para visualizar los productos del PCR se realizó una electroforesis en gel de agarosa al 1.3% teñido con bromuro de etidio, aplicando 10 u.l del producto de amplificación en cada pozo. La visualización se realizó en un transiluminador de luz UV.
Secuenciación. Se realizó la secuenciación en sentido reverso (cebador Hrev) para cuatro muestras de campo elegidas aleatoriamente más las vacunas n°1 y n°2. Las secuencias fueron realizadas en el Centro de Investigación en Biología Celular y Molecular (CIBCM), Universidad de Costa Rica (UCR),
Análisis filogenético. Las secuencias de cada cepa fueron alineadas mediante ClustalW 1.6, la historia de evolución fue inferida usando el método de Neighbor-Joining, mientras que las divergencias evolutivas fueron calculadas aplicando el método de máxima verosimilitud y se presentan como unidades de bases de sustituciones por sitio. Se realizó la prueba bootstrap (500 repeticiones) como estadístico de significancia y se muestra como el porcentaje de aparición de cada taxón en el árbol filogenético 11. El análisis se llevó acabo con ayuda del software Molecular Evolutionary Genetics Analysis Version 6 (MEGA 6) 12. La identificación del genotipo se determinó en base al aminoácido correspondiente al codón 426 del gen VP2 5. Las secuencias fueron enviadas al National Center for Biotechnology Information (NCBI) utilizando Banklt 13, obteniendo los números de acceso MN517818, MN517819, MN541177, MN541178 para las muestras, los números MN541179 y MN541180 fueron asignados a la vacuna n°1 y vacuna n°2 respectivamente.
Análisis estadístico. Los resultados se presentan en porcentajes y números absolutos, mientras que, para la búsqueda de asociación de variables categóricas se aplicó la prueba exacta de Fisher y T de Student para la edad en meses.
Consideraciones éticas. La toma de muestra se realizó siempre y cuando el dueño estuviera de acuerdo una vez explicado el motivo, sin causarle ningún daño o alteración al cachorro.
RESULTADOS
En el presente estudio el 28.9% (13/45) de los cachorros analizados fueron positivos a parvovirus canino en la PCR. El 66.7% (30/45) de los perros muestreado presentaron diarrea, el 43.3% (13/30) de los perros con diarrea amplificaron el gen VP2 y el 100% (15/15) de los perros sin diarrea fueron negativos a parvovirus, (p<0.01)(Tabla 1).
Tabla 1 Asociación del PCR con variables de los caninos muestreados.
| Variables | Resultado PCR | Total | Sig | ||
|---|---|---|---|---|---|
| Positivo | Negativo | ||||
| Si Diarrea No Total | Si | 13 | 17 | 30 | p<0.01a |
| No | 0 | 15 | 15 | ||
| Total | 13 | 32 | 45 | ||
| Si Hematoquecia No Total | Si | 11 | 7 | 18 | p<0.01 a |
| No | 2 | 25 | 17 | ||
| Total | 13 | 32 | 45 | ||
| Si Antecedentes No de Vacunación* Total | Si | 7 | 11 | 18 | p>0.05 a |
| No | 4 | 15 | 19 | ||
| Total | 11 | 26 | 37 | ||
| Edad en meses | 2.70 | 5.05 | p<0.01 b | ||
Sig= Significancia;
*Solo se obtuvo información de 37 caninos
a:Según la prueba de Fisher; b:Según la prueba de T de Student
Las muestras que resultaron positivas para el fragmento de Vp2, provenían de animales con un promedio de edad de 2.7 meses, los animales en los que no se detectó parvovirus, tenían un promedio de edad de 5.05 meses (p<0.01) (Tabla 1).
Los perros positivos en PCR presentaron una media de 2.70 meses de edad, en cambio los negativos presentaron promedio de edad de 5.05 meses (p<0.01) (Tabla 1).
De los 45 perros muestreados, solo en 37 se obtuvo alguna información relacionada con la vacunación contra parvovirus. De estos, 18 propietarios afirmaron haber vacunado sus cachorros, y 19 no recibieron vacunación. No obstante, el 38.9% (7/18) de los perros vacunados, resultaron positivos, y el 21.0% (4/19) de los no vacunados resultaron infectados con parvovirus (p>0.05) (Tabla 1).
Basado en la secuencia nucleotídicas de la posición 426 del gen VP2, las cuatro muestras de campo secuenciadas fueron identificadas como genotipo CPV-2C y las dos cepas vacunales se identificaron como genotipo CPV-2A (Figura 1).
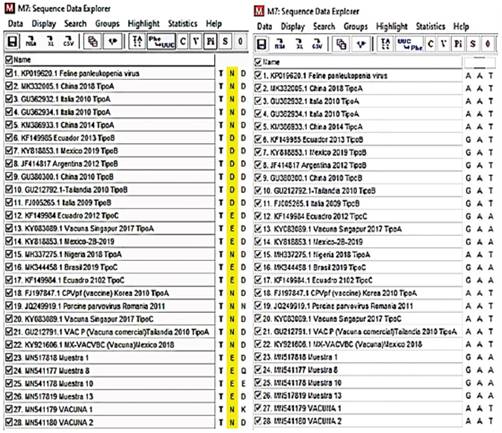
Figura 1.Genotipos de CPV según codón 426 de VP2. En la izquierda se muestran los aminoácidos Aparagina (N) para el genotipo A, Aspartato (D) para el genotipo B y Glutamato (E) para el genotipo C, en la derecha la secuencia de nucleotídicas.
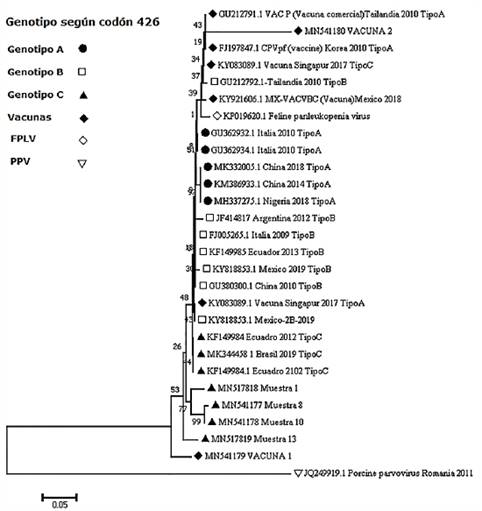
Figura 2 Árbol filogenético basado en la secuencia nucleotídicas del gen VP2 de parvovirus canino, se presentan las cuatro muestras de campo de Nicaragua (MN517818, MN541177, MN541178, MN517819) y las dos vacunas (MN541179, MN541180), comparadas con secuencias obtenidas del GenBank para parvovirus canino, Virus de la Panleucopenia Felina (FPLV) y Parvovirus Porcino (PPV).
La inferencia evolutiva de las secuencias nucleotídicas de cepas de campo alineadas mostró divergencia entre 7.7 y 8.9% respecto a la vacuna n°1 y entre 18.0 y 19.0% respecto a la vacuna n°2. La divergencia entre ambas vacunas fue de 17.7% y entre las cuatro muestras presentó valores entre 0.5 y 5.4%, mientras que entre las muestras de campo respecto a las secuencias de CPV encontradas en otros países (GenBank) fue desde 3.2% hasta 6.3%, mientras que respecto al FPLV se observó una divergencia entre 5.7 y 6.5% (Figura 2).
La historia evolutiva se infirió utilizando el método de Neighbor-Joining, se muestran junto a las ramas el porcentaje de árboles replicados en los que los taxones asociados agrupados en la prueba bootstrap (1000 repeticiones). Las distancias evolutivas se calcularon utilizando el método Maximum Composite Likelihood, el análisis involucró 28 secuencias de nucleótidos en un total de 383 posiciones en el conjunto de datos final.
DISCUSIÓN
Las variantes genotípicas presentes en las cuatro muestras secuenciadas fueron identificadas como genotipo CPV-2C según el codón 426 de gen VP2. Este resultado demuestra que en Nicaragua se observa un comportamiento similar al de otros países en los que se observa que CPV-2C es la variante más frecuente, coincidiendo con el estudio realizado en Argentina por Calderón et al 7, quienes encontraron un 91% de CPV-2C entre los años 2003 y 2010, además de haber sido el genotipo prevalente desde 2008, mientras que CPV2A y CPV2B representaron en el 3.6 y 5.4% de la población, respectivamente. Cabe mencionar que el haber encontrado CPV-2C en este estudio, no es excluyente de que se pueda encontrar las otras dos variantes genotípicas.
Se observó asociación de presencia de diarrea con la positividad en PCR, dado que en todos los perros con CPV presentaron diarrea y en ningún canino sin diarrea se encontró el virus, indicando que el síntoma diarreico está presente en todos los animales infectados, tal como se refleja en el estudio realizado por Zhao et al 14, quienes encontraron que todos los perros infectados experimentalmente presentaron síntomas de diarrea cuatro días post infección.
En el análisis de la asociación parvovirosis con la presencia de hematoquecia se observó que el 61.1% (11/18) de los perros con hematoquecia mostraron amplificación de la porción del gen VP2, en ausencia de hematoquecia el 7.2% (2/27) de caninos resultaron positivos. Estos resultados coinciden con lo descrito por Mauro et al 15, según sus registros, sólo en 7 de cada 10 casos con sospecha clínica, fueron positivos al virus, por tanto, se deberían de considerar otras etiologías como agentes causales.
Respecto a edad, se observó que la edad promedios de perros positivos fue menor que la edad de los negativos. En el estudio realizado por Nandi et al 1. las mayores tasas de infección se observan en cachorros mayores de 1.5 meses. Al igual que con otras enfermedades infecciosas de los caninos, los cachorros que nacen de perras inmunizadas, quedan protegidos durante la primera semana gracias al consumo de calostro rico en anticuerpos, pero luego los títulos disminuyen hasta alcanzar un período crítico en el que los anticuerpos maternos ya son insuficientes para proteger al cachorro 16.
La divergencia evolutiva observada en las vacunas con respeto a las cepas de campo lleva a realizar el planteamiento sobre la eficacia de las dos vacunas utilizadas en este estudio, en resultados obtenidos en este trabajo se encontró asociaciones de las sintomatologías de diarrea, hematoquecia con perros cuyas muestras presentaban CPV, los cuales tenían antecedentes de vacunación. Como se puede observar en el estudio realizado por Ohshima et al 17, los anticuerpos producidos por perros vacunados con la nueva cepa de vacuna CPV-2B reaccionaron más eficientemente a los aislamientos de CPV de campo recientes en comparación a los vacunados con CPV-2 convencional, por lo que, las mutaciones en la proteína VP2 pueden ocasionar fallos en la vacunación. La secuencia de la vacuna n°2 mostró alta similitud con la descrita para vacunas analizadas en Corea por Yoon et al 18, así como en México (ID KY921606), mientras que la vacuna n°1 mostró más similitud con las muestras de campo analizadas, sin embargo, se requiere de un estudio que evalué la estimulación de anticuerpos neutralizantes para los virus circulantes que determinar la efectividad de la vacunación














