INTRODUCCIÓN
Los ovinos, al igual que otros animales domésticos, no son originarios del continente Americano, arribaron desde la península Ibérica; primero en calidad de alimento por los navegantes y conquistadores, luego como pie de cría por los primeros colonos y religiosos; constituyéndose en la base racial del ganado lanar en América. A causa del reducido número de cabezas y de las condiciones ambientales, los animales se criaron sin un preciso esquema de selección, lo que produjo un mestizaje que perduró durante siglos, dando origen a las denominadas razas ovinas criollas que persisten hoy en día en forma de pequeños núcleos, generalmente en zonas marginales. Está demostrado que la difusión de las razas deslanadas en el continente americano se desarrolló desde las bases caribeñas. Las mayores características positivas del Criollo son su rusticidad, su adaptación a medios marginales, climas difíciles y su longevidad.
En Colombia, los animales que entraron por la costa del Caribe, probablemente por la Guajira, dieron origen al denominado Ovino Criollo Colombiano (OCC). Su amplia adaptación hace que fácilmente se encuentre desde las zonas áridas de la Guajira hasta los páramos húmedos de la zona Andina. La población ovina criolla colombiana está conformada por: Ovino Criollo de Lana (CL) de importancia económica para Boyacá, Cundinamarca, Nariño y Santander, la raza ovina sintética Mora Colombiana (MC) en los departamentos de Boyacá, Cundinamarca y Santander y el Ovino Criollo de Pelo o Africano (CP), que se encuentra principalmente en la Costa Atlántica, Llanos Orientales, Tolima, Valle del Cauca y Huila. En algunas zonas del país se reconocen tres variedades de CP: ovino criollo colombiano Sudán (CS) cuya capa va del amarillo al blanco, Etíope (CE) de color rojo cereza y Abisinio (CA) de color negro; esta denominación por variedades es tenida en cuenta principalmente por los productores del departamento de Córdoba y Valle del Cauca. El Instituto colombiano agropecuario (ICA) reporta que en Colombia existen 1.423.274 de animales. La producción en Colombia es de bajo uso de insumos y generalmente está relacionada con sistemas tradicionales y artesanales de producción tanto en el caso de ovinos de lana (OL) como en ovinos de pelo (OP) 1.
En el presente trabajo se evaluó la diversidad genética de los ovinos criollos colombianos, se estimaron las diferencias entre las variedades de ovinos criollos de pelo y se estudiaron las relaciones genéticas del criollo colombiano con ovinos de origen europeo y africano, mediante el uso de marcadores moleculares microsatélites.
MATERIALES Y MÉTODOS
Muestreo. Se tomaron muestras de sangre de 169 ovinos CP y 30 ovinos CL, en 40 fincas (Tabla1), según criterio de los productores se muestrearon animales no emparentados, en tubos vacutainer con EDTA se tomaron 10 ml de sangre a cada animal mediante punción en la vena yugular. El CP aportó el mayor tamaño de muestra, debido a que este es el primer trabajo en el país, que se realiza para conocer su diversidad genética. También se muestrearon ovinos criollos cruzados (Mes), con Pelibuey de origen mexicano (n=16), con Hampshire (n=2), con Katahdin (n = 5) y con Romney Marsh (n=2). Se utilizaron como muestras de referencia, razas ovinas foráneas en producción de carne (FC): Katahdin y Pelibuey mexicano y en producción de lana (FL), Corriedale y Hampshire. Se incluyeron muestras de Merino Español (n=5), Merino Precoz de origen francés (n=5), Merino Fleischschaf de origen alemán(n=5), Segureño (n=5) de origen español y Uda (n = 10) de origen nigeriano.
Tabla1. Número de muestras por grupo genético, abreviatura (Abrev.), tamaño de muestra (N) y localidad de los ovinos utilizados.
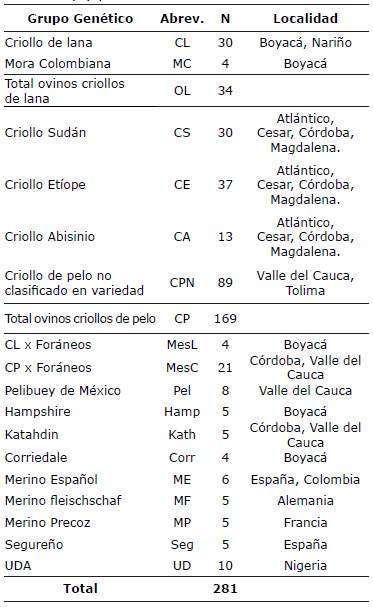
En el departamento de Córdoba los CP se clasificaron por variedades Sudán (CS), Etíope (CE) y Abisinio (CA).
Marcadores Microsatélites. A continuación los 15 microsatélites utilizados y su posición en el genoma: OarCP34(3), McM527(5), D5S2(5), RM006(5), OarAE129(5), ETH225(9), INRA35(12), TGLA53(12), INRA63(14), TGLA126(16), BM8125(17), OarFCB48(17), OarFCB304(19), OarCP20(21), BM6526(26), de un panel de 32 utilizados en el proyecto Biovis, elegido entre los propuestos por la FAO (Food and Agricultural Organization) e ISAG (International Society for Animal Genetics)2.
Extracción, PCR y Amplificación. La caracterización molecular se realizó en los laboratorios de Genética Animal y de Biología Molecular de la Universidad Nacional de Colombia, sede Palmira. Para la extracción de ADN se utilizó el protocolo Salting-Out (1988). Para la PCR se utilizaron 2.0 X de Tampón Taq; 4 mM de MgCl2; 0.4 mM de DNTPs; 0.2 mg/ml de BSA; 0.2 uM de cebadores; 0.75 U de Taq y 40 ng de ADN para un volumen final de 25 ul. El perfil térmico de PCR fue: denaturación inicial 5 min a 95°C, seguido de 35 ciclos de 30 segundos a 95°C, 30 segundos a 58°C, 90 segundos a 72°C y una extensión final a 72°C por 15 minutos. Para la visualización de los productos amplificados se utilizaron geles de poliacrilamida al 6%(29:1 acrilamida - bisacrilamida), en cámaras de secuenciación Fisher de 35 x 45 cm y marcador 10 bp.
Análisis Estadístico. El número de alelos (NA), el número promedio de alelos por locus (NPA), los valores de heterocigosidad observada, esperada y FIS, la prueba de desviación del equilibrio de Hardy-Weimberg (EHW), se estimaron mediante el software ARLEQUIN 3.11; el contenido de información polimórfica (PIC), con el programa Microsatellite Toolkit software para Excel; la riqueza alélica, mediante el programa FSTAT, ver. 2.9.3. Se realizó análisis de varianza molecular (AMOVA) con el programa ARLEQUIN ver 3.11.
Para estimar las distancias y relaciones filogenéticas entre grupos se utilizó la mínima distancia de Nei, mediante TFPGA® y el FST por pares de poblaciones con ARLEQUIN ver 3.11. Se construyeron dos dendogramas, el primero con todos los grupos genéticos y el segundo con los ocho departamentos muestreados utilizando únicamente los datos de las razas criollas, mediante el algoritmo UPGMA (Unweighted Pair Group Method with Arithmetic mean), mediante TFPGA®.
Se exploró la estructura de las poblaciones con el método del análisis de cluster, basado en modelos implementados en el programa STRUCTURE v.2.3.1, que calcula un valor de probabilidad para un número de K poblaciones (o cluster) pre-determinado, y asigna la parte del genoma de cada individuo que deriva de cada cluster. La estructura poblacional fue probada desde K = 1 hasta K = 16 con 15 marcadores microsatélites para analizar las relaciones entre ovinos criollos colombianos y razas foráneas. Cuando se incluyó la raza nigeriana UDA se probó con K=1 hasta K= 17 con siete marcadores microsatélites.
El número total de interacciones fue de 500000 después de 100000 burning-in con tres replicas para cada K. Se asumió que las frecuencias alélicas de las poblaciones actuales estaban correlacionadas y que podían haberse originado a partir de más de una población ancestral. El valor más probable de K fue determinado según el método de Evanno implementado en el programa Structure Harvester v0.6.94.
RESULTADOS
Para los 15 marcadores, el número de alelos osciló entre cinco y nueve, siendo los marcadores ETH225, INRA63, RM006 y OarFCB48 los que presentaron mayor número de alelos. Los valores de PIC fueron muy informativos, puesto que oscilaron entre 0.62 (OarFCB304) y 0.84 (INRA63, BM6526); el promedio para todos los marcadores fue de 0.76. En general, para cada marcador la heterocigosidad esperada (He) fue menor que la heterocigosidad observada (Ho). La Ho en todos los locis fue alta con excepción de OarCP34 (0.40) y OarFCB304 (0.44).
La diversidad genética encontrada por marcador fue alta (0.78±0.01). El OarFCB304 fue el único marcador que no se encontró en equilibrio de HW. El índice de fijación (FIS) no presentó valores significativos para la mayoría de los marcadores; únicamente los sistemas OarCP34 y OarFCB304 presentaron una reducción en el número de heterocigotos (p<0.001)(Tabla 2).
Tabla 2.|Número de alelos (NA), riqueza alélica (RA), contenido de información polimórfica (PIC), heterocigosidad observada (Ho), heterocigosidad esperada (He) e índice de fijación (FIS) para 15 marcadores.
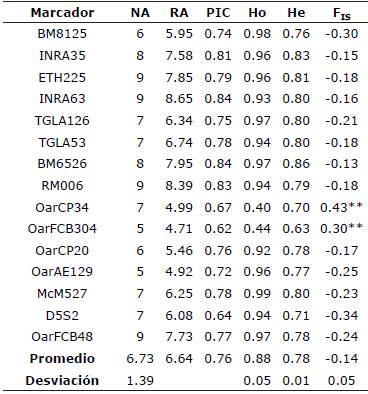
**p<0.001
En la tabla 3 se observa la estadística descriptiva entre los ovinos criollos colombianos de lana y pelo. Se hallaron 93 alelos en CL y 109 alelos en CP, este último presentó un promedio más alto en el número de alelos (7.27±1.39) con respecto a CL (6.20±1.48), lo cual puede estar afectado por el tamaño de muestra. Para ambos grupos genéticos, la He fue menor que la Ho. Los grupos genéticos CP y CL presentaron valores similares tanto de Ho (0.90±0.18 y 0.88±0,21) como de He (0.79±0.06 y 0.78±0.07) respectivamente. Se halló alta diversidad genética, superior al 75%.
Tabla 3 Estadística descriptiva para ovinos criollos de lana y pelo, variedades de pelo y mestizos: tamaño de muestra (N), número de alelos (NA), número de alelos totales por marcador (NTA), número promedio de alelos (NPA), número efectivo de alelos (Ne), heterocigosidad observada (Ho), heterocigosidad esperada (He) y (FIS).
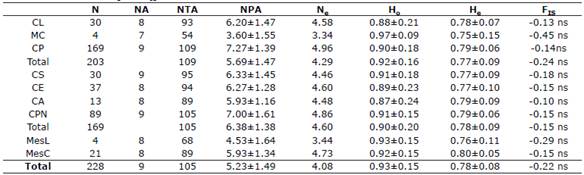
ns: no significativo. FST= 0.02**; FIS= -0.16; FIT= -0.14
En otro análisis donde se separaron por variedad los ovinos de pelo, en estos se encontró un máximo de 105 alelos. El grupo CPN, obtuvo un promedio de alelos de 7.00±1.61, siendo el más alto de todos los grupos de ovinos criollos; además, las poblaciones CS (6.33), CE (6.27) y CL (6.2), presentaron el número promedio de alelos más alto con respecto a los grupos MC, CA, MesL y MesC (5.93).
Se encontró un alelo privado en la población CA (124bp) en el marcador OarCP34 y en la población CPN (167bp) en el marcador McM527, con una frecuencia de 5.9% y 1.7% respectivamente.
La Ho presentó valores más altos que la He en todas las poblaciones ovinas (Tabla 3). La diversidad genética (He) en todas las poblaciones criollas fue alta, superior al 75%. Los valores de He oscilaron entre 0.75±0.15 y 0.80±0.05. Las variedades CS (0.77±0.09), CE (0.77±0.010) y CA (0.79±0.09) presentaron altos valores de diversidad genética, al igual que el grupo CPN (0.79±0.06).
Los valores de FIS (Tabla 3), no fueron significativos en ninguno de los grupos, mientras que el valor de FST encontrado fue de 0.02 siendo altamente significativo, indicando que existe poca diferenciación genética entre los grupos.
Los FST entre pares de poblaciones (Tabla 4), fueron bajos pero significativos en casi todas las combinaciones, excepto entre MC con CL y MesL; CA con CE y CPN con MesC. El grupo CL se diferenció de las poblaciones criollas de pelo (CS, CE, CA, CPN) puesto que todos los FST presentaron valores altamente significativos (p<0.001).
Tabla 4 Estimaciones de diferenciación genética para las poblaciones criollo de lana (CL), Mora Colombiana (MC), criollo de pelo Sudán (CS), criollo de pelo Etíope (CE), criollo de pelo Abisinio (CA), criollo de pelo no clasificado (CPN), criollo mestizo lana (MesL) y criollo mestizo carne (MesC).
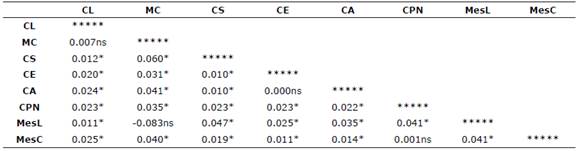
**(p<0.001); **(p<0.05); ns: no significativo
En la figura 1 se presenta el dendograma realizado con la mínima distancia de Nei, el cual incluye todos los grupos genéticos criollos, mestizos y foráneos muestreados. Se puede observar que los ovinos criollos colombianos se separan genéticamente de las razas Hampshire, Corridale y Mora Colombiana. Las razas ME, MP, MF y Seg, quedaron en un grupo diferente a las razas criollas. Las razas criollas colombianas y las razas Pelibuey y Katahdin se agruparon, lo anterior puede deberse a que tienen en común el mismo origen. Al observar a los ovinos criollos de pelo, los CPN y los MesC se ubicaron en un mismo grupo, mientras que en otra agrupación se ubicaron las variedades de pelo CE, CA y CS. Sugiriendo diferencias genéticas entre los diferentes biotipos de pelo.
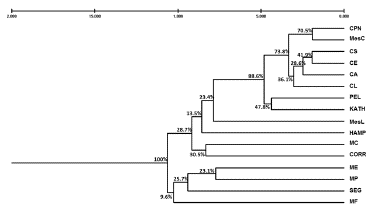
Figura 1 Dendograma realizado con la mínima distancia de Nei, mediante el método de clasificación UPGMA, utilizando los datos de 15 marcadores microsatélites en ovinos criollos colombianos y razas foráneas.
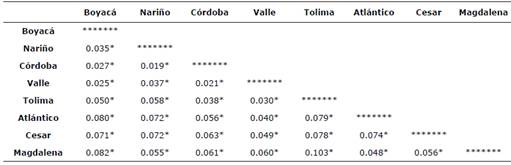
Para conocer la estructura genética por departamentos de los grupos genéticos CL fue: 0.039 (p<0.0001), que indica poca diferenciación genética entre los animales muestreados por departamento (Tabla 5). Se encontró estructura poblacional en todas las combinaciones realizadas, la mayor estructura genética se encontró entre los departamentos de Tolima y Magdalena (0.103), (diferenciación genética moderada).
En general, en casi todas las combinaciones de la tabla 5, los valores de estructura genética fueron pequeños, lo anterior sugiere que existe poca diferenciación por departamentos a pesar de la diferencia en clima y fenotipo ovino (CL y CP). Iguales resultados se observaron en el dendograma (Figura 2), donde se agruparon tanto las razas de lana como las de pelo en un mismo grupo.
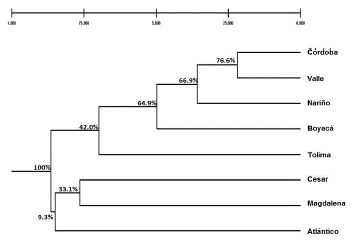
Figura 2 Dendograma realizado con la mínima distancia de Nei, mediante el método de clasificación UPGMA utilizando15 marcadores microsatélites en ocho departamentos colombianos.
Se realizó un análisis de la estructura de la población, mediante el algoritmo bayesiano. En la figura 3, cuando se asume la existencia de dos poblaciones ancestrales (K=2), se puede apreciar un ancestro común para todos los ovinos criollos CL, CS, CE, CA también para las razas Pel, Hamp, Corr y para los MesL, todos los anteriores se diferenciaron de las demás agrupaciones.
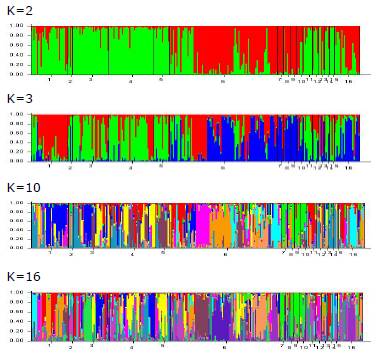
Figura 3 Representación gráfica de los resultados del análisis de la estructura genética de seis poblaciones ovinas criollas colombianas comparadas con razas foráneas y mestizos (K= 2 hasta K= 16), usando 15 marcadores microsatélites.1: CL; 2: MC; 3: CS; 4: CE; 5: CA; 6: CPN; 7: MF; 8: ME; 9: MP; 10: Seg; 11: Pel; 12: Hamp; 13: Kath; 14: Corr; 15: MesL; 16: MesC.
Cuando se asume que existen tres poblaciones ancestrales (K=3) se puede encontrar un ancestro común para todos los ovinos criollos, lo que concuerda con el origen mixto de estas poblaciones; se diferencian los CL, CS, CE, CA y Pel (color verde), compartiendo una mayor parecido en su estructura el CPN y el MesC con las razas foráneas MF, ME, MP, Seg (color azul), se puede apreciar que principalmente el CPN y el MesC poseen una mezcla tanto de razas foráneas como criollas, al igual que los MesL, Hamp, Kath y Corr. Cuando K=10 y K=16 se observa que en general todas las poblaciones tanto criollas como foráneas muestreadas en el país presentan una gran composición mixta.
DISCUSIÓN
El número promedio de alelos (NPA) encontrados en los ovinos CL y CP fue de 6.20 y 7.27 respectivamente, lo anterior pudo estar influenciado por el tamaño de muestra de los grupos. Los valores encontrados en el promedio de alelos en CL también se acerca a lo encontrado en razas criollas mexicanas: Chiapas (NPA= 6.10), Chamula (NPA= 6.90) y Café (NPA= 7.80), 3 y a lo reportado en el proyecto de biodiversidad ovina iberoamericana - Biovis (NPA= 6.90). Igualmente, el promedio de alelos encontrado en este trabajo es similar a los criollos de Paraguay 4, México 5 y Estados Unidos 6.
La He en ambos grupos fue similar y reveló altos niveles de variabilidad tanto para CL (0.78) como para CP (0.79).
Los altos valores de He encontrados en estos dos grupos genéticos colombianos coinciden con los hallados con microsatélites en ovinos criollos de América: en México con las razas ovinas de Chiapas 3, en la región oriental de Paraguay con ovejas de los humedales 4, en la raza ovina Chilota de Chile 7, en ovinos criollos y mejorados de los Estados Unidos 6.
Los altos niveles de variabilidad encontrados, podrían explicarse por su origen diverso puesto que es probable que los ovinos hubiesen seguido la misma ruta de entrada al país que los bovinos. De acuerdo a diferentes autores, los bovinos entraron por tres rutas: desde la costa del Caribe, desde Ecuador y también provenían de Nicaragua, por la vía Panamá-Buenaventura.
De acuerdo con Delgado et al 8, durante el siglo XV se realizaron varias introducciones ovinas, por lo tanto las poblaciones Iberoamericanas se fundaron sobre una base de animales muy diversos, importados de España y Portugal; posteriormente, los efectos de la selección natural y artificial, la deriva genética y las continuas migraciones de genotipos procedentes de la propia Península Ibérica, así como de otros países europeos, de África y Asia, constituyeron una alta riqueza genética. Por otro lado, a lo largo de la historia en los ovinos criollos se ha ejercido baja presión de selección.
El valor de FIS que estima el exceso o déficit de heterocigotos no fue significativo, presentó valores negativos tanto en CL (-0.13), como en CP (-0.14), sugiriendo presencia de exogamia en las producciones ovinas criollas del país. La alta diversidad genética y ausencia de endogamia intrapoblacional puede obedecer principalmente al uso constante de cruzamientos indiscriminados entre ovinos criollos y razas foráneas lo cual fue observado en campo.
Es importante resaltar que los criollos de lana (CL) y pelo (CP) presentaron muy poca diferenciación genética (FST= 0.014 (p<0.0001)) debido probablemente a la introducción de ovejas canarias en América. Las Islas Canarias jugaron un papel muy importante en la distribución de recursos genéticos animales posteriores al descubrimiento de América, al convertirse en cruce de rutas para los navegantes españoles y de otros países europeos con sus colonias de ultramar y existen múltiples evidencias históricas que confirman la participación de la oveja canaria en la primera colonización de América. Según Delgado et al 8, la oveja Canaria podría tener su origen en la hibridación entre los ovinos de pelo prehispánicos y los ovinos de lana introducidos en Canarias por los españoles procedentes de los puertos del sur de la península Ibérica o de la costa africana.
La cercanía genética entre CP y CL se observó también en el análisis por departamentos donde se encontró que Boyacá y Nariño que corresponden a trópico alto se agruparon con Valle del Cauca, Tolima y Córdoba (trópico bajo) independientemente de la ubicación geográfica y del fenotipo de ovinos (lana o pelo), mientras que los departamentos de la Costa norte de Colombia (Atlántico, Cesar y Magdalena), conformaron otro grupo solo de ovejas de pelo.
El número promedio de alelos encontrados en los ovinos criollos de pelo CS (NPA= 6.33), CE (NPA= 6.27) y CA (NPA= 5.93) fue similar, a pesar de la diferencia en el tamaño de muestra utilizado en cada variedad: 30, 37 y 13 respectivamente; fueron menores a los reportados en la oveja de pelo cubana (NPA= 7.2), 9, similares a la raza brasilera Morada Nova (NPA= 5.09), 10. Las tres variedades de ovinos de pelo mostraron altos valores de heterocigosidad, mayores al 75%, indicando alta diversidad genética. No se encontraron valores significativos para el índice de fijación FIS en los grupos CS, CE y CA.
La diferenciación fenotípica de las variedades criollas de pelo constituye una dificultad, pues los ejemplares CS, CE, CA se encuentran muy cruzados y sólo en unos pocos rebaños se pueden encontrar ejemplares puros, no obstante cuando se observa su color, tipo de cabeza, orejas y otras características fisionómicas se puede distinguir una variedad de otra.
Lo anterior es corroborado con los resultados encontrados en este trabajo, donde la estructura poblacional fue altamente significativa para estas tres variedades (FST=0.02), indicando que existe diferenciación genética según la escala de valores propuestos por Wright 11. La comparación CS con CE y CA difiere significativamente, pero CE y CA son similares. Por lo tanto, se puede concluir que únicamente en el país se encuentran dos biotipos de ovinos criollos de pelo el CE y CS mientras que la variedad CA posiblemente sea la misma CE. Las variedades CS y CE han sido reportadas por diferentes autores 1.
Es importante aclarar que los nombres de las variedades de ovinos de pelo colombianos Etíope, Sudán y Abisinio no tienen relación con los países de origen, no se encontró en la literatura una razón que lo justifique, puesto que estos países pertenecen al África oriental. Con excepción de Venezuela tampoco se halló otro país que utilice esta denominación. En 1940 llegó un núcleo de oveja de pelo, procedente de Abisinia al departamento del Tolima; es probable que estos nombres se hayan originado a partir de este evento.
Los CPN son el reflejo de la producción ovina del país, pues representan en este trabajo el mayor tamaño de muestra de diferentes departamentos (Magdalena, Atlántico, Cesar, Valle del Cauca y Tolima). Las distancias genéticas agrupan a los criollos de pelo no clasificados (CPN) con los mestizos (Mes), lo cual es confirmado en el análisis bayesiano y en los altos valores de diversidad (0.79±0.06). Este hallazgo genera una alerta, debido a que es una población vulnerable por la constante hibridación a la que está expuesta, lo que puede conllevar en un futuro no muy lejano la absorción por razas foráneas, teniendo en cuenta el bajo intervalo generacional.
Las variedades criollas de pelo CS, CE y CA formaron un grupo diferente que se aleja de los CPN y MesC, lo que sugiere que los primeros se mantienen relativamente libres de introgresión, con respecto a los criollos de pelo del resto del país.
Los ovinos criollos representan un recurso importante para el pequeño agricultor. Su alto valor como recurso genético, se refleja en el aumento constante de la población que se ha desarrollado sin ningún estímulo o apoyo de asociaciones u organizaciones gubernamentales.
Se encontró según lo observado en el análisis bayesiano y en el dendograma que el grupo de todos los ovinos criollos colombianos tanto de lana como de pelo (CL, CS, CE, CA, CPN) y también el grupo de razas foráneas muestreadas en Colombia (Corr, Hamp, Pel, Kath) presentan: 1. Similitud genética entre ellos lo que sugiere introgresión en los grupos genéticos criollos, presentándose en mayor proporción en los CPN como se explicó anteriormente. 2. Los ovinos criollos presentan diferencias con las razas foráneas ME, MF, MP, Seg y UD, lo que sugiere que los criollos se han separado genéticamente de sus ancestros.
Al observar los resultados en los ovinos criollos de pelo colombiano, es importante reconocer la diferencia encontrada entre las variedades de pelo (CS, CE y CA) y los CPN, los primeros presentan una clara tendencia a ser un grupo homogéneo y diferente del segundo. Las variedades CS, CE y CA presentan menor grado de introgresión con otras razas al compararse con el grupo CPN, este último grupo se acerca notablemente al grupo de ovinos criollos mestizos (Mes) y se observa claramente la introgresión que presenta de otras razas en el análisis bayesiano tanto en el FST, y en la mínima distancia de Nei. Los CPN son ovinos que pertenecen a los departamentos de Atlántico, Cesar, Magdalena, Tolima y Valle del Cauca y son el reflejo de la población ovina del país, que en su mayoría presentan cruces, únicamente los animales muestreados en Córdoba presentaron poca introgresión.
En general, la información que existe en las regiones tropicales del mundo acerca del recurso genético ovino criollo y su importancia es limitado; teniendo en cuenta que los OCC han tenido un proceso adaptativo al ambiente tropical del país 12,13,14; es necesario fomentar su reconocimiento y producción: las asociaciones ovinas, la academia y el estado deben incentivar en el productor el conocimiento y la conservación de estos grupos genéticos, puesto que su proceso de adaptación mayor a 500 años, les ha permitido sobrevivir al clima tropical convirtiéndolos en una fuente de valiosa riqueza y diversidad genética que se debe seguir explorando.
El conjunto de los 15 marcadores microsatélites utilizados brindó información que resultó de importancia para el análisis de diversidad genética y estructura poblacional de los ovinos criollos colombianos y su relación con algunas razas foráneas. Aporta un punto de partida de futuras acciones encaminadas a preservar la raza como control genealógico, asignación de individuos a poblaciones, trazabilidad de productos etc. Es uno de los primeros trabajos en Diversidad Genética de Ovinos Criollos de Pelo Colombianos del país. Se encontró alta diversidad genética debido a hibridaciones constantes de las razas criollas con otras razas, por lo tanto los biotipos criollos son vulnerables a la absorción por razas foráneas, esto se debe principalmente, a que en el país se utilizan razas introducidas, razas criollas y cruzamientos alternos con el fin de fortalecer el vigor híbrido para la producción 15. Se encontró alta cercanía genética entre los ovinos criollos de lana y pelo, debido probablemente a un origen común en las islas Canarias y al cruzamiento entre ambos biotipos en épocas recientes.
En el grupo de criollos de pelo no clasificados en variedades (CPN) se encontró alto grado de introgresión lo que conlleva a la pérdida de identidad genética. Los ovinos de pelo del departamento de Córdoba CS, CE y CA se diferencian del grupo CPN porque poseen identidad genética, que los distanció de los CPN. En los ovinos criollos de pelo del departamento de Córdoba, el CS se diferenció del CE y CA, mientras que estos dos últimos genéticamente fueron muy similares. Los ovinos de pelo del departamento de Córdoba poseen identidad genética propia manteniéndose relativamente más puros que los del resto del país. Se encontraron diferencias genéticas entre los ovinos criollos y las razas ME, MP, MF, Seg y Uda.