Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Médica de Risaralda
Print version ISSN 0122-0667
Revista médica Risaralda vol.23 no.2 Pereira July/Dec. 2017
Artículo original
Perfil de resistencia antimicrobiana en bacilos Gram negativos no fermentadores aislados en fuentes hídricas
José Alejandro Pulido Beltrán1. Rodríguez Ximena Andrea1. Méndez Iván Alberto1, ivan.mendez@ummilitar.edu.co
1 Grupo de epidemiología y salud colectiva, Facultad de Medicina y Ciencias de la Salud. Universidad Militar Nueva Granada
Fecha de envío: 28/04/2017
Fecha de correcciones 23/05/2017
Fecha de aceptación 30/07/2017
Fecha de publicación 31/08/2017
Resumen
La amplia distribución de los bacilos gram negativos no fermentadores en medios ambientales como el agua y especies vegetales cobra importancia al ser reconocidos como agentes causales de enfermedades en pacientes inmunocomprometidos, de allí la relevancia del porque debemos conocer la prevalencia y perfil de susceptibilidad de estos microorganismos en ambientes no hospitalarios. Materiales y Métodos: Estudio transversal, realizado en muestras hídricas de fuentes naturales y artificiales de almacenamiento para el consumo humano en la ciudad de Bogotá y municipios aledaños. La identificación se realizó a través de pruebas IMVIC y el perfil de resistencia a través del método de kirby bauer o E-TEST®. Resultados: Se obtuvieron 42 muestras, 7 (16,6%) con aislamientos de interés: 3 (60%) Pseudomonas spp, 2 (20%) Acinetobacter spp, 1 (10%) Sphingomonas paucimobilis y 1 (10%) Pantoea spp. El 70% presento resistencia a la ceftriaxona, el 30% a cefoxitina, 20% a gentamicina, 10% a ciprofloxacina y 10% a piperacilina-tazobactam. No se presentó resistencia a imipenem. Conclusión: 5 de 7 aislamientos revelaron un BGNNF de importancia en infección en humanos, siendo importante la resistencia encontrada a la ceftriaxona.
Palabras clave: Pseudomonas; Acinetobacter, Resistencia antimicrobiana.
Copyright © Facultad de Ciencias de la Salud de la Universidad Tecnológica de Pereira. 1995-2017. Todos los derechos reservados ®
Antimicrobial resistance profile in non-fermenting gram negative bacilli isolated in water resources
Abstract
The wide distribution of non-fermenting gram negative bacilli in environmental media such as water and plants becomes important as they are recognized a cause of diseases in immunocompromised patients, that’s the reason why we should to know the prevalence and the susceptibility profile of these microorganisms in non-hospital environments. Materials and Methods: Cross-sectional study done with samples of natural and artificial water storage for human consumption in the city of Bogotá and surrounding municipalities. The identification was made through IMVIC tests and the resistance profile through the kirby bauer or E-TEST® method. Results: 42 samples were obtained, 7 (16.6%) with isolates of interest: 3 (60%) Pseudomonas spp, 2 (20%) Acinetobacter spp, 1 (10%) Sphingomonas paucimobilis and 1 (10%) Pantoea spp. The 70% had resistance to ceftriaxone, 30% to cefoxitin, 20% to gentamicin, 10% to ciprofloxacin and 10% to piperacillin-tazobactam. No resistance to imipenem was shown. Conclusion: 5 of 7 isolates revealed a BGNNF of importance in infection in humans, with an important resistance to ceftriaxone.
Key words: Pseudomonas; Acinetobacter, Antimicrobial resistance.
Copyright © Facultad de Ciencias de la Salud de la Universidad Tecnológica de Pereira. 1995-2017. All rights reserved ®.
Introducción
Los bacilos gram negativos no fermentadores (BGNNF) son un grupo complejo y amplio de microorganismos aerobios, no esporulados, incapaces de fermentar hidratos de carbono, ubicuos del medio ambiente, siendo fuentes comunes de aislamiento el suelo, las plantas y el agua [1-3], aunque pueden también encontrarse distribuidos ampliamente en el ambiente hospitalario tanto en instrumentos como humidificadores, ventiladores mecánicos, colchones e incluso en la piel de los trabajadores sanitarios [4], representando aproximadamente el 15% del total de los aislamientos provenientes de muestras clínicas [5].
De las 120 especies reconocidas hasta el momento, sobresalen algunas como Pseudomonas aeruginosa, Acinetobacter baumannii, Burkholderia cepacia, Stenotrophomonas maltophilia, Ralstonia pickettii y Sphingomonas paucimobilis las cuales son identificadas como causantes de infecciones en pacientes con enfermedades significativas de base e inmunocomprometidos especialmente (6,7), además de ser causantes de brotes epidemiológicos nosocomiales gracias a su gran adaptabilidad en ambientes hospitalarios debido a su versatilidad nutricional, capacidad para diseminarse desde reservorios exógenos o endógenos, resistencia intrínseca a múltiples antimicrobianos de gran espectro y su facilidad para adquirir resistencia a diferentes antimicrobianos [8-9].
En los últimos 10 años, ha ido en aumento la incidencia de infecciones por este tipo de microorganismos, con perfiles que evidencian multiresistencia a diversos antibióticos, así como el aumento en las tasas de morbimortalidad, especialmente en aislamientos de Pseudomonas aeruginosa y Acinetobacter baumannii [10-13] que han venido tomando importancia por ser los principales causantes de enfermedades infecciosas severas que incluso pueden llevar a la muerte a las personas que lo padecen, razón por la cual es de gran ayuda identificar perfiles de resistencia de los mismos.
Actualmente en Colombia son insuficientes los datos que se pueden encontrar acerca del aislamiento de estos microorganismos en el medio ambiente donde el ser humano puede tener contacto en cualquier momento de su vida, por lo cual, el objetivo del presente artículo es dar a conocer la prevalencia de bacilos gram negativos no fermentadores en fuentes hídricas naturales y artificiales y su respectivo perfil de resistencia antimicrobiano.
Materiales y métodos
Estudio transversal. Se realizó un muestreo no probabilístico donde se recolectaron 42 muestras provenientes de piscinas, fuentes hídricas naturales y tanques de almacenamiento de agua, siendo estos últimos el número predominante de muestras, en Bogotá y municipios cercanos. Se consideraron como criterios de inclusión: 1. Muestras provenientes de tanques de almacenamiento de agua sin mantenimiento previo de 6 meses, 2. Muestras recolectadas de piscinas privadas o cuyo carácter no represente estándares estrictos de mantenimiento, 3. Muestras recolectadas en lagos o ríos que tengan condiciones favorables para el aislamiento de este tipo de microorganismos. Como criterios de exclusión se consideraron: 1. Muestras provenientes de recipientes que almacenan agua en conjuntos residenciales dado al constante mantenimiento al cual son sometidos lo cual dificulta el aislamiento de microorganismos. Cada muestra comprendió un total de 50 mL, almacenados en tubos Falcon y llevados al laboratorio para su procesamiento.
De las muestras recolectadas, se transfirió 1 mL, por dispersión en agar sangre y MacConkey, los cuales se incubaron por 48 horas a 37°C. Posteriormente se realizó tinción de gram a las colonias que crecieron en los agares nutritivos, con el fin de identificar su morfología. Aquellas identificadas como bacilos gram negativos se les realizaron pruebas bioquímicas tales como: fermentación de azúcar (Triple Sugar Iron), oxidasa, motilidad (Sulfuro Indol Motilidad), ureasa, citrato, Voges Proskauer (VP) y rojo metilo (RM), las cuales se incubaron a 37°C por 24 horas. Luego, cada prueba se interpretó para realizar la identificación de especies bacterianas.
La sensibilidad a antibióticos se realizó utilizando el método de Kirby-Bauer. Este proceso incluyo la toma de muestras en los aislamientos positivos para bacilos gram negativos no fermentadores, los cuales fueron inoculados en agar Mueller-Hinton e incubados 24 horas a 37°C. Se utilizaron los siguientes antibióticos: ciprofloxacina, cefoxitina, piperacilina-tazobactam, ceftriaxona y gentamicina. Además, se analizó la sensibilidad a imipenem mediante E-TEST®. Los procedimientos se realizaron según los lineamientos del Comité Nacional para Estándares de Laboratorio Clínico (NCCLS por sus siglas en inglés) [14].
Resultados
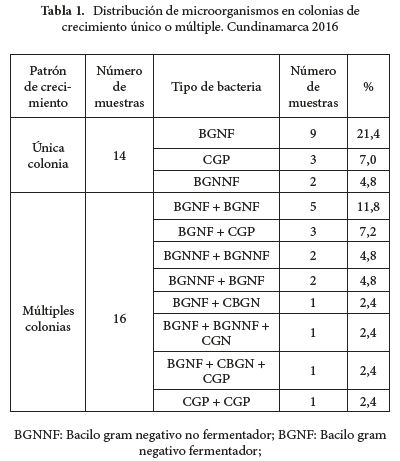
Se recolectaron un total de 42 muestras, de las cuales en 12 muestras no se evidenció crecimiento de microorganismos. En 30 muestras se observó crecimiento de microorganismos de la siguiente manera: 8 muestras (9 colonias) cocos gram positivos, 1 muestra (1 colonia) diplococos gram negativos, 2 muestras (2 colonias) cocobacilos gram negativos, 22 muestras (30 colonias) bacilos gram negativos termentadores y 7 muestras (10 colonias) bacilos gram negativos no termentadores. No se obtuvieron resultados sobre crecimiento de bacilos gram positivos (Tabla 1). Lo anterior teniendo en cuenta que 16 muestras tuvieron crecimiento de múltiples colonias.
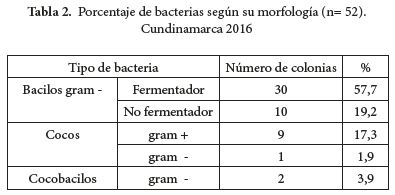
El microorganismo más prevalente en las fuentes hídricas son los bacilos gram negativos fermentadores (57.7%), seguidos por los BGNNF (19.2%) y los cocos gram positivos (17.3%), en menor proporción se encontró cocobacilos gram negativos (3.9%) y cocos gram negativos (1.9%) (Tabla 2).
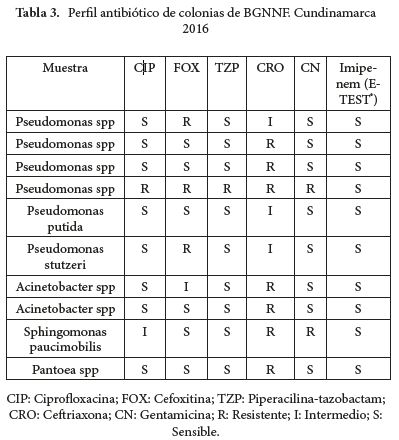
El microorganismo que se aisló con mayor frecuencia en el grupo de los BGNNF fue Pseudomonas spp (n=6) ocupando un 60% del total de los mismos, en menor proporción Acinetobacter spp (n=2, 20%) y Sphingomonas paucimobilis (n=1) y Pantoea spp (n=1) con igual porcentaje (10%). La fuente con mayor número de aislamientos fueron los tanques de agua que surten del líquido a las viviendas.
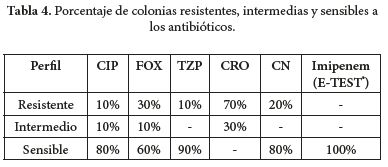
La mayoría de las colonias (60%) fueron sensibles a cefoxitina, 30% resistentes y 10% intermedio. Se observó además que la gentamicina y la piperacilina - tazobactam tienen poca resistencia en las colonias identificadas, solo el 10% presentó resistencia. Contrario a lo anteriormente mencionado, de las colonias aisladas ninguna fue sensible a ceftriaxona, siendo así un 70% resistente al antimicrobiano y un 30% presentó un perfil de resistencia intermedio (Tabla 3).
El 80% de las colonias no mostraron resistencia alguna a la ciprofloxacina, 10% presentaron un perfil de resistencia intermedio y el otro 10% fue resistente. Finalmente ninguna de las colonias aisladas fue resistente al imipenem estudiado mediante E-TEST® (Tabla 4).
Una de las colonias estudiadas (Pseudomonas spp) que se obtuvo de una alberca de casa presentó resistencia a todos los antibióticos utilizados en el estudio excepto al imipenem (E-TEST®). Además, se evidenció que entre las colonias que presentaron perfiles de resistencia a varios de los antibióticos utilizados se encuentran más frecuentemente Pseudomonas spp y Sphingomonas paucimobilis.
Discusión
Según el informe nacional de la calidad del agua para el consumo humano año 2013, reporta que el 41.2% de la población total del país se encuentra catalogada en el grupo “sin riesgo para la salud” respecto al Índice de Riesgo de la Calidad del Agua para Consumo Humano que evalúa el grado de riesgo de ocurrencia de enfermedades relacionadas con el no cumplimiento de las características físicas, químicas y microbiológicas del agua, el 29.3% se cataloga en el grupo de “riesgo bajo”, 13.6% en “riesgo medio”, 12.5% en “riesgo alto” y el 1.4% se categoriza en el grupo de población la cual recibe agua “inviable sanitariamente”. Aun así, si se compara la calidad de agua de ciudades o municipios respecto a su número de habitantes, es notoria la diferencia en el riesgo al cual se exponen, por ejemplo, poblaciones con más de 100.000 habitantes, se exponen a un riesgo alto en un 5%, comparado con el 29.8% en poblaciones con menos de 30.000 habitantes, adjudicado a los altos índices de población rural en las poblaciones con menor cantidad de habitantes. Bogotá D.C. y Cundinamarca, sitios de los cuales provienen las muestras recolectadas en este estudio, se encuentran categorizados en el grupo “Sin Riesgo”, es decir, entre las poblaciones con la mejor calidad de agua [15]. Ahora pues, a nivel mundial, se conocen diferentes reportes de infecciones causadas por microorganismos presentes en agua potable, los cuales llegan a ser numerosos, debido a la contaminación presente de fuentes hídricas tanto naturales como artificiales por microorganismos coliformes como Escherichia coli, especies de Enterococos, Pseudomonas spp y Aeromonas spp [16,17]. Los resultados encontrados en este estudio revelan que los aislamientos de microorganismos provenientes de fuentes hídricas destinadas para el consumo y uso humano alcanzó un 71.4%, de los cuales 19.2% corresponde a BGNNF y el 60% de estos corresponde a especies de Pseudomonas spp, resultados extrapolables al estudio realizado por Zannetti et al en los cuales aislaron, de muestras hídricas provenientes de dispositivos para la filtración del agua, BGNNF en 41.5% de las muestras, de los cuales el 20.5% correspondían al género Pseudomonas spp [18].
La relevancia que se encuentra en los aislamientos de BGNNF en fuentes hídricas radica en la capacidad de estos microorganismos para causar infecciones oportunistas en pacientes inmunocomprometidos o con patologías como cáncer, fibrosis quística o condiciones que propician el desarrollo de infecciones nosocomiales como la ventilación mecánica, el uso de catéter o procedimientos invasivos [19-21]. Ejemplo de ello, el estudio realizado por Damas et al en el cual, entre 595 casos de neumonía adquirida durante la estancia en UCI, se encontró que la incidencia de aquellas que eran causadas por BGNNF era del 34.3% (11.9% por cepas resistentes a cefalosporinas de tercera generación y 22.4% no) siendo el segundo grupo más prevalente después de las Enterobacteriaceae con un 60%. En cuanto a la severidad, 128 casos conllevaron a shock séptico y 99 al desarrollo de sepsis severa, de ellos el grupo de BGNNF resistentes a cefalosporinas de tercera generación fue el que con mayor frecuencia conllevó al desarrollo del shock séptico (p<0,001) y tuvo un mayor puntaje en la escala SOFA (Sequential Organ Failure Assessment scale) con un puntaje de 12.8±4.8 previo al desarrollo y durante el episodio de neumonía (p<0,0001), indicando así que la colonización por este tipo de microorganismos fue mayor en pacientes cuya condición era más crítica. Por último el porcentaje de mortalidad entre las infecciones causadas por cepas resistentes a cefalosporinas de tercera generación se ubicó en 96.7% (45.2% durante la estancia en UCI y 51.6% durante la estancia hospitalaria) y en 92.3% en aquellas sensibles (37% durante la estancia en UCI y 55.4% durante la estancia hospitalaria), cifras que solo fueron superadas por el grupo de las enterobacterias productoras de beta-lactamasas de espectro extendido en los cuales la mortalidad llego al 100% de los casos [22].
La alta resistencia intrínseca de los BGNNF a los antimicrobianos hace que el tratamiento de infecciones causadas por ellos sea difícil y costoso [19]. La aparición de bacterias resistentes a antimicrobianos es ampliamente conocida en zonas en las cuales se utilizan antibióticos, sin embargo, se ha reconocido también a los ambientes acuáticos como reservorios tanto de bacterias resistentes a antimicrobianos como de genes que inducen resistencia a los mismos [23-25], incluso llegando a aislarse en mayor cantidad en muestras provenientes de grifos que en muestras de fuentes naturales, sugiriendo que el tratamiento hecho al agua para que sea apta para el consumo humano podría incrementar la supervivencia de colonias resistentes a antimicrobianos o inducir la transferencia de genes que inducen resistencia entre determinadas poblaciones bacterianas. Por lo tanto, las fuentes naturales y las artificiales, como las fuentes de almacenamiento y dispensadores de agua, podrían llegar a tener un impacto considerable, aun no del todo claro, tanto en la incubación como en la distribución de cepas bacterianas resistentes a diferentes agentes antimicrobianos [23,24].
El antibiótico con mayor resistencia con respecto a las colonias de Pseudomonas spp estudiadas fue la ceftriaxona, ya que ninguna fue sensible, seguida por la cefoxitina (50%), la ciprofloxacina, la gentamicina y la piperacilina-tazobactam con un 83% de sensibilidad cada una y el antibiótico con mayor sensibilidad fue el imipenem (100%). Estos resultados son comparables a reportes en los que las especies de Pseudomonas se registraron un alto nivel de resistencia para ceftazidima (50%), aztreonam (50%) mientras que al cefepime mostraron en general una gran sensibilidad (83.3%) [19]. En el estudio realizado por Kalidas et al los antibióticos más activos fueron imipenem (91.08%) y amikacina (69.30%) [26-28], igual que el estudio realizado por Deepak et al donde se encontró que el 82% de P. aeruginosa fueron sensibles a imipenem, el 79% eran sensibles a Cefipime- sulbactam, el 69% eran sensibles a ceftazidima-sulbactam, el 34% fueron sensibles a amikacina y 46% para la piperacilina [27].
Contrario a lo encontrado en el presente estudio, el realizado por Veena et al evidencio un 60-70% de resistencia de P. aeruginosa a la amikacina, ceftazidima y ciprofloxacina [26].
Las colonias de Acinetobacter spp aisladas fueron 100% resistentes a ceftriaxona, el 50% fue sensible a cefoxitina y finalmente el 100% fue sensible a ciprofloxacina, piperacilina-tazobactam, gentamicina e imipenem. Igualmente en el estudio realizado por Kalidas et al se encontró que el 90% de las colonias eran sensibles a imipenem y el 62% a amikacina [28]. Contrario a nuestros resultados el estudio realizado por Gupta et al las cepas de Acinetobacter spp mostraron una mayor tasa de resistencia a la ciprofloxacina, amikacina, ceftazidima y piperacilina [29].
Sphingomonas paucimobilis fue totalmente resistente a la ceftriaxona y gentamicina. Mientras que la ciprofloxacina mostro una efectividad intermedia, el imipenem, piperacilina-tazobactam y cefoxitina fueron totalmente efectivos contra este microorganismo. Resultado comparables con lo publicado por Baruah et al donde encontraron que S. paucimobilis fue una cepa bastante sensible, con la resistencia observada sólo a ceftazidima y aztreonam [19].
Si bien poco se encuentra en la literatura sobre el género Pantoea, la especie más estudiada ha sido P. agglomerans, de la cual se conocen reportes acerca de brotes de septicemia nosocomiales en hospitales estadounidenses y canadienses, pro ducto de la colonización de botellas que contenían líquidos para infusión, medicamentos anestésicos como el propofol y componentes sanguíneos para transfusiones. El aislamiento de la colonia de Pantoea spp fue resistente a ceftriaxona, sensible a ciprofloxacina, cefoxitina, piperacilina-tazobactam, gentamicina e imipenem, este perfil de resistencia coincide al presentado por Delétoile et al, quienes realizaron el aislamiento de 36 colonias de Pantoea spp de las cuales 28 correspondieron a la especie P. agglomerans y las demás a otras especies de Pantoea. Algunas colonias solo presentaron susceptibilidad intermedia o resistencia a cefalosporinas de primera generación (cefalotina) o a la amoxicilina. Las demás presentaron sensibilidad a B-lactamicos, trimetoprim sulfametoxazol, gentamicina y ciprofloxacina, todos sus aislamientos fueron sensibles a imipenem y a cefalosporinas de espectro extendido (cefotaxime) [30], dato que contrasta con la resistencia a ceftriaxona presentada por la colonia aislada en nuestro estudio.
En el desarrollo del presente estudio se observó que 5 de 7 aislamientos revelaron un BGNNF, que como fue expuesto es de gran importancia en infecciones en humanos. En general se encontró gran porcentaje de colonias sensibles a los antibióticos utilizados, sin embargo, se destaca el alto porcentaje de colonias que fueron resistentes a la ceftriaxona, asi como el aislamiento de una colonia de Pseudomonas spp la cual fue resistente a todos los fármacos excepto al imipenem. Por lo demás, se puede concluir que la cefoxitina, la ciprofloxacina, piperacilina-tazobactam, gentamicina e imipenem son antimicrobianos que presentan una gran cobertura antibiótica contra los BGNNF autóctonos de ambientes acuáticos de los cuales fueron tomadas nuestras muestras. Estos microorganismos que están en permanente contacto con el ser humano, cobran importancia en infecciones nosocomiales que pueden llegar al desarrollo de septicemia e incluso la muerte de pacientes infectados por estos microorganismos, es por esta razón que es de gran importancia para el personal médico conocer sobre el perfil de resistencia antibiótica que presentan los BGNNF.
Agradecimientos
A Iveth Hernández por su disposición y colaboración, a la Universidad Militar Nueva Granada por su apoyo y recursos durante la elaboración de este proyecto de investigación.
Conflicto de intereses
Los autores declaran no tener conflictos de intereses en el presente manuscrito.
Referencias
1. Rojas T, Márquez E, Lugo R, Machado M, Vásquez Y, Fernández Y, et al. Bacilos gramnegativos no fermentadores en agua embotellada: suceptibilidad antimicrobiana y formación de biopeliculas. Revista de la Sociedad Venezonala de Microbiología 2014;34:64-69. [ Links ]
2. Radice M, Marín M, Giovanakis M, Vay C, Almuzara M, Limansky A, et al. Criterios de ensayo, interpretación e informe de pruebas de sensibilidad a los antibióticos en los bacilos gram negativos no fermentadores de importancia clínica: recomendaciones de la Subcomisión de Antimicrobianos de la Sociedad Argentina de Bacteriología, Micología y Parasitología Clínicas, Asociación Argentina de Microbiología. Rev Argent de Microbiol 2011;43(2):136-153. [ Links ]
3. Malini A, Deepa EK, Gokul BN, Prasad SR. Nonfermenting Gram-Negative Bacilli Infections in a Tertiary Care Hospital in Kolar, Karnakata. J Lab Physicians 2009;1(2):62-66. [ Links ]
4. Chawla K, Vishwanath S, Munim FC. Nonfermenting Gramnegative Bacilli other than Pseudomonas aeruginosa and Acinetobacter spp. Causing Respiratory Tract Infections in a Tertiary Care Center. J Glob Infect Dis 2013;5(4):144-148. [ Links ]
5. Su CS, Vaneechoutte M, Dijkshoorn L, Wei YF, Chen YL, Chang TC. Identification of non-fermeting Gram-negative bacteria of clinical importance by an oligonucleotide array. J Med Microbiol 2009;58:596-605. [ Links ]
6. Ryan MP, Adley CC. Sphingomonas paucimobilis: a persistent Gram-negative nosocomial infectious organism. J Hosp Infect 2010;75:153-157. [ Links ]
7. Carrillo R, Martínez J, Mendoza J. Infección de tejidos blandos por Stenotrophomonas maltophilia en un paciente con anemia aplásica. Med Int Mex. 2012;28:621-625 [ Links ]
8. Vincenti S, Quaranta G, DeMeo C, Bruno S, Ficarra MG, Carovillano S, et al. Non-fermentative gram-negative bacteria in hospital tap water and water used for haemodialysis and bronchoscope flushing: Prevalence and distribution of antibiotic resistant strains. Sci Total Environ 2014;499:47-54. [ Links ]
9. Fernández F. López LE, Rodríguez J. Contribución del laboratorio de microbiología en la vigilancia y el control de brotes nosocomiales producidos por bacilos gramnegativos no fermentadores. Enferm Infecc Microbiol Clin 2011;29(3):40-46. [ Links ]
10. Sharma D, Vyas N, Sinha P, Mathur A. Non fermentative gram negative bacilli as nosocomial pathogens: Identification and antibiotic sensitivity in clinical samples of indoor patients. Nepal Journal of Medical Sciences 2014;3(2):101-105. [ Links ]
11. Fariñas MC, Martínez-Martínez L. Infecciones causadas por bacterias gramnegativas multirresistentes: enterobacterias, Pseudomonas aeruginosa, Acinetobacter baumannii y otros bacilos gramnegativos no fermentadores. Enferm Infecc Microbiol Clin 2013;31(6):402-409. [ Links ]
12. Slama T. Gram-negative antibiotic resistance: there is a price to pay. Crit Care 2008;12(4):S4. [ Links ]
13. Samonis G, Maraki S, Vouloumanou EK, Georgantzi GG, Kofteridis DP, Falagas ME. Antimicrobial susceptibility of nonfermenting Gram-negative isolates of isepamicin in a region with high antibiotic resistance. Eur J Clin Microbiol Infect Dis 2012;31:3191-3198. [ Links ]
14. Cockerill FR, Patel JB, Alder J, Et al. Performance Standars for Antimicrobial Susceptibility Testing; Twenty - Third Informational Supplement. Clinical and Laboratory Standards Institute 2013;32:32-192. [ Links ]
15. República de Colombia. Ministerio de Salud y Protección Social, Subdirección de Salud Ambiental. Informe nacional de la calidad del agua para el consumo humano año 2013 con base en el IRCA. Bogotá, D.C. Diciembre de 2014. [ Links ]
16. Mulamattathil SG, Bezuidenhout C, Mbewe M, Ateba CN. Isolation of Environmental Bacteria from surface and Drinking Water in Mafikeng, South Africa, and Characterization Using Their Antibiotic Resistance Profiles. J Pathog 2014;2014:371208. [ Links ]
17. Staradumskyte D, Paulauskas A. Non-Fermentative GramNegative Bacteria in Drinking Water. J Water Resource Prot 2014;6:114-119. [ Links ]
18. Zanetti F, de Luca G, Leoni E, Sacchetti R. Occurrence of nonfermenting gram negative bacteria in drinking water dispensed from point-of-use microfiltration devices. Ann Agric Environ Med. 2014;21(1):29-34. [ Links ]
19. Baruah FK, Hussain AN, Kausalya, Grover RK. Antibiotic resistance profile of non-fermenting Gram-negative bacilli isolated from the blood cultures of cancer patients. J Glob Infect Dis 2015;7(1):46-47. [ Links ]
20. Prabash K, Medhekear A, Ghadyalpatil N, Noronha V, Biswas S, Kurkure P, et al. Blood stream infections in cancer patients: A single center experience of isolates and sensitivity pattern. Indian J Cancer 2010;47(2):184-188. [ Links ]
21. Memish ZA, Shibl AM, Kambal AM, Ohaly YA, Ishaq A, Livermore DM. Antimicrobial resistance among non-fermenting Gram-negative bacteria in Saudi Arabia. J Antimicrob Chemother 2012;67:1701-1705. [ Links ]
22. Damas P, Layios N, Seidel L, Nys M, Melin P, Ledoux D. Severity of ICU-acquired pneumonia according to infectious microorganism. Intensive Care Med 2011;37(7):1128-1135. [ Links ]
23. Berendonk TU, Manaia CM, Merlin C, Fatta-Kassinos D, Cytryn E, Walsh F, et al. Tackling antibiotic resistance: the environmental framework. Nat Rev Microbiol 2015;13(5):310-317. [ Links ]
24. Xi C, Zhang Y, Marrs CF, Ye W, Simon C, Foxman B, et al. Prevalence of antibiotic resistance in drinking water treatment and distribution systems. Appl Environ Microbiol 2009;75(17):5714-5718. [ Links ]
25. Wright, GD. Antibiotic resistance in the environment: a link to the clinic?. Curr Opin Microbiol 2010;13(5):589-594. [ Links ]
26. Kalidas R, Falguni N, Hirak JR, Maity PK. Prevalence and susceptibility profiles of nonfermenting gram-negative bacilli infection in a tertiary care hospital of eastern India. Indian J Clin Practice 2013;24(5):451-455. [ Links ]
27. Deepak J, Rajat P, Shamanth AS, Munesh S, Vikrant N, Neelam S. Prevalence of non-fermenting gram negative bacilli and their in vitro susceptibility pattern in a tertiary care hospital of Uttarakhand: A study from foothills of Himalayas. Saudi J Health Sci 2013;2:8-12. [ Links ]
28. Veena-Kumari HB, Nagarathna S, Chandramuki A. Antimicrobial resistance pattern among aerobic gram-negative bacilli of lower respiratory tract specimens of intensive care unit patients in a neurocentre. Indian J Chest Dis Allied Sci 2007;49(1):19-22. [ Links ]
29. Gupta N, Gandham N, Jadhav S, Mishra RN. Isolation and identifi cation of Acinetobacter species with special reference to antibiotic resistance. J Nat Sc Biol Med 2015;6159-62. [ Links ]
30. Delétoile A, Decré D, Courant S, Passet V, Audo J, et al. Phylogeny and identification of Pantoea species and typing of Pantoea agglomerans strains by multilocus gene sequencing. J. Clin. Microbiol. 2009;47 (2):300-310. [ Links ]