Introducción
Staphylococcus aureus es un microorganismo patógeno para el hombre, capaz de causar una multitud de infecciones con un amplio rango de gravedad, desde infecciones localizadas de piel e intoxicaciones alimentarias, hasta infecciones invasoras y potencialmente mortales como neumonía necrosante, osteomielitis y sepsis.[1].
Staphylococcus aureus ha desarrollado resistencia a prácticamente todas las clases de antimicrobianos, principalmente a los β-lactámicos. El mecanismo más frecuente de resistencia a los β-lactámicos radica en la penicilinasa, enzima que hidroliza a la penicilina y a otros compuestos sensibles. Los primeros β-lactámicos estables frente a la acción de penicilinasa como las cefalosporinas y meticilina estuvieron disponibles a finales de 1950 sólo unos años después se reportó la aparición de las primeras cepas resistentes a meticilina [2,3].
Las Enfermedades Transmitidas por Alimentos (ETA) son el síndrome originado por la ingestión de alimentos y/o agua, que contengan agentes etiológicos en cantidades tales que afecten la salud del consumidor a nivel individual o grupos de población. Estas enfermedades se dividen en Infecciones alimentarias e Intoxicaciones alimentarias. Las Infecciones Alimentarias son las ETA producidas por la ingestión de alimentos y/o agua contaminados con agentes infecciosos específicos tales como bacterias, virus, hongos, parásitos, que en la luz intestinal pueden multiplicarse o lisarse y producir toxinas o invadir la pared intestinal y desde allí alcanzar otros aparatos o sistemas. Las Intoxicaciones alimentarias son las ETA producidas por la ingestión de toxinas formadas en tejidos de plantas o animales, o de productos metabólicos de micro-organismos en los alimentos, o por sustancias químicas que se incorporan a ellos de modo accidental, incidental o intencional en cualquier momento desde su producción hasta su consumo [4].
El sector agroindustrial en Colombia se posiciona como el sector más importante de la industria manufacturera colombiana, con una producción bruta de US$ 226,2 Billones, un consumo intermedio de $140,5 billones y un valor agregado de $85,7 billones. Entre 2014 y 2015, el consumo intermedio del sector manufacturero presentó un crecimiento de 4,5%, La principal contribución a este aumento del consumo intermedio correspondió a productos alimenticios, que aportó 3,6 puntos porcentuales positivos a la variación total. El país es un gran productor de lácteos, bebidas, productos de molinería, cárnicos, aceites, entre muchas otras. [5]. [DANE, 2015].
El propósito de esta investigación es caracterizar la susceptibilidad a antibióticos y perfil plasmídico en cepas de staphylococcus aureus procedentes de quesos costeños de Valledupar.
La hipótesis de este estudio es determinar, si en los individuos de la ciudad de Valledupar se incrementan las enfermedades transmitidas por alimentos, cuando consume queso costeño.
Materiales y métodos
Tipo y diseño de estudio
El tipo de estudio es descriptivo se busca especificar las propiedades, características y los perfiles importantes de personas, grupos, comunidades o cualquier otro fenómeno que se someta a un análisis. El diseño utilizado es experimental,
Muestra de estudio
Se estudiaron 25 cepas de S. aureus aisladas de quesos tipo costeño, de manufactura industrial y artesanal. Las muestras de quesos se obtuvieron de diferentes puntos de ventas en el municipio de Valledupar, tomadas de forma aleatoria del universo presente en cada establecimiento. Se procesaron tres tipos de quesos en total, tres artesanales (costeño blando, costeño duro, costeño semiduro). Y uno industrial, de cada queso se procesaron 3 muestras para un total de 12 muestras.
Homogenización y aislamiento de la muestra
De cada muestra se pesó, asépticamente, 25 g de queso y se adicionaron a un vaso de licuadora estéril que contuvo 225 mL de solución salina peptonada (SSP) estéril (NaCl 0,85% p/v con 0,1% p/v de peptona), a fin de obtener una dilución inicial (10-1). Hay que indicar que la muestra se procesó hasta lograr homogeneizarla mecánicamente con el uso de una licuadora estéril de 2 a 3 minutos. Posteriormente 1 ml de esta solución se llevó asépticamente a un tubo con 9 ml de SSP 0,1%, y se obtuvo una dilución 10-2, tal procedimiento se repitió hasta que se obtuvo las diluciones 10-3, 10-4 y 10-5[6-9].
Para el aislamiento de S. aureus, se extendió con espátula de Drigalsky 0,1 mL de cada dilución sobre toda la superficie de placas con agar Baird Parker (BPA) por duplicado. Éstas se incubaron por un período de 24 a 48 horas a 37º C.
Caracterización de la morfología celular y colonial, y bioquímica de las cepas de Staphylococcus aureus así como la preservación de las muestras.
Se tomaron entre 3-5 UFC caracterizadas por ser negras, con borde estrecho blanquecino y rodeado de un halo claro, típico de cepas de S. aureus crecidas sobre agar BPA por su capacidad de reducir telurito, producir proteasas y lecitinasas respectivamente. Las mismas se sometieron a pruebas bioquímicas confirmativas para identificar el género y especie en estudio. Estas incluyeron: morfología bacteriana, tinción de Gram, producción de ácido a partir de manitol, Voges-Poskauer y prueba de catalasa y para la evaluación de la patogenicidad del microorganismo, las pruebas de: coagulasa y nucleasa termoestable [10].
Las cepas que resultaron positivas para las pruebas bioquímicas confirmativas de S. aureus, se almacenaron a -20º C en solución de glicerol al 20 % v/v para su preservación, y se activaron en caldo nutritivo para la evaluación de la susceptibilidad a antibióticos y la determinación del perfil plasmídico. [11].
Determinación de la susceptibilidad a antibióticos en cepas de S. aureus.
La resistencia de las cepas bacterianas a los antibióticos se determinaron por el método de difusión del disco en agar descrito por Kirby-Baüer , así como la cuantificación de la actividad in vitro a algunos antimicrobianos, determinando la Concentración Inhibitoria Mínima (CIM) de los antibióticos de interés mediante dilución en placas con agar Müeller-Hinton [12,13].
Determinación de resistencia a antibióticos por el Método Difusión del Disco en Agar.
Primero se activaron las cepas en caldo nutriente e incubaron en la incubadora BD 56 (www. Binder-world.com) a 37ºC por 24h posteriormente se sembraron en medios nutritivos, enriquecidos (agar sangre al 5%) no selectivos, incubando nuevamente las placas a 37 ºC por 24h para obtener colonias aisladas, las cuales fueron resuspendidas en solución salina 0,85% estéril hasta que alcanzaron una turbidez equivalente al tubo estándar 0,5 de McFarland, que contuvo aproximadamente de 1-2 x 103 UFC/ml. Posterior a la preparación del inóculo, se sembraron las cepas en placas conteniendo 4 mm de espesor de agar Müeller-Hinton (HiMedia® Laboratories) con hisopo estéril, rayando en tres direcciones, rotando las placas cada vez con giros de 60º. Se colocaron los discos asépticamente con una pinza esterilizada. Para estas cepas se utilizaron los siguientes antibióticos manufacturados por BD BBLTM (Sensi-DiscTM): Penicilina (10U/mL), Tetraciclina (30µg/mL), Trimetoprim/Sulfametoxazol (1,25-23,75µg/mL), Ciprofloxacina (5µg/mL), Gentamicina (10µg/mL), Cloranfenicol (30µg/mL), Rifampicina (5µg/mL), Vancomicina (30µg/mL), Clindamicina (2µg/mL), Eritromicina (15µg/mL), Linezolid (30µg/mL), Oxacilina (1µg/mL). Para verificar la calidad del antibiograma o de los discos se empleó la cepa de S. aureus ATCC 25923 como control.
Las placas se incubaron de manera invertida a 37º C en atmósfera aeróbica por 18 horas para medir los halos de inhibición y se determinó: Resistencia (R), Resistencia intermedia (I) o Susceptibilidad (S) de la bacteria frente a cada antibiótico, Teniendo en cuenta que VA y OX se leen a las 24 horas exactas.
Determinación producción de β-lactamasas en cepas de Staphylococcus aureus, mediante el método de la cefalosporina cromogénica (Nitrocefina).
Para la prueba se utilizaron bastoncillos impregnados con nitrocefina (marca OXOID®) que previamente fue humedecida con agua destilada estéril y se roto sobre la superficie de la colonia a partir de crecimiento fresco de 24 horas en agar sangre. Luego se esperó hasta 1 hora el cambio de color (rojo), este efecto es indicativo de una prueba positiva. Seguidamente, se examinó el extremo para detectar el cambio de color dentro de los cinco minutos seguidos a la inoculación. Algunas especies de estafilococos tardan hasta una hora para producir una reacción positiva [14].
Este ensayo estuvo controlado con las cepas Staphylococcus aureus ATCC 25923 (ß-lactamasa -) y ATCC 29213 (ß-lactamasa +).
Determinación de la Concentración Mínima Inhibitoria a los antibióticos Oxacilina, y Penicilina G, empleando el método de dilución en agar.
Para esto se prepararon tubos con 20mL de agar Müeller Hinton (MH) estériles y se llevaron a 45° C, luego se les adicionó 1mL del antibiótico en solución con una concentración conocida, obteniendo al final en la placa ya vertida la concentración que se desea.
Los antibióticos que se utilizados son Oxacilina (Sigma-Aldrich®), y Penicilina G Sódica® (Bristol-Myers Squibb). Las concentraciones de antibiótico utilizadas se encuentran dentro de los criterios establecidos por el instituto de normas clínicas y de laboratorio (CLSI). Se utilizó a cepa de Staphylococcus aureus ATCC 29213 como control negativo.
Determinación de fenotipos de resistencia a la familia macrólidos, lincosamidas, estreptomicina B (MLSB).
La técnica se realizó siguiendo las recomendaciones del CLSI. Dicha técnica consistió en colocar un disco de Eritromicina (15 μ g) y otro de Clindamicina (2 μ g) separados a una distancia entre 15 - 26 mm uno del otro, de borde a borde en una placa de agar Mueller-Hinton que ha sido previamente inoculada con una suspensión (0,5 McFarland) del microorganismo. Después de 16-18 h de incubación a 35° C, el achatamiento en la zona de inhibición de la Clindamicina próxima al disco de Eritromicina (efecto zona D) indica un fenotipo de resistencia inducible, la resistencia a Eritromicina y a Clindamicina indica un fenotipo de resistencia constitutivo y la sensibilidad a Clindamicina es definida por la ausencia de inducción de resistencia a Clindamicina en la zona próxima al disco de Eritromicina. Las cepas controles fueron la ATCC® 25923 sensible a ambos antibióticos (No formadora de la zona D). Las condiciones del ensayo fueron las mismas descritas para la determinación de resistencia a antibióticos por el Método Difusión del Disco en agar.
Extracción de DNA plasmídico de cepas de Staphylococcus aureus.
De un cultivo fresco se inoculó la cepa bacteriana en placas de agar nutriente, por rayado en cuadricula mediante hisopado. Las placas se incubaron en la incubadora BD 56 (www. Binder-world.com) a 37º C durante 24 horas. Para recoger el volumen celular, transcurrido el período de incubación se adicionó a la placa 0,5 mL de solución salina estéril al 0,85%, esta operación se repitió una vez más para cosechar la mayor cantidad de colonias posible y se transfirió a tubos eppendorf.
El volumen celular se centrifugo en la microcentrifuga Mkro 220 (www.sartrius.es/), por 5 minutos a 10.000 r.p.m y se descartó el sobrenadante. El pellet obtenido se resuspendió en 500 µL de buffer Tris HCl- EDTA-Glucosa (TES), para nuevamente ser centrifugado por 3,5 minutos a 10.000 r.p.m y se descartó el sobrenadante. Este nuevo pellet se resuspendió en 500µL de buffer Tris HCl- EDTA (TE) glucosa y se le agrego 20 µl de lisostafina (0,5 mg/mL). Se incubó en la incubadora BD 56 (www. Binder-world.com) a 37º C por 1 hora, se agregó 75 µL de buffer de lisis y se mezcló suavemente por inversión. La reacción se incubó a temperatura ambiente por un período de 10 minutos. Se procedió a adicionar 400 µL de acetato de potasio 5M, se mezcló por inversión el eppendorf, y se incubó en hielo por 30 minutos. Se centrifugó enseguida por 20 minutos, a temperatura ambiente a 10000 r.p.m. El sobrenadante obtenido fue transferido a un tubo eppendorf estéril. La obtención del ADN precipitado se llevó a cabo agregando 555 µL de etanol absoluto frío e incubando toda la noche a -20º C. Se centrifugó por 10.000 r.p.m por 10 minutos a temperatura ambiente y el sobrenadante se descartó, el pellet obtenido se lavó con etanol frío al 70%, seguido de 5 minutos de centrifugación. El lavado del pellet se repitió dos veces, y finalmente se secó a temperatura ambiente por 15 minutos [15].
Observación de plásmidos crípticos en geles de agarosa al 0,7% p/v.
La separación y observación de los plásmidos se realizó según especificaciones de Ausubel y colaboradores por electroforesis el equipo utilizado fue blotter semi seco enduro S2020 (www.cientificasenna.com) en geles de agarosa al 0,7% con EthBr (hasta una concentración final de 0,5µg/mL) en una cámara horizontal sumergida a 50 voltios durante 7-8 horas. El ADN fue visualizado en un transiluminador UVP, Chromato-Vue TM-20 (www.ebay.com) El tamaño de los plásmidos se determinó utilizando el programa computacional ORIGIN versión 6.0 para Windows, basado en la migración de las bandas de ADN de las muestras en estudio, en comparación con las bandas del marcador de peso molecular (PM) λHindlll, relacionando de esta manera el PM con la movilidad de cada banda en el gel de agarosa [16,17].
Análisis Estadístico
Se realizaron cálculos porcentuales para determinar la cantidad de cepas aisladas de cada queso, las bacterias resistentes por cada antibiótico ensayado, así como el número de antibióticos a los que es resistente cada cepa. También se correlacionaron los aislados de cada queso con los fenotipos de resistencia a antibióticos, multiresistencia, resistencia a meticilina, producción de ß- lactamasa con los plásmidos presentes en las cepas. Para estas pruebas se aplicó la prueba de correlación de Pearson con un valor de p>0,05.
Para el cálculo de los tamaños aproximados de las bandas plasmídicas de las cepas de S. aureus estudiadas se utilizó la aplicación Origin Lab, Origin Pro 7,5 para Windows. Para estos análisis se utilizaron los paquetes estadísticos Statistic for Windows versión 4,1.
Resultados
De las 12 muestras analizadas para determinar S aureus se observa que sobrepasa casi 5 veces el valor promedio máximo permitido, de esta manera se afirma que el alto recuento de este microorganismo comprueba las malas practicas higienicas en la manipulación, venta y distribución del producto, es decir, de los quesos.
Se observó que los quesos con mayor frecuencia de aislados de S. aureus fueron quesos costeños blandos (75%), seguidos por los quesos semiduros (25%), ambos de origen (artesanal), los cuales son de alto consumo en Valledupar. En los quesos duros e industrial no se aislaron S. aureus.
El presente estudio muestra un porcentaje alto (76%) de cepas sensibles a casi todos los antibioticos probados, el 20% muestra resistencia a un antibiotico (Tetraciclina) (Cloranfenicol) y el 4% con resistencia a tres antibioticos (Penicilina,Tetraciclina y Eritromicina).No se observo multirresistencia ( resistencia a 5 o mas antibioticos de grupos diferentes).
Se determinaron 4 patrones diferentes de resistencia en las cepas de S. aureus aisladas de los quesos, PR-TER-EI y CR siendo el patron TER común para dos cepas en cada caso,en el resto de los patrones no se observó similitud (Tabla 1).
Tabla 1 Patrón de resistencia y producción de B lactamasa en cepas de s aureus aislada de quesos. Laboratorio de biología marzo del 2012.

QB: Queso blando, QS: Queso semiduro, P: Penicilina; TE: Tetraciclina; E: Eritromicina; C: Cloranfenicol; R: Resistencia; I: Resistencia Intermedia; NA: No aplica.
La determinación de la producción β-lactamasas se le realizó a una cepa que resultó resistente a la penicilina y fue productora de β-lactamasa (Tabla 1).
Con la finalidad de corroborar el fenotipo de resistencia a la Penicilina se procedió a determinar la CMI a este antibiótico a la cepa 20QB2BA2. Este resultado también permitirá seleccionar, en futuras investigaciones, aquella concentración del antibiótico ideales para la inducción de la replicacion de los plásmidos involucrados en dicha resistencia. La concentración mínima inhibitoria obtenida fue de 32 ug/mL, catalogándose dicha cepa como resistente a la penicilina. El mecanismo de resistencia utilizado por esta cepa para alcanzar dicho fenotipo es la producción de β-lactamasa, enzima detectada en este trabajo mediante tiras de nitrocefina.
Perfil plasmídico
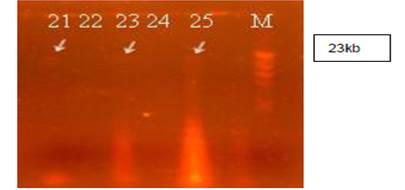
En relación a la determinación del perfil de bandas plasmídicas de las 25 cepas de S. aureus estudiadas, se puede mencionar que el 12% (3 cepas) mostró bandas de DNA plasmídico, cuyo tamaño fue de 23 kb Tabla 2, Figura 1. El 88% de cepas restantes [22 cepas] no mostraron bandas de ADN plasmídico.
Tabla 2 Perfiles plasmídicos en las cepas de S aureus aisladas de quesos. Laboratorio de biología marzo del 2012.
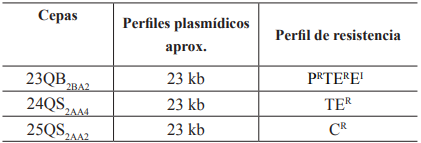
Figura Bandas plasmídicas observadas en las cepas: QB2BA2, QSd2AA3, Sd2AA2.M: λ Hind III.
Con respecto a la presencia de las bandas plasmídicas y el perfil de resistencia encontrado en los aislados estudiados, se observa que el 12% de los aislados (3 cepas: QSd2AA2, QSd2AA3 y QB2BA2) con alguna resistencia mostró por lo menos una banda de DNA plasmídico (Tabla 2, y Figura).
La resistencia a TE se debe a la metilación ARN ribosomal de la subunidad 50s y la presencia de estas bandas puede deberse a la presencia de plásmidos que le confieran resistencia a la bacteria. Con respecto a la cepa (QSd2AA2) que mostró resistencia al Cloranfenicol, se encontró que sólo contenía también una banda de 23 kb (Tabla 2, Figura).
Como se observa en la Tabla 2 y en la Figura existe coincidencia entre la presencia de las bandas de DNA plasmídico y el perfil de resistencia encontrado. Aunque no se demostró durante este estudio que estos elementos extracromosomales estén involucrados con la resistencia a los antimicrobianos, no se descarta esta posibilidad, ya que la resistencia para muchos de los antibacterianos ensayados, se hayan codificadas a nivel plasmídico.
Discusión
Los mayores porcentajes de aislamientos en este trabajo se obtuvieron a apartir de quesos artesanales, resultados similares se obtuvieron en investigaciónes realizadas en la region zuliana efectuada por Rivera a partir de los quesos no pasteurizados [18].
Nuestro estudio presento mas muestras sensibles que resistentes y sin multirresistencia a cinco antibioticos, esto difiere con los obtenidos por Mujica y colaboradores y Rivera, en aislados de alimentos, quienes mostraron patrones de resistencia a antibioticos variables.
El patron mas comun fue TER para dos cepas, este semejante a los conseguidos por Fuentes que obtuvo 11 cepas productoras de β-lactamasa en aislados de muestras de quesos utilizando el método de la cefalosporina cromogénica (nitrocefina, Difco).
Esta investigación permitirá seleccionar, en futuras investigaciones, aquella concentración del antibiótico ideal para la inducción de la replicacion de los plásmidos involucrados en dicha resistencia.
Los tamaños de los plásmidos relacionados al perfil coinciden con los obtenidos por Rivera, donde obtuvo cepas que presentaban bandas plasmídicas aproximadamente de 23, 27 y 34kb. Este trabajo presento cepas que llevaban consigo entre 1 a 4 bandas plasmidicas.
Los datos obtenidos en esta investigación no permiten dilucidar la ubicación genética de los marcadores de resistencia a antibióticos, ni los posibles fenotipos codificados en dichas bandas de DNA, por tal razón deberán aplicarse otras metodologías que permitan su estudio adecuado, profundizando el conocimiento del genotipo de las bacterias autóctonas de la región de Valledupar.
La determinación de los perfiles plasmídicos en cepas de S. aureus provenientes de alimentos, es de importancia biológica, clínica y comunitaria, toda vez que estos elementos genéticos son fuente de diseminación de múltiples genes de resistencia.