Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Universitas Scientiarum
versión impresa ISSN 0122-7483
Univ. Sci. v.14 n.3 Bogotá sep./dic. 2009
Caracterización e identificación de aislados de levaduras carotenogénicas de varias zonas naturales del Ecuador
Characterization and identification of isolates of carotenogenic yeast strains from several natural zones of Ecuador
Caracterização e identificação de isolados de leveduras carotenogénicas de diferentes áreas naturais do Equador
Investigación de la Biomasa, Colección de Levaduras Quito-Católica (CLQCA), Quito, Ecuador.
Av. 12 de Octubre 1076 y Roca, Casilla Postal 17-01-2184, Quito, Ecuador. Tel.: 1593 2 299 1700, ext. 1175; fax 1593 2 299 1687
Recibido: 13-10-2009; Aceptado: 03-12-2009
Resumen
Objetivo. Identificar y caracterizar levaduras pigmentadas aisladas de ambientes naturales del Ecuador que sean productoras de metabolitos de interés para la industria y puedan ser utilizadas en posteriores aplicaciones biotecnológicas. Materiales y métodos. Se trabajó con 26 aislados pigmentados de la Colección de Levaduras Quito-Católica (CLQCA) que fueron sometidos a pruebas fisiológicas y moleculares para su caracterización, tipificación e identificación. Se evaluó la capacidad de asimilación de azúcares. Se emplearon las técnicas moleculares RFLP-ITS, MSP-PCR, amplificación parcial del segmento 26S del ADNr y secuenciamiento para identificación a nivel de especie. Se incubaron todos los aislados a 20, 30 y 37°C para determinar la temperatura en la que presenten mejor crecimiento. Posteriormente, fueron determinadas las curvas de crecimiento para cada aislado. Resultados. Se identificaron las especies: Rhodotorula mucilaginosa, Rhodosporidium babjevae, Sporidiobolus ruineniae, Rhodotorula glutinis, Rhodotorula slooffiae y una especie nueva del género Rhodotorula. Conclusiones. Se seleccionaron como cepas promisorias para futuros usos industriales los aislados de Rhodotorula mucilaginosa (CLQCA-12-013 y CLQCA-12-008) y Rhodosporidium babjevae (CLQCA-10-188), por presentar características de crecimiento rápido, alta asimilación de azúcares, producción de biomasa e intensidad en la pigmentación.
Palabras clave: levaduras pigmentadas, tipificación molecular y fisiológica, curva de crecimiento.
Abstract
Objective. To identify and characterize pigmented yeasts isolated from natural environments in Ecuador, producing metabolites with industrial importance and with potential use in further biotechnological applications. Materials and methods. Twenty-six pigmented isolates from the "Colección de Levaduras Quito-Católica" (CLQCA) were exposed to physiological and molecular tests for their characterization, typification and identification. Sugar assimilation ability was evaluated. Molecular techniques such as ITS-RFLP, MSP-PCR, partial amplification of the segment 26S of the rDNA, and sequencing were used for their identification at the species level. Growth curves for each isolate according to their specific optimum growth temperatures. Results. The following species were identified: Rhodotorula mucilaginosa, Rhodosporidium babjevae, Sporidiobolus ruineniae, Rhodotorula glutinis, Rhodotorula slooffiae, as well as a new species of the genus Rhodotorula. Conclusions. Rhodotorula mucilaginosa (CLQCA-12-013 and CLQCA-12-008) and Rhodosporidium babjevae (CLQCA-10-188) isolates were selected as promising strains for industrial applications, because they showed characteristics such as rapid growth, high assimilation of sugars, biomass production, and pigment synthesis.
Key words: pigmented yeasts, molecular and physiological typification, growth curve.
Resumo
Objetivo. Identificar e caracterizar leveduras pigmentadas isoladas de ambientes naturais do Equador, que produzem metabólitos de interesse para a indústria e podem ser usadas em subseqüentes aplicações biotecnológicas. Materiais e métodos. Trabalhou-se com 26 isolados pigmentados da Coleção de Leveduras Quito-Católica (CLQCA) que se submeteram a provas fisiológicas e molecular para sua caracterização, tipificação e identificação. Foi avaliada a capacidade de assimilação de açúcares. Foram utilizadas as técnicas moleculares RFLP-ITS, MSP-PCR, amplificação parcial do segmento 26S do ADNr ribossomal e seqüenciamento para sua identificação em nível de espécie. Determinaramse as curvas de crescimento para cada isolado de acordo com a sua temperatura ótima de crescimento. Resultados. Foram identificadas as espécies: Rhodotorula mucilaginosa, Rhodosporidium babjevae, Sporidiobolus ruineniae, Rhodotorula glutinis, Rhodotorula slooffiae e uma nova espécie do gênero Rhodotorula. Conclusões. Foram selecionados como cepas promissoras para futuros usos industriais os isolados de Rhodotorula mucilaginosa (CLQCA-12-013 e CLQCA-12-008) e Rhodosporidium babjevae (CLQCA-10-188), por apresentar características de crescimento rápido, alta assimilação de açúcares, produção de biomassa e síntese de pigmentos.
Palavras-Chave: leveduras pigmentadas, tipificação molecular e fisiológica, curva de crescimento.
Introducción
Los pigmentos carotenoides presentes en algunos géneros de levaduras confieren colores característicos a las colonias y protección contra efectos dañinos de la radiación UV (1). La identificación de especies basada en criterios de morfología está fuertemente influenciada por las condiciones de cultivo por lo que, en la actualidad, es complementada con datos obtenidos por técnicas de biología molecular, que se fundamentan en el análisis de fragmentos de las moléculas de ácidos nucleicos, con particular énfasis en la comparación de secuencias de ADNr (2, 3).
Levaduras productoras de carotenoides pertenecientes a los géneros Rhodotorula, Rhodosporidium, Sporobolomyces, Sporidiobolus y la especie Phaffia rhodozyma han sido estudiadas en otros trabajos similares, las cuales se caracterizan por asimilar un amplio espectro de fuentes de carbono (4). Los géneros Rhodosporidium y Sporidiobolus, son productores de carotenoides como: â- caroteno, ã caroteno, toruleno y torularodina (5). Mientras que, Phaffia rhodozyma ha sido utilizada industrialmente en la producción de astaxantina. Este pigmento es utilizado en la avicultura para la alimentación de gallinas de postura y pollos de engorde para mejorar el color de la yema de huevo y la piel de pollo; además, en la acuacultura se utiliza como pigmento de carne, piel o exoesqueleto en salmónidos y crustáceos (6).
Estas levaduras pueden ser aisladas de ambientes naturales ya que presentan una amplia distribución, y son capaces de colonizar múltiples sustratos naturales y artificiales. Además, presentan capacidad de adaptación a ambientes extremos como glaciares y ambientes ácidos (10).
Algunas especies de levaduras, como las que son objeto del presente estudio, han desarrollado estrategias para la disminución del daño producido por la radiación solar.
Los mecanismos de fotoprotección que se conocen en levaduras incluyen la síntesis de compuestos antioxidantes como pigmentos carotenoides y de absorción UV, como las micosporinas (7, 8).
La función más importante de los carotenoides, en las levaduras es la protección contra la combinación dañina del oxígeno singlete y la luz visible o UV (6, 9). Su acción consiste en desactivar los radicales libres que se producen durante el metabolismo normal de las células, tales como el oxígeno singlete (1O2), hidroxilo (OH-), peróxidos y otros oxidantes mediante un proceso en el que se transfiere energía de altos niveles de excitación a un triplete de la molécula de carotenoide, el cual puede regresar a su estado basal liberando calor. Se ha reportado que en organismos como Phaffia rhodozyma, el 1O2 y los radicales peróxidos inducen la biosíntesis de su pigmento característico. Otros factores que inducen la síntesis de astaxantina incluyen a la antimicina y otros inhibidores de la cadena respiratoria (6).
Ecuador es un país megadiverso debido a la gran variedad de ecosistemas y microclimas como resultado de su ubicación geográfica, misma que además determina una alta incidencia de rayos UV, especialmente en la cordillera de los Andes. Esto hace del Ecuador un sitio de interés para estudios de biodiversidad de levaduras productoras de pigmentos. El presente estudio es el primero realizado sobre levaduras carotenogéncias de algunas regiones de Ecuador con un enfoque biotecnológico.
Las levaduras pigmentadas que se presentan en este estudio podrían ser utilizadas como fuentes naturales de carotenoides, lo que ha generado interés por las posibles aplicaciones industriales, en especial de alimentos y cosméticos. Esta investigación se enfoca en el estudio y selección de aislados promisorios con miras a futuras aplicaciones industriales.
El presente trabajo fue realizado entre enero de 2008 y abril de 2009 en lo que se refiere específicamente a la búsqueda de aislados de interés biotecnológico preservados en la CLQCA, sean estos aislados identificados en anteriores estudios o aislados por ser identificados dentro de esta investigación. La nueva especie del género Rhodotorula fue hallada por Carvajal y James en 2007.
Materiales y métodos
Aislados de levaduras carotenogénicas
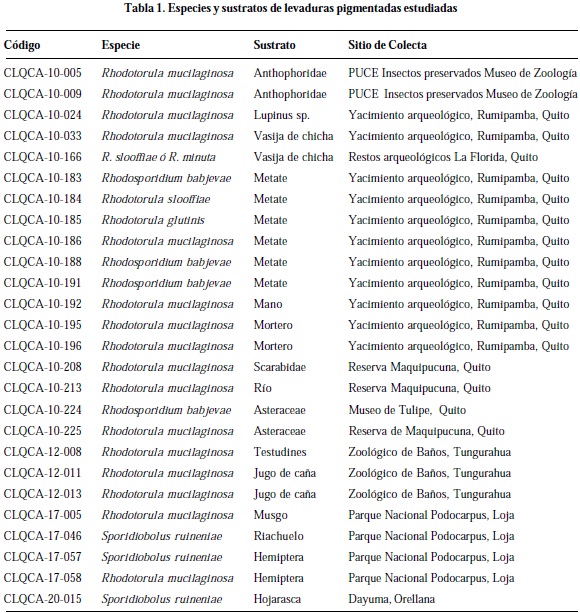
Se trabajó con 26 aislados de levaduras pigmentadas de la Colección de Levaduras Quito Católica (CLQCA) (Tabla 1).
Se realizaron diluciones seriadas de cada aislado (102, 103, 104, 105 y 106) a partir de cultivos líquidos previamente obtenidos con 24 horas de crecimiento. Las diluciones fueron sembradas por duplicado con esferas de vidrio en placas con medio CPM agar (Carotenoid Production Medium) (g L-1): glucosa, 20; fosfato de potasio, 8; sulfato de magnesio, 0,5; extracto de levadura, 4,7; agar, 20 a un pH 5,8 (16). Se monitoreó el crecimiento de los cultivos hasta las 72 horas (11) y se evaluó visualmente el número de colonias conseguidas en placa, lo que permitió conocer el número de células viables por volumen de solución, expresando en UFC mL-1. Todas las placas fueron incubadas a 20, 30 y 37°C con lo que se determinó cuál de estas temperaturas ofrece mejores condiciones para el crecimiento de cada aislado.
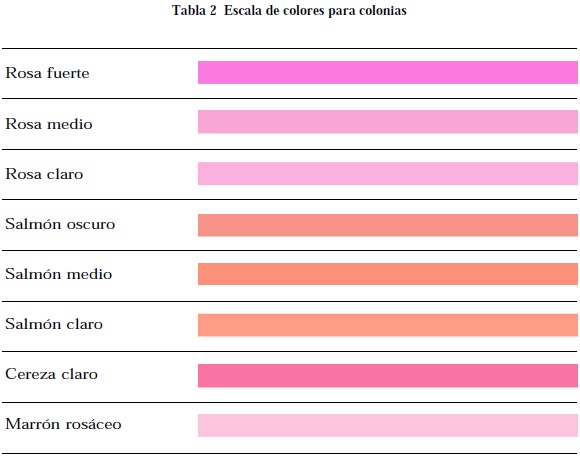
La caracterización morfológica de los 26 aislados se hizo tomando en cuenta: forma, brillo, tamaño, elevación, bordes, superficie, consistencia y color de las colonias que van de tonalidades salmón claro a rosa oscuro. A partir de esta caracterización se agruparon los aislados para posteriores análisis moleculares.
Evaluación de respuestas de crecimiento en compuestos carbonados
Para determinar la asimilación de azúcares se empleó medio YNB líquido (Yeast Nitrogen Base) al que se le añadió: glucosa, fructosa, xilosa, galactosa, sacarosa, lactosa, maltosa y rafinosa, de manera independiente, a una concentración final del 2% (p/v). Para determinar la actividad se evaluó la turbidez del medio; además, se utilizó el indicador rojo fenol para indicar cambio de pH. Los resultados fueron codificados mediante signos: (+), asimilación positiva; L, asimilación lenta; D, asimilación débil; (-), asimilación negativa (12). Y, se utilizó un control negativo para evaluar la asimilación de cada azúcar.
Identificación molecular
Extracción de ADN
La extracción de ADN se realizó mediante los protocolos descritos por Libkind et al. (2003); Hartung et al, (2005) para la amplificación de Micro/Minisatellite Primed PCR (MSP-PCR); y el protocolo descrito por Guthrie y Fink (2002) para la amplificación de la región ITS y 26S del ADNr con algunas modificaciones.
Amplificación de la región ITS mediante PCR
Para la amplificación de la región ITS se utilizaron los oligonucleótidos sintéticos ITS1 (5'–TCC GTA GGT GAA CCT GCG G-3') e ITS4 (5'–TCC TCC GCT TAT TGA TAT GC-3'). La reacción de PCR tuvo lugar añadiendo en un microtubo de 200 ul: 10 µl de buffer GoTaq® 5 X; 3 µL de MgCl2 25 mM; 4 µL mix dNTPs 10mM; 21,6 µL de agua libre de nucleasas; 0,4 µL de GoTaq® polimerasa (5 u µL-1); 3 µL de oligonucleótido ITS1 5 µM; 3 µL oligonucleótido ITS4 5 µM; y, 5 µL de ADN total previamente diluido 2:1000. El ADN fue amplificado en un termociclador TC-412 (Techne) mediante un paso inicial de desnaturalización de 5 min a 94°C seguido por 40 ciclos de 60 s a 93°C; 2 min de anillamiento a 55°C; 2 min de extensión a 72°C y, una extensión final de 10 min a 72°C. El producto de PCR se evidenció mediante electroforesis en geles de agarosa al 0,8% corridos a 100 V durante 30 min en buffer TBE 0,5 X (Tris 50 mM, ácido bórico, EDTA 50 mM, pH 8). Los geles fueron teñidos con bromuro de etidio. Se corrieron los amplicones obtenidos con un marcador de peso molecular con intervalos de 100 pb TrackltTM DNA Ladder (Invitrogen, 0,1 µg µL-1). Las bandas fueron evidenciadas mediante un transluminador Spectroline Ultraviolet Translluminator y las imágenes se registraron por un sistema de documentación digital (Fisher Bioblock Scientific).
Polimorfismos de Longitud de Fragmentos de Restricción (RFLP-ITS)
Para el análisis de los polimorfismos de longitud de fragmentos de restricción (RFLP-ITS) se emplearon tres enzimas de restricción: HhaI, HaeIII y HinfI. La reacción de digestión fue realizada en un volumen final de 15 µL conteniendo 5 µL del mix para RFLP-PCR y 10 µl del producto de amplificación de ITS. El mix RFLP-PCR para cada enzima fue preparado con 2 µL de buffer de reacción REact® 2 10x; 2,2 µL de H2O libre de nucleasas y 0,8 µL de enzima de restricción 10 u µL-1; según el protocolo descrito por Guillamón et al. (1998) (13). El patrón de bandas fue comparado con la base de datos de la Universidad de Valencia que incluye los tamaños de los fragmentos de restricción para diferentes especies de levaduras.
El producto de digestión se evidenció mediante electroforesis en geles de agarosa al 1% corridos a 100 V durante 90 minutos en buffer TBE 0,5 X, teñidos con bromuro de etidio. Los patrones de bandas fueron analizados mediante el programa PhotoCaptMw versión 10,0.
A partir de esta técnica se agruparon los 26 aislados de acuerdo con su patrón de digestión. Todos los aislados fueron analizados mediante MSP-PCR y amplificados en su región 26S ADNr para su secuenciamiento, mismo que fue efectuado únicamente en uno o dos aislados representantes de cada grupo.
MSP-PCR (Micro/Minisatellite-Primed PCR)
En el análisis de MSP-PCR, se emplearon los primers sintéticos (GTG)5 en experimentos de micro-satélites y la secuencia central del fago M13 (5'-GAG GGT GGC GGT TCT-3') en experimentos de mini-satélites (3). Las reacciones de PCR fueron realizadas en un volumen final de 25 µL, conteniendo 5 µL de buffer GoTaq® _5X; 1,5 µL de MgCl2 50 mM; 2,5 µL mix dNTPs 10 mM; 4 µL de primer M13 5 µM ó 4 µL de primer (GTG)5, 5µM; 0,4 µL de GoTaq® polimerasa, 5 u µL-1; 6,6 µL de agua libre de nucleasas y 5 µL de dilución de ADN 3:1000 (µL) previamente obtenido en la extracción de ADN. Los primers se utilizaron de manera independiente para cada reacción. El programa de amplificación constó de una desnaturalización inicial de 5 min a 95°C; seguido por 40 ciclos de 45s a 93°C; 60 s a 55°C; 60 s a 72°C; y, una extensión final de 6 min a 72°C.
El producto de PCR se evidenció mediante electroforesis en geles de agarosa al 1%, corridos a 100 V durante 90 minutos en buffer TBE 0,5 X. Los geles fueron teñidos con bromuro de etidio. El producto fue evidenciado mediante transluminador (Spectroline Ultraviolet Translluminator) y las imágenes se registraron por un sistema de documentación digital (Fisher Bioblock Scientific).
Amplificación del dominio D1/D2 del segmento 26S ADNr mediante PCR
Para el secuenciamiento que llevó a la identificación de especies, se amplificó el dominio D1/D2 del segmento 26S del ADNr mediante los primers NL1 (5'–GCATATCA ATAAGCGGAGGAAAAC-3') y NL4 (5'–GGTCCGT GTTTCAAGACGG-3') (14). La reacción de PCR se realizó en un volumen final de 50 µL conteniendo 10 µL de buffer 5 X GoTaq®; 1 µL mix dNTPs 10 mM; 1,5 µL de MgCl2, 25 mM; 1 µL de primer NL1 0.1 µM; 1 µL de primer NL4 0.1 µM; 30,1 µL de agua libre de nucleasas; 0,4 µL de GoTaq® polimerasa, 5µL-1; y, 5 µL de ADN templado. Las condiciones utilizadas en el termociclador consistieron en una desnaturalización inicial de 17 min a 94°C; 40 ciclos de 30 s a 95°C; 45 s a 60°C y, extensión final de 45 s a 72°C.
Los amplicones fueron enviados a secuenciar a la National Collection of Yeast Cultures (NCYC) en Norwich (Reino Unido) y en el Instituto de Ciencias Biológicas de la Universidad Federal de Minas Gerais (Brasil). La secuenciación en ambos casos se realizó usando un kit de ciclo terminador ABI BigDye versión 3,1 (Applied Biosystems) con los primers NL1 y NL4.
Curvas de crecimiento
Se inoculó 0,05 g de levadura liofilizada en 150 mL en YPD caldo (Yeast Peptone Dextrose) (g L-1): glucosa, 20; extracto de levadura, 10; peptona, 20 y agar, 20 (16); y se incubó durante 24 horas a la temperatura favorable previamente determinada para cada aislado con agitación de 260 rpm, en un agitador rotatorio (Microprocessor Shaker Bath, LAB-Line). Se midió la densidad óptica (DO600) de cada cultivo cada hora hasta obtener la gráfica de la curva de crecimiento, para lo que se empleó un espectrofotómetro HEëIOS â (Thermo Spectronic) (11).
Resultados y discusión
De las diferentes especies de levaduras pigmentadas que han sido identificadas en el presente estudio dentro de los géneros Rhodotorula, Rhodosporidium y Sporobolomyces, se evidencia variabilidad morfológica, bioquímica y molecular dependiendo de cada especie.
Se evidencia en general que existe una correlación entre el tipo de sustrato y la diversidad de especies que pueden encontrarse en ambientes naturales. De esta manera, especies que difieren de Rhodotorula mucilaginosa, como son Rhodosporidium babjevae, Rhodotorula glutinis, R. slooffiae y Rhodotorula sp. provienen, en su mayoría, de vasijas y metates de excavaciones arqueológicas realizadas en las localidades de La Florida y el yacimiento arqueológico de Rumipamba (Quito); mientras que, Sporidiobolus ruineniae es una especie que fue hallada en bosques de las provincias de Loja y Orellana..
Los aislados de Rhodotorula mucilaginosa se caracterizan por presentar colonias mucoides en diferentes tonalidades salmón y rosa (Tabla 2, Figura 1). Asimilan de manera positiva la sacarosa y fructosa en relación al resto de azúcares utilizados donde se da una asimilación lenta o débil (Tabla 3).
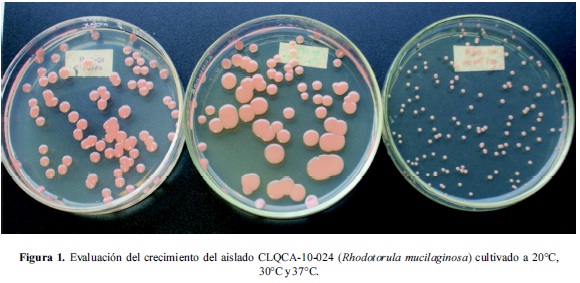
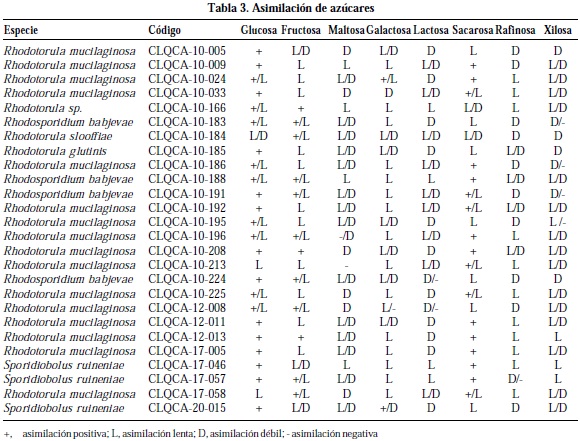
Las colonias que pertenecen a Rhodosporidium babjevae se caracterizan por tener una consistencia butirosa o cremosa, y una predominancia del color salmón. Asimilan de manera positiva la fructosa (Tabla 3). La temperatura que resultó más favorable para su crecimiento fue 20°C.
Sporidiobolus ruineniae presenta colonias cremosas color rosa claro y salmón. El azúcar que asimila de mejor manera es la sacarosa, en comparación con el resto de azúcares que los asimila de manera lenta o débil.
Al comparar la base de datos de levaduras pigmentadas actualmente identificadas de la CLQCA con la base de datos de levaduras NCYC "National Collection of Yeast Cultures"; CBS "Centraalbureau voor Schimmelcultures" Holanda y Barnett et al. (2007) "Yeasts: Characterization and Identification" se puede evidenciar que la especie Rhodotorula mucilaginosa es la más abundante en lo que se refiere a levaduras pigmentadas en todas las colecciones, incluida la CLQCA, donde fue realizado este trabajo.
La segunda especie más representativa en la CLQCA fue Rhodosporidium babjevae (4 aislados) con mayor número de ejemplares que en las otras colecciones anteriormente mencionadas. Sporidiobolus ruineniae está presente en la CLQCA con 3 aislados; mientras que Rhodotorula glutinis, Rhodotorula slooffiae y Rhodotorula sp. se encuentran representadas por un solo aislado.
De los 26 aislados con los que se trabajó, se observó un crecimiento conspicuo de 13 aislados a 30°C (50%); 9 aislados a 20°C (34,6%); y en los 4 aislados restantes se observó un mejor crecimiento a 37°C (15,3%).
Evaluación de respuestas de crecimiento en compuestos carbonados
Las levaduras pigmentadas en general asimilaron de mejor manera los azúcares: glucosa, sacarosa y fructosa, en comparación con el resto de azúcares que fueron asimilados de manera débil o lenta. Al comparar los resultados obtenidos en asimilaciones negativas sobre maltosa, galactosa y lactosa; los datos coinciden con la bibliografía sobre la ambigüedad que puede presentarse en distintos aislados para asimilar estos azúcares (12).
Identificación molecular de los aislados estudiados
Mediante RFLP-ITS, se evidenció un patrón de digestión para cada especie debido a que los organismos difieren en la distancia de los sitios de clivaje para cada enzima de restricción en diferentes especies. Por otro lado, la similitud de los patrones generados permitió establecer diferencias dentro de las seis especies y entre ellas (2). Los patrones de digestión que coincidieron con las especies se muestran en la tabla 4.
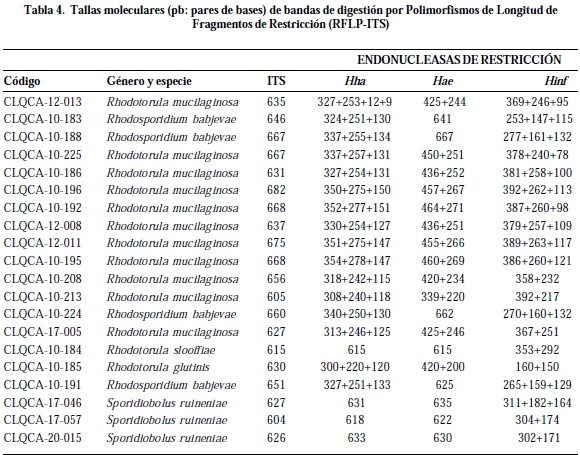
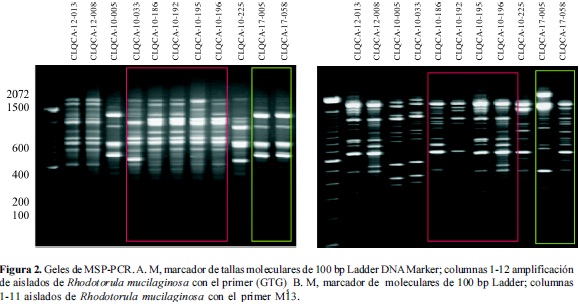
Según la técnica de RFLP, para los aislados de R. mucilaginosa el patrón de digestión fue similar dentro de la especie. Esta identificación preliminar fue confirmada por medio de la secuenciación obtenida del fragmento 26S ADNr. Por la técnica de MSP-PCR se agruparon los aislados evidenciando que existe un perfil homogéneo en la mayoría de aislados de los mismos sustratos o localidades. Por ejemplo, aislados extraídos de vasijas y metates tienen un perfil de ADN similar entre ellos, sugiriendo que se trata de aislados que corresponden a la misma cepa (CLQCA-10-186, CLQCA-10-192, CLQCA-10-195, CLQCA-10-196), así como aislados de la provincia de Loja (CLQCA-17-005, CLQCA-17-058) (Figura 2).
Para los aislados pertenecientes a Rhodosporidium babjevae y Sporidiobolus ruineniae analizados mediante las técnicas de RFLP y MSP-PCR, el perfil de ADN fue similar a nivel intraespecífico para cada una de estas especies. Mientras que, el resto de especies no pudo ser evaluado mediante estas técnicas debido a que se encontró solo un aislado, por lo que los resultados de secuenciación de su región 26S ADNr fueron determinantes para su identificación.
MSP-PCR (Micro/Minisatellite-Primed PCR)
A través de la técnica de MSP-PCR y empleando individualmente los primers (GTG)5 y M13, se obtuvo los perfiles de ADN para los 26 aislados. Se evidenció polimorfismo para la especie R. mucilaginosa con el primer (GTG)5. Estos datos contrastan con estudios realizados por Libkind (2007) donde el primer (GTG)5 permitió diferenciar a la especie R. mucilaginosa de otras, mientras que los resultados obtenidos con el primer M13 dieron perfiles homogéneos. Esto sugiere que el primer M13 puede amplificar regiones más conservadas y, por lo tanto, tendría menor grado de resolución para diferenciar especies emparentadas con R. mucilaginosa (10). Como se puede ver en las figuras 2A y 2B, en el caso de la amplificación con el primer (GTG)5 existen patrones muy similares, mientras que en el caso de la amplificación con el primer M13, los patrones evidencian diferencias importantes que entre los diferentes aislados.
Amplificación del dominio D1/D2 del segmento 26S ADNr mediante PCR
A partir de la amplificación de la región 26S rADN se realizó el secuenciamiento de los aislados en dos grupos. El primer grupo fue secuenciado en la NCYC en Norwich- Inglaterra (julio de 2007), donde los aislados CLQCA-10-005, CLQCA-10-009, CLQCA-10-024, CLQCA-10-033, CLQCA-12-013 y CLQCA-17-058 fueron identificados como Rhodotorula mucilaginosa (99-100%), mientras que el aislado CLQCA-10-166 fue identificado como Rhodotorula slooffiae ó Rhodotorula minuta con una similitud del 97,7%-97,6% respectivamente.
El segundo grupo de amplicones a secuenciar correspondió a los aislados seleccionados de acuerdo a los grupos formados según el patrón de bandas de las técnicas moleculares previamente empleadas.
Curvas de crecimiento
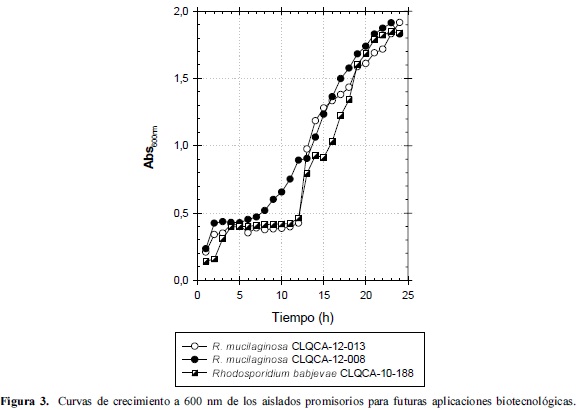
El tiempo en que se dan las diferentes fases de crecimiento varía entre cada aislado y entre especies. Los aislados de R. mucilaginosaespecie altamente representada en la colecciónpresentaron una fase de latencia corta de 8 horas, con una duración de 7 horas para llegar a su fase estacionaria. Los siguientes aislados que muestran una potencial habilidad de crecer en este medio es la perteneciente a la especie Rhodotorula sp., así como también Rhodotorula glutinis y Rhodosporidium babjevae, seguidos por Sporidiobolus ruineniae al presentar una fase de latencia más larga. Esta fase tiene gran importancia en la microbiología industrial porque se puede predecir la evolución de un cultivo microbiano bajo factores que influyen en esta etapa de crecimiento, tales como medio y condiciones iniciales del inóculo (15), (Figura 3).
Conclusiones
La predominancia de levaduras pigmentadas en la CLQCA está determinada por R. mucilaginosa, como también en otras colecciones como NCYC y CBS.
Los aislados de R. mucilaginosa con los códigos: (CLQCA-12-013 y CLQCA-12-008) y el aislado de Rhodosporidium babjevae con el código (CLQCA-10-188), fueron encontrados como los aislados con mejores características de crecimiento y producción de intensidad de color; por lo que se sugiere, podrían ser escogidos para futuros estudios que lleven a la producción industrial de pigmentos. El aislado CLQCA-12-013 al presentar características de crecimiento rápido, asimilación de azúcares y mayor intensidad de su coloración, es considerado como un aislado promisorio para futuras aplicaciones industriales.
Agradecimientos
A la Pontificia Universidad Católica del Ecuador por el financiamiento para esta investigación. Al Institute of Food Research, Nacional Collection of Yeast Cultures y Universidade Federal de Minas Gerais por su colaboración. Al Dr. Carlos Rosa y Dr. Diego Libkind por su apoyo para la realización de este proyecto.
Financiación
El proyecto fue financiado con fondos de la Pontificia Universidad Católica del Ecuador, entre el año 2007 y el 2009.
Conflictos de intereses
Los autores no tienen conflicto de intereses relacionados con la investigación aquí presentada.
Referencias
1. Amitabha D, Sang-Hwal Y, Sook-Hee Lee, Jae-Yean K, Deok-Kun O, Seon-Won K. An update on microbial carotenoid production: application of recent metabolic engineering tools. Applied Microbiology and Biotechnology 2007; 77:505-512. [ Links ]
2. Orberá T. Métodos moleculares de identificación de levaduras de interés biotecnológico. Revista Iberoamericana de Micología 2004; 21(1):15-19. [ Links ]
3. Hierro N, González A, Mas A, Guillamón J. New PCRbased methods for yeast identification. Journal of Applied Microbiology 2004; 97:792–801. [ Links ]
4. Libkind D, Diéguez M, Moline M, Pérez P, Zagarese H, Van Broock M. Occurrence of Photoprotective Compounds in Yeasts from Freshwater Ecosystems of Northwestern Patagonia (Argentina). Photochemistry and Photobiology 2006; 82:972–980. [ Links ]
5. Buzzini P, Innocenti M, Turchetti B, Libkind D, Van Broock M, Molinacci N. Carotenoid profiles of yeast belonging to the genera Rhodotorula, Rhodosporidium, Sporobolomyces, and Sporidiobolus. Canadian Journal of Microbiology 2007; 53:1024-1031. [ Links ]
6. Sánchez A, Flores L, Langley E, Martín R, Maldonado G, Sánchez S. Carotenoides: Estructura, Función, Biosíntesis, Regulación y Aplicaciones. Revista Latinoamericana de Microbiología 1999; 41:175-191. [ Links ]
7. Bandaranayake W. Mycosporines: are they nature's sunscreens? Natural Product Reports 1998; 15:159-172. [ Links ]
8. Libkind D, Moliné M, Van Broock M. Posibles mecanismos de fotoprotección en levaduras. Revista electrónica de Radiobiología 2004; 4:84-88. [ Links ]
9. Libkind D, Pérez P, Sommaruga R, Diéguez M, Ferraro M, Brizzio S, Zagarese H, Van Broock M. Constitutive and UV-inducible synthesis of photoprotective compounds (carotenoids and mycosporines) by freshwater yeasts. Photochemical and Photobiological Sciences 2004, 3(3):281-286. [ Links ]
10. Libkind D. Evaluación de la técnica de MSP-PCR para la caracterización molecular de aislamientos de Rhodotorula mucilaginosa provenientes de la Patagonia noroccidental. Revista Argentina de Microbiología 2007; 39:133-137. [ Links ]
11. Margesin R. Effect of temperature on growth parameters of psychrophilic bacteria and yeasts. Extremophiles 2009; 13:257-262. [ Links ]
12. Barnett J, Payne R, Yarrow D. YEASTS. Characteristics and identification. Third Edition. Printed in the United Kingdom at the University Press, Cambridge. United States of America. 2007, 1139 p. [ Links ]
13. Guillamón J, Sabaté J, Barrio E, Cano J, Querol A. Rapid identification of wine yeast species based on RFLP analysis of the ribosomal internal transcribed spacer (ITS) region. Archives in Microbiology 1998; 169:387–392. [ Links ]
14. James S, Carvajal E, Bond C, Cross K, Núñez N, Portero P, Roberts I. Candida carvajalis sp.nov., an ascomycetous yeast species from the Ecuadorian Amazon jungle. FEMS Yeast Research 2009; 1: 1–5. [ Links ]
15. Cattaneo C, Larcher L, Togo S, Chaillou L. Aplicación de métodos de Monte Carlo para estudio de crecimiento de bacterias y levaduras. Mecánica Computacional 2007; 36:3380-3393. [ Links ]
16. Libkind D. Levaduras carotenogénicas de ambientes acuáticos de la Patagonia Noroccidental Argentina. Aplicaciones biotecnológicas. Trabajo para la obtención del grado académico superior de doctor en bioquímica. 2006. Argentina. [ Links ]














