Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Ciencia y Tecnología Agropecuaria
versão impressa ISSN 0122-8706
Corpoica cienc. tecnol. agropecu. vol.13 no.2 Mosquera jun./dez. 2012
MICROBIOLOGÍA DEL SUELO
1 Centro de Investigación Tibaitatá, Corporación Colombiana de Investigación Agropecuaria (Corpoica). Mosquera (Colombia). rvalencia@corpoica.org.co
2 Centro de Biotecnología y Bioindustria, Centro de Investigación Tibaitatá, Corporación Colombiana de Investigación Agropecuaria (Corpoica). Mosquera (Colombia).
Fecha de recepción: 23-03-2012. Fecha de aceptación: 02-11-2012
RESUMEN
El cultivo de la soya (Glycinemax (L.) Merril), en oxisoles de la Orinoquia colombiana presenta restricciones por la alta saturación de aluminio y en general, baja capacidad de intercambio catiónico (CIC) y de materia orgánica (MO), que afectan la fijación biológica del nitrógeno (N) y el crecimiento normal de las plantas. Una alternativa ambiental, a este problema es inocular las plantas con diferentes cepas de Bradyrhizobium japonicum. Las cepas de B. japonicum J-96 y J-98 se constituye en una opción de fijación biológica de nitrógeno altamente efectivas para las variedades de soja, en la Orinoquia colombiana. En este trabajo se determinaron los patrones moleculares de cepas J-01, J-96 y J-98, a partir de los primers Box-PCR y Eric- PCR. Se logró establecer diferencias genéticas entre estas, las cuales pueden ser de utilidad para complementar futuros programas de identificación de individuos, estudios poblacionales y selección asistida. Las cepas de B. japonicum J-96 y J-98, constituyen nuevas opciones de variedades biológicas para fijación de nitrógeno de soya altamente eficaces en el Orinoco colombiano.
Palabras clave: variedad, polimorfismo, oxisol, iniciador
ABSTRACT
The cultivation of soybeans (Glycine max (L.) Merril), in oxisols of the Colombian Orinoco has restrictions due to high aluminum saturation and generally low CEC and OM, which affect the biological fixation of N and the normal growth of plants. In response to this problem, the plants are inoculated with different strains of Bradyrhizobium japonicum. The B. japonicum strains J-96 and J-98 are a highly effective choice for biological nitrogen fixation in soybean varieties in the Colombian Orinoco. This study determined the molecular patterns of the strains J-01, J-96 and J-98 with the primers Box- PCR and Eric-PCR; genetic differences were established, which can be useful for future programs of identification of individuals, population studies and assisted selection. The B. japonicum strains J-96 and J-98 are highly effective new options of biological varieties for nitrogen fixation of soybeans in the Colombian Orinoco.
Keywords: variety, polymorphism, oxisol, initiator
INTRODUCCIÓN
El cultivo de la soya (Glycine max (L.) Merril), en oxisoles de la Orinoquia colombiana presenta restricciones, debido a la condiciones propias de estos suelos, es decir, un pH promedio de 4,5, baja la disponibilidad de P, Ca, Mg, K (Amézquitaet al., 2005; Rivas et al., 2004; Molina et al., 2003), baja capacidad de intercambio catiónico, materia orgánica y alta fijación de fosfatos (Borkert y Sfredo, 1995). Además poseen fuertes problemas de fitotoxicidad por aluminio (Bianchi et al., 2000), todo estos factores limitan el crecimiento de las plantas, su productividad agrícola (Von y Mutert, 1995) y la fijación biológica de nitrógeno. Este último requerimiento es suplido a partir de asociaciones simbióticas con cepas de Bradyrhizobium japonicum, ofertadas en pequeña escala por empresas privadas, las cuales fueron adoptadas por los agricultores para su uso en este tipo de suelos. La soya tiene la habilidad de asociarse en forma simbiótica con estas bacterias para obtener a través de la fijación biológica del nitrógeno (FBN) gran parte del N que requiere para su crecimiento (Hungria et al., 1998; Perticari et al., 2003), con aportes hasta del 94% del N en los cultivares más productivos (Hungria et al., 2006). Sin embargo esta situación puede estar restringiendo la expresión del potencial genético de las nuevas variedades por la baja efectividad simbiótica de las cepas. Fernández (2003), asegura que la ocurrencia de factores ambientales adversos (estrés por temperatura, humedad, acidez, etc.) afecta la supervivencia del rizobio, disminuyendo el número y peso de nódulos de la población de cepas, reduciendo la producción de biomasa vegetal.
La especificidad del Rhizobium por un determinado genotipo varietal está determinada en su código genético. En la evaluación de cepas de Rhizobium se ha sugerido la existencia de una amplia variación genética intraespecífica. Se ha encontrado que el 70% del ADN es común entre cepas, mientras que el resto es variable de cepa a cepa (López et al., 2009). La variación intraespecífica está relacionada con la capacidad de las cepas para adaptarse y sobrevivir en distintas condiciones. Numerosos estudios han demostrado que la transferencia horizontal juega un papel importante en la evolución y grado de especificidad de los rizobios (López et al., 2009). Además, la diversidad genómica y la transferencia horizontal de genes en las poblaciones del suelo incrementan la plasticidad de adaptación y la supervivencia de rhizobacterias en el medio ambiente, en comparación con la cepa inoculada, que es normalmente un solo genotipo (López et al., 2009).
El uso de técnicas moleculares ha provisto una descripción precisa de Rhizobium. Los estudios de identificación de individuos y diversidad genética basados en las técnicas de PCR, se logran generalmente con iniciadores o cebadores universales que generan fragmentos que con frecuencia son llamadas huellas dactilares genómicas o fingerprints genómico. Tres protocolos para la generación de huellas genómicas, han sido comúnmente empleados en bacterias, incluidos los preparados arbitrariamente AP-PCR o los RAPDs (Randomly Amplified Polymorphic), los AFLP (Amplified Fragment Length Polymorphism) y las secuencias repetidas basadas en rep-PCR. Cada uno de estos protocolos genera una colección de fragmentos del genoma a través de PCR, que se visualizan como patrones de bandas que ofrecen un alto nivel de resolución taxonómica (Louws et al., 1999).
La utilidad del método rep-PCR, desarrollado para clasificar a las bacterias con base en sus patrones de huellas dactilares genómicas, se fundamenta en el uso de iniciadores de ADN correspondientes a los elementos repetitivos naturales intercalados en las bacterias, como el REP (Repetitive Extragenic Palindromic), ERIC (Entero-bacterial Repetitive Intergenic Consensus) y BOX (Entero-bacterial Repetitive Sequences), y la reacción de PCR, que colectivamente reciben la denominación de rep-PCR. Estas secuencias repetitivas conservadas en diversos géneros de bacterias, han sido estudiadas en más detalle por Versalovic et al. (1994) y sus secuencias parecen estar situadas en distintas posiciones intergénicas de todo el cromosoma y en ambas orientaciones.
Estudios realizados por De Bruijn et al. (1996), demostraron que el método de huellas digitales rep-PCR, es simple, altamente reproducible y no es absolutamente dependiente de ADN puro para distinguir cepas estrechamente relacionadas, o para deducir las relaciones filogenéticas entre cepas bacterianas y estudiar su diversidad en una variedad de ecosistemas. Igualmente, Judd et al. (1993) y Schloter et al. (2000), determinaron que las secuencias repetidas REP y ERIC utilizadas en conjunto con la técnica de reacción en cadena de la polimerasa (REP y ERIC-PCR) son altamente eficaces para diferenciar y clasificar genéticamente cepas de B. japonicum relacionadas. La amplificación por PCR de regiones genómicas entre copias REP o ERIC produce una colección de fragmentos distintos o patrón de huella digital típico, en un gel de agarosa (De Bruijn, 1992). Similares propiedades han sido observadas con las secuencias repetidas tipo BOX, las cuales son de gran utilidad para realizar discriminaciones a nivel de cepa (Giongo et al., 2008). Las secuencias repetidas REP, ERIC y BOX, son altamente conservadas en los genomas de rizobios. Xue et al. (1999), encontraron que las regiones intergénicas 16S y 23S ADNr pueden ser usadas para caracterizar rizobios de soya a nivel de especie, subespecie, respectivamente. Sin embargo, para la diferenciación intraespecífica de B. japonicum, las secuencias repetidas rep-PCR han demostrado ser de mayor utilidad (Ferreira y Hungria, 2002; Giongo et al., 2008).
La falta de nuevas alternativas de fijadores biológicos de nitrógeno, el escaso conocimiento de la capacidad simbiótica de las cepas de rizobio en suelos de la Altillanura y el incremento progresivo de las aplicaciones de fertilizantes nitrogenados para abastecer los requerimientos de la planta, hacen necesario la caracterización molecular de las cepas de B. japonicum J-01, J-96 y J-98, de esta forma, poder establecer las diferencias y similitudes moleculares entre estas y construir los perfiles moleculares. Lo anterior es de gran utilidad para futuros estudios de diversidad y selección de cepas superiores en la fijación simbiótica de N. Los nódulos de donde fueron aisladas las bacterias de estudio, fueron obtenidas en oxisoles de la Orinoquia colombiana, en latitudes entre 04° 03' N y 73° 29' O, con altitudes entre 150 y 336 msnm.
MATERIALES Y MÉTODOS
Cepas de B. japonicum
Las cepas de Bradyrhizobium en estudio pertenecen a la siguiente nomenclatura: ICA J-98 o SEMIA 5079, también denominada CPAC 15 y conocida en algunos serogrupos como SEMIA 566; la cepa J-96 o SEMIA 5080 o variante CPAC 7; la ICA J-01 o variante de USDA 110, denominada también 3I1b110, TAL102, RCR3427, 61A89 o SEMIA 5032 (Chen et al., 2000). ICA J-01 fue generada por el ICA (Salamanca y Ramírez, 2000) y las cepas J-96 y J-98 del banco de germoplasma de Corpoica fueron introducidas del Brasil. Las dos primeras cepas son utilizadas como inoculante comercial en Brasil (Boddey y Hungria, 1997) y la ICA J-01 de uso comercial en Colombia (Salamanca y Ramírez, 2000).
Aislamiento de rizobios y de ADN bacteriano
Para el aislamiento de rizobios de las cepas de B. japonicum ICA J-01, J-96 y J-98 y crecimiento bacteriano, se siguió el protocolo estandarizado por el Laboratorio de Microbiología de suelos del Centro de Biotecnología y Bioindustría (CBB) de Corpoica en el Centro de Investigación Tibaitatá. Se usaron tres nódulos tomados de campo por cepa para extraer el contenido nodular mediante maceración, estos fueron previamente desinfectados con etanol al 95% por 30 s e hipoclorito de sodio al 3,25% por 2 min y luego enjuagados cinco veces con agua destilada estéril. El contenido nodular en suspensión con agua estéril se sembró en cajas de Petri con extracto de levadura y manitol (YEM) suplementado con rojo congo 0,0025% (Vincent, 1970). Las cepas aisladas se purificaron después de repiques sucesivos y se almacenaron en tubos inclinados con medio YEM a 4°C. El procedimiento anterior se hizo en condiciones asépticas de laboratorio. Para la extracción del ADN genómico de las cepas de referencia del banco de germoplasma y sus correspondientes aislamientos a partir de nódulos, se utilizó el método del fenol cloroformo descrito por Rakhi et al. (2006).
rep-PCR
Las reacciones PCR con secuencias repetidas altamente conservadas se realizaron con los iniciadores ERIC1R (3'-CACTTAGGGGTCCTCGAATGTA-5'), ERIC2(5'-AAGTA AGTGACTGGGGTGAGCG-3'), y BOXA1 (CTACGGC AAGGCGACGCTGACG), descritos por Versalovic et al. (1994). Estas fueron realizadas a un volumen final de 25 mL, que contenían 50 ng de ADN, 1 U de Taq ADN polimerasa, 1,5 mM de MgCl2, 2,0 mM de dNTPs y 0,1 mM de cada iniciador. El proceso de amplificación de los iniciadores BOX y ERIC consistió de 1 ciclo inicial de desnaturalización 95°C por 6 min, 35 ciclos de desnaturalización con la siguiente secuencia: 94°C por 1 min, 50°C por 1 min y 65°C por 8 min, y un 1 ciclo final de extensión de 65°C por 16 min. La PCR fue corrida en un termociclador PT100 (MJ-Research, Watertown, MA) y los fragmentos fueron visualizados por electroforesis sobre un gel de agarosa al 1,5% (P/V) teñido con bromuro de etidio al 0,1 mg mL-1 y corrido a 100 V por 3 h. El tamaño de las bandas generadas fue establecido con base en el marcador de peso molecular 1 kb Plus DNA ladder (Gibco® BRL). Cada amplificación y visualización se realizó por triplicado para verificar su reproducibilidad y la consistencia del patrón de bandas.
Análisis de la información
Las bandas obtenidas por cada iniciador fueron transformadas en una matriz binaria basada en la presencia (1) o ausencia (0) de cada fragmento amplificado. Con el paquete estadístico NTSYpc versión 2.02 (Rohlf, 1997), se determinó el índice de similitud de Dice (1945).
RESULTADOS Y DISCUSIÓN
Se obtuvieron perfiles o patrones moleculares de las cepas de referencia del Banco de Germoplasma de Corpoica de B. japonicum J-01, J-96 y J-98 y de aislamientos de nódulos. Cada perfil obtenido mediante los protocolos BOX y ERIC-PCR, presentó múltiples bandas de amplificación con resultados reproducibles en diferentes pruebas. Las diferencias moleculares entre cepas se hicieron evidentes con los dos protocolos de rep-PCR.
Para las cepas de referencia, el patrón más complejo se obtuvo con ERIC-PCR al generarse 16 fragmentos totales. Resultados similares fueron obtenidos por Ferreira y Hungria (2002), quienes encontraron un promedio de 15 bandas en cepas de B. japonicum. Las bandas amplificadas tuvieron tamaños entre 420 y 3.500 pb, con la presencia de una banda común de 1.200 bp, entre cepas. El mayor bandeo se presentó en la J-96 y el menor en la J-01 (Figura 1A). El mayor polimorfismo se observó entre 3.000 y 3.500 pb y entre 420 y 900 pb.
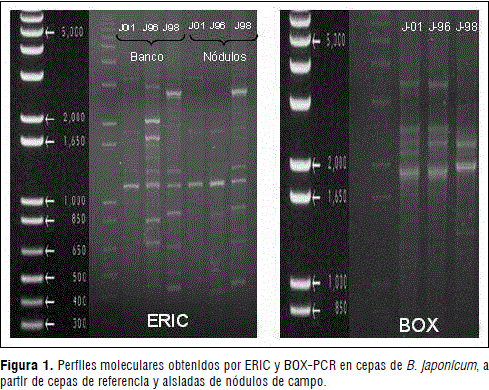
Con BOX-PCR las bandas presentaron un tamaño entre 1.900 pb y 3.800 pb, en un patrón molecular de seis fragmentos. Las cepas J-01 y J-96 compartieron cuatro de las seis bandas, mientras que J-98 solo compartió una banda de 2.500 pb con las otras dos cepas (Figura 1B).
Aunque ERIC presentó mayor fragmentación y polimorfismo que BOX, los dos sistemas permitieron establecer diferencias genéticas intraespecíficas entre cepas de B. japonicum. Sin embargo, los patrones moleculares ERIC-PCR obtenidos a partir de nódulos con respecto a las cepas de referencia del Banco de Germoplasma fueron diferentes. Las mayores diferencias se observaron con la cepa J-96, seguida por la J-01, mientras que la J-98 fue muy estable. Estos resultados concuerdan con los resultados obtenidos por Batista et al. (2007), quienes encontraron que la variabilidad entre B. japonicum y B. elkanii puede ser tal que incluso dentro de un grupo genéticamente más estable (como la CPAC 15), sólo el 6,4% de los aislamientos mostraron una alta similitud a la cepa inoculante, mientras que ninguno fue similar a la CPAC 7. La variabilidad genética parece ser el resultado de una variedad y combinación de eventos, incluyendo la dispersión de la cepa, la recombinación genómica y la transferencia horizontal de genes. Además, la variabilidad genética de las cepas parece que se asocia principalmente con la adaptación, capacidad saprofita y competitividad, y no con la eficacia simbiótica, ya que la similitud de los genes simbióticos fue superior a la de regiones conservadas del ADN.
Con la combinación de los patrones moleculares producidos por ERIC y BOX, se determinaron los índices de similitud entre cepas. Se observó una mayor cercanía entre J01 y J96, con un índice de Dice de 0,45 y distantes de J98, con una similitud de 0,33 y 0,27, respectivamente. Investigaciones realizadas por Batista et al. (2007), demuestran mediante perfiles rep-PCR menor variabilidad genética en cepa CPAC 15 (origen de la cepa J-98) y mayor en CPAC 7 (origen de la cepa J-96). La alta capacidad saprofita y competitiva de la cepa CPAC 15 después de varios años de cultivo, contrastó con la pobre supervivencia y competitividad de la cepa CPAC 7. Existe evidencia de las diferencias genéticas entre cepas parentales y las aisladas a partir de sus rizobios en campo, como consecuencia de largos períodos con alta temperatura y estrés hídrico (Hungria y Vargas, 2000), aunque la trasferencia horizontal de genes puede también estar obrando en esta diversidad (Barcellos et al., 2007).
El presente estudio permitió encontrar diferencias moleculares entre cepas y con capacidad diferencial para nodular, ratificando que los análisis de variabilidad genética usando los marcadores BOX y ERIC-PCR son útiles para genotipificar bacterias, siendo aplicados con éxito para caracterizar intraespecíficamente a B. japonicum (Judd et al.,1993; Xue et al., 1999; Ferreira y Hungria, 2002; Giongo et al., 2008).
Estos resultados podrían ser útiles para la identificación de nuevas cepas y formulación de nuevos inoculantes con mayor capacidad para fijar N, lo que permitirá incrementar la productividad en el cultivo de la soja en oxisoles de la Orinoquia colombiana. Varios trabajos de investigación (Hungria et al., 1996; Santos et al., 1999), reportan la existencia de diferencias entre cepas parentales y sus variantes, en formación de moco, síntesis de IAA, perfil del factor Nod y el patrón molecular de ADN obtenido con iniciadores arbitrarios o específicos. Godoy et al. (2008), encontraron en estudios genómicos diferencias genéticas notorias entre las cepas CPAC 15 y USDA 110, de las cuales se derivaron las cepas J-98 e ICA J-01, respectivamente, en aspectos de nodulación, relación planta huésped y adaptación, y genes relacionados con el sistema de secreción y transporte.
Es importante considerar que, no existen cepas indígenas o nativas en Brasil (Ferreira, y Hungria, 2002), ni en Colombia, por no ser centros de origen. Sin embargo, la siembras sucesivas de soyas inoculadas con rizobios introducidos, han generado en estos países cepas naturalizadas que han logrado adaptarse a las nuevas condiciones edafoclimáticas y que se constituyen en fuente de diversidad biológica para procesos de selección de cepas más efectivas para fijar N.
CONCLUSIONES
El método de genotipificación basado en secuencias ERIC y BOX presentes en bacterias de B. japonicum, mediante procesos de PCR fue altamente efectivo para diferenciar cepas estrechamente relacionadas. Por ello, se constituye en una herramienta genética de alto valor para estudios de identificación y diversidad de B. japonicum.
El sistema ERIC-PCR, generó mayor polimorfismo que BOX-PCR en las cepas bajo estudio, lo que le confirió mayor precisión en la diferenciación de capas B. japonicum.
AGRADECIMIENTOS
Los autores expresan sus agradecimientos a la Corporación Colombiana de Investigación Agropecuaria (Corpoica) por el apoyo, para la realización de esta investigación, especialmente a los Laboratorios de Genética Molecular Vegetal y Microbiología de Suelos (Tibaitatá).
REFERENCIAS BIBLIOGRÁFICAS
Amézquita E, Hoyos Ph, Molina D. 2005. Mejoramiento de la calidad del suelo en rotación maíz-soya en la altillanura colombiana. En: Resúmenes III Seminario Regional Agrociencia y Tecnología Siglo XXI Orinoquia Colombiana. Villavicencio, Colombia: Corpoica. p. 45. [ Links ]
Barcellos F, Menna P, Da Silva Batista J, Hungria M. 2007. Evidence of horizontal transfer of symbiotic genes from a Bradyrhizobium japonicum inoculant strain to indigenous diazotrophs Sinorhizobium (Ensifer) fredii and Bradyrhizobium elkanii in a Brazilian Savannah soil. Appl Environ Microbiol 73:2635-2643. [ Links ]
Batista J, Hungria M, Barcellos F, Ferreira M, Mendes I. 2007. Variability in Bradyrhizobium japonicum and B. elkanii seven years after introduction of both the exotic microsymbiont and the soybean host in a Cerrados soil. Microbial Ecol 53:270-284. [ Links ]
Bianchi C, Carter T, Bailey Jr M, Mian M, Rufty T, Ashley D, Boerma H, Arellano C, Hussey R, Parrott W. 2000. Aluminum tolerance associated with quantitative trait loci derived from soybean PI 416937 in hydroponics. Crop Sci 40:538-545. [ Links ]
Boddey L, Hungria M. 1997. Phenotypic grouping of Brazilian Bradyrhizobium strains which nodulate soybean. Biol Fertil Soils 25:407-415. [ Links ]
Borkert C, Sfredo G. 1995. La fertilización de los suelos tropicales para el cultivo de la soya. En: El cultivo de la soya en los trópicos. Mejoramiento y producción. Roma: Colección FAO: Producción y Protección Vegetal No. 27. pp. 175-200. [ Links ]
De Bruijn F. 1992. Use of repetitive (repetitive extragenic palindromic and enterobacterial repetitive intergenic consensus) sequences and the polymerase chain reaction to fingerprint the genomes of Rhizobium meliloti isolates and other soil bacteria. Appl Environ Microbiol 58(7):2180-2187. [ Links ]
De Bruijn F, Rademaker J, Schneider M, Rossbach U, Louws F. 1996. Rep-PCR genomic fingerprinting of plant-associated bacteria and computer-assisted phylogenetic analyses. En: Stacey G, Mullin B, Gresshoff P, editores. Biology of plant-microbe interaction; Proceedings of the 8th International Congress of Molecular Plant-Microbe Interactions. Saint Paul, MN: APS Press. pp. 497-502. [ Links ]
Dice LR. 1945. Measures of the amount of ecologic association between species. Ecology 26:297-302. [ Links ]
Fernández CM. 2003. Manual de nodulación. Buenos Aires: Editorial Hemisferio Sur. [ Links ]
Ferreira M, Hungria M. 2002. Recovery of soybean inoculant strains from uncropped soils in Brazil. Field Crops Res 79:139-152. [ Links ]
Giongo G, Ambrosini A, Vargas L, Freire J, Bodanese M, Passaglia L. 2008. Evaluation of genetic diversity of bradyrhizobia strains nodulating soybean [Glycine max (L.) Merrill] isolated from South Brazilian fields. Appl Soil Ecol 38:261-269. [ Links ]
Godoy, L, Vasconcelos A, Chueire L, Souza R, Nicolas M, Barcellos F, Hungria M. 2008. Genomic panorama of Bradyrhizobium japonicum CPAC 15, a commercial inoculant strain largely established in Brazilian soils and belonging to the same serogroup as USDA 123. Soil Biol Biochem 40:2743-2753. [ Links ]
Hungria M, Boddey L, Santos M, Vargas M. 1998. Nitrogen fixation capacity and nodule occupancy by Bradyrhizobium japonicum and B. elkanii strains. Biol Fertil Soils 27:393-399. [ Links ]
Hungria M, Vargas M. 2000. Environmental factors affecting N2 fixation in grain legumes in the tropics, with an emphasis on Brazil. Field Crops Res 65(2-3):151-164. [ Links ]
Hungria M, Campo R, Mendes I, Graham P. 2006. Contribution of biological nitrogen fixation to the nitrogen nutriton of grain crops in the tropics: the success of soybean (Glycine max L. Merr) in South america. En: Singh RP, Shankar N, Jaiwal PK, editores. Nitrogen nutrition in plant productivity. Houston, TX: Studium Press; LLCC, pp. 43-93. [ Links ]
Judd A, Schneider M, Sadowsky M, Bruijn F. 1993. Use of repetitive Sequences and the polymerase chain reaction technique to classify genetically related Bradyrhizobium japonicum serocluster 123 strainst. Appl Environ Microbiol 59(6):1702-1708. [ Links ]
López, S, Perticari, A, Piccinetti, C, Ventimiglia L, Arias A, De Battista J, Althabegoiti M, Mongiardini E, Pérez J, Quelas J, Lodeiro A. 2009. In-Furrow inoculation and selection for higher motility enhances the efficacy of Bradyrhizobium japonicum nodulation. Agron J 101:357-363. [ Links ]
Louws F, Rademaker J, Bruijn F. 1999.The three Ds of PCR-based genomic analysis of phytobacteria: diversity, detection, and disease diagnosis. Annu. Rev. Phytopathol. 37:81-125. [ Links ]
Molina D, Amézquita E, Hoyos Ph. 2003. Construcción de capas arables en suelos oxisoles de la Altillanura colombiana. Evaluación de parámetros y procesos hidrológicos en el suelo. En: VII Escuela Latinoamericana de Física de Suelos. La Serena, Chile. pp. 113-117. [ Links ]
Perticari A, Arias N, Baigorri H, De Battista J, Lett, L, Montecchia M, Pacheco J, Simonella A, Toresani S, Ventimiglia L, Vicentini R. 2003. Inoculación y fijación biológica de nitrógeno en el cultivo de soja. En: El libro de la soja. Quito: SEMA. pp. 69-76. [ Links ]
Rakhi A, Bajoria S, Pareek P. 2006. DNA Isolation from Rhizobium by phenol chloroform method. En: Birla Institute of Scientific Research, http://www.protocol-online.org; consulta: noviembre, 2012. [ Links ]
Rivas L, Hoyos Ph, Amezquita E, Molina D. 2004. Manejo y usos de los suelos de la altillanura colombiana. Proyecto de evaluación de impacto. Palmira, Colombia: MADR; CIAT. [ Links ]
Rohlf F. 1997. Software NTSYS-pc. Numerical taxonomy and multivariate analysis system versión 2.02. New York: Exeter Software Publishers. [ Links ]
Salamanca C, Ramírez M. 2000. ICA J 01 Biofertilizante para soya. Inoculante de rizobio para los Llanos Orientales. Villavicencio, Colombia: Corpoica; Ministerio de Agricultura. FOCC. [ Links ]
Santos M, Vargas M, Hungria M. 1999. Characterization of soybean Bradyrhizobium strains adapted to the Brazilian savannas. FEMS Microbiol Ecol 30:261-272. [ Links ]
Schloter M, Lebuhn M, Heulin T, Hartmann A. 2000. Ecology and evolution of bacterial microdiversity. FEMS Microbiol Rev 24(5):647-660. [ Links ]
Versalovic J, Schneider M, De Bruijn F, Lupski J. 1994. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction. Meth Mol Cell Biol 5,25-40. [ Links ]
Vincent J. 1970. A manual for the practical study of root nodule bacteria. Oxford, UK: Blackwell Scientific. [ Links ]
Von H, Mutert E. 1995. Global extent, development and economicimpact of acid soils. Plant Soil 171:1-15. [ Links ]
Xue Z, Muilenburg M, Lie T, Jun Z, Fu L. 1999. Characterization of soybean rhizobia at different levels using PCR based techniques. Biodiv Sci 7(4):277-284. [ Links ]













