Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Ciencia y Tecnología Agropecuaria
versión impresa ISSN 0122-8706
Corpoica cienc. tecnol. agropecu. vol.17 no.2 Mosquera mayo/ago. 2016
Artículo de investigación
Estudio de variabilidad genética en aislamientos de Leptospira spp., en sistemas bovinos de carne y doble propósito
Genetic variability study of field Leptospira spp. isolates in beef and double purpose bovine production systems
Estudo de variabilidade genética em isolamentos de Leptospira spp., em sistemas bovinos de carne e duplo propósito
Elizabeth Regina Cassalett-Bustillo,1 Rocío Esperanza Patiño-Burbano,2 José Luis Rodríguez-Bautista,3 José Luis Parra-Arango4
1 MSc, Universidad Nacional de Colombia. Investigadora máster, Corpoica. Villavicencio, Colombia. ecassalett@corpoica.org.co.
2 MSc, Pontificia Universidad Javeriana. Investigador máster, Corpoica. Mosquera, Colombia. rpatino@corpoica.org.co.
3 MSc, Washington State University. Investigador máster, Corpoica. Mosquera, Colombia. jlrodriguez@corpoica.org.co.
4 MSc, Universidad Nacional de Colombia. Investigador máster, Corpoica, Villavicencio, Colombia, jparra@corpoica.org.co.
Fecha de entrega: 28/07/2015 Fecha de aceptación: 20/01/2016
Para citar este artículo: Cassalett-Bustillo ER, Patiño-Burbano RE, Rodríguez-Bautista JL, Parra-Arango JL. Estudio de variabilidad genética en aislamientos de Leptospira spp., en sistemas bovinos de carne y doble propósito. Corpoica Cienc Tecnol Agropecuaria. 17(2):229-236
Resumen
A partir de riñón y orina de bovinos, se aislaron en campo serovares de Leptospira spp., en los departamentos de Cundinamarca y Meta, Colombia. Los aislamientos fueron clasificados mediante análisis filogenético y ribotipificación, utilizando como marcador el gen 16S ADNr. El análisis filogenético permitió clasificar los aislamientos en dos de las tres genomoespecies reconocidas: patógeno e intermedio, siendo este último de gran importancia, dado que no se conoce su patrón de comportamiento inmunológico ante un huésped, lo que podría generar respuestas variables. En la ribotificación se obtuvieron ribopatrones de cuatro aislamientos de leptospira y cinco cepas de referencia con mayor identidad y su análisis mostró la presencia de dos perfiles predominantes dentro de los cuatro aislamientos. Un perfil coincidió con la cepa de referencia intermedia y otro perfil fue similar a la cepa de referencia patógena serovares Copenhageni y Lai. El análisis filogenético del aislamiento, fue agrupado dentro de leptospiras tipo intermedio y su patogenicidad se encuentra todavía en estudio. Los análisis filogenéticos de especies de leptospiras basados en secuencias comparativas del gen 16S ADNr, permiten confirmar e identificar tres grupos según su estatus de patogenicidad (patógeno, saprofítico e intermedio), donde el propósito taxonómico de los marcadores genera resultados consistentes en obtener secuencias del gen 16S ADNr agrupados en un árbol filogenético.
Palabras clave: gen, ADN Ribosomal, leptospira, ganado bovino, filogenia.
Abstract
Leptospira spp. serovars were isolated from bovine kidney and urine in the Cundinamarca and Meta departments of Colombia. The isolates were classified by both, phylogenetic analysis and ribotyping, using 16SDNAr as a marker gene. The Phylogenetic analysis allowed the classification of the isolates into two of the three genomaspecies recognized: pathogenic and intermediate. The latter is of great importance because its immunological behavior in a host is unknown and this could generate variable answers. Ribotyping yielded “ribopatterns” of four Leptospira isolates and five reference strains with major identity; the analysis showed the presence of two predominant profiles in the four isolates. One profile was in line with the intermediate reference strain and the other profile was similar to the pathogenic reference strain, Copenhageni and Lai serovars. The isolate by phylogenetic analysis was placed within the intermediate type Leptospiras and its pathogenicity is still under study. The phylogenetic analysis of Leptospira species based on comparative sequences of 16SDNAr gene confirmed the possibility of identifying three groups according to their pathogenic status (pathogenic, intermediate and saprophytic), where the taxonomic purpose of the markers, showed consistent results in obtaining sequences of the 16SDNAr gene, grouped in a phylogenetic tree.
Keywords: Gen, Ribosomal ADN, Leptospira, Cattle, Phylogeny.
Resumo
A partir de rim e urina de bovinos, se isolaram em campo serovares de Leptospira spp., nos departamentos de Cundinamarca e Meta, Colômbia. Os isolamentos foram classificados mediante análise filogenética e ribotipificação, utilizando como marcador o gene 16S ADNr. A análise filogenética permitiu classificar os isolamentos em dois das três genomo-espécies reconhecidas: patógeno e intermédio, sendo este último de grande importância, dado que não se conhece o seu padrão de comportamento imunológico perante um hóspede, o que poderia gerar respostas variáveis. Na ribotificação obtiveram-se ribopatrões de quatro isolamentos de leptospira e cinco cepas de referência com maior identidade e a sua análise mostrou a presença de dois perfis predominantes dentro dos quatro isolamentos. Um perfil coincidiu com a cepa de referência intermédia e outro perfil foi similar à cepa de referência patógena serovares Copenhageni e Lai. A análise filogenética do isolamento, foi agrupada dentro de leptospiras tipo intermédio e a sua patogenicidade se encontra ainda em estudo. As análises filogenéticas de espécies de leptospiras baseados em sequências comparativas do gene 16S ADNr, permitem confirmar e identificar três grupos segundo o sue status de patogenicidade (patógeno, saprofítico e intermédio), onde o propósito taxonômico dos marcadores gera resultados consistentes em obter sequências do gene 16S ADNr agrupados em uma árvore filogenética.
Palavras chave: gene, ADN Ribosomal, leptospira, gado bovino, filogenia.
Introducción
La leptospirosis es una enfermedad zoonótica con altas tasas de morbimortalidad en humanos y animales causada por una espiroqueta que se encuentra en el medioambiente y una amplia gama de animales (Díaz et al. 2011; Organización Mundial de Sanidad Animal 2008). La bacteria genera trastornos reproductivos en los bovinos y porcinos y está clasificada en subgrupos y serovares determinados por sus funciones antigénicas (Zuluaga 2009). El género Leptospira está dividido en especies patógenas y saprófitas con 20 genomoespecies, basado en el estudio del gen 16S; a su vez, se han dividido en tres grupos filogenéticos: patógenas, intermedias y saprofíticas, y cada uno de ellos está conformado por diferentes serovares, considerados de importancia para monitoreo diagnóstico y epidemiológico (Fenner et al. 2010; Cerqueira y Picardeau 2009). En estudios epidemiológicos realizados en el país (Díaz et al. 2011) se examinaron serológicamente 2.140 sueros bovinos del Eje Cafetero y se encontró que 681 muestras (32 %) resultaron positivas al serovar L. hardjo; 390 muestras (18,2 %), al serovar L. icterohaemorrhagiae; 207 muestras (9,6 %) al serovar L. pomona y 182 (8,5 %) al serovar L. canícola.
Evidencias serológicas y aislamientos realizados en el país, indican que L. hardjo tiene importancia epidemiológica en los bovinos de los Llanos Orientales con prevalencias del 63,5 %, en hatos del Valle del Cauca, con el 80,9 % y en la costa Caribe, con el 89,1 %. Así mismo, estudios de las regiones Caribe, piedemonte llanero y Andina mostraron prevalencias de leptospirosis serovar L. hardjo del 32,8 %, 24,8 % y 14,4 %, respectivamente, con un promedio para el país del 21,7 %. En el país, se han realizado estudios de ribotipificación y análisis filogenético de especies de Leptospira en diferentes especies de animales, así: en bovinos, Torres et al. (2013), obtuvieron ribopatrones de 19 aislamientos de Leptospira y 10 cepas de referencia (serovares Pomona, Tarassovi, Wolffi, Pyrogenes, Hardjo, Grippotyphosa, Canicola, Ballum Icteroahemorragiae y Patoc). Romero-Vivas et al. (2013) obtuvieron de aislamientos en cerdos relacionamiento con Leptospira interrogans serovariedad Pomona; un aislamiento de rata fue indistinguible de Leptospira interrogans serovariedades Icterohaemorrhagiae o Copenhageni, mientras que aislamientos de perro y agua no se relacionaron con alguna de las 200 cepas de referencia; las más cercanas son L. noguchii serovares Nicaragua y Orleans.
El objetivo de este trabajo fue clasificar aislamientos de campo de Leptospira a través del análisis filogenético y de los ribopatrones basados en el polimorfismo generado por los fragmentos de restricción de longitud polimórfica (RFLP), utilizando como marcador molecular el gen 16S ADNr, con el fin de poder identificar especie, grupo de patogenecidad, serovar y variantes genéticas dentro de estos aislamientos
Materiales y métodos
Las ecorregiones escogidas para desarrollar el trabajo fueron valles interandinos, costa Caribe y piedemonte llanero. Se tomaron 140 muestras de riñón y 20 de orina de bovinos, directamente de vejiga, en cinco mataderos ubicados en Zipaquirá (Cundinamarca), Ibagué-Espinal (Tolima), Guamal (Meta) y Ciénaga de Oro (Córdoba). Así mismo, se obtuvieron 367 muestras de orina por micción espontánea en 11 hatos bovinos ubicados en el altiplano cundiboyacense (3), Tolima (2), Córdoba (2), Meta (2) y Casanare (2). Para el trabajo de toma de muestras, se utilizaron dos formatos de toma de información en campo y, para los aislamientos, se implementaron los protocolos de aislamiento de leptospira a partir de riñón y de orina que hacen parte del Banco de Germoplasma de Microorganismos, Virus y Bacterias de Corpoica.
En la planta de sacrificio, se retiró el riñón completo y se transportó sin refrigeración a un sitio de menor contaminación; el área de extracción de la muestra se esterilizó con una espátula caliente y ayuda de una pipeta Pasteur estéril. Se sembró en medio líquido Ellinghausen-McCullough-Johnson-Harris (EMJH) y después de 48 horas de incubación a 30 °C, se tomaron 3 ml del cultivo inicial que fue filtrado a través de un filtro milipore (0,22 µm) a un medio semisólido con 0,1% de agar. La incubación a 30 °C se mantuvo hasta por 90 días, tiempo en el cual se descartó como negativo o se determinó como positivo, al observar la aparición de un anillo en la porción media del tubo, indicativo de crecimiento de Leptospira spp.
El aislamiento de Leptospira spp. a partir de orina recolectada de micción espontánea o directamente de vejiga de animales sacrificados, se realizó pasando 3 ml de la orina a través de un filtro milipore (0,22 µm) y sembrado en medio semisólido Ellinghausen-McCullough-Johnson-Harris (EMJH) con 0,1% de agar a 30 °C. Se revisaron los cultivos cada 8 días para observar el crecimiento y verificar que no existiera contaminación. Si se contaminaban, el cultivo era filtrado con membrana de 0,22mm y llevado nuevamente a incubación a 30 °C, hasta observar o no el crecimiento de leptospira por un lapso no mayor a 90 días.
Para el análisis filogenético, a los aislamientos se les realizó extracción de ADN por el método de fenol cloroformo, de acuerdo al protocolo Extracción de ADN para bacterias del género Leptospira Código IN-R-104. La amplificación de la región 16S del ADNr fue realizada por PCR (protocolo código IN-R-107), utilizando iniciadores universales externos (Morey et al. 2006; Janda y Abbott 2007). Los productos de PCR fueron clonados, utilizando un kit comercial TOPO TA 4 Cloning® Kit (Invitrogen) y el ADN plasmídico fue secuenciado. Las secuencias del 16S ADNr fueron alineadas y comparadas con las bases de datos del GenBank, usando el algoritmo Basic Local Alignment Search Tool (BLAST) para la construcción de los árboles filogenéticos. Para la ribotipificación, se utilizó la enzima de restricción EcoRV; este polimorfismo fue evidenciado por Southern Blot, utilizando sondas marcadas, homólogas a secuencias de ADN del gen 16S ADNr y el proceso de detección fue realizado por quimioluminiscencia.
El análisis de la secuencia del gen 16S ADNr de los aislamientos de leptospira permitió la identificación de especie y clasificarlos dentro de los tres grupos de patogenicidad previamente establecidos: patógeno, saprofito e intermedio. Este análisis se realizó con los siguientes criterios:
- Porcentaje de cubrimiento. Por medio del cual se determina el porcentaje de homología entre la secuencia problema y las reportadas en las bases de datos.
- Porcentaje de identidad. Por medio del cual se determina el grado de identidad que existe entre la secuencia problema y las reportadas en las bases de datos. Los porcentajes por debajo de 95 % no fueron tenidos en cuenta, puesto que cepas con menor porcentaje de identidad en las secuencias de la región del gen 16S ADNr es improbable que lleguen a estar relacionadas a nivel de especie.
- Valor E (E-Value). Por medio del cual se determina la probabilidad de que el resultado obtenido sea el más acertado.
Resultados y discusión
Una vez realizado el seguimiento bacteriológico, se determinaron 69 aislamientos como sospechosos, los cuales fueron sometidos a pruebas de reacción en cadena de la polimerasa (PCR) amplificando el gen 16S del ADNr, para analizar si estas espiroquetas están agrupadas en un ancestro común de protospirocheta, como lo determinan Paster et al. (1991). Se obtuvieron cuatro productos de amplificación de la región del 16S del ADNr (figura 1), que mostraron un fragmento cuyo tamaño fue aproximadamente de 1.500 pb. Así se determinó la presencia de Leptospira spp. en esos aislamientos, lo que confirma la sensibilidad y especificidad de la prueba, de esta manera se concordó con lo obtenido anteriormente por varios investigadores que amplificaron el gen 16S (Grimont y Grimont 1986; Paster et al. 1991; Cerqueira y Picardeau 2009; Torres et al. 2013; Romero-Vivas et al. 2013).
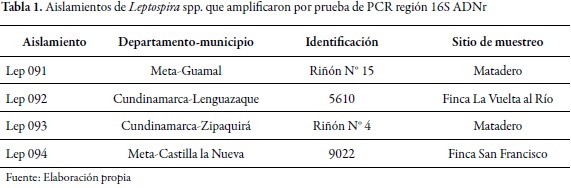
Los asilamientos que amplificaron a leptospira pertenecían los departamentos del Meta y Cundinamarca, según se describe en la tabla 1. Se confirmó que en Colombia, al igual que en otros países como Brasil, India y Nicaragua, existe circulación de cepas de Leptospira spp. en el sistema de producción bovino (Moreno y Agudelo Flórez 2010). Estos aislamientos de leptospira en las dos zonas del país, son determinantes para estudios de respuesta inmunológica, dada la característica de patogenicidad de las especies encontradas.
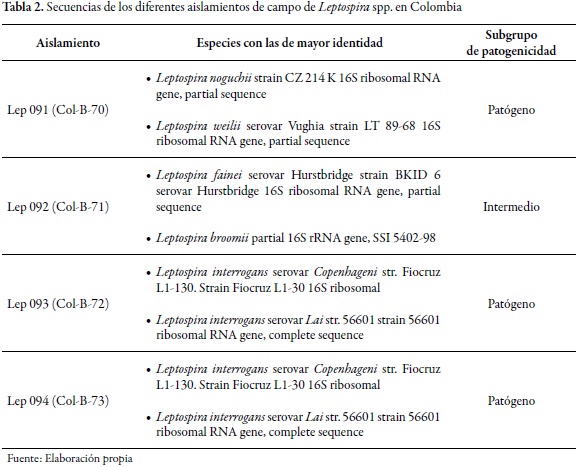
El análisis de secuencia de la región 16S ADNr de los cuatro aislamientos de leptospira permitió su clasificación en dos grupos de patogenicidad (tabla 2). Según los resultados obtenidos del análisis filogenético de este gen, tres aislamientos fueron agrupados dentro de las especies patógenas y uno se relaciona filogenéticamente dentro del grupo de Leptospiras que presentan una patogenicidad de tipo intermedia; es importante destacar que los árboles filogenéticos de estos aislamientos muestran mayor cercanía con las especies patógenas que con las saprófitas (Postic et al. 2000).
En la ribotificación se obtuvieron ribopatrones de cuatro aislamientos de leptospira y cinco cepas de referencia con mayor identidad (serovares Vughia, Hurstbridge, Copenhageni y Lai); el análisis de estos ribopatrones mostró la presencia de dos perfiles predominantes dentro de los cuatro aislamientos (tabla 2). Un perfil coincidió con la cepa de referencia intermedio y otro perfil fue similar a la cepa de referencia patógena serovares Copenhageni y Lai. La patogenicidad del aislamiento que, por análisis filogenético fue agrupado dentro de las leptospiras de tipo intermedia, se encuentra todavía en estudio (Cerqueira y Picardeau 2009, Torres et al. 2013; Moreno y Agudelo-Flórez 2010). El aislamiento de cepas de carácter intermedio cobra gran importancia, dado que no se sabe el patrón de comportamiento inmunológico ante un huésped, lo que podría generar respuestas variables ante la presencia de este tipo de especies, lo que hace más complejos los estudios epidemiológicos.
Con los resultados obtenidos del análisis de las secuencias del gen 16S ADNr se procedió a construir los árboles filogenéticos que reflejan de forma esquemática (figura 1) el grado de parentesco genético entre las bacterias comparadas utilizando el algoritmo Blast y el método de clústers UPGMA (Unweighted Pair Group Method using Arithmetic averages). El método evidenció mayor parentesco entre los aislamientos Lep093 y Lep094, estando, a su vez, estos dos más alejados de los Lep091 y Lep092, con un porcentaje de cubrimiento en todos los casos del 99 y 100 % (figura 2). El resultado del árbol filogenético de estos aislamientos mostró efectivamente lo que se esperaba: mayor acercamiento de los Lep093 y Lep094, dado que estos tenían mayor afinidad por las especies Leptospira interrogans serovar Copenhageni y Leptospira interrogans serovar Lai, como también mayor distanciamiento del Lep091 que tiene mayor afinidad con la Leptospira noguchii y Leptospira weilii serovar Vughia. El aislamiento del grupo intermedio se encuentra más cercano a los Lep093 y Lep094 (L. interrogans), lo que permite inferir que posiblemente su comportamiento ante un huésped podría ser más hacia patógeno que saprofítico.
Los análisis filogenéticos de especies de leptospiras, basados en secuencias comparativas del gen 16S ADNr, permiten confirmar lo asegurado previamente (Paster et al. 1991; Schmid et al. 1986; Matthias et al. 2008), sobre la posibilidad de identificar tres grupos basados en el estatus de patogenicidad (patógeno, saprofítico e intermedio), donde el propósito taxonómico de los marcadores genera resultados consistentes en obtener secuencias del gen 16S ADNr, agrupados en un árbol filogenético. Así mismo, la agrupación de las bacterias por ribotipificación es frecuentemente usada para propósitos taxonómicos y caracterización de subgrupos de microorganismos de diferentes especies de leptospira (Grimont y Grimont 1986). Lo importante del uso de estas herramientas moleculares es poder clasificar adecuadamente los microorganismos de interés en salud pública con el fin de tomar medidas profilácticas y curativas adecuadas.
Conclusiones
El uso de la ribotipificación de las especies de Leptospira spp. circulantes en el sistema de producción bovino es una herramienta primordial y fundamental para las entidades de control sanitario y para los profesionales clínicos, que manejan el diagnóstico de la Leptospira spp. como causal de problemas reproductivos. El análisis filogenético del gen 16S permitió clasificar los aislamientos dentro de las especies reconocidas hasta el momento y definir su estatus de patogenicidad.
La ribotificación permitió confirmar estos resultados y discriminar variantes genéticas; sin embargo, no se observó heterogeneidad dentro de los aislamientos analizados. Los métodos de análisis filogenético y ribotipificación son complementarios para la identificación de aislamientos patógenos de leptospira provenientes de diferentes fuentes.
Descargos de responsabilidad
El presente trabajo es producto de la investigación realizada por los autores, quienes no manifiestan conflictos de interés con respecto a este artículo.
Referencias
Cerqueira GM, Picardeau M. 2009. A century of Leptospira strain typing. Infect Genet Evol. 9(5)760-768. [ Links ]
Díaz OL, Orjuela JE, Ortiz J, Patiño A, Linares C, González PM. 2011. Colombia, sanidad animal 2009. Bogotá, Colombia: ICA. [ Links ]
Fenner JS, Anjum MF, Randall LP, Pritchard GC, Wu G, Errington J, Dalley CG, Woodward MJ. 2010. Analysis of 16S rDNA sequences from pathogenic Leptospira serovars and use of single nucleotide polymorphisms for rapid speciation by D-HPLC. Res Vet Sci. 89(1)48-57. [ Links ]
Grimont F, Grimont PA. 1986. Ribosomal ribonucleic acid gene restriction patterns as potential taxonomic tools. Ann Inst Pasteur Microbiol. 137(2):165-175. [ Links ]
Janda JM, Abbott SL. 2007. 16S rRNA gene sequencing for bacterial identification in the diagnostic laboratory: pluses, perils, and pitfalls. J Clin Microbiol. 45(9):2761-2764. [ Links ]
Matthias MA, Ricaldi JN, Cespedes M, Diaz MM, Galloway RL, Saito M, Steigerwalt AG, Patra KP, Ore CV, Gotuzzo E, et al. 2008. Human leptospirosis caused by a new, antigenically unique Leptospira associated with a Rattus species reservoir in the Peruvian Amazon. PLoS Negl Trop Dis. 2(4)e213. [ Links ]
Moreno N, Agudelo-Flórez P. 2010. Aplicación de las pruebas de PCR convencional simple y múltiple para la identificación de aislamientos de Leptospira spp. en Colombia. Rev Peru Med Exp Salud Publica. 27(4):548-556. [ Links ]
Morey RE, Galloway RL, Bragg AL, Steigerwalt AG, Levett PN. 2006. Species-specific identification of Leptospiraceae by 16S rRNA gene sequencing. J Clin Microbiol. 44(10):3510-3516. [ Links ]
Organización Mundial de Sanidad Animal. 2008. Manual de la OIE sobre animales terrestres 2008. Paris, Francia: OIE. Capítulo 2.1.9. Leptospirosis; pp. 251-260. [ Links ]
Paster BJ, Dewhirst FE, Weisburg WG, Tordoff LA, Fraser GJ, Hespell RB, Stanton TB, Zablen L, Mandelco L, Woese CR. 1991. Phylogenetic analysis of the spirochetes. J Bacteriol. 173(19):6101-6109. [ Links ]
Postic D, Riquelme-Sertour N, Merien F, Perolat P, Baranton G. 2000. Interest of partial 16S rDNA gene sequences to resolve heterogeneities between Leptospira collections: application to L. meyeri. Res Microbiol. 151(5): 333-341. [ Links ]
Romero-Vivas CM, Thiry D, Rodríguez V, Calderón A, Arrieta G, Mattar S, Cuello M, Levett P, Falconar AK. 2013. Serovar characterization at the molecular level of Leptospira spp. isolates from animals and water in Colombia. Biomédica. 33(Supl. 1):179-184. [ Links ]
Schmid GP, Steere AC, Kornblatt AN, Kaufmann AF, Moss CW, Johnson RC, Hovind-Hougen K, Brenner DJ. 1986. Newly recognized Leptospira species ("Leptospira inadai" serovar lyme) isolated from human skin. J Clin Microbiol. 24(3):484-486. [ Links ]
Torres L, Rodríguez JL, Jiménez SC, Patiño RE. 2013. Genotipificación de aislamientos Colombianos de Leptospira. Resumen presentado en: II Congreso Brasileiro de Recursos Genéticos. Belem, Brasil. [ Links ]
Zuluaga AG. 2009. Factores de riesgo asociados a leptospirosis en hatos bovinos de Pereira, 2002-2005. Investig Andina. 11(19):109-117. [ Links ]