Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Ciencia y Tecnología Agropecuaria
Print version ISSN 0122-8706
Corpoica cienc. tecnol. agropecu. vol.18 no.1 Mosquera Jan./Apr. 2017
https://doi.org/10.21930/rcta.vol18_num1_art:562
Artículo de investigación científica y tecnológica
DOI: http://dx.doi.org/10.21930/rcta.vol18_num1_art:562
Estandarización de un método de detección molecular del Cucumber mosaic virus (CMV) en banano ecuatoriano
Estandarización de un método de detección molecular del Cucumber mosaic virus (CMV) en banano ecuatoriano
Standardization of a molecular diagnostic method for Cucumber mosaic virus (CMV) in Ecuadorian bananas
Padronização de um método de detecção molecular do ICucumber mosaic virus (CMV) em banana equatoriana
Johanna Liseth Buitrón-Bustamante,1 Luis Eduardo Morillo-Velastegui2
1 Ingeniera en Biotecnología, ESPE. Investigadora Instituto Nacional de Investigaciones Agropecuarias (Iniap), Departamento Nacional de Biotecnología. Quito, Ecuador. johanna.buitron@iniap.gob.ec
2 PhD, Montpellier SupAgro. Responsable del Departamento Nacional de Biotecnología, Instituto Nacional de Investigaciones Agropecuarias (Iniap). Quito, Ecuador. eduardo.morillo@iniap.gob.ec
Fecha de recepción: 19/04/2016 Fecha de aceptación: 28/06/2016
Para citar este artículo: Buitrón-Bustamante JL, Morillo-Velastegui LE. 2017. Estandarización de un método de detección molecular del Cucumber mosaic virus (CMV) en banano ecuatoriano. CORPOICA Cienc Tecnol Agropecuaria. 18(1):113-124
Resumen
El cultivo del banano en el Ecuador se ve afectado por una serie de enfermedades, de las cuales el Cucumber mosaic virus (CMV) es uno de los fitopatógenos más importantes. Con este estudio se buscó estandarizar una técnica molecular para la detección sensible y altamente específica de este agente viral en el banano ecuatoriano. Para este fin, se realizó el diseño de primers específicos, a partir de la secuencia que se codifica para la proteína de la cápside del virus. Los primers PC-F1, PC-RD1 y K-F, usados en cDNA replicado a partir de ARN de banano infectado, permitieron detectar eficientemente la presencia del virus con la aplicación de reacciones de retrotranscripción y hemi-nested PCR. Se reportó la detección del virus incluso en plantas asintomáticas, lo cual evidencia que esta técnica resulta de alta sensibilidad para su aplicación en diagnósticos rutinarios del sector bananero.
Palabras clave: Musa acuminata, cucumovirus del mosaico del pepino, control de enfermedades de plantas, transcripción inversa.
Abstract
Several pests and diseases affect banana crop in Ecuador and Cucumber mosaic virus (CMV) is one of the most important pathogens. The aim of this research was to standardize a new molecular approach to achieve a sensitive and highly specific detection of CMV in Ecuadorian bananas. Specific primers were designed from the sequence encoding for the virus capsid protein. PC-F1, PC-RD1 and K-F primers, obtained from cDNA replicated from RNA of infected banana, allowed accurate virus detection by Reverse transcription and Hemi-nested PCR. Virus detection was possible even in asymptomatic plants, providing a technology with potential use for the Ecuadorian banana producers.
Keywords: Musa acuminata, Cucumber mosaic virus, plant disease control, reverse transcription.
Resumo
A cultura da banana no Equador vê-se afetada por uma série de doenças, das quais o cucumber mosaic vírus (CMV) é um dos fitopatógenos mais importantes. Com este estudo procurou-se padronizar uma técnica molecular para a detecção sensível e altamente específica deste agente viral na banana equatoriana. Para este fim, realizou-se o desenho de primers específicos, a partir da sequência que se codifica para a proteína da cápside do vírus. Os primers PC-F1, PC-RD1 e K-F, usados em cDNA replicado a partir de ARN de banana infectada, permitiram detectar eficientemente a presença do vírus com a aplicação de reações de retrotranscrição e hemi-nested PCR. Reportou-se a detecção do vírus mesmo em plantas assintomáticas, o qual evidencia que esta técnica resulta de alta sensibilidade para a sua aplicação em diagnósticos rotineiros do setor bananeiro.
Palavras chave: Musa acuminata, cucumo vírus do mosaico do pepino, controle de doenças de plantas, transcrição inversa.
Introducción
La producción de banano en América Latina representa un importante ingreso económico (Soto 2011). La exportación de esta fruta a nivel mundial está liderada por Ecuador, con el 21,95% para el 2012 (DICI 2013). Para este país, la producción de banano aporta de manera considerable a su producto interno bruto (PIB): representa el 2% del PIB general, el 26% del PIB agrícola, el 8% de las exportaciones generales y el 27% de las exportaciones agropecuarias (dici 2013; Fierro y Villacres 2014; Moina y Torres 2015). El banano ecuatoriano (Musa acuminata, grupo AAA) es conocido a nivel mundial por sus características en cuanto a calidad y sabor, razón por la cual es ampliamente cotizado en los mercados internacionales (Vaca y Mesías 2014). La disminución de su producción incide directamente y de forma negativa en la economía del país según lo comentado arriba sobre el PIB.
Desafortunadamente, los problemas fitosanitarios se encuentran dentro de las principales causas de la disminución de su producción, lo cual desencadena una reducción de la competitividad en los mercados por descenso en la producción por hectárea y el deterioro de la fruta (Mena 2014). Diversos patógenos son los causantes de dichos efectos, ya que el banano se ve afectado por virus, bacterias y hongos, entre otros (Bautista et al. 2015); sin embargo, los virus patógenos son el mayor problema para el cultivo (Vishnoi et al. 2013).
La incidencia del CMV (Cucumber mosaic virus), también llamado virus del mosaico del pepino, tiene un gran impacto económico, ya que aparte de infectar al banano, afecta a una amplia gama de hospedantes. Su incidencia, en efecto, está reportada en más de 1.300 especies vegetales, lista a la que se añaden nuevas especies cada año (Arafati et al. 2013). En el Ecuador, aunque son pocas las enfermedades virales reportadas en el banano, el CMV, que se encuentra diseminado en amplias zonas bananeras del país (Niblett et al. 1994; Orellana et al. 2008), representa una amenaza grave para el cultivo. Recientemente ha sido reportado también en la provincia de Manabí (MAGAP 2014).
El CMV es un virus del género Cucumovirus, de la familia Bromoviridae. Posee un genoma que se compone de tres moléculas de ARN de sentido positivo de una sola hebra y dos RNA subgenómicos. El ARN 1 y el ARN 2 codifican proteínas que intervienen en la replicación, además de codificar proteínas multifuncionales. El ARN 3 codifica las proteínas de la cápside y del movimiento (Nouri et al. 2014). Los ARN subgenómicos (RNA 4 y RNA 4A) codifican para la CP (proteína de la cápside) y para la 2b (proteína supresora del silenciamiento génico) (Ding et al. 1994; Jacquemond 2012). Toda esta información ha contribuido a establecer mecanismos de detección de este patógeno (Reichel et al. 1996).
Las técnicas tradicionales de detección del virus son limitadas, puesto que por tratarse de un parásito endonuclear obligado se hace imposible su aislamiento en medios de cultivo artificiales. Por otro lado, las técnicas de observación que podrían utilizarse requieren de la microscopía electrónica, que es una tecnología de alto costo y sofisticación (Pupiro et al. 2003). Las técnicas serológicas, que no requieren de un aislamiento previo o presencia de síntomas en el hospedante para su detección, tienen una sensibilidad limitada, lo cual genera posibles inconvenientes para un control totalmente eficiente (Macías Et al. 2006).
Hoy día, las técnicas moleculares superan las limitaciones de las técnicas tradicionales y serológicas para la detección de patógenos. Las primeras permiten una detección más eficiente y de mayor fiabilidad, además de ser muy variadas y aplicables a casi cualquier diagnóstico asociado con agentes biológicos (Corvalán et al. 2003; Rodríguez y Barrera 2004; Mumford et al. 2006). En el caso del CMV, existen reportes de marcadores moleculares para su identificación, por ejemplo, Berniak et al. (2009) reportan una mayor sensibilidad de la PCR [Polymerase Chain Reaction (reacción en cadena de la polimerasa)] en la detección de CMV con respecto al método ELISA. En el mencionado estudio, la sensibilidad del método ELISA se verificó con extractos vegetales con el virus sin diluir o en preparaciones con el extracto diluido hasta 100 veces. La sensibilidad de la RT-PCR [Reverse-Transcription PCR (reacción en cadena de la polimerasa con transcriptasa inversa)]fue superior, pues se detectó el virus en extractos de plantas infectadas diluidas hasta 106 veces.
La alta tasa de variabilidad genética de los virus, especialmente, del ARN, provoca la aparición de nuevas variantes virales, específicas de un hospedero. En consecuencia, en muchos casos se requiere la generación de marcadores específicos que permitan la detección de estas variantes virales (Reichel et al. 1996).
En este trabajo se describe la estandarización de un método de detección molecular de CMV para banano ecuatoriano. Esta herramienta podrá emplearse como método rutinario de detección y control del virus, lo cual permitirá tomar decisiones oportunas en el manejo de las plantaciones bananeras afectadas.
Materiales y métodos
Diseño de primers de detección
Se realizó una revisión bibliográfica para la búsqueda de primers reportados para detectar el virus en banano americano. Para el diseño de nuevos primers de detección, se utilizaron los polimorfismos del gen que codifica la proteína de cápside del virus, obtenidos de la base de datos del GenBank del NCBI (National Center for Biotechnology Information). Varios estudios reportan este gen para la detección de CMV (Rizos et al. 1992; Reichel et al. 1996; Mariño 1997; Gutiérrez et al. 2004), el cual es de gran interés por guardar relación directa con los factores de virulencia para la mayoría de las plantas hospedantes del virus (Mochizuki y Ohki 2011; Mochizuki et al. 2014). Las secuencias disponibles reportadas en GenBank para CMV aislado en banano americano tienen los siguientes números de accesión: AM055602.1, DQ640743.1, AM158321.1, DQ152254.1, U32858.1, U32859.1, M98501.1, M57602.1. Para alinear las secuencias se utilizó el programa BioEdit® (versión 5.0.9.1) y se buscaron secuencias de consenso de 18 a 25 pb (Nouri et al. 2014). Para el diseño de cebadores se utilizó el programa Primer3 (versión 0.4.0) y se configuraron parámetros para descartar primers con alta formación de dímeros o con hibridaciones inespecíficas (Medrano et al. 2006). Finalmente, el cebador K-F (5'ATGGACAAATCTGAATCAACC3') del estudio de Khan et al. (2011), que presentó complementariedad con las secuencias del CMV en banano americano, fue utilizado con otros nuevos cebadores diseñados para los ensayos de detección (tabla 1).

Obtención del material vegetal infectado
Las plantas de banano infectadas se obtuvieron en una plantación en el sector de El Carmen (provincia de Manabí) y se establecieron en la Estación Experimental Tropical Pichilingue. La presencia del virus se verificó mediante una prueba serológica de ELISA con el kit de Agdia® (Cat. N.° PSA 44501/0096). Para disponer de suficiente material para los ensayos de detección molecular, las plantas infectadas se propagaron in vitro. Para los ensayos de detección se utilizaron como control dos plantas que resultaron positivas en el test ELISA (códigos CP1/CP2). Además, se incluyeron como controles negativos plantas que resultaron negativas en el test ELISA.
Extracción y cuantificación del arn genómico
Para la extracción de ARN genómico se emplearon entre 50 y 100 mg de hojas frescas de banano infectado con CMV. La muestra se maceró con nitrógeno líquido utilizando TRIzol® (Thermo Fisher Scientific, Cat. N.° 15596-026), conforme al protocolo indicado en Chou et al. (2008). La cuantificación del ARN se realizó mediante el fluorímetro Qubit® RNA Assay (Thermo Fisher Scientific, Cat. N.° Q10210).
Retrotranscripción
La retrotranscripción se realizó con el Strand cDNA Synthesis Kit para RT-PCR (AMV) de Roche (Cat. N.° 11483188001). Para este proceso se empleó el primer PC-RD1.
Estandarización y optimización de la metodología de detección molecular
A partir del cDNA obtenido se realizó una heminested PCR (tabla 2). Las concentraciones de los componentes PCR y los programas de amplificación se realizaron de acuerdo con Khan et al. (2011) con modificaciones recomendadas por Espinosa (2007).

Para estandarizar y optimizar el método de detección, se realizaron ensayos de amplificación mediante la modificación de la temperatura de annealing (50 °C; 53,5 °C; 56 °C; 60° C), la modificación de la concentración de cloruro de magnesio (1,5 mM; 2 mM; 2,5 mM; 3 mM) y la reducción en el volumen final (25 µL a 10 µL). La tabla 3 detalla las concentraciones empleadas para la primera PCR estandarizada y optimizada en la detección del CMV.
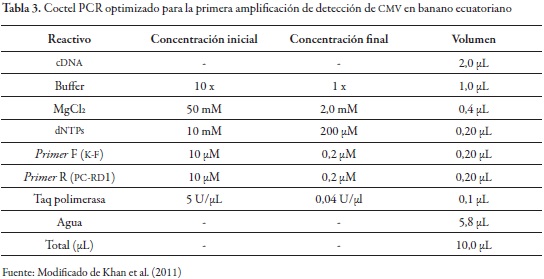
Las temperaturas y los tiempos de amplificación empleados para la primera reacción de PCR se detallan en la tabla 4.
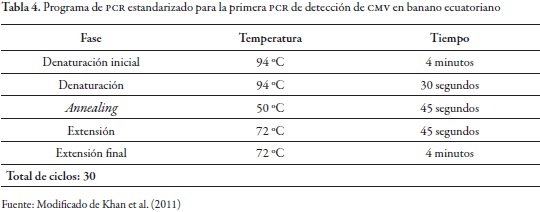

Reproducibilidad en la detección de CMV
La reproducibilidad de la detección se realizó con base en las sugerencias de Villarreal et al. (2008). Este ensayo consistió en efectuar cinco repeticiones de la prueba de detección, utilizando las condiciones estandarizadas y optimizadas de la reacción previamente descritas.
Detección de CMV a partir de muestras vegetales infectadas
Para la validación, se realizaron ensayos de detección en plantas infectadas sintomáticas y asintomáticas disponibles en invernadero, utilizando el control positivo de la fase de estandarización.Resultados y discusión
Detección de CMV
Con el protocolo de extracción establecido, se obtuvieron al menos 6 fg de ARN del virus, cantidad requerida para la RT-PCR. Con el ARN extraído se iniciaron las pruebas y ensayos para la detección del CMV. Los ensayos de detección del virus se realizaron con los primers reportados por Rizos et al. (1992), Gutiérrez et al. (2004) y Khan et al. (2011), con los cuales no se obtuvo la amplificación del fragmento esperado (figura 1). Estos marcadores resultaron, por lo tanto, ineficientes para detectar el virus en el material de control disponible. Según Nouri et al. (2014) y Pita et al. (2015), la diversidad de ARN de los virus conduce a la aparición de nuevas cepas y genotipos, los cuales surgen por recombinación genética o mutaciones. Por esto, resulta probable que las cepas de CMV que se encuentran en el Ecuador difieran de las reportadas por Rizos et al. (1992) y por Gutiérrez et al. (2004), lo cual explicaría que no sean detectadas por los marcadores disponibles.
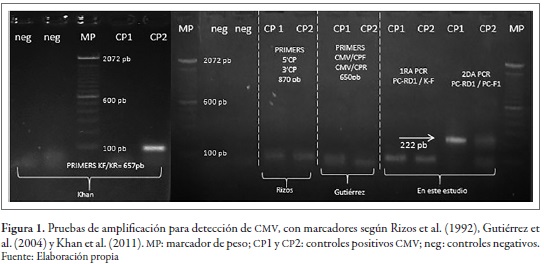
Con el alineamiento de secuencias del GenBank, se validó únicamente el primer K-F reportado por Khan et al. (2011). Además, se evidenció una gran cantidad de variaciones a lo largo de las secuencias alineadas, de acuerdo con lo mencionado por Nouri et al. (2014) y Pita et al. (2015) (figura 2). Se identificaron dos secuencias de consenso para todos los aislados, lo que facilitó el diseño de nuevos cebadores. De este alineamiento se sintetizaron dos juegos de cebadores para la detección y se obtuvieron los mejores resultados con los primers PC-F1 y PC-RD1. Cabe indicar que el primer PC-RD1 es degenerado en una base con el fin de amplificar el polimorfismo observado en esta posición (figura 3).

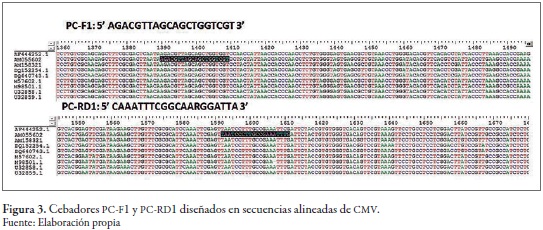
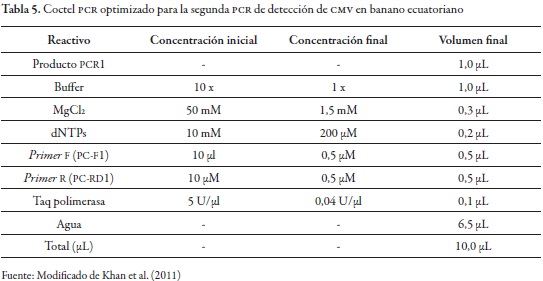
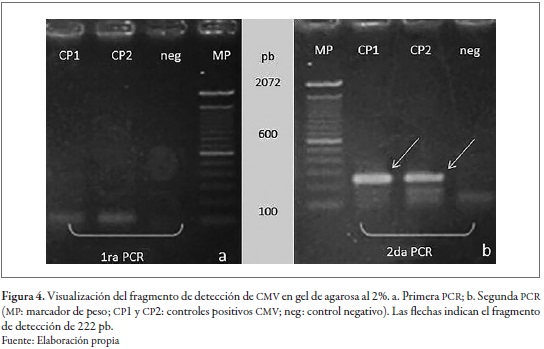
Dado que el virus es monocatenario, la retrotranscripción se realizó con el primer reverse PC-RD1, razón por la cual únicamente se empleó el cebador reverso. Con el objeto de aumentar la sensibilidad de la detección y conforme a la validez de tres marcadores, se realizó una segunda PCR, en la que se conservó uno de los primers (hemi-nested PCR). Esta variante de PCR tuvo una mayor sensibilidad y especificidad de detección, gracias a lo cual se corroboró lo reportado por Pulido et al. (2006), Batista et al. (2008) y Acosta et al. (2011). Además, debido a la degeneración del primer PC-RD1, el aumento en la especificidad resultó ser otro punto importante para la elección de esta técnica de amplificación. El fragmento amplificado tuvo un peso de 349 pb en la primera PCR (no visible en gel) y, en la segunda, de 222 pb (figura 4).
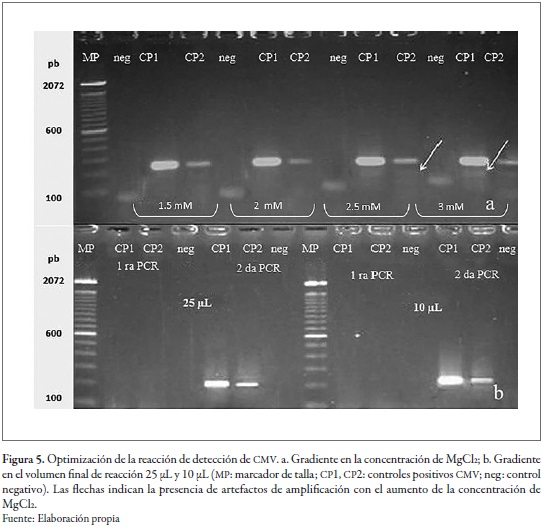
Las reacciones PCR se basaron en las recomendaciones de Espinosa (2007) y se optimizaron en volumen de reacción, concentración de cloruro de magnesio y temperatura de annealing. Como se observa en la figura 5, se estandarizó la reacción con una concentración final de 1,5 mM de MgCl2 en un volumen final de 10 µL.
Reproducibilidad y validación del método
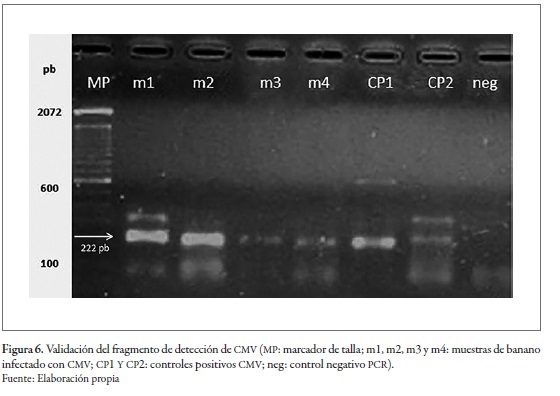
Se comprobó que el método de detección desarrollado es altamente reproducible. No se observaron diferencias en la eficiencia de la detección en las repeticiones realizadas, resultado que concuerda con lo reportado en trabajos anteriores (Poutou et al. 2005; Villarreal et al. 2008). Por otro lado, la validación del método de detección resultó eficiente. Los resultados de los ensayos permitieron la validación del marcador de detección en plantas asintomáticas que resultaron infectadas (figura 6), así como en un screening con plantas sanas e infectadas (figura 7). Resultados similares se reportan en los estudios de Rizos et al. (1992), Gutiérrez et al. (2004) y Khan et al. (2011), quienes reportan la presencia del virus en plantas asintomáticas.
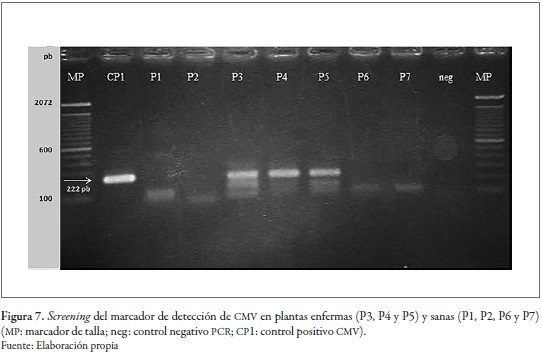
Conclusiones
La tecnología de detección reportada permite dentificar la presencia de CMV en plantas de banano ecuatoriano y representa una alternativa frente a los métodos serológicos existentes. Las pruebas con otros marcadores publicados anteriormente no permitieron realizar la detección en las muestras de control, por lo que en este trabajo se reporta el desarrollo de nuevos primers para la detección en plantas infectadas con CMV.
La sensibilidad del método se basa en una reacción de hemi-nested PCR en el cDNA de la cápside del virus. Esta técnica fue capaz de detectar hasta seis femtogramos de ARN viral presente en la muestra, resultado que indica una alta sensibilidad del marcador de detección desarrollado. El método presenta un elevado potencial para el screening de muestras en las que el virus se encuentra en estadios de incidencia temprana, inicial o en bajas concentraciones. En el corto plazo, será conveniente adaptar esta tecnología a la PCR en tiempo real (qPCR).
Agradecimientos
Los autores dejan constancia de su reconocimiento al personal del Centro de Investigación de la Caña de Azúcar del Ecuador (Cincae), en especial, a Raúl Castillo y a Alexandra Gómez, por su colaboración para la extracción de ARN. Asimismo, agradecen a Emmerick Motte y al personal de la Estación Experimental Tropical Pichilingue (EETP), Iván Garzón e Ignacio Sotomayor, por el apoyo logístico.
Descargos de responsabilidad
Este estudio se financió con fondos del proyecto Fortalecimiento-Componente Biotecnología del Instituto Nacional de Investigaciones Agropecuarias (Iniap). Los autores manifiestan que no existe ningún conflicto de interés que ponga en riesgo la validez de los resultados presentados.
Referencias
Acosta K, Martínez Y, Zamora L, Fernández A, Santos-Cervantes ME, Leyva-López NE. 2011. PCR múltiple anidada para detección de fitoplasmas y rickettsia asociados con los síntomas del cogollo arrepollado (BTS) en papayo. Rev Protección Veg. 26(3):156-163. [ Links ]
Arafati N, Farzadfar S, Pourrahim R. 2013. Characterization of coat protein gene of Cucumber mosaic virus isolates in Iran. Iran J Biotech. 11(2):109-114. [ Links ]
Batista L, Peña I, López D, Pérez J, Llauger R. 2008. Técnicas de diagnóstico de enfermedades que afectan a los cítricos. Consejo Estatal Citrícola (Concitver); [consultado 2016 ene 24]. http://goo.gl/SzRhfq. [ Links ]
Bautista L, Bolaños M, Asakawa N, Villegas B. 2015. Respuesta de fitonematodos de plátano Musa AAB Simmonds a estrategias de manejo integrado del suelo y nutrición. Luna Azul. 40:69-84. [ Links ]
Berniak H, Malinowski T, Kaminska M. 2009. Comparison of ELISA and RT-PCR assays for detection and identification of Cucumber mosaic virus (CMV) isolates infecting horticultural crops in Poland. J Fruit Ornam Plant Res. 17(2):5-20. [ Links ]
Chou C-N, Chen C-E, Wu M-L, Su H-J, Yeh H-H. 2008. Biological and molecular characterization of Taiwanese isolates of Cucumber mosaic virus associated with banana mosaic disease. J Phytopathol. 157(2):85-93. [ Links ]
Corvalán A, Aguayo F, Lévican J, Corvalán I. 2003. Biología molecular en Infectología. Parte II: diagnóstico molecular de agentes infecciosos. Rev Chil Infect. 20(1):26-38. [ Links ]
[DICI] Dirección de Inteligencia Comercial e Inversiones. 2013. Análisis del sector banano. Guayaquil, Ecuador: DICI, Pro Ecuador. [ Links ]
Ding SW, Anderson BJ, Haase HR, Symons RH. 1994. New overlapping gene encoded by the Cucumber mosaic virus genome. Virology. 198(2):593-601. [ Links ]
Espinosa, L. 2007. Las herramientas moleculares, capítulo 17. [INECC] Instituto Nacional de Ecología y Cambio Climático; [consultado 2016 ene 16]. http://goo.gl/8WFzqD. [ Links ]
Fierro I, Villacres C. 2014. Diagnóstico de la cadena logística de exportación del banano ecuatoriano hacia Estados Unidos de América. Saber Cienc Lib. 8(2):77-90. [ Links ]
Gutiérrez C, Ruiz R, Piedra E, De la Torre R. 2004. Caracterización de una variante del virus del mosaico del pepino (CMV) asociada con los síntomas de moteado amarillo de la azucena (Hippeastrum×hybridum Leopoldii) en México. Agro-cienc. 38(3):343-354. [ Links ]
Jacquemond M. 2012. Cucumber mosaic virus. Adv Virus Res. 84:439-504. [ Links ]
Khan S, Tasleem A, Aquil B, Mohd Q, Haq R. 2011. Coat protein gene based characterization of Cucumber mosaic virus isolates infecting banana in India. J Phytol. 3(2):94-101. [ Links ]
Macías O, Delgado Y, Peña E, León R, Barreto R. 2006. Técnicas para el diagnóstico y determinación de variabilidad genética de fitopatógenos. Universidad de Matanzas; [consultado 2016 ene 16]. http://goo.gl/RW8ib3. [ Links ]
[MAGAP] Ministerio de Agricultura, Ganadería, Acuacultura y Pesca (Ecuador). 2014. Boletín situacional plátano; [consultado 2016 ene 23]. http://sinagap.agricultura.gob.ec/phocadownloadpap/cultivo/2014/nboletin-situacional- de-platano-2014-actualizado.pdf. [ Links ]
Mariño L. 1997. Clonación del gen de la cápside protéica de una cepa colombiana del virus del mosaico del Pepino (CMV) para su expresión en plantas por transformación mediante Agrobacterium. CORPOICA Cienc Tecnol Agropecuaria. 2(1):58-59. [ Links ]
Medrano G, Hung A, Rubio N. 2006. Detección molecular temprana de Sarcocystis en el animal vivo y su estudio filogenético basado en el análisis del gen SSU rRNA en alpacas en Perú. Mosaico Cient. 3(2):5-9. [ Links ]
Mena J. 2014. Herramientas biotecnológicas empleadas para controlar el hongo (Mycosphaerella fijiensis) causante de la enfermedad sigatoka negra en plátano y banano [tesis de especialización]. [Turbo, Colombia]: Universidad Nacional Abierta y a Distancia. [ Links ]
Mochizuki T, Ohki ST. 2011. Single amino acid substitutions at residue 129 in the coat protein of Cucumber mosaic virus affect symptom expression and thylakoid structure. Arch Virol. 156(5):881-886. [ Links ]
Mochizuki T, Yamazaki R, Wada T, Ohki ST. 2014. Coat protein mutations in an attenuated Cucumber mosaic virus encoding mutant 2b protein that lacks RNA silencing suppressor activity induces chlorosis with photosynthesis gene repression and chloroplast abnormalities in infected tobacco plants. Virology. 456-457:292-299. [ Links ]
Moina M, Torres K. 2015. Estudio de factibilidad para la exportación de puré de banano de la Empresa Oro Banana S. A. de la ciudad de Machala hacia el mercado de Alemania [tesis doctoral]. [Guayaquil, Ecuador]: Universidad Católica de Santiago de Guayaquil. [ Links ]
Mumford R, Boonham N, Tomlinson J, Barker I. 2006. Advances in molecular phytodiagnostics-new solutions for old problems. Eur J Plant Pathol. 116:1-19. [ Links ]
Niblett CL, Pappu S, Bird J, Lastra R. 1994. Infectious chlorosis, mosaic and heart rot. En: Ploetz R, Zentmyer G, Nishijima WT, Rohrbach KG, Ohr HD, editores. Compendium of tropical fruit disease. St Paul, EE. UU.: APS Press. [ Links ]
Nouri S, Arevalo R, Falk BW, Groves RL. 2014. Genetic structure and molecular variability of Cucumber mosaic virus isolates in the United States. PloS One. 9(5):e96582. [ Links ]
Orellana H, Solórzano H, Bonilla A, Salazar G, Falconí-Borja C, Velasteguí R. 2008. El cultivo de banano. Edifarm; [consultado 2016 ene 16]. http://www.edifarm.com.ec/edifarm_quickagro/pdfs/manual_cultivos/BANANO.pdf. [ Links ]
Pita J, Morris V, Roossinck M. 2015. Mutation and recombination frequencies reveal a biological contrast within strains of Cucumber mosaic virus. J Virol. 89(13):6817-6823. [ Links ]
Poutou R, Burbano M, Sierra S, Torres K, Carrascal AK, Mercado M. 2005. Estandarización de la extracción de ADN y validación de la PCR múltiple para detectar Listeria monocytogenes en queso, leche, carne de res y pollo. Univ Scient. 10(2):61-78. [ Links ]
Pulido A, Mogollón JD, Morales HJ, Rincón MA. 2006. Estandarización y aplicación de la técnica de PCR anidado para la detección de Mycoplasma hyopneumoniae. Rev Med Vet Zoot. 53:22-32. [ Links ]
Pupiro M, Malagón L, Rodríguez L. 2003. Técnicas del diagnóstico de las enfermedades en las plantas. Trabajo de Patología Vegetal. La Habana, Cuba: Universidad Agraria de La Habana. [ Links ]
Reichel H, Mariño L, Kummert J, Belalcázar S, Narváez J. 1996. Caracterización del gen de la proteína de la cápside de dos aislados de virus del mosaico del pepino (CMV), obtenidos de plátano y banano (Musa spp.). CORPOICA Cienc Tecnol Agropecuaria. 1:1-5. [ Links ]
Rizos H, Gunn LV, Pares RD, Gillings MR. 1992. Differentiation of Cucumber mosaic virus isolates using the polymerase chain reaction. J Gen Virol. 73(8):2099-2103. [ Links ]
Rodríguez I, Barrera, H. 2004. La reacción en cadena de la polimerasa a dos décadas de su invención. Ciencia UANL. 7(3):323-334. [ Links ]
Soto M. 2011. Situación y avances tecnológicos en la producción bananera mundial. Rev Bras Frutic. 33(spl. vol. especial):13-28. [ Links ]
Vaca SI, Mesías FJ. 2014. Percepciones de los consumidores españoles hacia las frutas de Ecuador: un estudio preliminar cualitativo con técnicas proyectivas. Inf Tec Econ Agrar. 110(1):89-101. [ Links ]
Villarreal J, Soto Z, Pereira N, Varela L, Jaramillo R, Villanueva D, Mendoza E. 2008. Reacción en cadena de la polimerasa para la detección de Salmonella sp. en leche en polvo: optimización del método en 12 horas. Rev Cient Salud Uninorte. 24(2):215-225. [ Links ]
Vishnoi R, Kumar S, Raj S. 2013. Molecular characterization of a Cucumber mosaic virus isolate associated with mosaic disease of banana in India. Phytoparasitica. 41(5):545-555. [ Links ]














