Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista de Medicina Veterinaria
versión impresa ISSN 0122-9354
Rev. Med. Vet. no.25 Bogotá ene./jun. 2013
Características, ventajas y desventajas
de la hibridización in situ para la identificación
de agentes patógenos
Martha Lilia Franco Mesa1
1 Médica veterinaria, Universidad de La Salle, Bogotá, Colombia. MSc (C) en Salud Animal, Universidad Nacional de Colombia. Investigadora del Grupo de Patobiología Veterinaria, Universidad Nacional de Colombia. Subgerente del Centro Especializado de Diagnóstico Veterinario-Microvet.
marthafra@hotmail.com
Recibido: 10 de diciembre de 2012. Aceptado: 19 de marzo de 2013
Resumen
La citogenética molecular y los métodos de hibridización in situ (HIS) han revolucionado la comprensión de la estructura, función, organización y evolución de los genes y el genoma, además de permitir identificar la presencia y expresión de agentes patógenos dentro de las células afectadas. La HIS es una técnica que combina la biología molecular y las técnicas de histoquímica para estudiar la expresión de genes en secciones de tejido y preparados citológicos, de tal manera que el ADN o el ARN puedan localizarse rápidamente en una célula específica. La HIS localiza la secuencia específica de un gen in situ y visualiza el producto de la expresión de dicho gen preservando al tiempo la integridad de la célula dentro del tejido que la rodea, lo cual permite dictar interpretaciones anatómicas significativas. Esta técnica es el resultado de una reacción en la cual una sonda marcada se une a una secuencia de ácido nucleico complementarias entre sí. Los métodos de HIS son aplicables en investigación clínica y en patología diagnóstica, siendo muy utilizados para buscar expresión de genes cromosomales o para detectar la presencia de bacterias o virus en tejidos infectados, ya que permiten diferenciar los agentes contaminantes de los verdaderos agentes patógenos en un proceso infeccioso.
Palabras clave: ADN, ARN, hibridización in situ, sondas.
Features, Advantages and Disadvantages of in situ
Hybridization to Identify Pathogenic Agents
Abstract
Molecular cytogenetics and in situ hybridization (ISH) methods have revolutionized the understanding of the structure, function, organization and evolution of genes and genomes, and allow identifying the presence and expression of pathogens within the affected cells. ISH is a technique that combines molecular biology and histochemical techniques to study gene expression in tissue sections and cytological preparations, so that the DNA or RNA can be quickly located in a specific cell. ISH locates the specific sequence of an in situ gene and displays the result of the expression of said gene while preserving the integrity of the cell in the surrounding tissue, which allows for significant anatomical interpretations. This technique is the result of a reaction in which a labeled probe binds to a nucleic acid sequence, which is complementary. ISH methods are applicable in clinical research and diagnostic pathology, being widely used to search for chromosomal gene expression or to detect the presence of bacteria or viruses in infected tissues, as it makes it possible to differentiate pollutants from real pathogens in an infectious process
Keywords: DNA, RNA in situ hybridization, probes.
Características, vantagens e desvantagens da hibridização
in situ para identificar agentes patogênicos
Resumo
A citogenética molecular e os métodos de hibridização in situ (HIS) têm revolucionado a compreensão da estrutura, função, organização e evolução dos genes e o genoma, além de que permite identificar a presença e expressão de agentes patógenos dentro das células afetadas. A HIS é uma técnica que combina a biologia molecular e as técnicas de histoquímica para estudar a expressão de genes em seções de tecido e preparos citológicos, de tal modo que o ADN ou o ARN possam localizar-se rapidamente em uma célula específica. A HIS localiza a sequência específica de um gene in situ e visualiza o produto da expressão deste gene preservando ao mesmo tempo a integridade da célula dentro do tecido que a rodeia, o que permite ditar interpretações anatômicas significativas. Esta técnica é o resultado de uma reação na qual uma sonda marcada se une a uma sequência de ácido nucleico complementares entre si. Os métodos de HIS são aplicáveis em pesquisa clínica e em patologia diagnóstica, sendo muito utilizados para buscar expressão de genes cromossômicos ou para detectar a presença de bactérias ou vírus em tecidos infectados, já que permite diferenciar os agentes contaminantes dos verdadeiros agentes patogênicos em um processo infeccioso.
Palavras chave: ADN, ARN, hibridização in situ, sondas.
INTRODUCCIÓN
La hibridización in situ (HIS) se realizó por primera vez en 1969 usando ARN 28S o ADN radiomarcado, que hibridiza preparados citológicos de oocitos de Xenopus y fuera detectado por medio de microautorradiografía; esta técnica permitió desde ese momento examinar secuencias de ácidos nucleicos dentro de las células sin alterar su morfología o la integridad de sus componentes. A través de los años la técnica ha sido modificada para estudios de evolución cromosomal, estudios moleculares de tumores y leucemias, y estudios citogenéticos en una gran variedad de especies. En 1988 se diseñó la primera sonda para la detección microscópica de bacterias; recientemente el desarrollo de sondas noisotópicas ha generado una gran expansión en el uso de HIS para diagnóstico clínico que permite identificar la expresión de genes oncológicos y la presencia de agentes patógenos infecciosos como virus, bacterias, hongos y parásitos (1, 2).
La hibridización involucra el apareamiento de hebras complementarias de ácidos nucleicos (ADN o ARN). Esta reacción es reversible y las hebras híbridas pueden ser separadas mediante tratamientos con calor o sustancias alcalinas para interrumpir los puentes de hidrógeno que se forman entre las bases nitrogenadas complementarias. Esta es una de las herramientas más importantes en el análisis molecular; si se adiciona un marcador a una de las hebras de ácido nucleico, puede ser utilizada como sonda para el análisis de HIS, que permite la visualización de las señales hibridizadas por medio de métodos isotrópicos o colorimétricos. La principal ventaja de los métodos de HIS incluye la especificidad por células individuales en un tejido heterogéneo o una población celular, y su alta sensibilidad para determinar la expresión, incluso a bajo nivel, de un gen específico en células o en mapeo genético cromosomal. La sensibilidad de HIS no depende tanto de la especie animal sino del tipo de tejido que se va a investigar, teniendo en cuenta su obtención, fijación y tratamiento (1-5).
Este artículo de revisión se enfoca en las bases generales de la técnica de HIS y muestra las ventajas y desventajas de su uso para la identificación de agentes patógenos en diagnóstico clínico o en investigación veterinaria.
SONDAS
Una sonda es un ácido nucleico (ADN o ARN) cuya secuencia de nucleótidos es complementaria a la del ácido nucleico de interés (6). Cualquier fuente de ADN o ARN puede ser utilizada para obtener sondas de HIS. La HIS de ADN aporta información acerca de la organización, localización, distribución, número de copias, cambios evolutivos y mezclas con otras secuencias de ácidos nucleicos, mientras que la HIS de ARN aporta información acerca de la localización y el grado de expresión de un gen en particular (7). Se deben tener en cuenta varios factores para elegir las sondas de HIS, entre ellos se tienen la especificidad y sensibilidad, facilidad en la penetración de los tejidos, estabilidad de los híbridos y repetitividad de la técnica (8).
TIPOS DE SONDAS
Sondas ADN
Las sondas de ADN pueden generarse por: a) vectores de clonación, en donde los clones tienen una porción del ADN de un organismo determinado; b) amplificación de secuencias específicas de ADN mediante una reacción en cadena de la polimerasa (PCR) utilizando un ADN molde y los primers o cebadores apropiados, y c) a partir de ADN genómico aislado del núcleo de un organismo; estas sondas se utilizan en investigación viral, bacteriana o genómica (1, 2, 7, 9-15).
Sondas ARN
Las ribosondas se obtienen mediante un proceso de transcripción in vitro a partir de un ADN molde linear que incorpora nucleótidos marcados. Son de una sola hebra y más susceptibles a la degradación por acción de ARNsas; sin embargo, las sondas ARNc tienen la ventaja de tener híbridos ARN-ARN más estables que los ADN-ADN o los ADN-ARN (7, 12, 16-20).
Sondas de oligonucleótidos
Las sondas de oligonucleótidos están compuestas por 20 a 50 bases que pueden ser generadas con un sintetizador de ADN automático. Estas sondas penetran las células más rápidamente y generan excelentes señales de hibridización (10, 21-27).
MARCADORES Y DETECCIÓN DE LA SEÑAL
Para la HIS los nucleótidos en las sondas tienen que ser modificados o marcados para que se hagan evidentes después del proceso de hibridización con el material genético de interés (1, 7).
Marcadores radioisótopos
Es la forma más tradicional de HIS por ser la más sensible; las desventajas de estas sondas incluyen una vida media corta, peligro biológico, y el hecho de que toman un largo tiempo en el proceso para obtener resultados (1, 7, 27-30).
Marcadores noisotópicos
Las ventajas de este método son una mayor estabilidad de las sondas marcadas, resultados rápidos y una mejor resolución. Las sustancias más utilizadas en los protocolos de HIS son la biotina (vitamina H) para marcar la sonda y su afinidad con la avidina o la estreptavidina para detectar los sitios de hibridización; también se utilizan comúnmente la digoxigenina (un esteroide de la Digitalis purpurea) y la fluoresceína (FITC), que son muy fáciles de detectar en los tejidos (1, 2, 7, 8, 31-34). Las sondas marcadas con digoxigenina tienen una mayor sensibilidad y menos marcación inespecífica de fondo que las biotiniladas, además no existe en los tejidos animales (2, 35, 36). Las sondas noisotópicas se consideran menos sensibles que las radiactivas por lo que los resultados de hibridización son difíciles de cuantificar (1).
SEVERIDAD
La hibridización entre la sonda marcada y el ADN o ARN blanco se consigue mediante puentes de hidrógeno e interacciones hidrofóbicas en equilibrio. La homología entre la sonda (ácido nucleico marcado) y el blanco (por ejemplo, ADN cromosomal o ARN celular) que se requiere para que se forme una molécula híbrida (doble hélice) y se mantenga estable se calcula utilizando la temperatura de fusión Tm, del ADN o del ARN, que hace referencia al punto en el cual el 50% de las dobles cadenas de ácido nucleico se separan; es decir, la temperatura en la cual las moléculas de doble cadena y de una sola hebra son equivalentemente estables. La HIS generalmente se lleva a cabo en un rango entre 70% (baja severidad) hasta 90% (alta severidad) (1, 7) y se ve afectada por varios factores como la concentración de cationes monovalentes, formamida, naturaleza de la sonda y el blanco, pH, longitud de los fragmentos de la sonda y el contenido de guanina y citocina (1, 2, 7, 37, 38).
PREPARACIÓN Y FIJACIÓN DE TEJIDOS
La HIS es aplicable en células (frotis, centrifugados), secciones de tejidos (congelados, embebidos en parafina, semidelgados, en cortes ultradelgados de plástico) y en montajes de embriones frescos o de archivo (1, 2, 7). El material embebido en parafina presenta una excelente preservación en la estructura de los tejidos y permite un estudio morfológico tridimensional del mismo, además se pueden tomar varias muestras al tiempo para utilizar diferentes coloraciones y tipos de sonda (2, 7, 9, 21, 36, 39, 40). La fijación ideal para HIS debe preservar tanto el ARN como el ADN y la morfología de los tejidos, además debe permitir la penetración de las sondas. Por regla general la fijación no debe ser superior a 24 horas, incluso 15 a 60 min son suficientes para secciones de tejido delgadas (1, 2, 7, 37). Hay varios pasos que se deben seguir antes de la hibridización para aumentar su eficiencia y eliminar la marcación no específica de fondo. Estos son (figura 1):
Tratamientos con proteasas. El tratamiento con proteasas y en especial la proteinasa K es el paso más importante para incrementar la disponibilidad de los ácidos nucleicos de interés, especialmente en el caso de tejidos embebidos en parafina tratados con sondas noisotópicas, además ayudan a remover proteínas que incrementan marcaciones de fondo indeseables (2, 7, 41, 42).
Detergentes o ácidos. La hidrólisis con ácidos débiles y una incubación en una solución con detergentes aumentan el acceso de la sonda a las secuencias del ácido nucleico blanco (7, 43).
Pretratamiento con microondas. Esta técnica incrementa la sensibilidad de la HIS, es especialmente útil para tejidos de archivo embebidos en parafina (21, 39, 41-43).
Tratamientos de acetilación. La acetilación de los tejidos reduce la unión electrostática de la sonda a los tejidos mediante la acetilación de los grupos aminos con cargas positivas y previene la unión inespecífica de la sonda a las láminas cargadas positivamente o cubiertas con poly-L-lysina (1, 2, 7).
FIJACIÓN DE LOS TEJIDOS
La marcación de los ácidos nucleicos celulares por medio de HIS requiere una muy buena preservación de las moléculas blanco dentro del tejido permitiendo al mismo tiempo un máximo acceso de la sonda a las secuencias específicas que se buscan dentro de las células individuales; sin embargo, este proceso dificulta el acceso de la sonda al ácido nucleico, especialmente si el proceso de fijación es prolongado (21, 39, 44, 45). Es necesario evaluar los mecanismos de acción así como las ventajas y desventajas de cada producto para elegir el apropiado de acuerdo al uso que se le va a dar a un tejido en particular.
Formaldehído
La formalina bufferada al 10 % es el fijador más utilizado ya que preserva un gran rango de tejidos y sus componentes; no obstante, se ha probado que incluso un tratamiento corto con este fijador reduce significativamente la solubilidad del ADN o ARN (21, 39, 45-48). El formaldehído inicia la desnaturalización del ácido nucleico creando sitios para la interacción química y estableciendo así alteraciones y uniones eslabonadas que pueden dañar el ácido nucleico y hacerlo inaccesible, particularmente si se realiza a temperatura ambiente. Hay que recordar que si el tiempo de fijación es prolongado mayores serán los efectos adversos sobre el ADN tisular; en general, se recomienda una duración de 3 a 6 horas usando formalina buferada al 10% fría (4 °C) (46, 49, 50).
Glutaraldehído
Al igual que el formaldehído forma enlaces eslabonados entre proteínas para proteger la integridad de los tejidos; es generalmente usado para microscopía electrónica ya que su baja penetración y necesidad de renovación constante lo limitan como fijador biológico. Sin embargo, se ha demostrado que el glutaraldehído a pH 7,0 tiene una mejor preservación del ADN de alto peso molecular que la formalina bufferada al 10 % (48).
Etanol y metanol
Los fijadores que no generan enlaces eslabonados son mejores que los aldehídos para preservar ácidos nucleicos, el etanol y el metanol al 100 % no generan cambios químicos, son de bajo peso molecular y penetran rápidamente en los tejidos, lo cual contribuye a una fijación uniforme y una mínima pérdida de los componentes tisulares (48).
HIBRIDIZACIÓN
La hibridización propiamente dicha consiste en la unión de la sonda con la diana.
Prehibridización
Para la HIS el material es prehibridizado incubándolo en un buffer de hibridización (que no contenga la sonda) durante una o dos horas a la temperatura de hibridización, con el fin de asegurar la penetración del tejido y bloquear los sitios de unión inespecíficos. La ventaja de este paso es que disminuye la marcación inespecífica y la desventaja es que disminuye la sensibilidad de la técnica (2, 7).
Hibridización
Al buffer de prehibridización se le adiciona la sonda marcada y se incuban las láminas en cámara húmeda para lograr la hibridización del ácido nucleico deseado con la sonda marcada. Para obtener un acoplamiento óptimo entre la sonda y la diana se precisan alrededor de una a dos horas para sondas biotiniladas o conjugadas con fluorocromos y hasta doce horas para sondas conjugadas con digoxigenina (51-53).
Lavados poshibridización
Los lavados después de la hibridización remueven la sonda que no se unió o que tiene uniones débiles con el ADN molde o con la mezcla de hibridización; generalmente, los lavados se realizan con la misma severidad de la hibridización. Cuando se utilizan oligonucleótidos los lavados deben ser suaves, ya que estas sondas son cortas y hay que asegurarse de que permanezcan hibridizadas al blanco (1, 2, 7, 37).
Detección colorimétrica de los sitios de hibridización
La detección de los sitios de hibridización es una parte importante de la HIS, ya que permite la visualización de los híbridos formados entre la sonda y el blanco. Para la detección de los sitios de hibridización ARN o ADN en células, tejidos o embriones se prefieren sistemas cromogénicos mediados por enzimas que generan precipitados colorimétricos insolubles. Anticuerpos (o avidina), conjugados a una enzima, pueden unirse a la sonda marcada ya hibridizada, y luego incubarse con un sustrato cromogénico adecuado para la enzima. Comúnmente la enzima es la fosfatasa alcalina (PA) y se conjuga a la (estrepto) avidina, antidigoxigenina o antifluoresceína; y el sistema de detección que se usa es el NBT/BCIP (4-Nitroazul tetrazolio cloruro y 5-bromo-4-cloro-3-indoil-fosfato). Dependiendo de la abundancia del blanco que se quiere detectar la reacción se deja por unos minutos, horas o incluso días (2, 7, 54-56).
Contraste, montaje y evaluación
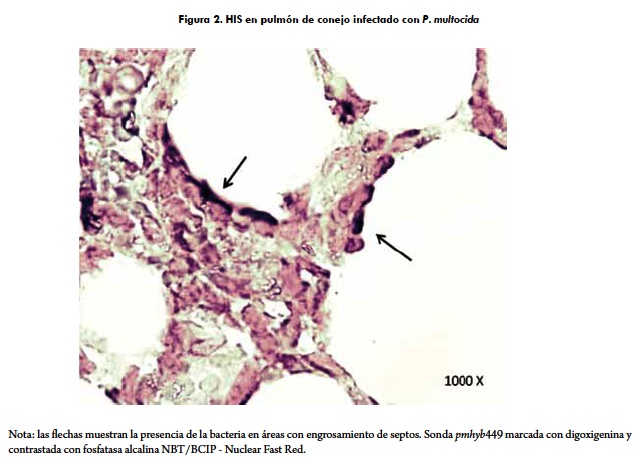
Se pueden utilizar muchos protocolos de coloración para identificar y contrastar los componentes celulares contra el precipitado catalizado por la enzima (p. ej. azul de toluidina, hematoxilina, eosina, verde metilo, nuclear fast red, safranina y giemsa). Como regla general la coloración debe ser débil y contrastar con la señal de HIS. Los montajes basados en xilol pueden inducir formación de cristales de NBT/BCIP, por esto se recomienda el uso de soluciones acuosas a base de glicerol, sin embargo se debe recordar que este no es un montaje permanente (2, 7, 37). La evaluación y el análisis de las láminas dependen de cada experimento y del método de marcación utilizado. La HIS puede ser evaluada por medio de microscopía de luz, microscopía electrónica de transmisión o de barrido, epifluorescencia, microscopía confocal, etc. (1, 19, 57, 58) (figura 2).
APLICACIONES DE LA HIBRIDIZACIÓN IN SITU
Los métodos de HIS son aplicables en tejidos vivos o fijados y procesados de biopsias o material de necropsia, y han demostrado ser preferibles a otras técnicas convencionales como la reacción en cadena de la polimerasa (PCR), inmunohistoquímica o cultivos bacterianos para el diagnóstico y la identificación de agentes patógenos (tabla 1). En el caso de la PCR ambas técnicas tienen buena sensibilidad y especificidad; sin embargo, la PCR no permite identificar los microorganismos dentro del tejido lo cual limita su aplicabilidad en investigación y en oncología, donde se prefiere tener la ubicación exacta de las células cancerígenas para un mejor tratamiento y un pronóstico más certero. La inmunohistoquímica, por su parte, permite identificar la molécula blanco dentro de los tejidos, pero tiene una sensibilidad más baja que HIS y una alta incidencia de marcación inespecífica que puede generar resultados engañosos; por último, los cultivos bacterianos son poco sensibles, se contaminan fácilmente y toman varios días e incluso semanas para mostrar un resultado; a pesar de ser una de las técnicas más utilizadas en el diagnóstico de agentes infecciosos como bacterias u hongos, es una de las menos confiables (1, 7, 37, 59, 60). A continuación se muestran algunas de las aplicaciones de HIS en el diagnóstico de agentes patógenos.
Virus
En el campo de las enfermedades infecciosas la HIS ha sido muy utilizada para detectar la presencia de virus o definir la extensión de la infección sistémica en secciones histológicas o preparados citológicos. Anteriormente se sospechaba la presencia de un virus durante un proceso patológico por los síntomas o por efectos citopáticos observados en láminas histopatológicas, sin embargo, con esta técnica ahora es posible ver y confirmar la presencia de estos patógenos en tejidos o preparados citológicos; ha sido especialmente usada en humanos para el diagnóstico de cytomegalovirus (CMV), papilomavirus (HPV), Eipsten-Barr virus (EBV) y herpes virus (HPV) entre otros. Para este último, HIS no solo permite identificar la presencia del virus sino también diferenciar si es un subtipo de alto o bajo riesgo (1, 6).
Bacterias
La HIS se ha usado para estudiar fisiología bacteriana, enumerar bacterias viables y examinar biofilms (61-64). En medicina veterinaria se ha aplicado para estudiar bacterias como Streptococcus suis en cerdos (65), Chlamydia trachomatis en cerdos y ratones (66), Campylobacter spp. (25) Haemophilus somnus (67), Streptococcus agalactiae, Streptococcus uberis, Arcanobacterium pyogenes (68), Actinobacillus pleuropneumoniae (69) y Listeria spp. (70), P. multocida (12, 71, 72). En el caso del Bacillus Calmette-Guérin involucrado en el cáncer de vejiga, la HIS, y más específicamente FISH (Fluorescent in situ hybridization), no solo ayuda para el diagnóstico de la enfermedad activa, sino que ayuda a identificar pacientes en riesgo de recurrencia del tumor, mientras se llevan a cabo las inmunoterapias que permiten a combatir este tipo de cáncer (73).
Hongos
La identificación de hongos y levaduras en láminas histopatológicas o preparados citológicos se basa en las características morfológicas de los organismos presentes, sin embargo, este tipo de estructuras tiende a tener una baja densidad con morfologías similares y se sobreponen unas a otras, es por esto que los diagnósticos se limitan a "morfología consistente con... "; HIS permite un excelente y rápido método diagnóstico que con certeza identifica el tipo de hongo presente en una muestra determinada sin tener que esperar más de un mes como en el caso de los cultivos (1).
Parásitos
Hay algunos parásitos que son difíciles de identificar en preparados citológicos o en muestras coproparasitológicas, es por esto que la técnica de HIS ha sido muy útil en el diagnóstico y estudio microbiológico de parásitos como helmintos y tremátodos (1, 7), mycoplasmas (74), hemoparásitos como la Haemobartonella felis en anemia infecciosa felina (15) o la Babesia gibsoni (33), y protozoarios como microsporidios, tripanosomas, plasmodios, trichomonas (1, 75).
VENTAJAS Y DESVENTAJAS
La HIS es una prueba mucho más sensible que otras (6, 67, 76), la principal ventaja de esta técnica se basa en la especificidad por células individuales manteniendo la morfología del tejido, lo cual la convierte en un procedimiento ideal para investigación; por otro lado, la HIS de ADN aporta información acerca de la organización, localización, distribución, número de copias, cambios evolutivos y mezclas con otras secuencias de ácidos nucleicos (76), mientras que la HIS del ARN aporta información acerca de la localización y el grado de expresión de un gen en particular (7, 12, 16, 26, 34, 37, 77-82). Sin embargo, es una técnica costosa que requiere de espacio, infraestructura y equipos adecuados para el trabajo con biología molecular, el personal debe ser capacitado y entrenado para realizar esta técnica y el tiempo de estandarización puede ser prolongado (1, 81, 83).
REFERENCIAS
1. Lloyd RV. Introduction to Molecular Methods. En: Morphology Methods. Cell and Molecular Biology Techniques. New Jersey: Humana Press; 2001. p. 1-10. [ Links ]
2. Morel G, Cavalier A. In situ hybridization in light microscopy. Florida: CRC Press LLC; 2001. p. 25-112. [ Links ]
3. Enomoto H, Inoue S, Matsuhisa A, Aizawa N, Imanishi H, Saito M et ál. Development of a new in situ hybridization method for the detection of global bacterial ADN to provide early evidence of a bacterial infection in spontaneous bacterial peritonitis. J Hepatol. 2012; 56: 85-94. [ Links ]
4. Gray JW, Pinkel D and Brown JM. Fluorescence in situ hybridization in cancer and radiation biology (Review). Radiat Res. 1994; 137: 275-289. [ Links ]
5. Pors S, Chadfield M, Sorensen D, Offenberg H, Heegard P, Bisgaard M, et ál. Pathology, tissue metalloproteinase transcription and haptoglobin responses in mice after experimental challenge with different isolates of Pasteurella multocida obtained from cases of porcine pneumonia. J Comp Path. 2011; 145: 251-260. [ Links ]
6. Sekiguchi M, Shibahara T, Miyazaki A, Tajima T, Shimizu S, Kabali E, et ál. In situ hybridization and immunohistochemestry for the detection of porcine cytomegalovirus. J Virol Meth. 2012; 179: 272-275. [ Links ]
7. Schwarzacher T, Heslop-Harrison P. Practical in situ Hybridization. New York: BIOS Scientific Publishers; 2000. P. 1-203. [ Links ]
8. Lloyd RV and Jin L. In situ hybridization analysis of chromogranin A and B mRNAs in neuroendocrine tumors with digoxigenin-labeled oligonucleotide probe cocktails. Diagn Mol Pathol. 1995; 4: 143-151. [ Links ]
9. Mertz L, Rashtchian A. Nucleotide imbalance and polymerase chain reaction: effects on DNA amplification and synthesis of high specific activity radiolabeled DNA probes. Anal Biochem. 1994; 221: 160-165. [ Links ]
10. Lloyd RV, Jin L, Kulig E, Fields K. Molecular approches for the analysis of chromogranins and secretogranins. Diagn Mol Pathol. 1993; 1: 2-15. [ Links ]
11. Poppert S, Essig A, Marre R, Wagner M and Horn M. Detection and Differentiation of Chlamydiae by Fluorescence In Situ Hybridization. Appl Environ Microbiol. 2002; 68: 4081-4089. [ Links ]
12. Mbuthia PG, Christensen H, Boyce M, Majken K, Petersen D, Bisgaard M, et ál. Specific Detection of Pasteurella multocida in Chickens with Fowl Cholera and in Pig Lung Tissues Using Fluorescent rRNA In Situ Hybridization. J Clin Microbiol. 2001; 39: 2627-2633. [ Links ]
13. Jauch A, Daumer C, Lichter P, Murken J, Schroeder KT, Cremer T. Chromosomal in situ supression hybridization of human genosomes and autosomes and its use in clinical cytogenetics. Hum Genet. 1990: 85: 145-150. [ Links ]
14. Lichter P, Cremer T, Borden J, Manuelidis L, Ward DC. Delineation of individual chromosomes in metaphase and interphase cells by in situ supression hybridization using recombinant DNA libraries. Hum Genet. 1988; 80: 224-234. [ Links ]
15. Berent L, Messick J, Cooper S, Cusick P. Specific in situ hybridization of Haemobartonella felis with DNA probe and tyramide signal amplification. Vet Pathol. 2000; 37: 47-53. [ Links ]
16. Brown C. Review Article. In Situ Hybridization with Riboprobes: An Overview for Veterinary Pathologists. Vet Path. 1998; 35: 159-167. [ Links ]
17. Chakravorty S, Helb D, Burday M, Connell N, Alland D. A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria. J Microbiol Methods. 2007; 69: 330-339. [ Links ]
18. Pereira M, Singer R, Meirellas M. Ultrastructural distribution of poly (A)+ RNA during Trypanosoma cruzi-cardiomyocyte interaction in vitro: a quantitative analysis of the total mRNA content by in situ hybridization. J Eukaryot Microbiol. 2000; 47: 264-270. [ Links ]
19. Sibon OC, Cremers FF, Boonstra HJ, Verkeij AJ. Localization of nuclear RNA by pre- and post-embedding in situ hybridization using different gold probes. Histochem J. 1995; 37: 35-45. [ Links ]
20. Ueda T, Kobori A, Yamayoshi A, Yoshida H, Yamaguchi M, Murakami A. RNA-based diagnosis in a multicellular specimen by whole mount in situ hybridization using an RNA-specific probe. Accepted Manuscript. Bioorg Med Chem [Internet], 2012. Disponible en: http://dx.doi.org/10.1016/).bmc.2012.08.028 [ Links ]
21. Oliver KR, Heavens RP, Sirinathsinghji DJ. Quantitative comparison of pretreatment regimens used to sensitize in situ hybridization using oligonucleotide probes on paraffin-embedded brain tissue. J Histochem Cytochem. 1997; 45: 1707-1713. [ Links ]
22. Lloyd RV, Cano M, Chandler WF, Barkan AL, Horvath E, Kovacs K. Human growth hormone and prolactine secreting pituitary adenomas analysed by in situ hybridization. Am J Pathol. 1989; 134: 605-613. [ Links ]
23. Pagani A, Cerrato, Bussolati G. Nonspecific in situ hybridization reaction in neuroendocrin cells and tumors of the gastrointestinal tract using oligonucleotide probes. Diagn Mol Pathol. 1993; 2: 125-130. [ Links ]
24. Stahl WL, Eakin TJ, Baskin DG. Selection of oligonucleotide probes for detection of mRNA isoforms. J Histochem Cytochem. 1993; 41: 1735-1740. [ Links ]
25. Lehtola M, Loades C, Keevil W. Advantages of peptide nucleic acid oligonucleotides for sensitive site directed 16S rRNA fluorescence in situ hybridization (FISH) detection of Campylobacter jejuni, Campylobacter coli and Campylobacter lari. J Microbiol Meth. 2005; 62: 211-219. [ Links ]
26. Kornreich B, Craven M, McDonough S, Nydam D, Scorza V, Assarasakorn S, et ál. Fluorescence in-situ hybridization for the identification of bacterial species in archival heart valve sections of canine bacterial endocarditis. J Comp Path. 2012; 146: 298-307. [ Links ]
27. Dagerlind A, Friberg K, Bean AJ Hokfelt T. Sensitive detection using unfixed tissues: combined radioactive and nonradioactive in situ hybridization histochemistry. Histochemistry. 1992; 98: 39-43. [ Links ]
28. Steel JH, Jeffery RE, Longcroft JM, Rogers LA, Poulsom R. Comparison of isotropic and non-isotropic labeling for in situ hybridization of various mRNA targets with cRNA probes. Eur J Histochem. 1998; 42: 143-150. [ Links ]
29. Kallio P, Syrjanen S, Tervahauta A, Syrjanen K. A simple method for isolation of DNA from formalin-fixed paraffin-embedded samples for PCR. J Virol Meth. 1991; 35: 39-47. [ Links ]
30. Steel JH, Jeffery RE, Longcroft JM, Rogers LA, Poulsom R. Comparison of isotropic and non-isotropic labeling for in situ hybridization of various mRNA targets with cRNA probes. Eur J Histochem. 1998; 42: 143-150. [ Links ]
31. Durrant I, Chadwick P. Hybridization of fluorescein-labeled oligonucleotide probes and enhanced chemiluminescence detection. En: Protocols for Nucleic Acid Analysis by Nonradioactive probes. Methods in Molecular Biology, vol. 28. USA: Humana Press; 1994. p. 141-148. [ Links ]
32. Sheldon S. Fluorescent in situ hybridization. En: Morphology Methods. Cell and Molecular Biology Techniques. USA: Humana Press; 2001. p. 67-89. [ Links ]
33. Yamasaki M, Kobayashi Y, Nakamura K, Sasaki N, Murakami M, Rajapakshage B, et ál. Babesia gibsoni: Detection in blood smears and formalin-fixed, paraffin embedded tissues using deoxyribonucleic acid in situ hybridization analysis. Experiment Parasitol. 2011; 127: 119-126. [ Links ]
34. Bouvier T, del Giorgio T. MiniReview. Factors influencing the detection of bacterial cells using fluorescence in situ hybridization (FISH): A quantitative review of published reports. FEMS Microbiol Ecol. 2003; 44: 3-15. [ Links ]
35. Schmitz G, Waiter T, Seibl R, Kessler C. Nonradioactive labeling of oligonucleotides in vitro with the hapten digoxigenin by tailing with terminal transferase. Anal Biochem. 1991; 192: 222-231. [ Links ]
36. Komminoth P, Werner M. Target and signal amplification: approaches to increase the sensitivity of in situ hybridization. Histochem Cell Biol. 1997; 108: 325-333. [ Links ]
37. Kadkol S, Gage W, Pasternack G. Molecular Diagnosis Reviews. In Situ Hybridization Theory and Practice. Mol Diag. 1999; 4 (3): 169-183. [ Links ]
38. Wetmer J, Ruyechen W, Douthart R. Denaturation and renaturation of Penicillium chrysogenum mycophage double-stranded ribonucleic acid in tetra alkylammonium salt solutions. Biochemistry. 1981; 20: 2999-3002. [ Links ]
39. Lan H, Mu Wei, NG Y, Nikolic-Paterson D, Atkins R. A simple, reliable, and sensitive Method for nonradioactive in situ hybridization: Use of microwave heating to improve hybridization efficiency and preserve tissue morphology. J Histochem Cytochem. 1996; 44 (3): 281-287. [ Links ]
40. Hayat M. Microscopy, Immunohistochemistry and antigen retrieval methods. New York: Kluwer Academic Plenum Publishers; 2002. [ Links ]
41. Shi S, Key M, Kalra K. Antigen retrieval in formalin-fixed, paraffin-embedded tissues: An enhancement method for immunohistochemical staining based on microwave oven heating of tissue sections. J Histochem Cvtochem. 199; 39: 741-748. [ Links ]
42. Shi S, Cote R, Taylor C. Antigen retrieval techniques: Current perspectives. J Histochem Cvtochem. 2001; 49: 931-937. [ Links ]
43. Kitayama Y, Igarashi H, Kozu T, Nagura K, Ohashi Y, Sugimura H. Repeated fluorescense in situ hybridization by a microwave-enhanced protocol. Path International. 2006; 56: 490-493. [ Links ]
44. Ben-Ezra J, Johnson D, Rossi J, Cook N, Wu A. Effect of fixation on the amplification of nucleic acids from paraffin-embedded material by the polymerase chain reaction. J Histochem Cytochem. 1991; 39: 351-354. [ Links ]
45. Greer C, Peterson S, Kiviat N, Manos M. PCR amplification from paraffin-embedded tissues: effects on fixative and fixation time. Am J Clin Pathol. 1991; 95: 117-124. [ Links ]
46. Shibata D, Martin W, Arnheim N. Analysis of DNA sequences in forty-year old paraffin-embedded thin-tissue sections: a bridge between molecular biology and classic histology. Cancer Res. 1988; 48: 4564-4566. [ Links ]
47. Speel E, Hopman AH and Komminoth P. Amplification methods to increase the sensitivity of in situ hybridization: play CARD(S). J Histochem Cytochem. 1999; 47: 281-288. [ Links ]
48. Srinivasan M, Sedmak D, Jewell S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. Am J Path. 2002; 161 (6): 1961-1971. [ Links ]
49. Alers J, Krijtenburg P, Vissers K, van Dekken H. Effect of bone decalcification procedures on DNA in situ hybridization and comparative genomic hybridization: EDTA is highly preferable to a routinely used acid decalcifier. J Histochem Cytochem. 1999; 47 (5): 703-709. [ Links ]
50. Whitlon D, Szakaly R , Greiner M. Cryoembedding and sectioning of cochleas for immunocytochemistry and in situ hybridization. Brain Res Protoc. 2001; 6: 159-166. [ Links ]
51. Gebeyehu G, Rao P, SooChan P, Simms D, Klevan L. Novel biotinylated nucleotide analogs for labeling and colorimetric detection of DNA. Nucleic Acids Res. 1987; 15: 4513-4534. [ Links ]
52. Kessler C, Hõltke H, Seibl R, Burg J, Mühlegger K. Nonradioactive labeling and detection of nucleic acids: I. A novel DNA labeling and detection system based on digoxigenin: anti-digoxigenin ELISA principle. Biol Chem. 1990; 371: 917-927. [ Links ]
53. Polak J, McGee J. In Situ Hybridization: Principles and Practice. Oxford: University Press; 1990. p. 10-30. [ Links ]
54. Vass K, Berger M, Nowak T, Welch W, Lassmann H. Induction of stress protein HSP 70 nerve cells after status epilepticus in rat. Neurosci Lett. 1989; 100: 259-264. [ Links ]
55. Strehl S, Ambros P. Fluorescent in situ hybridization combined with immunohistochemistry for highly sensitive detection of chromosome 1 aberrations in neuroblastoma. cytogenet. Cell Genet. 1993; 63: 24-28. [ Links ]
56. Ponder B, Wilkinson M. Inhibition of endogenous tissue alkaline phosphatase with the use of alkaline phosphatase conjugates in mmunohistochemistry. J Histochem. 1981; 29: 981-984. [ Links ]
57. Lloyd RV, Jin L, Song J. Ultrastructural localization of prolactin and chromogranin B messenger ribonucleic acids with biotinylated oligonucleotide probes in cultured pituitary cells. Lab Invest. 1990; 63: 413-319. [ Links ]
58. Bienz K and Egger D. Immunocytochemistry and in situ hybridization in the electron microscope: combined application in the study of virus-infected cells. Histochemistry. 1995; 103: 328-338. [ Links ]
59. Takahashi T, Ishiguro K. Development of an automatic machine for in situ hybridization and immunohistochemistry. Anal Biochem. 1991; 196: 390-402. [ Links ]
60. McNicol A, Farquharson M. In situ hybridization and its diagnostic applications in pathology. J Pathol. 1997; 182: 250-261. [ Links ]
61. Braun-Howland R, Danielsen S, Nierzwicki-Bauer S. Development of a rapid method for detecting bacterial cells using 16S rRNA-targeted probes. Biotechniques. 1992; 13: 928-934. [ Links ]
62. Manz W, Szewzyk U, Ericsson P, Amann R, Schleifer K, Stenstrom T. In situ identification of bacteria in drinking water and adjoining biofilms by hybridization with 16S and 23S rRNA-directed fluorescent oligonucleotide probes. Appl Environ Microbiol. 1993; 59: 2293-2298. [ Links ]
63. Shiaishi F, Zippel B, Neu T, Arp G. In situ detection of bacteria in calcified biofilms using FISH and CARD-FISH. J Microbiol Meth. 2008; 75: 103-108. [ Links ]
64. DeLong D, Manning PJ, Gunther R, Swanson DL. Colonization of rabbits by Pasteurella multocida: serum IgG responses following intranasal challenge with serologically distinct isolates. Lab Anim Sci. 1992; 42: 13-8. [ Links ]
65. Boye M, Feenstra A, Tegtmeier C, Andresen L, Rasmussen S, Bille-Hansen V. Detection of Streptococcus suis by in situ hybridization, indirect immunofluorescence, and peroxidase-antiperoxidase assays in formalin-fixed, paraffin-embedded tissue sections from pigs. J Vet Diagn Invest. 2000; 12: 224-232. [ Links ]
66. Chae C, Cheon D, Kwon D. In situ hybridization for the detection and localization of swine Chlamydia trachomatis. Vet Pathol. 1999; 36: 133-137. [ Links ]
67. Tegtmeier C, Angen 0, Ahrens P. Comparison of bacterial cultivation, PCR, in situ hybridization and immunohistochemistry as tools for diagnosis of Haemophilus somnus pneumonia in cattle. Vet Microbiol. 2000; 76: 385-394. [ Links ]
68. Werckenthin C, Gey A, Straubinger R, Poppert S. Rapid identification of the animal pathogens Streptococcus uberis and Arcanobacyerium pyogenes by fluorescence in situ hybridization (FISH). Vet Microbiol. 2012; 156: 330-335. [ Links ]
69. Kim C, Oh Y, Han K, Seo H, Kim D, Kang I et ál. Expression of secreted mucins (MUC2, MUC5AC, MUC5B and MUC6) and membrane-bound mucin (MUC4) in the lungs of pigs experimentally infected with Actinobacillus pleuropneumoniae. Res Vet Sci. 2012; 92: 486-491. [ Links ]
70. Zhang X, Wu S, Shuai J, Dong Q, Fang W. Peptide nucleic acid fluorescence in situ hybridization for identification of Listeria genus, Listeria monocygenes and Listeria ivanovii. Internal J Food Microbiol. 2012; 157: 309-313. [ Links ]
71. Pors S, Hansen M, Bisgaard M, Jensen H. a. Occurrence and associated lesions of Pasteurella multocida in porcine bronchopneumonia. Vet Microbiol. 2011; 150: 160-166. [ Links ]
72. Pors S, Hansen M, Christensen H, Jensen H, Petersen A, Bisgaard M c. Genetic Diversity and associated pathology of Pasteurella multocida from porcine pneumonia. Vet Microbiol. 2011; 150: 354-361. [ Links ]
73. Kamat A, Dickstein R, Messetti F, Anderson R, Pretzsch S, Noguera G et ál. Use of Fluorescence In Situ Hybridization to Predict Response to Bacillus Calmette-uérin Therapy for Bladder Cancer: Results of a Prospective Trial. J Urol. 2012; 187: 862-867. [ Links ]
74. Ha S, Jung K, Choi C, Ha Y, Song H, Lim J et ál. Development of In-situ hybridization for the detection of Mycoplasma haemosuis (Eperythrozoon suis) in formalin-fixed, paraffin wax-embedded tissues from experimentally infected splenectomized pigs. J Comp Path. 2005; 133: 294-297. [ Links ]
75. Mostegl M, Richter B, Nedorost N, Maderner A, Dinhopl N, Weissenböck H. Investigations on the prevalence and potential pathogenicity of intestinal trichomonads in pigs using in situ hybridization. Vet Parasitol. 2011; 178: 58-63. [ Links ]
76. Wu Q, Li Y, Wang M, Pan X, Tang Y. Fluorescence in situ hybridization rapidly detects three different pathogenic bacteria in urinary tract infection samples. J Microbiol Meth. 2010; 83: 175-178. [ Links ]
77. Cheon D, Chae C, Lee Y. Detection of nucleic acids of porcine reproductive and respiratory syndrome virus in the lungs of naturally infected piglets as determined by in situ hybridization. J Comp Path. 1997; 117: 157-163. [ Links ]
78. Boyce J, Chung J, Adler B. Pasteurella multocida capsule: composition, function and genetics. J Biotechnol. 2000; 83: 153-160. [ Links ]
79. Coleman J, Culley D, Chrisler W, Brockman F. mRNA-targeted fluorescent in situ hybridization (FISH) of Gram-negative bacteria without template amplification or tyramide signal amplification. J Microbiol Methods. 2007; 71: 246-255. [ Links ]
80. Svendsen C, Boye M, Struve C, Krogfelt K. A novel fluorescent in situ hybridization technique for detection of Rickettsia spp. In archival samples. J Microbiol Meth. 2009; 76: 301-304. [ Links ]
81. Stroot J, Leach K, Stroot P, Lim D. Capture antibody targeted fluorescence in situ hybridization (CAT-FISH); Dual labeling allows for increased specificity in complex samples. J Microbiol Meth. 2012; 88: 275-284. [ Links ]
82. Wagner M, Haider S. New Trends in fluorescence in situ hybridization for identification and functional analysis of microbes. Curr Op Biotech. 2012; 23: 96-102. [ Links ]
83. Lee Y, Ha Y, Ahn K, Cho K, Lee B, Kim S, Chae C. Comparison of a new synthetic, peptide-derived, polyclonal antibody-based, immunohistochemical test with in situ hybridization for the detection of swine hepatitis E virus in formalin-fixed, paraffin-embedded tissues. Vet J. 2009; 182: 131-135. [ Links ]