Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de Medicina Veterinaria
Print version ISSN 0122-9354
Rev. Med. Vet. no.30 Bogotá July/Dec. 2015
Diversidad molecular de candidatos vacunales en Leptospira spp.
Molecular Diversity of Vaccine Candidates in Leptospira spp.
Diversidade molecular de candidatos para vacina em Leptospira spp.
Patricia Hernández-Rodríguez1 / Mónica Baquero P.2 / Andrés Felipe Santander Torres3 / Arlen Gómez4
1 MSc. Universidad de La Salle, Departamento de Ciencias Básicas, Programa de Biología. Grupo de investigación Biología Molecular e Inmunogenética (Biomigen), Bogotá, Colombia.
phernandez@unisalle.edu.co
2 MSc. Universidad de La Salle, Facultad de Ciencias Agropecuarias, Programa de Medicina Veterinaria, Bogotá, Colombia.
mmbaquero@live.com
3 Universidad de La Salle, Facultad de Ciencias Agropecuarias, Programa de Medicina Veterinaria, Bogotá, Colombia.
andresfsantander@lasalle.edu.co
4 PhD. Universidad de La Salle, Facultad de Ciencias Agropecuarias, Programa de Medicina Veterinaria, Bogotá, Colombia.
agomez@unisalle.edu.co
Recibido: 16 de marzo del 2015 / Aceptado: 17 de abril del 2015
Cómo citar este artículo: Hernández-Rodríguez P, Baquero M, Santander Torres AF, Gómez A. Diversidad molecular de candidatos vacunales en Leptospira spp. Rev Med Vet. 2015;(30):95-106.
Resumen
El objetivo de este estudio fue determinar la diversidad molecular de las proteínas OmpL1, LipL32, LipL41, LigA y LigB y de los genes que las codifican mediante análisis bioinformáticos en diferentes cepas patógenas de Leptospira spp., a partir de la información disponible en las bases de datos. Se utilizaron las secuencias de aminoácidos de las proteínas OmpL1, LipL32, LipL41, LigA y LigB, así como las de los genes que las codifican en las cepas de Leptospira spp. registradas en The National Center for Biotechnology Information (NCBI). Los análisis de las proteínas y los genes se realizaron mediante los recursos Protein, Nucleotide y Gene del NCBI. La alineación de las secuencias consenso se realizó con las herramientas PSI-BLAST y BLASTn. El porcentaje de cobertura de las secuencias seleccionadas de los genes ompL1, lipL32, lipL41, ligA y ligB en cepas patógenas de Leptospira spp. es de 100% para ompL1, lipL32 y lipL41, 75% para ligA y 99% para ligB con porcentajes de identidad de 85, 98, 88, 90 y 80% respectivamente; el porcentaje de cobertura de las secuencias seleccionadas de las proteínas es de 100, 77, 99, 100 y 100% con porcentajes de identidad de 90, 99, 92, 63 y 60% respectivamente, lo cual indica que los genes y las proteínas, excepto las proteínas LigA y LigB, son bastante conservadas en los diferentes serovares patógenos de Leptospira spp. Según dichos resultados, se recomienda realizar análisis complementarios de estas proteínas con el fin de determinar si es viable su uso como candidatos vacunales.
Palabras clave: diversidad, OmpL1, LipL32, LipL41, LigA, LigB.
Abstract
The aim of this study was to determine the molecular diversity of OmpL1, LipL32, LipL41, LigA and LigB proteins and that of the genes that encode them using bioinformatic analysis in different pathogenic strains of Leptospira spp. based on the information available in databases. The amino acid sequences of OmpL1, LipL32, LipL41, LigA and LigB proteins were used, as well as the genes encoding them in strains of Leptospira spp. reported at The National Center for Biotechnology Information (NCBI). The analysis of proteins and genes were performed using the Protein, Nucleotide and Gene resources from the NCBI. The alignment of the consensus sequences was performed using the PSI-BLAST and BLASTn tools. The coverage percentage of the selected sequences ofthe ompL1, lipL32, lipL41, ligA and ligB genes in pathogenic strains of Leptospira spp. is 100% for ompL1, lipL32 and lipL41, 75% for ligA and 99% for ligB with identity percentages of 85, 98, 88, 90 and 80% respectively; the coverage percentage of the selected protein sequences is 100, 77, 99, 100 and 100% with identity percentages of 90, 99, 92, 63 and 60% respectively, indicating that genes and proteins, except LigA and LigB proteins, are highly conserved in various pathogenic serovars of Leptospira spp. According to these results, it is recommended that further analysis of these proteins be made in order to determine the feasibility of its use as vaccine candidates.
Keywords: diversity, OmpL1, LipL32, LipL41, LigA, LigB.
Resumo
El objetivo de este estudo foi determinar a diversidade molecular das proteínas OmpL1, LipL32, LipL41, LigA e LigB e dos genes que as codificam mediante análises bioinformáticas em diferentes cepas patógenas de Leptospira spp. A partir da informação disponível nas bases de dados. Utilizaram-se as sequências de aminoácidos das proteínas OmpL1, LipL32, LipL41, LigA e LigB, assim como as dos genes que as codificam nas cepas de Leptospira spp. Reportadas em The National Center for Biotechnology Information (NCBI). As análises das proteínas e dos genes se realizaram mediante os recursos Protein, Nucleotide e Gene do NCBI. O alinhamento das sequências consenso se realizou com as ferramentas PSI-BLAST e BLASTn. A porcentagem de cobertura das sequências selecionadas dos genes ompL1, lipL32, lipL41, ligA e ligB em cepas patógenas de Leptospira spp. É de 100% para ompL1, lipL32 e lipL41, 75% para ligA e 99% para ligB com porcentagens de identidade de 85, 98, 88, 90 e 80% respectivamente; A porcentagem de cobertura das sequências selecionadas das proteínas é de 100, 77, 99, 100 e 100% com porcentagens de identidade de 90, 99, 92, 63 e 60% respectivamente, o que indica que os genes e as proteínas, exceto as proteínas LigA e LigB, são altamente conservadas nos diferentes serovares patogênicos de Leptospira spp. Segundo esses resultados, se recomenda realizar análises complementares destas proteínas com finalidade de determinar se é viável o seu uso como candidatos para vacina.
Palavras chave: diversidade, OmpL1, LipL32, LipL41, LigA, LigB.
INTRODUCCIÓN
La leptospirosis es una enfermedad de distribución mundial (1) que afecta a humanos y animales de zonas tropicales (2,3). Es causada por una espiroqueta del género Leptospira de la cual se han registrado 20 especies y más de 300 serovares agrupados a su vez en 20 serogrupos (4), de las cuales 14 han sido identificadas como potencialmente patógenas (5).
Se han realizado varios trabajos en los que se ha identificado la porina OmpL1, las lipoproteínas LipL32 y LipL41 (6) y las adhesinas LigA y LigB como candidatos potenciales para el desarrollo de vacunas, debido a su alto grado de inmunogenicidad (7). Entre estas la LigB es de mayor interés por estar presente en todos los serovares patógenos, lo cual la convierte en un componente clave para la elaboración de una vacuna recombinante universal para leptospirosis (8).
La primera proteína transmembranal descrita en Leptospira interrogans fue la OmpL1 (9). Su estructura contiene por lo menos diez segmentos transmembranales β y canales de porinas en la bicapa lipídica; su función es permitir la difusión de solutos hidrofílicos a través de la membrana externa hacia el periplasma. Es una proteína de 31 kDa que se encuentra en pequeñas cantidades (10) en todas las cepas patógenas de Leptospira y está ausente en las cepas saprófitas (3,9).
También conocida como Hap1, LipL32 es un antígeno inmunodominante por su capacidad inmunogénica (11), y es el blanco principal de la respuesta inmune tanto en animales como en humanos (12). Es considerada un blanco para el desarrollo de vacunas y nuevos métodos diagnósticos (13,14). La expresión del gen que codifica a LipL32 fue detectada en seis cepas de Leptospira spp. que incluyen cinco cepas de L. interrogans y una de L. borgpetersenii, lo que indica que la proteína LipL32 recombinante es un candidato óptimo como antígeno molecular para el diagnóstico (15). LipL32 es la lipoproteína más abundante de las especies patógenas de Leptospira y se ubica en la superficie de la membrana externa (10,16); sin embargo, estudios recientes realizados por Pinne y Haake, en 2013, buscaron validar los resultados sobre la localización de LipL32 registrados con anterioridad (16), y encontraron que esta proteína, en su mayoría o totalmente, no se ubica en la superficie de la membrana externa. Los resultados que presenta este nuevo estudio fundamentan una localización bajo la superficie de la membrana a partir de la observación de LipL32 en los productos cromatográficos del experimento que incluyó proteólisis de la membrana externa; adicionalmente, se comprobó mediante inmunofluorescencia que LipL32 no fue detectada en la superficie de la membrana externa intacta de L. interrogans pero sí bajo diferentes métodos de permeabilización de membrana. De esta manera, un cambio en la localización de esta lipoproteína presenta una nueva perspectiva que promueve la formulación de trabajos para aclarar el verdadero papel de LipL32 en la leptospirosis, pues a pesar de los diferentes estudios su función sigue siendo desconocida (17).
La lipoproteína LipL41 tiene un peso molecular de 41 kDa y se encuentra exclusivamente en cepas patógenas (18). La secuencia de aminoácidos de esta proteína en L. interrogans serovar Pomona tiene una similitud del 99% con la secuencia de L. kirschneri serovar Grippotyphosa, lo cual sugiere que existe un alto grado de conservación (19). Además existe una correlación positiva entre la patogenicidad y la reactividad con el antisuero de LipL41 (20). Se ha propuesto como antígeno en la prueba de Elisa como herramienta de serodiagnóstico para la leptospirosis bovina (21), y junto con OmpL1 se expresan durante la infección de mamíferos, lo cual produce un efecto inmunogénico sinérgico (22).
Las proteínas LigA, LigB y LigC pertenecen a la familia de las proteínas similares a inmunoglobulinas (Lig). LigA y LigB son consideradas antígenos candidatos para el desarrollo de inmunógenos (23). Los genes que codifican para las proteínas Lig se expresan únicamente en cepas patógenas (24). La proteína LigA recombinante utilizada como vacuna confiere inmunoprotección contra una dosis letal de Leptospira interrogans serovar Pomona con el hámster como modelo experimental (25); esta proteína tiene un peso molecular de 130 kDa (23). La proteína LigB, con un peso molecular de 212 kDa (26), se ha propuesto para ser utilizada como antígeno en vacunas recombinantes debido a que se reveló su protección con una mayor tasa de supervivencia y una reducción en las lesiones histopatológicas de órganos vitales de hámster (27).
A pesar de los diferentes estudios que han publicado secuencias parciales y completas tanto de nucleótidos como de aminoácidos, de genes y proteínas respectivamente, no se han identificado secuencias consenso que se conservan en los serovares patógenos y que pueden llevar al desarrollo de vacunas recombinantes que confieran inmunoprotección contra Leptospira spp. Con la realización del presente trabajo se busca determinar la diversidad molecular de las proteínas OmpL1, LipL32, LipL41, LigA y LigB, y de los genes que las codifican mediante análisis bioinformáticos, para buscar secuencias conservadas en diferentes cepas patógenas de Leptospira spp.
MATERIALES Y MÉTODOS
El presente estudio descriptivo se realizó en las salas de sistemas de la Universidad de La Salle (sede norte), en Bogotá (Colombia). Se utilizaron las secuencias de aminoácidos de las proteínas: OmpL1 (GI: 51475352), LipL32 (GI: 302310467), LipL41 (GI: 56407625), LigA (GI: 218148452) y LigB (GI: 218148514) y las secuencias de nucleótidos de los genes: ompL1 (GI: 51475351), lipL32 (GI: 166084878), lipL41 (GI: 56407624), ligA (GI: 218148451) y ligB (GI: 218148533) de las cepas de Leptospira spp. registradas en el National Center for Biotechnology Information (NCBI) (28). Se emplearon los recursos Nucleotide y Gene para determinar la secuencia, el tamaño y la posición de los genes analizados dentro de las cepas de Leptospira spp. Además se utilizó el recurso Protein para la determinación del número de aminoácidos de las proteínas OmpL1, LipL32, LipL41, LigA y LigB registradas en esta base de datos genética. Así mismo, empleando estas mismas herramientas se consultó el número de registros de las proteínas y de los genes en las especies de L. weilii, L. nogu-chii, L. kirschneri, L. santarosai, L. borgpetersenii y L. interrogans. Las secuencias seleccionadas de los genes fueron alineadas con el recurso BLASTn y las secuencias seleccionadas de proteínas fueron alineadas con PSI-BLAST del NCBI, recursos creados para establecer la variabilidad molecular entre secuencias (29). Para establecer la diversidad molecular se tuvo en cuenta el valor E, que hace referencia a la significancia estadística de la alineación.
RESULTADOS
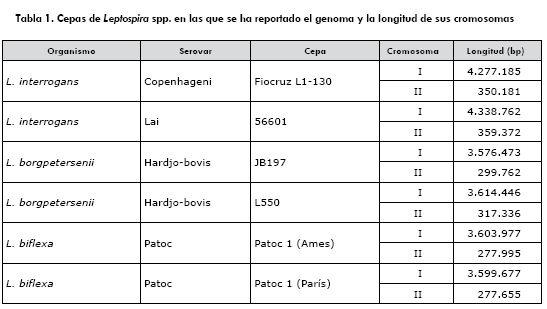
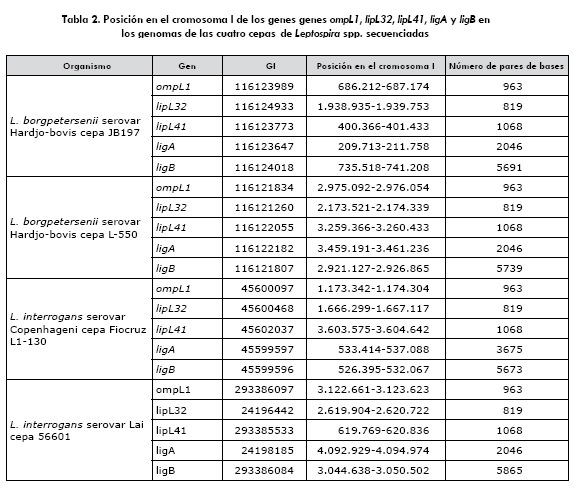
En el NCBI se encontró el reporte del genoma de cuatro cepas patógenas de Leptospira spp.: L. interrogans serovar Copenhageni cepa Fiocruz L1-130 (GI cromosoma I: 45655914; GI cromosoma II: 45655585), L. interrogans serovar Lai cepa 56601 (GI cromosoma I: 294827553; GI cromosoma II: 294653513), L. borgpetersenii serovar Hardjo-bovis cepa JB197 (GI cromosoma I: 116123488; GI cromosoma II: 116332445) y L. borgpetersenii serovar Hardjo-bovis cepa L550 (GI cromosoma I: 116119596; GI cromosoma II:116329556). Además, se identificó e incluyó en el estudio el genoma de dos cepas saprófitas de Leptospira spp.: L. biflexa serovar Patoc cepa "Patoc 1 (Paris)" (GI cromosoma I: 167777849; GI cromosoma II: 189913173) y L. biflexa seorvar Patoc cepa "Patoc 1 (Ames)" (GI cromosoma I: 167774246; GI cromosoma II: 167777524). En la tabla 1 se enmarca la clasificación y la longitud de las secuencias de nucleotidos -bp (base pair)- correspondiente a cada microorganismo. En la tabla 2 se indica la posición en el cromosoma I de los genes consenso de ompL1, lipL32, lipL41, ligA y ligB dentro del genoma de las cuatro cepas patógenas de Leptospira spp.
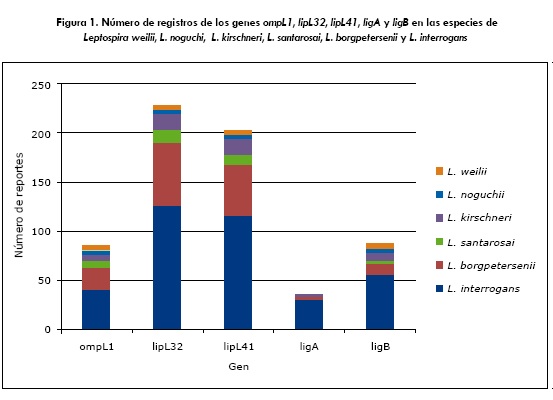
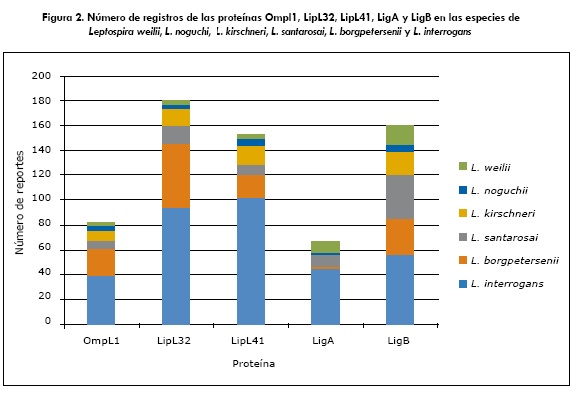
Luego de la revisión en el NCBI se identificó que las secuencias completas de los genes ompL1 , lipL32, lipL41 , ligA y ligB han sido registradas en 33, 40, 40, 12 y 12 cepas, respectivamente. Así mismo, en la figura 1 se indica el número de veces que se generan informes en los que se han identificado los genes ompL1, lipL32, lipL41, ligA y ligB en Leptospira spp. Por otra parte, en la figura 2 se muestra el número de veces que se han registrado las proteínas OmpL1, LipL32, Lipl41, LigA y LigB en las especies de Leptospira.
Al realizar con BLASTn la alineación de las secuencias seleccionadas de los genes ompL1, lipL32, lipL41, ligA y ligB, se observa que el porcentaje promedio de cobertura con las cepas patógenas de Leptospira spp. es de 100% para ompL1, lipL32 y lipL41, 75% para ligA y 99% para ligB. De la misma manera, se obtuvieron porcentajes promedios de identidad de los genes ompL1, lipL32, lipL41, ligA y ligB, frente a las cepas patógenas de Leptospira spp. de 85, 98, 88, 90 y 80% respectivamente (E < 0,5). En la tabla 3 se muestran las secuencias seleccionadas para los análisis de estos genes.
Las secuencias de aminoacidos registrados que se seleccionaron para las proteínas OmpL1, LipL32, LipL41, LigA y LigB tienen 135, 87, 133, 42 y 71 aminoácidos, respectivamente. Con esta información y la identificación de las secuencias, se realizó la alineación de las proteínas OmpL1, LipL32, LipL41, LigA y LigB con PSI-BLAST, lo que permite observar que el porcentaje promedio de cobertura con las cepas patógenas de Leptospira spp. fue de 100% para OmpL1, LipL41, LigA y LigB, y de 77% para LipL32. Así mismo, fueron obtenidos los porcentajes promedio de identidad de las proteínas OmpL1, LipL32, LipL41, LigA y LigB frente a las cepas patógenas de Leptospira spp., con lo cual se obtuvieron los siguientes porcentajes: 90, 99, 92, 63 y 60% respectivamente (E < 0,5).
DISCUSIÓN
Los genomas reportados indican que tanto las especies patógenas como las saprofitas de Leptospira spp. cuentan con dos cromosomas: uno grande y uno pequeño. Un estudio que comparó el genoma de L. interrogans, L. borgpetersenii y L. biflexa determinó que las tres especies comparten 2052 genes; L. biflexa comparte 93 y 44 genes con L. interrogans y L. borgpetersenii, respectivamente, quienes a su vez comparten 656 genes. Los genes específicos de L. biflexa, L. interrogans y L. borgpetersenii son 1348, 627 y 265, respectivamente. Los 2052 genes que comparten las tres especies tienen funciones de mantenimiento como metabolismo de ADN y ARN, procesamiento de proteínas y secreción, estructura celular, procesos celulares y metabolismo energético e intermediario (30).
Es evidente que a pesar de los numerosos estudios registrados en las bases de datos sobre los genes y las proteínas que se encuentran en cepas de Leptospira spp., aún no se conoce con exactitud la función que muchas de ellas desempeñan en la patogenicidad y en la interacción huésped-patógeno. De esta forma, se ha calculado que aproximadamente el 40% de los genes totales de este patógeno codifica para proteínas de función desconocida. Sin embargo, dentro de los genes con funcionalidad definida se incluyen los responsables de la biosíntesis de los lipopolisacáridos como LipL32, LipL41 y las proteínas de superficie Lig (31, 32). A pesar de conocerse otros genes conservados entre las especies patogenas de Leptospira, como lo son los codificantes para Loa22, HemO y LA1641, la función exacta dentro de la patogenicidad se desconoce y a pesar de tener resultados prometedores en su estudio para atenuación de cepas, falta investigación que clarifique de qué manera están involucrados en los procesos de infección y si son indispensables para el desarrollo de esta (31-33).
Al analizar las alineaciones de los genes ompL1, lipL32, lipL41, ligA y ligB, se evidencia que los porcentajes de identidad son similares; sin embargo, la cobertura de las secuencias frente a las cepas de Leptospira spp. variaron, en mayor medida la del gen ligA. Esta variación respecto a los genes lig se ha detectado en diferentes estudios (34,35), en los cuales se examinaron los patrones de variación de secuencia y de recombinación de estos genes frente a cepas patógenas de Leptospira, con lo cual se encontró que los genes ligB permanecían intactos, a diferencia del gen ligA encontrado exclusivamente en las cepas L. interrogans y L. kirschneri (35). El gen ligA deriva del gen ligB por un proceso parcial de duplicación de genes, por lo cual ambos se encuentran involucrados con los procesos de infección; de todas formas, no se requieren las dos copias, lo que explicaría la ausencia de ligA en algunas cepas de Leptospira y la variación detectada (35,36).
Por otra parte, al observar los porcentajes de alineación de las proteínas OmpL1, LipL32, LipL41, LigA y LigB, se identificó una variación en la identidad de estas últimas dos secuencias. Se ha encontrado que las proteínas Lig tienen un promedio de identidad bajo, como es el caso de LigA, la cual posee una identidad de secuencia de solo 37% (35); sin embargo, comparadas con otras proteínas que poseen secuencias completas más conservadas, la generación de anticuerpos en modelos animales como el hámster son muy similares (37). Con esto se indica que las proteínas Lig presentan una diversidad importante, por lo que se deben analizar cuidadosamente con el fin de seleccionar polipéptidos para uso en el diagnóstico o como candidatos vacunales (35).
Un análisis comparativo de los genomas de los serovares Copenhageni y Lai de L. interrogans concluye que existen diferencias en las vías de biosíntesis, adaptación, colonización y patogénesis entre serovares. Se identificó un gran número de lipoproteínas y proteínas de membrana externa específicas, que actuarían en la patogénesis (38); esto es acorde con estudios que indican que las vacunas realizadas con ciertas proteínas son serovar-específicas (39,40). Por otra parte, se determinó que existen proteínas de membrana externa que están conservadas entre los serovares patógenos. Estas proteínas son las recomendadas para el desarrollo de vacunas recombinantes (38).
Los análisis bioinformáticos realizados indican que los genes ompL1, lipL32, lipL41, ligA y ligB se encuentran exclusivamente en cepas patógenas de Leptospira spp., según lo registrado por diferentes autores (9,10,13,14,19,24,41). Los genomas de las cuatro especies patógenas secuenciadas indican que existe una variación en la localización de estos genes en el cromosoma I.
Para todos los genes se observa que el mayor número de reportes se ha realizado en diferentes cepas y serovares de L. interrogans, seguido por L. borgpetersenii, L. santarosai, L. kirschneri, L. noguchi y L.weilii. Se observó que las secuencias de los genes estudiados son poco variables entre las cepas registradas; esto concuerda con estudios que determinaron que entre los genes ompL1, lipL32 y lipL41 hay una equivalencia de 96-99% (19). En cuanto a los genes ligA y ligB, un estudio demostró que ligB está presente en todas las cepas patógenas de Leptospira spp. (35). Las proteínas LigA y LigB desempeñan un papel importante en la virulencia de la enfermedad debido a que actúan como adhesinas (26).
Un estudio evaluó la secuencia de los genes ompL1 y lipL32 y de sus respectivas proteínas en 38 cepas de Leptospira spp., para demostrar que existe mayor variabilidad en el ompL1 que en el gen lipL32. La variabilidad existente se debe a que los mecanismos de los genes de Leptospira spp. que codifican para proteínas de membrana externa incluyen mutaciones de nucleótidos que generan polimorfismos simples de nucleótidos y transferencia horizontal de ADN entre las diferentes especies de Leptospira spp. (42). Se considera que los mecanismos evolutivos de los genes ligA y ligB consisten en recombinación y transferencia horizontal de ADN (35).
Se sugiere que las secuencias seleccionadas son regiones invariables presentes en las proteínas de cepas patógenas de Leptospira spp. nominadas como candidatos vacunales, exceptuando LigA y LigB, proteínas con el menor porcentaje de identidad con las cepas patógenas utilizadas. Se recomienda realizar más investigaciones relacionadas con la identificación de una región invariable de estas proteínas, preferiblemente de la región C- terminal, que es la más conservada de esta proteína (35).
CONCLUSIONES
Mediante este estudio se confirmó, con ayuda de diferentes herramientas bioinformáticas, que existen regiones conservadas, tanto en los genes como en las proteínas entre las diferentes especies, serovares y cepas de Leptospira (excepto la secuencia identificada para LigB), que se proponen como secuencias de aminoácidos para la generación de vacunas recombinantes que confieran inmunoprotección a diferentes cepas de Leptospira spp. Se recomienda realizar estudios de secuenciación de estos genes en cepas de campo aisladas de animales enfermos, para verificar las secuencias consenso identificadas.
AGRADECIMIENTOS
Los autores expresan agradecimiento a la Universidad de La Salle por promover el fortalecimiento del trabajo en leptospirosis, con el fin de conocer una enfermedad que afecta a la salud pública y a la industria pecuaria en el mundo.
REFERENCIAS
1. Bharti AR, Nally JE, Ricaldi JN, Matthias MA, Díaz MM, Lovett MA, et al. Leptospirosis: a zoonotic disease of global importance. Lancet Infect Dis. 2003;3(12): 757-71. [ Links ]
2. Adler B, Moctezuma A. Leptospira and leptospirosis. Vet Microbiol. 2010;140(3-4): 287-96. [ Links ]
3. Hernández-Rodríguez P, Díaz C, Dalmau E, Quintero G. A comparison between Polymerase Chain Reaction (PCR) and traditional techniques for the diagnosis of leptospirosis in bovines. J Microbiol Methods. 2011;84(1):1-7. [ Links ]
4. Picardeau M. Diagnosis and epidemiology of leptospirosis. Med Mal Infect. 2013;43(1):1-9. [ Links ]
5. Rajapakse S, Rodrigo C, Handunnetti SM, Fernando SD. Current immunological and molecular tools for leptospirosis: diagnostics, vaccine design, and biomarkers for predicting severity. Ann Clin Microbiol Antimicrob. 2015;14(2):1-8. [ Links ]
6. Chen HW, Zhang Z, Halsey ES, Guevara C, Canal E, Hall E, et al. Detection of leptospira-specific antibodies using a recombinant antigen-based enzyme-linked immunosorbent assay. Am J Trop Med Hyg. 2013;89(6):1088-94. [ Links ]
7. Wang Z, Jin L, Wegrzyn A. Leptospirosis vaccines. Microb Cell Fact. 2007;6(39):1-10. [ Links ]
8. Dellagostin OA, Grassmann AA, Hartwig DD, Félix SR, Da Silva ÉF, McBride AJ. Recombinant vaccines against leptospirosis. Hum Vaccin. 2011;7(11):1215-24. [ Links ]
9. Dong H, Hu Y, Xue F, Sun D, Ojcius D, Mao Y, Yan J. Characterization of the ompL1 gene of pathogenic Leptospira species in China and crossimmunogenicity of the OmpL1 protein. BMC Microbiol. 2008;8:223-35. [ Links ]
10. Haake DA, Matsunaga J. Characterization of the leptospiral outer membrane and description of three novel leptospiral membrane proteins. Infect Immun. 2002;70(9):4936-45. [ Links ]
11. Li X, Zhao J, Qian J, Mao Y, Pan J, Li L, et al. Identification of immunodominant B- and T-Cell combined epitopes in outer membrane lipoproteins LipL32 and LipL21 of Leptospira interrogans. Clin Vaccine Immunol. 2010;17(5):778-83. [ Links ]
12. Haake DA, Chao G, Zuerner RL, Barnett JK, Barnett D, Mazel M, et al. The leptospiral major outer membrane protein LipL32 is a lipoprotein expressed during mammalian infection. Infect Immun. 2000;68(4):2276-85. [ Links ]
13. Guerreiro H, Croda J, Flannery B, Mazel M, Matsunaga J, Galvao Reis M. Leptospiral proteins recognized during the humoral immune response to leptospirosis in humans. Infect Immun. 2001;69(8):4958-68. [ Links ]
14. Zhang X, Yu Y, He P, Zhang YX, Hu BY, Yang Y, et al. Expression and comparative analysis of genes encondig outer membrane proteins LipL21, LipL32 and OmpL1 in epidemic leptospires. Acta Biochim Biophys Sin (Shanghai). 2005;37(10):649-56. [ Links ]
15. Flannery B, Costa D, Carvalho FP, Guerreiro H, Matsunaga J, Da Silva ED, et al. Evaluation of recombinant Leptospira antigen based enzyme-linked immunosorbent assays for the serodiagnosis of leptospirosis. J Clin Microbiol. 2001;39(9):3303-10. [ Links ]
16. Murray GL, Morel V, Cerqueira GM, Croda J, Sri-kram A, Henry R, et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infect Immun. 2009;77(2):810-6. [ Links ]
17. Pinne M, Haake DA. LipL32 Is a Subsurface Lipoprotein of Leptospira interrogans: presentation of new data and reevaluation of previous studies. PLoS One. 2013;8(1):e510-25. [ Links ]
18. Shang ES, Summers TA, Haake DA. Molecular cloning and sequence analysis of the gene encoding LipL41, a surface-exposed lipoprotein of pathogenic Leptospira species. Infect Immun. 1996;64(6):2322-30. [ Links ]
19. Vedhagiri K, Natarajaseenivasan K, Chellapandi P, Prabhakaran SG, Selvin J, Sharma S, Vijayachari P. Evolutionary implication of outer membrane lipoprotein-encoding genes ompL1, lipL32 and lipL41 of pathogenic Leptospira species. Genomics Proteomics Bioinformatics. 2009;7(3):96-106. [ Links ]
20. Hu Y, Guo ZQ, Sun BL, Yang P, Yan J. Study on the location of membrane and detection of antibody in the sera of genus-specific antigen LipL41s in patients with Leptospira interrogans. Zhonghua Liu Xing Bing Xue Za Zhi. 2007;28(8):776-80. [ Links ]
21. Mariya R, Chaudhary P, Kumar AA, Thangapandian E, Amutha R, Srivastava SK. Evaluation of a recombinant LipL41 antigen of Leptospira interrogans serovar Canicola in ELISA for serodiagnosis of bovine leptospirosis. Comp Immunol Microbiol Infect Dis. 2006;29(5-6):269-77. [ Links ]
22. Lin X, Sun A, Ruan P, Zhang Z, Yan J. Characterization of conserved combined T and B cell epitopes in Leptospira interrogans major outer membrane proteins OmpL1 and LipL41. BMC Microbiol. 2011;11(1):21. [ Links ]
23. Palaniappan RU, Chang YF, Jusuf SS, Artiushin S, Timoney JF, McDonough SP, Mohammed HO. Cloning and molecular characterization of an immunogenic LigA protein of Leptospira interrogans. Infect Immun. 2002;70(11):5924-30. [ Links ]
24. Palaniappan RU, Chang YF, Hassan F, McDonough SP, Pough M, Barr SC, et al. Expression of leptospiral immunoglobulin-like protein by Leptospira interrogans and evaluation of its diagnostic potential in a kinetic ELISA. J Med Microbiol. 2004;53(Pt 10):975-84. [ Links ]
25. Palaniappan RU, McDonough SP, Divers TJ, Chen CS, Pan MJ, Matsumoto M, Chang YF. Immunoprotection of recombinant leptospiral immunoglobulin-like protein A against Leptospira interrogans serovar pomona infection. Infect Immun. 2006;74(3):1745-50. [ Links ]
26. Choy HA, Kelley MM, Chen TL, Moller AK, Matsunaga J, Haake DA. Physiological osmotic induction of Leptospira interrogans adhesion: LigA and LigB bind extracellular matrix proteins and fibrinogen. Infect Immun. 2007;75(5):2441-50. [ Links ]
27. Yan W, Faisal SM, McDonough SP, Divers TJ, Barr SC, Chang CF, et al. Immunogenicity and protective efficacy of recombinant Leptospira immunoglobulin-like protein B (rLigB) in a hamster challenge model. Microbes Infect. 2009;11(2):230-7. [ Links ]
28. National Center for Biotechnology Information (NCBI). Nucleotide and protein tools. Tools [internet]. 2015 [citado 2015 feb 21]. Disponible en: http://www.nlm.nih.gov [ Links ]
29. Jones DT, Swindells MB. Getting the most from PSIBLAST. Thends Biochem Sci. 2001;27(3):161-4. [ Links ]
30. Picardeau M, Bulach DM, Bouchier C, Zuerner RL, Zidane N, Wilson PJ, et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLoS One. 2008;3(2):e1607. [ Links ]
31. Adler B, Lo M, Seemann T, Murray GL. Pathogenesis of leptospirosis: the influence of genomics. Vet Microbiol. 2011;153(1-2):73-81. [ Links ]
32. Zheng L, Ge Y, Hu W, Yan J. Expression changes of major outer membrane protein antigens in Leptospira interrogans during infection and its mechanism. Zhejiang Da Xue Xue Bao Yi Xue Ban. 2013;42(2):156-63. [ Links ]
33. Ristow P, Bourhy P, McBride FW, Figueira CP, Huerre M, Ave P, et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathogens. 2007;3(7):e97. [ Links ]
34. Matsunaga J, Barocchi MA, Croda J, Young TA, Sánchez Y, Siqueira I, et al. Pathogenic Leptospira species express surface-exposed proteins belonging to the bacterial immunoglobulin superfamily. Mol Microbiol. 2003;49(4):929-45. [ Links ]
35. McBride AJ, Cerqueira GM, Suchard MA, Moreira AN, Zuerner RL, Reis MG, et al. Genetic diversity of the leptospiral immunoglobulin-like (Lig) genes in pathogenic Leptospira spp. Infect Genet Evol. 2009;9(2):196-205. [ Links ]
36. Srimanote P, Wongdeethai N, Jieanampunkul P, Samonkiert S, Leepiyasakulchai C, Kalambaheti T, Prachayasittikul V. Recombinant LigA for leptospirosis diagnosis and ligA among the Leptospira spp. clinical isolates. J Microbiol Methods. 2008;72(1):73-81. [ Links ]
37. Coutinho ML, Choy HA, Kelley MM, Matsunaga J, Babbitt JT, Lewis MS, et al. A LigA three-domain region protects hamsters from lethal infection by Leptospira interrogans. PLoS Negl Trop Dis. 2011;5(12):1-10. [ Links ]
38. Nascimento AL, Ko AI, Martins EA, Monteiro-Vitorello CB, Ho PL, Haake DA, et al. Comparative genomics of two Leptospira interrogans serovars reveals novel insights into physiology and pathogenesis. J Bacteriol. 2004;186(7):2164-72. [ Links ]
39. Levett PN. Leptospirosis. Clin Microbiol Rev. 2001;14(2):296-326. [ Links ]
40. Roach JM, Van Vuuren M, Picard JA. A serological survey of antibodies to Leptospira species in dogs in South Arica. J S Ar Vet Assoc. 2010;81(3):156-9. [ Links ]
41. Hernández-Rodríguez P, Gómez A, Baquero M, Quintero G. Identification of ompl1 and lipl32 genes to diagnosis of pathogenic Leptospira spp. isolated from cattle. Open J Vet Med. 2014;4(5):102-12. [ Links ]
42. Haake D, Suchard M, Kelley M, Dundoo M, Alt D, Zuerner R. Molecular evolution and mosaicism of leptospiral outer membrane proteins involves horizontal DNA transfer. J Bacteriol. 2004;186(9):2818-28. [ Links ]