Introducción
El gato doméstico (Felis catus) es un mamífero pequeño con un peso alrededor de 5 kg; generalmente, los machos son más grandes que las hembras. Con una longitud de 50 cm sin incluir la cola, es un carnívoro de la familia de los félidos con garras retráctiles. Los gatos domésticos procederían de cinco líneas maternas diferentes de gatos monteses de Oriente Próximo (Felis silvestris lybica), que habrían sido domesticados hace más de 10.000 años, y de los gatos silvestres (Felis libyca) (1). Su área natural incluía el norte de África, China, India, sur de Europa, Gran Bretaña e islas del Mediterráneo. Gracias a sus características adaptativas, en la actualidad se encuentran ampliamente distribuidos, tanto en ambientes continentales como insulares, y ocupan una gran variedad de hábitats (2).
Los gatos presentan polimorfismos como el color, el patrón y la textura de la capa, particularidades probables de reconocer a simple vista; por lo cual, la recolección de datos es un procedimiento sencillo. Sumado a esto, los gatos son una especie de elección para este tipo de estudios poblacionales, por ser animales cosmopolitas y ser una población panmíctica (3). Los análisis genético-poblacionales en gatos son necesarios para revelar la historia de la evolución de estos y para la construcción de hipótesis filogenéticas sobre las relaciones entre los alelos. Sin embargo, a pesar de la importancia de este tipo de estudios, la información en el contexto mundial es escasa y en algunos lugares, inexistente.
Coveñas puede ser considerada una de las ciudades más importantes del departamento de Sucre, por su estratégica posición geográfica, por la abundancia de recursos naturales y por ser desde tiempos ancestrales, desde la Conquista y la Colonia españolas, uno de los principales puertos marítimos de Colombia en el mar Caribe. Esto pudo haber incidido en la diversidad genética de poblaciones de Felis catus. Sin embargo, no existe ningún tipo de información sobre la variabilidad de los marcadores fenotípicos en los genes del pelaje en las poblaciones de gato doméstico en esta ciudad. Por tal motivo, el objetivo de este estudio fue conocer la variabilidad genética de las poblaciones de gatos domésticos (Felis catus) mediante marcadores fenotípicos que codifican para el pelaje en Coveñas.
Materiales y métodos
Área de estudio
La recolección de datos se realizó en barrios de zonas urbanas del municipio de Coveñas, perteneciente al departamento de Sucre, Colombia, ubicados a 9° 24' 05" latitud norte y 75° 40' 48" de longitud oeste.
Recolección de datos
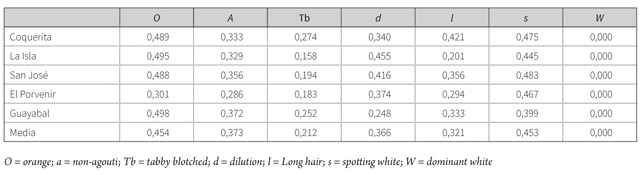
Se realizaron muestreos mediante excursiones urbanas y observación directa en cinco barrios de Coveñas: Coquerita, La Isla, San José, El Porvenir y Guayabal, donde se realizó una clasificación fenotípica de cada uno de los individuos adultos encontrados (n = 187). Cada ruta se utilizó solo una vez, a fin de evitar el remuestreo, y atendió a la presencia o ausencia de los marcadores autosómicos: a = non-agouti; Tb = tabby blotched; d = dilution; l = long hair; s = spotting white; W = dominant white y el locus ligado al sexo O (Orange) (tabla 1); por último, se tomaron registros fotográficos de cada individuo.
Variables de estudio
Para el estudio de la diversidad genética de las poblaciones de gatos domésticos (Felis catus) se tuvieron en cuenta marcadores fenotípicos (tabla 1) propuestos por el Committee on Standardized Genetic Nomenclature for Cats (4): O (Orange, carácter ligado al sexo) y los loci autosómicos, A (A, a; agouti vs. non-agouti), T (Ta, t+, tb; abyssinian tabby vs. mackerel o atigrado vs. blotched tabby), D (D, d; full color vs. dilution), L (L, l; pelo corto vs. long hair), S (s+, S; no manchado de blanco vs. manchado de blanco) y W (w+, W; color normal vs. dominante blanco).
Análisis poblacional
Se estimaron mediante el programa PopGene 1.31 (5) las frecuencias alélicas; la diversidad genética de Nei, correspondientes a la heterocigosidad esperada (He) y heterocigosidad de la población total (HT); el coeficiente de diferenciación genética (GST); el flujo génico (Nm); el equilibrio Hardy-Weinberg y la distancia genética de Nei (6) entre poblaciones.
Para determinar las relaciones genéticas entre las poblaciones analizadas se construyeron árboles filogenéticos que se obtuvieron mediante el algoritmo matemático UPGMA (Unweighted Pair Group Methodwith Arithemetic Mean), utilizando el programa MEGA 5.2 (7), a partir de la matriz de distancias genéticas.
Resultados
Frecuencias alélicas
Se muestrearon un total de 187 individuos. Se encontró que la frecuencia alélica más alta la obtuvo el alelo non-agouti, especialmente para las poblaciones La Isla (0,793) y Guayabal (0,774); mientras que para el marcador dominant white no se observó ningún individuo (tabla 2).
Diversidad genética
La población que presentó el valor más alto de heterocigosidad esperada en Coveñas fue Guayabal, para el marcador orange, seguido por la población La Isla para el mismo marcador, mientras que los marcadores dominante blanco y tabby blotched presentaron los valores más bajo de heterocigocidad (tabla 3).
El test de equilibrio Hardy-Weinberg (tabla 4) globalmente mostró ausencia de equilibrio para las poblaciones estudiadas.
Diferenciación genética y flujo génico
La diversidad genética total (HT) de la población de Coveñas es moderada, siendo el promedio igual a 0,316, con un intervalo de 0,192 en el locus tabby blotched a 0,498 en el locus spotting white. La mayor parte de la diversidad genética se encuentra dentro de las poblaciones (HS = 0,257) y poca entre las poblaciones (DST = 0,038). El coeficiente de diferenciación genética (GST = 0,057) indica que el 5,7 % de la diversidad genética se encuentra entre poblaciones y el 94,3 % dentro de las poblaciones. El valor de Nm mayor a 1 indica que existe alto flujo genético entre las subpoblaciones locales estudiadas de Felis catus en Coveñas (tabla 5).
Distancia genética
La distancia genética entre las poblaciones fue baja, siendo Coquerita y La Isla las poblaciones más cercanas; mientras que La Isla y Guayabal presentaron mayor distancia génica (tabla 6).
En el dendrograma UPGMA elaborado a partir de los valores de distancia genética (6) para las cinco poblaciones de Coveñas se puede apreciar claramente que las poblaciones más cercanas son Coquerita y La Isla, a la que se ha unido San José; la población más alejada fue Guayabal (figura 1).
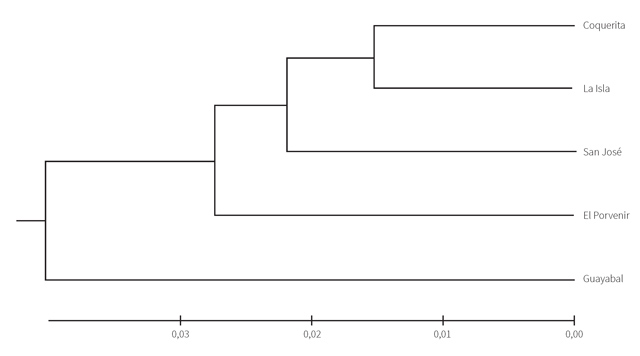
Figura 1 Dendrograma construido con el método UPGMA basado en la distancia genética de Nei (1972) de poblaciones de Felis catus de Coveñas.
El dendrograma UPGMA elaborado a partir de los valores de distancia genética de Nei (6) para poblaciones de Felis catus de Colombia revela la conformación de dos grupos: en un primer clúster se agrupan las poblaciones del interior (Leticia, Popayán, Pasto, Bogotá, Bucaramanga, Ibagué, Duitama y Cali), mientras las poblaciones costeñas (Montería, Coveñas, San Antero, Tolú, Lorica, Santa Marta y Riohacha) se unen en un segundo cluster (figura 2).
Discusión
La elevada frecuencia del alelo non-agouti en Coveñas podría estar relacionada con factores ambientales como temperaturas elevadas, que posiblemente estarían favoreciendo no solo la presencia, sino también el aumento de individuos portadores de dicho gen (2,8). A esto se une la posible existencia de sucesos selectivos, asociados a una progresiva melanización del pelaje, lo que genera alternativas alélicas que posibilitan que las coloraciones más melánicas se vean favorecidas en el medio urbano (9). Entre los sucesos selectivos, cabe destacar las altas densidades poblacionales y la injerencia humana, debido a que estos individuos son más sociables con otros congéneres, lo que aumenta la posibilidad de coexistir y adaptarse a entornos urbanos (10-12). Algunos estudios realizados en ratas (13,14), en visones (15) y en zorros (16,17) revelan una correlación entre genes de la coloración y ciertos rasgos del comportamiento, de lo cual resultan los individuos más melánicos, menos temerosos, menos agresivos y más resistentes al estrés que producen las zonas urbanas.
El locus spotting white (s) también presentó altas frecuencias, lo que podría estar relacionado con factores ambientales, ya que este gen se asocia a zonas de altas temperaturas y exposición solar; se postula como un posible claro ejemplo de selección natural in situ en nuevos hábitats colonizados, ya que en general estos municipios son muy calurosos debido a las altas temperaturas, que posiblemente estarían favoreciendo no solo la presencia, sino también el aumento de individuos portadores de dicho gen (2,8).
En relación con las frecuencias obtenidas para los alelos tabby blotched y dominant white, estos concuerdan con los resultados de Ruiz-García y Álvarez (18), quienes afirman que en Latinoamérica las frecuencias de tabby blotched resultan ser bajas o muy bajas. Por otro lado, la ausencia del alelo dominant white en Coveñas podría deberse, primero, a que las condiciones climáticas no son las adecuadas para que se desarrolle este gen, pues es de esperarse que lo haga en lugares donde la temperaturas son mucho menores a las presentadas en esta zona, donde las temperaturas oscilan entre 28-39 °C (19); y, segundo, puede atribuirse a la particular condición que caracteriza a este gen, ya que propicia efectos pleiotrópicos sobre la audición y la visión (20), hechos que ponen en desventaja y provocan la reducción sustancial de los individuos que portan este gen.
La presencia de la mayoría de los marcadores estudiados en la población de gatos domésticos de Coveñas demuestra la gran variedad de genes disponibles en la zona. Esta situación posiblemente se puede atribuir a la ubicación geográfica estratégica de dicha población, pues desde hace mucho tiempo es paso obligado de transeúntes, lo que favorece el movimiento de migrantes y, en consecuencia, facilita un considerable flujo génico.
La desviación del equilibrio de Hardy-Weinberg para los alelos O (orange) y S (spottingwhite) puede deberse a diversas causas; como los resultados arrojan un exceso de heterocigotos y déficit de homocigotos, podría atribuirse en este caso a algunos factores evolutivos como la selección artificial, ya que los humanos tienen predilección por cierto tipo de carácter en los gatos, lo que ha favorecido algunas característica fenotípicas más que a otras. Otro aspecto que podría afectar esta desviación es la cercanía geográfica entre las subpoblaciones estudiadas, lo que ocasiona un aumento sustancial del flujo génico que hay entre estas, dado que la existencia de un alto intercambio de genes previene eventos de endogamia en las poblaciones (21); esto provoca, por lo tanto, un aumento de genotipos heterocigotos en la población.
La diversidad genética total (HT) encontrada en Coveñas fue moderada. La mayor parte de la diversidad génica se encuentra dentro de las poblaciones (Hs) y poca entre las poblaciones (DST), lo cual indica que las poblaciones locales comparten una gran proporción de la diversidad total. El flujo de genes Nm resultó alto; en general, se considera que los valores de Nm cercanos o mayores de 4 son considerados suficientes para mantener una relativa homogeneidad del pool de genes (22). Estos resultados indican que en apariencia no hay diferenciación entre las poblaciones muestreadas. La falta de diferenciación genética entre poblaciones es considerada, por lo general, como el resultado de un suficiente flujo de genes, típicamente dado por migraciones, lo cual ocurre en todas las poblaciones para contrarrestar los efectos de la selección o la deriva genética, pues según Lowe (23), cuando Nm es mayor a 1 se espera que las poblaciones conserven conectividad genética, ya que el flujo genético sobrepasa los efectos de la deriva e impide la diferenciación local.
El elevado grado de flujo génico permite inferir que las poblaciones se encuentran muy relacionadas genéticamente y se comportan como una metapoblación, situación a la cual se atribuye la aproximación de todas las poblaciones desde el punto de vista estructural (2).
En el dendrograma UPGMA elaborado a partir de los valores de distancia genética de Nei (6) para las cinco poblaciones de Coveñas se aprecia que la cercanía genética obedece antes a la cercanía geográfica y a la antigüedad de los barrios, que es donde se supone que llegaron los primeros gatos al municipio (24).
En el dendrograma UPGMA elaborado a partir de los valores de distancia genética de Nei (6) para las ciudades de Colombia, se encontró que las ciudades del interior, como Leticia, Popayán, Pasto, Bogotá, Bucaramanga, Ibagué, Duitama y Cali, se reunieron en un cluster, y en otro cluster se agruparon: Coveñas, Tolú, Cartagena, San Antero, Montería, Santa Marta y Rio-hacha. Esta elevada homogeneidad genética hallada en estas ciudades puede estar relacionada con el vertiginoso desplazamiento de los conquistadores españoles por el río Magdalena, los cuales establecieron muchas ciudades y villas en poco tiempo (2,24). Además, este gran parecido genético entre las poblaciones puede corresponder a un evento fundador común, las cuales se originaron a partir de las poblaciones españolas (2), al ser una especie ausente en América hasta la inmigración de los europeos; su entrada debió estar asociada a las rutas colonizadoras de diferentes naciones europeas en el Nuevo Mundo (25). Sumado a esto, cabe resaltar la similitud en las condiciones ambientales del Caribe, como temperatura y humedad relativa, entre otras.
Conclusiones
Mediante este estudio se identificó la variabilidad genética de las poblaciones de gatos (Felis catus) en Coveñas, Sucre. Se encontró que el gen non-agouti fue el de mayor frecuencia, mientras los marcadores tabby blotched y dominant white exhibieron los valores menores. La mayor parte de la diversidad genética se encontró dentro de las poblaciones (HS) y poca entre las poblaciones (D); además se observó un exceso de heterocigotos a nivel poblacional y ausencia de equilibrio Hardy-Weinberg.