Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Boletín de Investigaciones Marinas y Costeras - INVEMAR
Print version ISSN 0122-9761
Bol. Invest. Mar. Cost. vol.45 no.1 Santa Marta Jan./June 2016
EVALUACIÓN DE AMPLIFICACIÓN CRUZADA DE MICROSATÉLITES PARA ESTUDIOS DE GENÉTICA POBLACIONAL DEL CAZÓN ANTILLANO RHIZOPRIONODON POROSUS (CARCHARHINIDAE) EN EL CARIBE COLOMBIANO
EVALUATION OF MICROSATELLITE CROSS-AMPLIFICATION FOR POPULATION GENETIC STUDY OF THE CARIBBEAN SHARPNOSE SHARK RHIZOPRIONODON POROSUS (CARCHARHINIDAE) IN THE COLOMBIAN CARIBBEAN
Mónica Almanza-Bernal1,3, Edna J. Márquez2 y Luis Chasqui3
1 Centro de Estudios en Ciencias del Mar- CECIMAR. Universidad Nacional de Colombia - Sede Caribe, Calle 25 No. 2-55, Playa Salguero, El Rodadero, Santa Marta, Colombia. monialmanza@hotmail.com
2 Universidad Nacional de Colombia - Sede Medellín, Facultad de Ciencias, Escuela de Biociencias, Calle 59ª No. 63-20, Bloque 19 A, Laboratorio 310, Medellín, 050034 Colombia. ejmarque@unal.edu.co
3 Instituto de Investigaciones Marinas y Costeras - (INVEMAR), Calle 25 No. 2-55, Playa Salguero, El Rodadero, Santa Marta, Colombia. luis.chasqui@invemar.org.co
RESUMEN
El tiburón cazón antillano, Rhizoprionodon porosus, constituye un recurso importante para pesquerías artesanales de pequeña escala; es uno de los tiburones costeros más abundantes dentro de su ámbito de distribución y desempeña una importante función como depredador en los ecosistemas marino-costeros. Por sus hábitos costeros, la especie es susceptible de sufrir extracción intensiva, especialmente en estados juveniles. Para darle un manejo apropiado y conservar las poblaciones explotadas de R. porosus se necesita conocer la diversidad genética y la estructura de las poblaciones dentro de sus rangos de distribución. En este estudio se probó la habilidad de cebadores heterólogos desarrollados para otras especies de tiburones de la familia Carcharhinidae para amplificar marcadores nucleares microsatélites en R. porosus (amplificación cruzada). La variación de la frecuencia alélica de genotipos de cuatro loci microsatélites sirvió para evaluar la estructura genética de R. porosus en siete localidades de cuatro departamentos del Caribe colombiano. El análisis de varianza molecular Amova y análisis de estructura poblacional mediante el estadístico Фst para las frecuencias genotípicas, indican que hay diferenciación genética baja pero significativa entre R. porosus provenientes de los departamentos analizados (Фst (3,165)= 0.002; p = 0.000). Además, el análisis pareado indica que existe una diferenciación genética significativa entre el cazón antillano de La Guajira y las demás muestras analizadas (todos los p = 0.000). La información obtenida ayuda a entender la dinámica de las poblaciones naturales del cazón antillano, sirve de línea base para la formulación y desarrollo de estrategias de conservación y manejo de este recurso pesquero; sin embargo, dado el bajo número de loci heterólogos útiles para estudios genético poblacionales, se deben enfocar los esfuerzos de investigación en el desarrollo de marcadores específicos para futuros estudios de genética poblacional de esta especie.
PALABRAS CLAVES: Estructura genética, Microsatélites, Amplificación cruzada, Rhizoprionodon, Carcharhinidae.
ABSTRACT
The Caribbean sharpnose shark, Rhizoprionodon porosus is an important resource for artisanal small-scale fisheries. It is one of the most abundant coastal sharks within its distribution range, and plays an important role as a predator in coastal marine ecosystems. For its coastal habits, it is susceptible to intensive extraction, especially the juveniles. To accomplish proper management and conservation of exploited Rhizoprionodon populations, knowledge about its genetic diversity and its population structure within their distribution range is needed. The ability of heterologous primers developed for other requiem sharks to amplify microsatellite molecular markers in R. porosus was tested in this study (cross amplification). The change in allele frequency of four microsatellite loci served to assess the genetic structure of R. porosus in the Colombian Caribbean. Analysis of molecular variance Amova and population structure analysis using the Фst statistical of genotype frequencies indicate low but significant genetic differentiation among R. porosus from the departments analyzed (Фst (3,165) = 0.002; p = 0.000). Besides, the analysis of pairs of departments indicates that there is significant genetic differentiation among La Guajira and the other samples analyzed of the Antillean sharpnose shark (all p values = 0.000). The information obtained helps to understand the dynamics of natural populations of the Caribbean sharpnose shark, serving as a baseline for the formulation, development of conservation strategies and management of this fishery resource; however, due the low number of heterologous loci useful for population genetics studies, research efforts on the development of specific markers for the species should be done for further population genetic studies of this species.
KEY WORDS: Genetic structure, Microsatellites, Cross amplification, Rhizoprionodon, Carcharhinidae.
INTRODUCCIÓN
Los tiburones del género Rhizoprionodon son abundantes a lo largo de su ámbito de distribución y desempeñan un papel importante como depredadores tope de la cadena trófica, a la vez que representan un recurso económico importante para pesquerías artesanales de pequeña y mediana escala en diferentes localidades alrededor del mundo (Simpfendorfer, 1993; Márquez-Farias y Castillo-Géniz, 1998; Mattos et al., 2001; Motta et al., 2005; Henderson et al., 2007; Andrade et al., 2008; Silva-Junior et al., 2008; Vishnoff, 2008; Almanza, 2009; Mendonça et al., 2011). Los requerimientos costeros de las especies del género las hacen susceptibles a la extracción intensiva, principalmente en estados juveniles, lo que genera un riesgo para sus poblaciones debido a la reducción de la variabilidad genética.
Proporcionar las bases para el desarrollo de estrategias para el manejo efectivo de las pesquerías costeras a través de la región Caribe es indispensable para la conservación de las especies, las poblaciones naturales y la variabilidad genética en el medio marino, además del mantenimiento de las funciones ecosistémicas, y en general para la conservación de la biodiversidad marina (Allendorf et al., 1987; Lessa et al., 2006; Mendonça et al., 2009). La dinámica y estructura poblacional de Rhizoprionodon porosus en el suroeste del Mar Caribe es desconocida, a pesar de ser una información necesaria para el establecimiento de estrategias de conservación adecuadas de especies explotadas o sobreexplotadas, y de poblaciones fragmentadas o en peligro de extinción (González, 2003).
La estructura poblacional de las dos especies del género Rhizoprionodon presentes en la región del Atlántico suramericano ha sido evaluada anteriormente por Mendonça et al. (2011; 2013) a partir de muestras provenientes del Caribe venezolano y varios puntos a lo largo de la costa de Brasil, basados en el análisis de la región control del ADNmt. Los estudios revelan que tanto R. porosus (Mendonça et al., 2011) como R. lalandii (Mendonça et al., 2013) presentan una fuerte estructura poblacional que separa cada especie en dos grandes unidades de manejo distintas, delimitadas por la corriente ecuatorial. La diferenciación genética existente entre las poblaciones brasileras y las del Caribe de R. porosus y R. lalandii posiblemente está modulada por la distancia geográfica, aunque barreras geográficas producidas por las corrientes marinas en Suramérica promueven la diferenciación en especies de peces costeros (Santos et al., 2003; Mendonça et al., 2013).
Los marcadores moleculares mitocondriales han contribuido y han sido fuertemente usados en análisis a nivel de población; sin embargo, con la disponibilidad de tecnología de secuenciación de alto rendimiento, los métodos de análisis de secuencia a escala fina, como los microsatélites, dan una nueva dimensión a los estudios de población, mediante el análisis de múltiples genes, ya que son menos sensibles a genealogías de genes específicos que podrían no reflejar correctamente la historia de la población (Hajibabaei et al., 2007). Los marcadores nucleares microsatélites tienen características como la herencia mendeliana, la codominancia (diferenciación de homocigotos y heterocigotos) y el ser altamente polimórficos, esto los convierte en los marcadores por excelencia en estudios de genética poblacional. Además, su genotipificación es relativamente fácil y automatizable, con resultados sencillos de analizar, fiables y repetitivos (González, 2003).
Aunque para R. porosus no se han desarrollado los cebadores específicos para marcadores microsatélites, en este estudio se realizó una aproximación al conocimiento de la genética poblacional del stock del cazón antillano (o Caribbean sharpnose shark) en la región del Caribe colombiano mediante la evaluación de la amplificación cruzada de microsatélites con cebadores que han sido desarrollados para otras especies de carcharhínidos, probando el uso de esta técnica por primera vez en la región para una especie de tiburón. Este estudio permitió ampliar el conocimiento sobre la dinámica de las poblaciones y la caracterización de la diversidad genética del tiburón cazón antillano R. porosus (Carcharhinidae) en el Caribe colombiano. Los objetivos planteados fueron: i) evaluar la habilidad de cebadores desarrollados para otras especies de carcharhínidos, en los análisis genético-poblacionales de R. porosus en la región Caribe; ii) determinar los patrones de diversidad genética de R. porosus a partir del análisis de marcadores moleculares microsatélites y iii) establecer si existen diferentes stocks genéticos de la especie en el Caribe colombiano.
ÁREA DE ESTUDIO
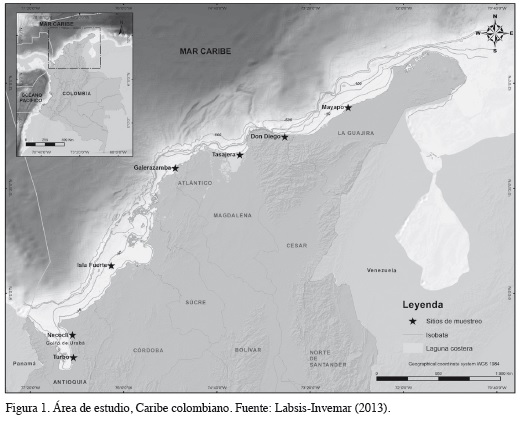
Morfológicamente el Caribe colombiano (Figura 1) presenta sectores contrastantes donde se tiene una plataforma continental amplia (hasta 50 km costa afuera en el golfo de Morrosquillo) y otros sectores donde su desarrollo es casi nulo (área adyacente a la Sierra Nevada de Santa Marta). La morfología submarina es diversa y está influenciada en los sectores central y suroccidental por la presencia del delta del río Magdalena y del cinturón o prisma acrecionario del Sinú, los cuales se caracterizan por exhibir geoformas como colinas, escarpes, cañones, sistemas de canales, leeves, depósitos de flujo de masa y formas dómicas producto de diapirismo de lodo (Rangel-Buitrago e Idárraga-García, 2010).
El sector nororiental, correspondiente al margen continental de La Guajira, presenta una plataforma amplia que aumenta su tamaño hacia el oriente y cuyo rasgo más significativo es el Cañón de Ranchería. Desde el punto de vista sedimentológico, en la plataforma continental de La Guajira y del Sinú se tiene sedimentación areno-lodosa predominantemente calcárea asociada a la presencia de formaciones arrecifales y de algas; en los sectores Tayrona y delta del Magdalena predominan los sedimentos terrígenos areno-lodosos (Rangel-Buitrago e Idárraga-García, 2010).
En el Caribe colombiano dominan la corriente superficial del Caribe, que fluye este-noroeste a través del noreste, y la circulación ciclónica del giro Panamá-Colombia (GPC) al suroeste del mar Caribe. El extremo sur del giro, que colinda la costa, es conocido como la contracorriente Panamá-Colombia (CPC), la cual forma un flujo superficial hacia el este, atrapado contra la costa en todas las secciones de las costas colombiana y panameña (Andrade et al., 2003). Dependiendo de la época del año, la CPC varía su extensión a lo largo de la costa colombiana. Durante la época seca esta corriente se extiende desde el golfo del Darién hasta cercanías de la desembocadura del río Magdalena. En la época de lluvias, cuando se disminuye el efecto de los vientos alisios, esta contracorriente se extiende hasta la península de La Guajira (Lozano-Duque et al., 2010).
En La Guajira persiste una sub-corriente hacia el este. La corriente hacia el este también se evidencia fuera de la costa de Venezuela, donde alcanza la superficie. Existe una pequeña circulación ciclónica superficial fuera de la costa central llamada el giro venezolano, la cual es probablemente contigua con la contracorriente general. El flujo hacia el este parece ser una característica permanente de la circulación en la región del sur del mar Caribe, uniendo la CPC y la contracorriente de Venezuela por debajo de las aguas superficiales dirigidas al oeste, producidas por la surgencia de La Guajira (Andrade et al., 2003).
MATERIALES Y MÉTODOS
Se utilizaron 83 individuos de R. porosus provenientes de varias localidades a lo largo de la costa Caribe de Colombia: Mayapo en La Guajira, Don Diego y Tasajera en Magdalena, Galerazamba en Bolívar, Isla Fuerte frente a la costa de Córdoba y cerca de Necoclí en el golfo de Urabá. Las muestras de tejido hacen parte de la colección de referencia del Museo de Historia Natural Marina de Colombia (MHNMC) y se encuentran preservadas en etanol al 96% para garantizar la integridad del ADN.
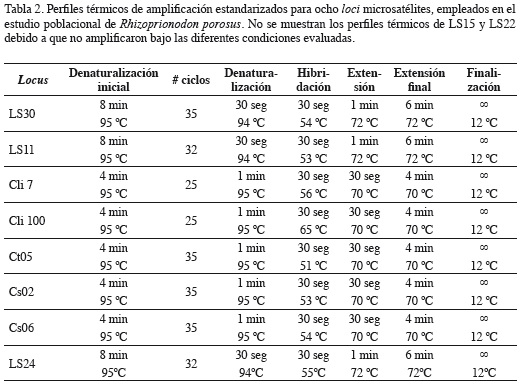
El ADN se extrajo empleando el kit DNeasy® Blood and Tissue de QIAGEN, siguiendo las instrucciones del fabricante. Para algunas muestras el ADN se extrajo modificando el protocolo de extracción con Chelex® 5% descrito por Ward et al. (2008). La verificación de la cantidad y calidad del ADN extraído se realizó mediante electroforesis en geles de agarosa 1%, a partir de la intensidad de la fluorescencia emitida por el Gel Red® irradiado con luz UV, comparada con la intensidad del marcador de peso molecular Hyperladder IVTM (Bioline). Se seleccionaron diez cebadores de marcadores microsatélites desarrollados para otras especies de carcharhínidos, teniendo en cuenta el nivel de polimorfismo, la simplicidad de la secuencia de repetición y el número de alelos de cada locus. De los diez cebadores, cinco fueron aislados por Feldheim et al. (2001) a partir de una librería genómica de Negaprion brevirostris (Ls11, Ls15, Ls22, Ls24 y Ls30), dos por Keeney y Heist (2003) para Carcharhinus limbatus (Cli7 y Cli100); Ovenden et al. (2006) aislaron un microsatélite para C. sorrah y dos para C. tilstoni (Tabla 1).
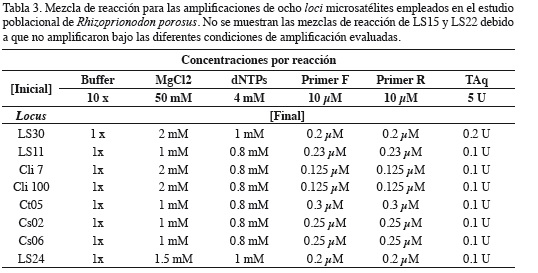
Para la estandarización de las reacciones de amplificación se partió de los protocolos descritos por los autores. La mezcla de reacción contenía amortiguador PCR en una concentración 1x, 1 a 2 mM de MgCl2, 800 a 1000 µM de mezcla de dNTPs (Bioline), 0.1 a 0.3 µM del cebador correspondiente, 0.1 unidades por reacción de BiolaseTM DNA Polymerase, 2 µL de ADN molde y agua grado molecular hasta un volumen final de 15 µL. Las reacciones de amplificación se realizaron en un termociclador de gradiente BioRad T100TM, bajo las siguientes condiciones: desnaturalización cuatro a ocho minutos a 95 ºC, seguida de 25 a 35 ciclos de desnaturalización de un minuto a 94 ºC, hibridación y extensión final de cuatro a seis minutos a 72 ºC. La hibridación tuvo un ajuste de la temperatura partiendo de la temperatura descrita por los autores para N. brevirostris, C. limbatus, C. sorrah, y C. tilstoni, utilizando un gradiente entre dos grados centígrados por debajo y dos grados por encima de lo registrado en la literatura; y una extensión de 30 segundos a 70 ºC. La temperatura de finalización fue 12 ºC por tiempo indefinido. Todas las estandarizaciones incluyeron controles negativos para monitorear posibles problemas de contaminación. Las comprobaciones de amplificación se realizaron mediante electroforesis en geles de agarosa 3%, con un marcador de peso molecular HyperLadderTM V (Bioline). Una vez estandarizadas las condiciones de amplificación para los microsatélites (Tablas 2 y 3), se procedió a amplificarlos con cebadores fluoromarcados (6-FAM, VIC, NED y PET), para su posterior detección en un secuenciador automático mediante el procedimiento de análisis de fragmentos. La genotipificación automática se llevó a cabo mediante el proceso de electroforesis capilar en un analizador genético Applied Biosystems 3500, utilizando mezclas post-amplificación de los productos de PCR obtenidos a partir de los cebadores fluoromarcados.
Con el programa GeneMapper® V4.1 (Applied Biosystems) se realizó la asignación automática de los genotipos, analizando los electroferogramas, que registran la intensidad de la señal fluorescente para cada alelo a partir de información suministrada sobre el ámbito de peso obtenido de la amplificación y la medida de repetición. El tamaño de los alelos se estimó con respecto a un marcador de peso molecular GeneScan LIZ-600® que se agregó a cada muestra durante la electroforesis. Los genotipos se analizaron en el programa MicroChecker (Oosterhout et al., 2004) para detectar errores de genotipificación (e.g. alelos nulos). La diversidad genética fue estimada por locus y por regiones, calculando el número promedio de alelos por locus, número de loci polimórficos, heterocigosidad media observada, heterocigosidad media esperada y el índice de fijación utilizando el programa GenAlEx V6.41 (Peakall y Smouse, 2006). Las desviaciones del equilibrio de Hardy-Weinberg y el desequilibrio de ligamiento fueron estimados con el test de Probabilidad de Fisher descrito por Raymond y Rousset (1995) usando GenepopOn-the-web V4.2 (Rousset, 2008).
Dado que las hipótesis de equilibrio de Hardy-Weinberg y desequilibrio de ligamiento no fueron satisfechas, la diferenciación genética entre las muestras de acuerdo con su origen geográfico (entre y dentro de poblaciones) fue evaluada usando el estadístico Фst y el análisis molecular de varianza AMOVA (Excoffier et al., 1992). Las comparaciones pareadas entre departamentos se hicieron mediante el Test Exacto de Diferenciación basado en frecuencias genotípicas (Raymond y Rousett, 1995), utilizando el programa Arlequin V3 (Excoffier et al., 2005). Las comparaciones múltiples fueron ajustadas con el Test de Bonferroni (Dunn-Sidak) al 95% para estimar niveles críticos de significancia (Sokal y Rohlf, 1995).
RESULTADOS
Evaluación de los cebadores
De los diez marcadores microsatélites diseñados para diferentes especies de carcharhínidos que se usaron para realizar amplificación cruzada en R. porosus, fue posible estandarizar las condiciones de amplificación de ocho loci (Cli7, Cli100, Cs02, Cs06, Ct05, Ls11, Ls24 y Ls30) (Tablas 2 y 3). Los cebadores de los microsatélites Ls15 y Ls22 no favorecieron la elongación del ADN, a pesar de someterlos a pruebas con varias concentraciones de MgCl2 y diferentes temperaturas de hibridación.
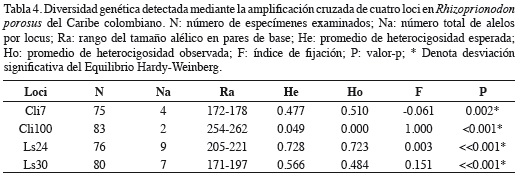
De los ocho loci estandarizados solo fue posible amplificar cinco de manera masiva y consistente con los cebadores fluoromarcados (Cli7, Cli100, Cs06, Ls24 y Ls30), resultando tres polimórficos (Cli7, Ls24 y Ls30). El cebador Cs06 amplificó una región monomórfica (i.e. alelo único) para las muestras de todo el Caribe colombiano, y el cebador Cli100 fue monomórfico en muestras provenientes de dos departamentos (Magdalena y Córdoba).
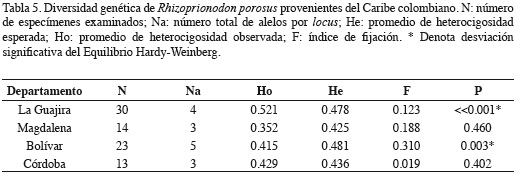
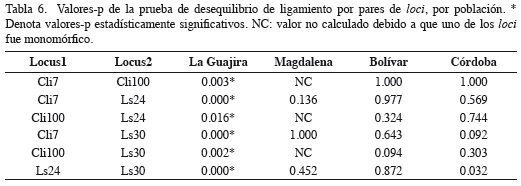
Se analizó un promedio de 80 muestras por locus. El ámbito alélico estuvo entre 171 -262 pares de bases, siendo Ls24 el locus con el mayor nivel de polimorfismo (Na = 9) (Tabla 4). En la muestra total, el número promedio de alelos por locus estuvo entre dos y siete, la heterocigosidad observada entre 0.000 y 0.723, y todos los loci se desviaron del Equilibrio de Hardy-Weinberg (Tabla 4). En los departamentos, el número promedio de alelos por locus por región estuvo entre tres y cinco, la heterocigosidad observada entre 0.352 (Magdalena) y 0.521 (La Guajira), con desviaciones significativas del Equilibrio Hardy-Weinberg en La Guajira y Bolívar (Tabla 5). Además, se detectó desequilibrio de ligamiento entre todos los pares de loci en La Guajira (Tabla 6).
Estructura genética
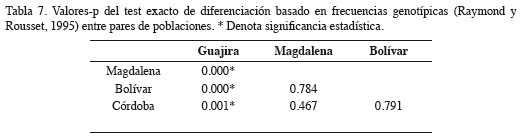
Mediante el estadístico Фst calculado para cuatro loci microsatélites se rechazó la hipótesis de homogeneidad genética de R. porosus. Entre muestras de los departamentos evaluados, se evidenció una baja pero significativa diferenciación genética en el Caribe colombiano (Фst (3,165)= 0.002; p = 0.000). Los valores del AMOVA mostraron que la mayor fuente de variación se da dentro de los departamentos, con 98% de variación, en contraste con 2% de diferencia entre las poblaciones. Las comparaciones del test exacto de diferenciación (Raymond y Rousset, 1995) por pares de departamentos, demuestran que las frecuencias alélicas en La Guajira son significativamente diferentes que el resto de las localidades (Tabla 7).
DISCUSIÓN Y CONCLUSIONES
Al evaluar la habilidad de diez cebadores heterólogos desarrollados para amplificar loci microsatélites en varias especies de carcharhínidos con muestras de R. porosus, cinco (Cli7, Ls24, Ls30, Cli100, Cs06) generaron amplificaciones masivas exitosas y solo tres (Cli7, Ls24, Ls30) pudieron aportar información a nivel de población para esta especie, dado su nivel de polimorfismo y su habilidad para mostrar individuos heterocigotos en frecuencias concordantes con el equilibrio de Hardy-Weinberg en varias de las poblaciones examinadas. El locus Cli100 fue monomórfico en R. porosus de dos departamentos y mostró un nivel bajo de heterocigosidad, pero podría tener utilidad potencial para discriminar poblaciones.
El nivel de polimorfismo del locus Ls24 en R. porosus contrasta con el monomorfismo observado en el tiburón limón Negaprion brevirostris, especie en la que fue originalmente identificado (Feldheim et al., 2001). De manera similar, la ausencia de polimorfismo de los loci Cli100 y Cs06 en R. porosus contrasta con el nivel de polimorfismo observado en especies de la misma familia, como C. limbatus o C. tilstoni (Keeney y Heist, 2003; Ovenden et al., 2006). Los resultados anteriores indican que cada locus exhibe tasas de cambio diferenciales dependiendo de la especie estudiada y limita el uso de la amplificación cruzada con cebadores heterólogos, sugiriendo que los esfuerzos se deben enfocar en el desarrollo de novo de marcadores especie-específicos para las especies de tiburones y otros recursos explotados en la región Caribe.
El valor del estadístico (Фst) empleado para el cálculo de la diferenciación genética con frecuencias alélicas de microsatélites de las poblaciones de R. porosus es significativo, y concuerda con los resultados encontrados con las regiones mitocondriales Citocromo oxidasa I (COI) y Región Control (RC) (Almanza, 2014) y con los resultados encontrados por Mendonça et al. (2011), quienes encontraron estructuración genética significativa en el tiburón cazón antillano debida a las posibles barreras al flujo genético que representan la Sierra Nevada de Santa Marta en el Caribe colombiano y la corriente ecuatorial en el Atlántico suroccidental (Mendonça et al., 2011; Almanza, 2014). Sin embargo, los resultados de este estudio están limitados por el bajo número de loci (cuatro) empleados para el análisis, por lo que se hace necesario desarrollar cebadores específicos para la especie, así como aumentar el número de microsatélites y el número de muestras con el fin de mejorar el grado de resolución que proveen los estadísticos.
Las muestras de La Guajira en comparación con las de los demás sitios analizados mostraron un significativo déficit de heterocigotos, desequilibrio de ligamiento en los tres loci microsatélites examinados y frecuencias alélicas significativamente diferentes del resto de las localidades, lo que sugiere que las muestras de esa localidad corresponden a una población de individuos que podría estar conformada por organismos emparentados. Las muestras de las demás localidades mostraron que los loci analizados satisfacen los supuestos del equilibrio de Hardy-Weinberg y/o ligamiento, sugiriendo que pueden representar una sola población pancmíctica. Sin embargo, estas hipótesis deben ser contrastadas en estudios posteriores con un número mayor de loci.
Una posible explicación a estos resultados es la ocupación de áreas de crianza, ya que especies costeras tienen complejas relaciones con sus hábitats cerca de la costa, lo cual puede limitar la dispersión (Heupel et al., 2007) y puede resultar en poblaciones subdivididas. Las zonas que utilizan las especies de Rhizoprionodon para reproducción, nacimientos y maduración son principalmente someras, cerca de las costas y son hábitats altamente productivos que probablemente proveen alimento y protección para tiburones juveniles. El grado de segregación de esos sitios, y principalmente la fidelidad de sitio (filopatría) pueden afectar directamente el nivel de subdivisión de la población y la divergencia genética ente regiones geográficas (Simpfendorfer y Milward, 1993; Hueter et al., 2005).
Además, considerando aspectos históricos, hay evidencias que indican relaciones entre los registros de distribución de tiburones Rhizoprionodon y las corrientes cálidas (Gallo et al., 2010). En todas las secciones de la costa Caribe de Colombia, existe un flujo superficial hacia el este, formado desde la contracorriente Panamá-Colombia (CPC), que se debilita en su recorrido, y en la región de La Guajira forma una subcorriente hacia el este. La CPC se une superficialmente con la contracorriente de Venezuela, por debajo de las aguas superficiales dirigidas al oeste por la surgencia de La Guajira (Andrade et al., 2003). Es posible que en el Caribe colombiano la distribución de R. porosus responda a este flujo de la corriente en dirección este, siendo interrumpida en La Guajira por la surgencia donde el flujo de agua se presenta en forma de subcorriente; esto explicaría el posible parentesco de los individuos en La Guajira, revelado por el déficit de heterocigotos, desequilibrio de ligamiento en los tres loci microsatélites examinados y frecuencias alélicas significativamente diferentes del resto de las localidades. Desde este punto de vista, y por la relación de las especies del género Rhizoprionodon con las corrientes cálidas (Gallo et al., 2010), el déficit de heterocigotos, el desequilibrio de ligamiento y las frecuencias alélicas divergentes en La Guajira con respecto al resto de las localidades, se podría sugerir que la surgencia de La Guajira, con su característica baja temperatura, representa en cierta medida un factor de aislamiento entre esta población y sus áreas de crianza, con las demás localidades analizadas de R. porosus. Adicionalmente, la interrupción de la plataforma costera en ambos costados de la Sierra Nevada de Santa Marta, podría representar una barrera al flujo para especies que habitan sobre la plataforma costera (Betancur et al., 2010), como las del género Rhizoprionodon.
La dependencia de hábitats costeros de R. porosus somete a la especie a una variedad de actividades antropogénicas deletéreas que, junto con la alta presión pesquera que tradicionalmente ocurre en áreas cercanas a la costa, hace que la especie sea vulnerable a la extirpación local y aumenta el potencial riesgo de extinción (Martin, 2005). Reconocer el patrón y grado de subdivisión de las poblaciones silvestres es importante para los reguladores del recurso, puesto que fallar en la detección de la estructura de la población puede resultar en sobreexplotación o extirpación localizada (Hueter et al., 2005). Actualmente existe una explotación extensiva de tiburones costeros por pesquerías artesanales y persiste una gran deficiencia en la documentación de esas capturas, por lo que mantener estas poblaciones en el medio marino requiere de medidas de conservación que salvaguarden el potencial adaptativo y evolutivo de las especies. Reconocer las unidades de manejo es fundamental para darle un manejo a corto plazo de unidades evolutivas significativas, para monitoreo de las poblaciones y estudios demográficos (Mendonça et al., 2013).
AGRADECIMIENTOS
Este estudio se realizó en el marco del Proyecto de Fortalecimiento de Laboratorios de Investigación Marina auspiciado por Colciencias (Contrato RC 5302012) en el Instituto de Investigaciones Marinas y Costeras Invemar, que brindó soporte financiero y logístico a través del Convenio de Cooperación Científica y Académica No. 018-12. Agradecemos a los investigadores del Invemar por el apoyo a lo largo del desarrollo de esta investigación. Igualmente a la sede Caribe de la Universidad Nacional de Colombia por el apoyo a la tesis de posgrado mediantela convocatoria de 2012, a la Fundación Alejandro Ángel Escobar por el apoyo mediante el fondo de becas Colombia Biodiversa, así como a los investigadores, monitores y pescadores en campo que colaboraron con la colecta de las muestras de tejido. Esta publicación forma parte del trabajo de grado de MA para optar por el título de Maestría en Ciencias, bajo la dirección de EMF. Contribución 406 del Programa de Posgrado en Ciencias-Biología, Línea Biología Marina, Centro de Estudios en Ciencias del Mar -CECIMAR, Universidad Nacional de Colombia, Sede Caribe.
BIBLIOGRAFÍA
Allendorf, F., N. Ryman y F. Utter. 1987. Genetics and fishery management: Past, present and future. 1-20. In: Ryman, N. y F.M. Utter (Eds.). Population genetics and fishery management. University of Washington, Washington. 440 p. [ Links ]
Almanza, M.A. 2009. Caracterización de la pesca artesanal de peces cartilaginosos, con énfasis en sus aspectos tróficos y reproductivos en Isla Fuerte, Caribe colombiano. Trabajo de grado Biol. Mar. Univ. Bogotá Jorge Tadeo Lozano, Santa Marta. 87 p. [ Links ]
Almanza, M.A. 2014. Análisis genético poblacional del tiburón cazón antillano, Rhizoprionodon porosus (Carcharhinidae), en el Caribe colombiano. Tesis Maestria. Univ. Nacional de Colombia-Sede Caribe. 78 p. [ Links ]
Andrade, A.C., L.C. Silva-Junior y M. Vianna. 2008. Reproductive biology and population variables of the Brazilian sharpnose shark Rhizoprionodon lalandii (Muller & Henle, 1839) captured in coastal waters of south-eastern Brazil. J. Fish Biol., 72: 473-484. [ Links ]
Andrade, C.A., E. Desmon y C.N.K. Mooers. 2003. Evidence for an eastward flow along the Central and South American Caribbean Coast. J. Geophys. Res., 108(C6): 3185. [ Links ]
Betancur, R., A. Acero P., H. Duque-Caro y S. Santos. 2010. Phylogenetic and morphologic analysis of a coastal fish reveals a marine biogeographic break of terrestrial origin in the southern Caribbean. Plos One, 5(7): e11566. [ Links ]
Excoffier, L., P.E. Smouse y J.M. Quattro. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data. Genetics, 131: 479-491. [ Links ]
Excoffier, L., G. Laval y S. Schneider. 2005. Arlequin V3.0: an integrated software packaged for population genetics data analysis. Journal of Evolutionary Bioinformatics Online, 1: 47-50. [ Links ]
Feldheim, K.A., S.H. Gruber y V. Ashley. 2001. Population genetic structure of the lemon shark (Negaprion brevirostris) in the western Atlantic: DNA microsatellite variation. Mol. Ecol., 10: 295-303. [ Links ]
Gallo, V., M.J. Cavalcanti, R.F.L Da Silva, H.M.A. Da Silva y D. Pagnoncelli. 2010. Panbiogeographical analysis of the shark genus Rhizoprionodon (Chondrichthyes, Carcharhiniformes, Carcharhinidae). J. Fish Biol., 76: 1696-1713. [ Links ]
González, G.E. 2003. Microsatélites: sus aplicaciones en la conservación de la biodiversidad. Graellsia, 59(2-3): 377-388. [ Links ]
Hajibabaei, M., G. Singer, P. Hebert y D. Hickey. 2007. DNA Barcoding: how it complements taxonomy, molecular phylogenetic and population genetics. Trends in Genetics, 23(4): 167-172. [ Links ]
Henderson, A., J.L. McIlwain, H.S. Al-Oufi y S. Al-Sheili. 2007. The Sultanate of Oman shark fishery: Species composition, seasonality and diversity. Fish. Res., 86: 159-168. [ Links ]
Heupel, M.R., J.K. Carlson y C.A. Simpfendorfer. 2007. Shark nursery areas: concepts, definition, characterization and assumptions. Mar. Ecol. Progr. Ser., 337: 287-297. [ Links ]
Hueter, R.E., M.R. Heupel, E.J. Heist y D.B. Keeney. 2005. Evidence of philopatry in sharks and implications for the management of shark fisheries. J. Northwest Atl. Fish. Sci., 35: 239-247. [ Links ]
Keeney, D.B. y E.J. Heist. 2003. Characterization of microsatellite loci isolated from the blacktip shark and their utility in requiem and hammerhead sharks. Molecular Ecology Notes, 3: 501-504. [ Links ]
Lessa, R., S.M. Quijano, F.M. Santana y J. Monzini. 2006. Rhizoprionodon porosus. En: IUCN (Ed.). IUCN Red list of threatened species. Version 2013.1. http://www.iucnredlist.org. 07/08/2013. [ Links ]
Lozano-Duque, Y., J. Medellín-Mora y G.R. Navas. 2010. Contexto climatológico y oceanográfico del mar Caribe colombiano. 53-84. En: Invemar (Eds.). Biodiversidad del margen continental del Caribe colombiano. Serie de Publicaciones Especiales del Invemar 20, Santa Marta. 458 p. [ Links ]
Márquez-Farias, J.F. y J.L. Castillo-Géniz. 1998. Fishery biology and demography of the Atlantic sharpnose shark, Rhizoprionodon terraenovae, in the southern Gulf of Mexico. Fish. Res., 39: 183-198. [ Links ]
Martin, R.A. 2005. Conservation of freshwater and eurihaline elasmobranchs: a review. J. Mar. Biol. Ass. UK, 85: 1049-1073. [ Links ]
Mattos, S.M.G., M.K. Broadhurst, F.H.V. Hazin y D.M. Jonnes. 2001. Reproductive biology of the Caribbean sharpnose shark, Rhizoprionodon porosus, from the Northern Brazil. Mar. Freshw. Res., 52(5): 745-752. [ Links ]
Mendonça, F.F., C. Oliviera, O.F.B. Gadig y F. Foresti. 2009. Populations analysis of the Brazilian sharpnose shark Rhizoprionodon lalandii (Chondrichthyes: Carcharhinidae) on the Sao Paulo coast, Southern Brazil: Inferences from mtDNA sequences. Neotrop. Ichth., 7(2): 213-216. [ Links ]
Mendonça, F.F., C. Oliviera, O.B.F. Gadig y F. Foresti. 2011. Phylogeography and genetic population structure of Caribbean sharpnose shark Rhizoprionodon porosus. Rev. Fish Biol. Fisheries, 21: 799-814. [ Links ]
Mendonça, F.F., C. Oliviera, O.B.F. Gadig y F. Foresti. 2013. Diversity and genetic population structure of the Brazilian sharpnose shark Rhizoprionodon lalandii. Aq. Cons.: Mar. Freshwater Ecosys., 23(6): 850-857. DOI: 10.1002/aqc.2342. [ Links ]
Motta, F.S., O.B.F. Gadig, R.C. Namora y F.M.S. Braga. 2005. Size and sex compositions, length-weight relationship, and occurrence of the Brazilian sharpnose shark, Rhizoprionodon lalandii, caught by artisanal fishery from southeastern Brazil. Fish. Res., 74: 116-126. [ Links ]
Oosterhout, C. Van, W.F. Hutchinson, D. Wills y P. Shipley. 2004. Micro-Checker: Software for identifying and correcting genotyping errors in microsatellite data. Mol. Ecol. Notes, 4(3): 535-538. [ Links ]
Ovenden, J.R., R. Street y D. Broderick. 2006. New microsatellite loci for carcharhinid sharks (Carcharhinus tilstoni and C. sorrah) and their cross-amplification in other shark species. Mol. Ecol. Notes, 6: 415-418. [ Links ]
Peakall, R. y P.E. Smouse. 2006. Genalex 6: genetic analysis in Excel. Population genetic software for teaching and research. Mol. Ecol. Notes, 6: 288-295. [ Links ]
Rangel-Buitrago, N. y J. Idárraga-García. 2010. Geología general, morfología submarina y facies sedimentarias en el margen continental y los fondos oceánicos del mar Caribe colombiano. 30-51. En: Invemar (Ed.). Biodiversidad del margen continental del Caribe colombiano. Serie de Publicaciones Especiales del Invemar No. 20, Santa Marta. 458 p. [ Links ]
Raymond, M. y F. Rousset. 1995. Genepop V1.2: population genetics software for exact tests and ecumenicism. J. Heredity, 86: 248-249. [ Links ] Rousset, F. 2008. Genepop'007: a complete reimplementation of the Genepop software for Windows and Linux. Mol. Ecol. Res., 8: 103-106. [ Links ]
Santos, S., H. Schneider e I. Sampaio. 2003. Genetic differentiation of Macrodon ancylodon (Sciaenidae, Perciformes) populations in Atlantic coastal waters of South America as revealed by mtDNA analysis. Genet. Mol. Biol., 26: 151-161. [ Links ]
Silva-Junior, L.C., A.C. Andrade y M. Vianna. 2008. Caracterização de uma pescaria de pequena escala em un área de importância ecológica para elasmobrânquios, no Recreio Dos Bandeirantes, Rio de Janeiro. Arq. Ciên. Mar, Fortaleza, 41(2): 47-57. [ Links ]
Simpfendorfer, C.A. 1993. Age and growth of the Australian sharpnose shark, Rhizoprionodon taylori, from Queensland, Australia. Environ. Biol. Fishes, 36: 233-241. [ Links ]
Simpfendorfer, C.A. y N.E. Millward. 1993. Utilization of a tropical bay as a nursery by sharks of the families Carcharhinidae and Sphyrnidae. Environ. Biol. Fishes, 37: 337-345. [ Links ]
Sokal, R.R. y F.J. Rohlf. 1995. Biometry: The principles and practice of statistics in biological research. W.H. Freeman and Co., Nueva York. 778 p. [ Links ]
Vishnoff, I.M.J. 2008. Conocimiento de la biología reproductiva de algunos carcharhínidos a través de las actividades de pesca artesanal en Isla Fuerte, Caribe colombiano (2006-2007). Trabajo de grado, Biol. Mar. Univ. Bogotá Jorge Tadeo Lozano, Santa Marta. 87 p. [ Links ]
Ward, R.D., B. Holmes, W. White, P.R. Last. 2008. DNA Barcoding Australasian chondrichthyans: results and potentials uses in conservation. Marine and Freshwater Research, 59: 57-71. [ Links ]
RECIBIDO: 16/12/2014 ACEPTADO: 21/12/2015