INTRODUCCIÓN
Las bacterias existen en el medio acuático bajo dos formas o estados: a) pelágicas, de libre flotación, y b) bacterias en biopelículas, formando colonias sésiles (Nazar, 2007). Las biopelículas son comunidades de microorganismos que crecen incluidos en una matriz de exopolisacáridos y adheridos a una superficie inerte o a un tejido vivo. En principio la formación de una biopelícula se establece como parte de los procesos que se pueden presentar en el mecanismo quorum sensing (QS) (Díaz et al., 2011). Las bacterias mantienen una comunicación permanente entre ellas, dentro de los diferentes ambientes o microambientes donde permanecen y conviven. Los mecanismos de comunicación les permiten reconocer cuando se alcanza un umbral o nivel de presencia para desarrollar nuevas funciones, especialmente un comportamiento social, simbiótico y de permanente reconocimiento, útil para nuevas tareas (Nowak, 2009).
Las biopelículas naturales en muchos ambientes, se caracterizan a menudo por presentar alta densidad celular y alta diversidad de especies microbianas, que les permiten estrechas interacciones célula-célula dentro o entre las especies, dando lugar a inevitables interacciones intra e inter-especies, incluyendo la cooperación y la competencia (Kolenbrander et al., 2002). Estas interacciones pueden desempeñar papeles muy importantes en el mantenimiento de la homeostasis de microbios en una comunidad de la biopelícula (Kuramitsu et al., 2007).
En los procesos de colonización de sustratos limpios sumergidos que involucra la adhesión, desde moléculas disueltas, a bacterias, diatomeas e invertebrados, intervienen algunos factores que pueden favorecer o dificultar la adhesión; por las fuerzas motrices, que pueden ser primarias o secundarias. La primera se relaciona con la abundancia relativa de cada tipo de molécula y microbiota en la columna de agua (Lau et al., 2002), mientras que las fuerzas motrices secundarias son las interacciones físicas, químicas y de comportamiento entre los incrustantes. Ejemplo de ello son las interacciones entre las bacterias asociadas a la superficie y las larvas de invertebrados que buscan asentarse (Rodriguez y Epifanio, 2000). Un caso puntual es el del poliqueto Hydroides elegans, que depende para su asentamiento de las bacterias, las cuales mediante la producción y posterior expulsión al medio de moléculas de comunicación contribuyen a que el poliqueto pueda seleccionar el área para asentarse; a las bacterias que se les han relacionado con este proceso son las Gram positivas de los géneros Bacillus, Brevibacterium, Micrococcus y Staphylococcus, y las Gram negativas de los géneros Alteromonas, Pseudoalteromonas y Vibrio (Lau et al., 2002; Hadfield, 2011). La interacción entre bacterias y eucariotas bentónicos se ha demostrado incluso con evidencias paleontológicas que revelan una relación entre metazoos y biopelículas muy temprano en la evolución de estos; por lo tanto, los receptores a las señales bacterianas en las larvas de invertebrados son muy antiguos y posiblemente únicos (Hadfield, 2011).
Las bacterias juegan un rol importante en los ambientes marinos, incluyendo la participación en los ciclos biogeoquímicos del sulfuro, nitrógeno y carbono (Paerl y Pinckney, 1996; Hawley et al., 2017) supliendo de materiales y energía a niveles tróficos superiores (Azam et al., 1983; de Carvalho y Caramujo, 2012). Las especies del género Roseobacter son reconocidas como las principales colonizadoras de superficies en ambientes acuáticos debido a su rápida respuesta a la presencia de nutrientes en la etapa de acondicionamiento bioquímico del sustrato y la formación de biopelícula (Dang et al., 2008). De hecho, se ha encontrado que estas bacterias son los microbios más importantes en las superficies marinas para iniciar la colonización; estas células pueden determinar la estructura y función de las biopelículas maduras (Dang et al., 2008; Dang y Lovell, 2016). Colonizan superficies naturales, artificiales, aceleran la corrosión (Little et al., 2008), favorecen el biofouling (Cao et al., 2011) y pueden influir en la flotabilidad del plástico polietileno (Lobelle y Cunliffe, 2011). No obstante, Lee et al. (2018) sugieren que en el medio marino la composición de las comunidades bacterianas cambia a través del tiempo; las colonizadoras primarias se caracterizan por ser poco abundantes en él, pero de crecimiento rápido y las poblaciones varían de acuerdo con la superficie o nutrientes disponibles. Por estas razones es de suma importancia conocer las comunidades bacterianas asociadas a los sedimentos marinos y evaluar su capacidad de formación de biopelículas en condiciones de cultivo in vitro que permitan plantear hipótesis sobre su desarrollo en ambientes naturales.
ÁREA DE ESTUDIO
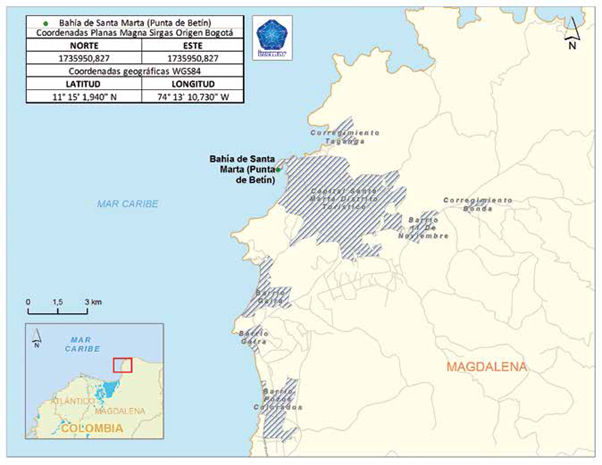
Punta de Betín se ubica en la costa Caribe colombiana y pertenece al área de Santa Marta, departamento del Magdalena. Es una península rocosa de pendientes escarpadas, localizada al norte de la bahía de Santa Marta, por lo que se ve directamente afectada por la descarga de aguas dulces (río Manzanares y emisario submarino) y el puerto (granos y polvillo de carbón) (Martínez y Acosta, 2016). La estación de muestreo se ubicó en las coordenadas 11°15'01,94''N y 74°13'10,73''W, recolectando muestras a tres profundidades: 5, 10 y 15 m (Figura 1).
MATERIALES Y MÉTODOS
Recolección de muestras.
Las muestras fueron recolectadas en Punta Betín (bahía de Santa Marta) con una draga Van Veen de 14 cm de diámetro con superficie de muestreo de 0,08 m2 y buceo autónomo, tomando la capa de sedimento superficial por triplicado en un rango de 5 hasta 15 m de profundidad. Las muestras recolectadas fueron almacenadas en frascos estériles forrados con papel aluminio para protegerlos de la luz y almacenados en una nevera plástica con hielo aproximadamente a 4 °C, para ser transportada al área de productos naturales marinos del Laboratorio de Bioprospección Marina del Invemar-LabBIM para su procesamiento.
Crecimiento y aislamiento de bacterias marinas
La obtención de bacterias cultivables a partir de las diferentes muestras, se realizó empleando medios sólidos con alta concentración de nutrientes, como el agar Tripticasa soja con 1% de NaCl (TSA-I), Agar Marino (AM), Agar ISP2 con 1% NaCl y Agar Actinobacterias (Difco) enriquecido con sales (Raver, 2000). Se adicionaron 20 g de sedimento de la muestra en cajas de Petri estériles y se sometió a un tratamiento térmico en un horno a 50 °C por 60 min, con el fin de eliminar la microbiota vegetal de la muestra (Rocha et al., 2013). A continuación, se realizaron diluciones seriadas (1/10, 1/100 y 1/1000) en agua de peptona suplementada con agua de mar artificial estéril (1:1) y se sembraron en placas de agar incubándolas a 26°C por un período de 15-20 días (León et al., 2007; Romero y Otero 2010; Torres et al., 2012).
Conservación de las cepas
Se aislaron las cepas del sedimento, que luego se conservaron en cultivos axénicos. Cada aislamiento se hizo en caldo nutritivo suplementado con 1 % de NaCl y glicerol a 20% (V/V). Las cepas cultivadas fueron codificadas y almacenadas a una temperatura de -80 °C. A partir del concepto que la producción de antimicrobianos está regulada por mecanismos quorum sensing, en este trabajo solamente se evaluaron once cepas que mostraron actividad biológica contra microorganismos resistentes de interés clínico en ensayos realizados por el Laboratorio de Bioprospección Marina del Invemar. Finalmente, la codificación de estas se basó en la identificación molecular de las mismas. Las cepas fueron depositadas en el Museo de Historia Natural Marina de Colombia - Makuriwa del Invernar, el cual asignó los siguientes códigos para las cepas identificadas: 64093 , 64094, 64095, 64096, 64097, 64100, 64101, 64181, 64186, 64170 y un aislado sin identificar 64156.
Reactivación de las cepas
La reactivación de los aislados evaluados en este trabajo se llevó a cabo a partir de un proceso de descongelamiento que consistió en pasar de -80 °C a -20 °C y dejar las cepas durante dos horas en esta temperatura, luego se pasaron a 4 °C y se mantuvieron una hora a esa temperatura. Posteriormente, se inocularon 100 μl (usando la técnica de crecimiento masivo) en cajas Petri con agar TSA a 28 °C durante 48 h; finalmente se mantuvieron a 4 °C para su uso durante este estudio.
Identificación genética
La identificación genética de las once cepas partió de un cultivo en agar TSA incubado a 28 °C durante 48 h.
Extracción del ADN
La extracción del ADN se realizó usando el kit PureLink Genomic DNA de Invitrogen. Se realizó elución en un volumen final de 50 μl. Al finalizar el proceso de extracción se realizó por espectrofotometría a longitudes de onda de 260/280 nm en un equipo Nanodrop (Thermo Fisher Scientific).
Amplificación del marcador ARN 16S
El ADN obtenido crudo se empleó como plantilla en la amplificación de la región 16S mediante reacción en cadena de la polimerasa (PCR) (Dashti et al., 2009; Sowmya et al., 2012). El gen 16S se amplificó empleando los iniciadores universales 27F (5'-AGA GTT TGA TCM TGG CTC AG-3') y 1492R (5'-GGT TAC CTT GTT ACG ACT T-3') (Weisburg et al., 1991) que amplifican un fragmento de 1500 pares de bases (pb) aproximadamente.
Las condiciones de la PCR fueron: desnaturalización inicial del ADN a 94 ° C durante 3 min, 35 ciclos de amplificación, desnaturalización a 94 ° C durante 30 s, recocido del cebador a 50 ° C durante 30 s, extensión del cebador a 72 ° C durante 2 min, seguido de una extensión final a 72 ° C durante 8 min (termociclador T-100 Bio-Rad). Los productos de PCR se migraron en gel de agarosa al 1% (p / v) a 100 V durante 80 min, tampón TBE 1X y 1 ll de tampón de carga EZ-vision (AMRESCO). Los resultados se visualizaron por exposición a la luz ultravioleta y se fotografiaron con un sistema de captura digital (ENDUROTM GDS - LABNET). Adicionalmente, la identidad de algunos aislados del género Bacillus fue complementada a nivel de especie mediante la amplificación del gen codificante para la chaperonina groEL empleando los iniciadores 550F (5'-GAGCTTGAAGTKGTTGAAGG-3') y 1497R (5'-TGAGCGTGTWACTTTTGTWG-3') (Rooney et al., 2009).
Secuenciamiento del marcador ARN 16S
El producto de amplificación se sometió a reacción de secuenciamiento por el método de Sanger/capilar y leyendo ambas cadenas para asegurar la fiabilidad de la secuenciación. Esto fue realizado en las instalaciones de la corporación Corpogen en Bogotá e instalaciones del Centro Nacional de Secuenciación Genómica de la Universidad de Antioquia en Medellín.
Ensamblaje y Clasificación basado en el marcador 16S
Las secuencias obtenidas fueron depuradas de los extremos con bases de baja calidad y ensambladas con el programa eBioX versión 1.5.1. La secuencia final fue analizada mediante comparación con las secuencias depositadas en la base de datos del servidor BLAST del National Center for Biotechnology Information (NCBI) y el rRNA Database Project (RDP II).
Las secuencias codificantes para el ARNr 16S fueron alineadas con el programa SINA (Silva Incremental Aligner v1.2.11), el cual usa una combinación entre las secuencias de los k-mer y la alineación de orden parcial (POA) con el fin de mantener una alta precisión (Pruesse et al., 2012). La identidad de las secuencias fue comparada con las secuencias de referencia alojadas por SILVA RDP, Greengenes and SILVA, empleando el método del ancestro común más bajo (ACB) y buscando cinco de las secuencias más cercanas con una identidad mínima de 0,97.
Análisis Filogenético
El análisis filogenético se llevó a cabo inicialmente con un análisis de distancias de las diez secuencias obtenidas durante el proceso de secuenciación y realizando una búsqueda en las bases de datos con secuencias reportadas del género Bacillus para el marcador ARN 16S. Para la construcción del árbol se usó el software MEGA 7 con el método de máxima verosimilitud con 1000 réplicas de Bootstrap, siguiendo el modelo de evolución Hasegawa, Kishino y Yano (1985).
Inducción a la formación de biopelículas y diseño experimental
Para la inducción a la formación de biopelículas en las once cepas estudiadas, se realizó un preinóculo de cada una de las cepas en caldo tripticasa de soya (TSB), y en frascos microbiológicos, adicionando 5 ml del medio de cultivo más 200 μl de la cepa y se dejaron en agitación durante 24 h a 28 °C. Posteriormente, se midió la absorbancia a una longitud de onda 600 nm con un espectrofotómetro Multiskan Go (Thermo Scientific), para confirmar que la absorbancia no fuera superior a 0,2 con el propósito de tener todas las cepas en la misma concentración para realizar el ensayo.
La formación de las biopelículas se llevó a cabo en placas de 96 pozos de fondo plano y en cada pocillo se adicionaron 100 μl de la cepa y 100 μl de medio TSB fresco, incubando todas las cepas inicialmente a 28 °C y en condiciones estáticas, para inducir a la formación de la biopelícula durante 24, 48, 72 h. Posteriormente, se seleccionaron dos cepas con mayor formación de biopelícula (64181 y 64186) para realizar ensayos a diferentes temperaturas (26, 28 y 30°C) por 48 h. Como control negativo se utilizó una cepa de Staphylococcus aureus ATCC 43300, la cual se puso a crecer en las mismas condiciones que las cepas estudiadas. Por cada cepa se realizaron cinco repeticiones, incluyendo el control negativo, y cada experimento de cuantificación se replicó tres veces. Todo esto para ambos métodos (Cristal violeta, XTT).
En las gráficas que se presentan se usa como código de cada cepa los dos últimos dígitos de los cinco utilizados.
Tinción con cristal violeta
La cuantificación de la formación de biopelículas a los ensayos de 24, 48 y 72 h, se llevó a cabo con la tinción con cristal violeta; para ello se extrajo el medio de cultivo de los pozos y se les realizaron tres lavados con 200 μl de PBS 1X; después se realizó un lavado con agua estéril y se tiñó con 200 μl de cristal violeta a una concentración de 0,2 %; se esperó 15 minutos y se extrajo el colorante para realizar nuevamente dos lavados con 200 μl de agua estéril, dejando secar al aire libre; a continuación se añadió una solución de ácido acético 10 %, etanol 50 % y agua 40 % (v/v/v) para diluir el cristal violeta y finalmente se midió la absorbancia a una longitud de onda 540 nm a los ensayos de 24, 48 y 72 h.
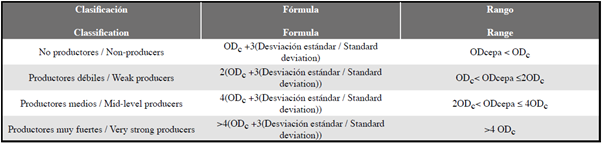
El análisis estadístico realizado a los datos fue la media, desviación estándar y se aplicó la fórmula de densidad óptica (OD) del control para clasificar las cepas de acuerdo con la formación de la biopelícula; esto se realizó con los resultados del ensayo a las 48 h. Las categorías seleccionadas fueron no productores, productores débiles, productores medios y productores muy fuertes (Tabla 1) (Stepanović et al., 2000).
Tinción y cuantificación de la actividad metabólica
Preparación de los reactivos
Para la cuantificación de la actividad metabólica de las biopelículas se usó como método la reducción del colorante cloruro de tetrazolio - XTT (Sigma-Aldrich) mediante respiración celular. Para ello se preparó una mezcla de XTT y menadiona. El XTT se preparó a 1 mg/ml y se disolvió en PBS 1X, luego se filtró mediante un filtro de 0,22 μm. La menadiona se preparó a una concentración de 400 mM y se disolvió en acetona para filtrarse posteriormente a 0,22 μm. La mezcla XTT/Menadiona se realizó a una proporción 5:1.
La tinción XTT se inició con el lavado de las biopelículas con 200 μl de PBS 1X (tres lavados) para eliminar bacterias planctónicas que pudieran estar en los pozos; luego se agregaron 200 μl de PBS y 12 μl de reactivo XTT/Menadiona y se incubó la microplaca a 30 °C durante dos h, sin exponerse a la luz y se midió la absorbancia a 490 nm. La reducción del colorante es una medida de la actividad metabólica.
Viabilidad celular - Tinción Live/Dead
Se realizaron ensayos de formación de biopelículas en cubreobjetos de vidrio durante 24 h para las cepas 64181 y 64186 y para el ensayo mixto. Para llevar a cabo lo anterior, los cubreobjetos estériles se colocaron en placas de Petri con 5 ml de cultivo fresco y 20 μl del medio con las cepas, incubándolos a 28 °C para el desarrollo de la biopelícula. Transcurrido el tiempo, los cubreobjetos fueron lavados con PBS 1X y se tiñeron con el sistema LIVE/DEAD® Viability Kit (Invitrogen), dejándolos en incubación durante 15 a 20 min a temperatura ambiente. La formación de las biopelículas se visualizó mediante un microscopio de epifluorescencia, lo que permitió reconocer mediante la tinción Live/Dead las células que estaban vivas (verde - amarillo) y las células muertas (rojas).
Análisis estadístico
El análisis estadístico de los datos se efectuó primero con el software Excel para realizar las gráficas de promedios y desviaciones estándar para cada experimento. Posteriormente el análisis inferencial de los datos se hizo con el software R; primero, a los datos de cada experimento se les realizó una prueba de normalidad y homogeneidad de varianzas; luego, de acuerdo con estos resultados, para comparar las medias entre tratamientos (cepas, tiempos, temperaturas) se hicieron análisis de varianza ANOVA en el caso de los datos paramétricos y Kruskal Wallis para los no paramétricos; en cada caso se realizaron pruebas de comparaciones múltiples Tukey y Nemenyi respectivamente con un p ≤ 0,05.
RESULTADOS
Identificación molecular
El análisis filogenético a partir de las secuencias del gen 16S ARN permitió clasificarlas dentro del género Bacillus; sin embargo solo fue posible identificar hasta especie la cepa 64181. Para esto se usó el marcador groEL (Rooney et al., 2009), fragmento que codifica para una proteína que permite tolerar cambios térmicos en especies de Bacillus. Este marcador indicó que dicha cepa corresponde a la especie Bacillus safensis con un porcentaje de identidad del 100% (Figura 3).
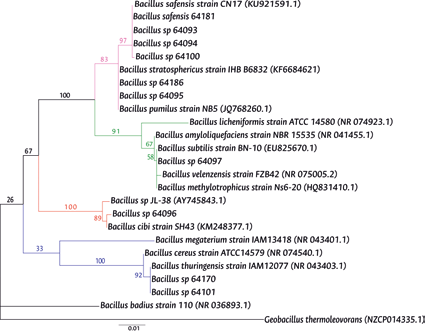
Figura 2 Árbol de máxima verosimilitud representando la relación filogenética de las cepas estudiadas basado en el gen 16S ARN. Los valores de bootstrap se muestran en porcentaje. En paréntesis se indica el código de acceso en el GenBank. Ramas en azul representan el grupo de Bacillus cereus, las rojas el grupo de Bacillus cibi, las verdes el grupo de Bacillus subtilis, las rosadas el grupo de Bacillus pumilus.
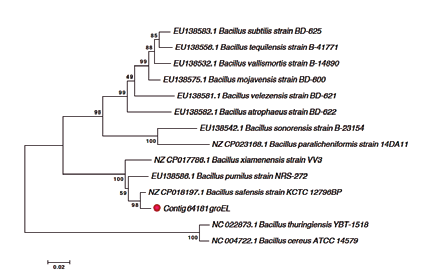
Figura 3 Árbol de Neighborjoining representando la relación filogenética de la especie Bacillus safensis cepa 64181 (punto rojo) basada en el gen groEL. Los valores de bootstrap se muestran en porcentaje.
Las otras cepas analizadas se agruparon según el árbol de máxima verosimilitud en los grupos de Bacillus subtilis, Bacillus cereus y Bacilluspumilus. Por ejemplo, la cepa 64097 se ubicó cercana a las secuencias de referencia del grupo Bacillus subtilis, formando un grupo hermano con valor de bootstrap de 83; las cepas 64101, 64170, por su parte, formaron grupos hermanos con las secuencias de referencia del grupo Bacillus cereus, ambas con Bacillus thuringiensis strain IAM12077 (NR043403.1) con valor de bootstrap de 92 y la cepa 64096 con Bacillus cibi strain SH43 (KM248377.1) mostrando un bootstrap de 89. Los aislados restantes se ubicaron en el grupo Bacillus pumilus, el cual se dividió en dos clados uno con bootstrap de 97 y el otro con 83 (Figura 2).
Inducción a la formación de biopelículas
Ensayo general con tinción de cristal violeta
Los ensayos para la inducción a la formación de biopelícula, realizados para todas las cepas a las 24 y 48 h mostraron que en condiciones estáticas y a temperatura de 28 °C, los bacilos aislados de sedimentos marinos en su mayoría son formadores en diferente medida de biopelícula, destacándose B. safensis 64181 y Bacillus sp 64186 como las de mayor adherencia, exceptuando Bacillus sp 64093, Bacillus sp 64100 y 64156, que no mostraron adherencia (Tabla 2). Esta clasificación se realizó a partir de dos cortes sobre los valores de absorbancia obtenidos de los resultados del ensayo de cristal violeta a las 48 h, tiempo en el que se evidenció la mayor formación de biopelícula por parte de las bacterias; como control negativo se usó la bacteria S. aureus por ser una bacteria con capacidad de formación de biopelícula que a menos de 37°C se le dificulta realizar este proceso.
Es importante resaltar que fue comparada la densidad óptica (O.D) de los aislados con la del control negativo y se consideró formadora de biopelícula cuando esta absorbancia fue superior a 0,4 (Figura 4).
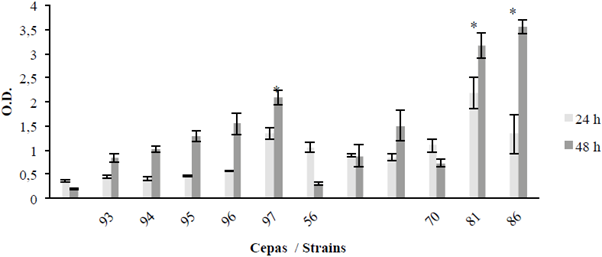
Figura 4 Formación de la biopelícula a 24 y 48 horas; cepas 64093, 64094, 64095, 64096, 64097, 64156, 64100, 64101, 64170, 64181 y 64186 usando como control la cepa de S. aureus (SA). Densidad óptica (O.D.); la barra es el ± 1 error estándar y los asteriscos diferencias significativas entre las cepas y el control, solamente para 48 horas (p ≤ 0,05).
En cuanto a la comparación del comportamiento todas las cepas entre las 24 y 48 h a 28 oC y en condiciones estáticas, la mayoría de las cepas aumentaron la biomasa de las 24 a las 48 h, a excepción de las cepas 64156 y 64170 (Figura 4). Las diferencias significativas que se muestran en la siguiente gráfica corresponden a las cepas 64097, 64181 y 64186 respecto al control negativo (-) (p ≤ 0,05) a las 48 h.
Formación de biopelículas cepas 64181 y 64186
De acuerdo con los resultados, B. safensis 64181 y Bacillus sp 64186fueron las de mayor formación de biopelículas (Tabla 2 y Figura 4), por lo tanto se procedió a su cuantificación por separado y juntas (mixto) a las 24, 48 y 72 h; luego de este tiempo se presentaron inconvenientes para la lectura de la absorbancia debido a la alta formación de biomasa, lo que hizo difícil obtener la densidad óptica (O.D.) del ensayo. En la Figura 5 se muestra que la mayor formación de biopelícula se da a las 48 h en condiciones estáticas, alcanzando un O.D. de 3,00, 3,64 y 2,58 para las cepas 64181, 64186 y el cultivo mixto. Después de este tiempo todas disminuyen.
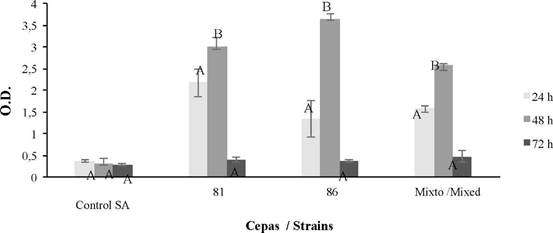
Figura 5 Formación de la biopelícula a las 24, 48 y 72 horas para las cepas 64181, 64186 y el cultivo mixto, usando como control la cepa de S. aureus (SA). Densidad óptica (O.D.); la barra promedio indica el ± 1 error estándar y las letras tiempos iguales entre sí, por separado para el control, cepas y mixto (p ≤ 0,05).
Para el ensayo de la biopelícula mixta, se observó que la formación de esta fue inferior a la que mostró el tratamiento de cada cepa individual, a pesar que las condiciones de crecimiento fueron las mismas para todos los tratamientos (Figura 5). En relación con los experimentos realizados a diferentes temperaturas para B. safensis 64181, Bacillus sp 64186 y juntas (mixto) (Figura 6) se determinó que a 26 °C y 30 °C existen diferencias significativas (p ≤ 0,05) en la formación de biopelícula respecto a la temperatura de 28°C.

Figura 6 Formación de la biopelícula a 26, 28 y 30 °C durante 48 horas para las cepas de mayor formación B. safensis 64181, Bacillus sp 64186 y Mixto (M). Densidad óptica (O.D.); la barra promedio indica el ± 1 error estándar promedio y las letras temperaturas iguales entre sí, por separado para las cepas y mixto (p ≤ 0,05).
Ensayo general de actividad metabólica con XTT
Para conocer la actividad metabólica de la biopelícula, también se realizaron ensayos con XTT a todas las cepas y el control negativo (-), observándose que todas tienen una actividad metabólica por encima de 0,6 y en algunas aumenta de las 24 a las 48 h, se resalta nuevamente que las cepas con mayor actividad fueron 64097, 64181 y 64186 (Figura 7). De nuevo, los ensayos con cada cepa mostraron que la mayor actividad metabólica se da a las 48 h, perdurando la actividad hasta las 96 h. El grupo A (64097,64181 y 64186) fue diferente a 64095, 64096 y 64156 a las 24 h, el grupo B (control) fue diferente a 64096 a las 48 h y el grupo C (64097, 64181 y 64186) fue significativamente diferente a las cepas 64095, 64096 y 64156 a las 48 h(p ≤0,05).
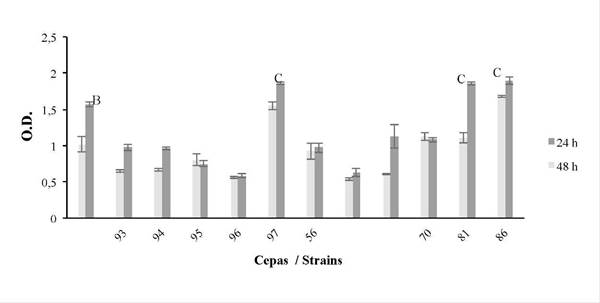
Figura 7 Actividad metabólica durante la formación de la biopelícula a 24 y 48 h (método XTT), para las cepas 64093, 64094, 64095, 64096, 64097, 64156, 64100, 64101, 64170, 64181, 64186, usando como control la cepa de S. aureus (SA). La variable respuesta fue densidad óptica (O.D.). La barra es promedio ± 1 error estándar y para las letras el resultado de comparaciones estadísticas múltiples entre cepas, por separado para cada tiempo (p ≤ 0,05).
Actividad metabólica de B. safensis 64181 y Bacillus sp 64186
La comparación de la actividad metabólica en las biopelículas de B. safensis 64181 y Bacillus sp 64186 a través del tiempo, permitió identificar las horas en que el metabolismo de las bacterias está más activo, observándose que a las 48 h se alcanza su máximo de actividad metabólica en experimentos in vitro y luego a partir de las 72 h empieza a descender. También se observó que en el ensayo mixto hay poca variación entre las 48 y 72 horas, a diferencia de las cepas individuales que presentaron una disminución en la actividad metabólica celular (Figura 8).
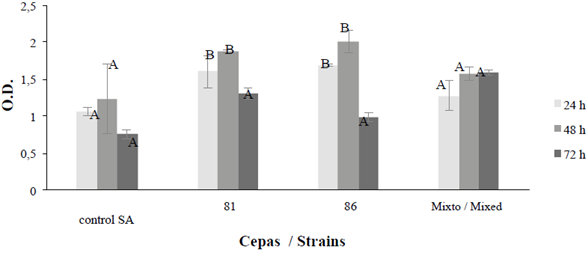
Figura 8 Formación de la biopelícula a las 24, 48 y 72 horas para las cepas B. safensis 64181, Bacillus sp 64186 y Mixto, usando como control la cepa de S. aureus (SA). Densidad óptica (O.D.); la barra promedio indica el ± 1 error estándar y las letras tiempos iguales entre sí, por separado para control, cepas y mixto (p ≤ 0.05).
Por último, los experimentos variando la temperatura, mostraron que a los 28 °C existe una mayor actividad metabólica en las bacterias que forman las biopelículas individuales a diferencia de la mixta donde se mantiene igual desde los 26 hasta los 28 °C e incluso sin diferencias significativas con 30 °C (Figura 9).
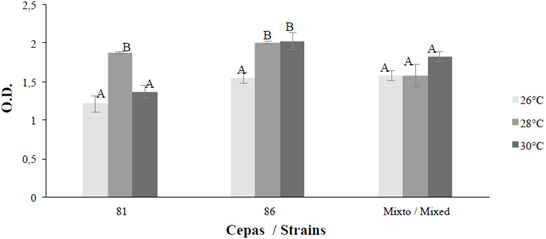
Figura 9 Formación de la biopelícula a 26, 28 y 30 °C durante 48 horas para las cepas de mayor formación B. safensis 64181, Bacillus sp 64186 y Mixto (M). La variable respuesta es densidad óptica (O.D.). La barra promedio indica el ± 1 error estándar y las letras temperaturas iguales entre sí para las cepas y el mixto (p ≤0.05).
Viabilidad celular Tinción LIVE/DEAD
En cuanto a la formación de biopelícula en cubreobjetos, se encontró que las bacterias estaban vivas en todos los experimentos (tinción LIVE/DEAD). Se observó que Bacillus sp 64186 mostró una gran cobertura de área, recubriendo la mayor parte de la superficie; sin embargo, en algunas zonas se visualizó que las bacterias se aglomeraban para seguir formado la biopelícula (Figura 10 e y f).
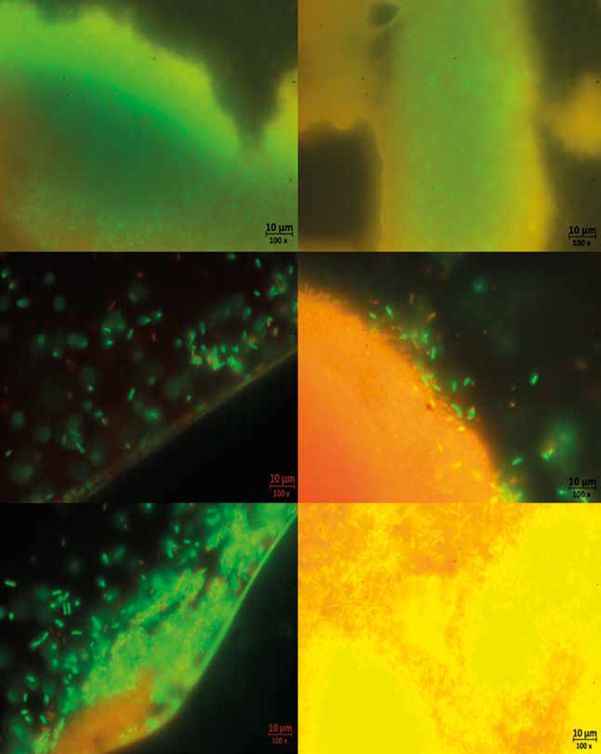
Figura 10 Tinción Live/Dead de las biopelículas después de 24 horas de cultivo sobre cubreobjetos. a. biopelícula de la cepa 64181; b. biopelícula con formaciones similares a canales, cepa 64181; c y d. biopelículas mixta, bacterias adhiriéndose a la matriz; e. bacterias formando cúmulos, cepa 64186; f. Biopelícula de la cepa 64186.
Incluso, en el ensayo con B. safensis 64181 (Figura 10 a) se observó que esta no se adhirió al vidrio, y la biopelícula se formó en la interfase líquido-aire dificultándose la visualización en el microscopio; a pesar de ello se logró ver una matriz donde se encontraban inmersas las bacterias vivas con espacios libres de células que parecían estar formando estructuras similares a canales (Figura 10 b). Finalmente, la biopelícula mixta se observó más densa que las anteriores y con gran cantidad de bacterias adheridas a la matriz, la cual tenía una estructura que presentaba poros y mostró que las bacterias vivas seguían adhiriéndose a ella (Figura 10 c y d).
DISCUSIÓN
Identificación de las cepas
De acuerdo a la identificación molecular se determinó que todos los aislados pertenecen al phylum Firmicutes, familia Bacillaceae. Del análisis filogenético se obtuvo que pertenecían al género Bacillus ubicándose las cepas en diferentes grupos, tales como el de Bacillus cereus, Bacillus pumilus y Bacillus subtilis con un porcentaje de identidad de alrededor de 99% con las cepas de referencia del NCBI. Las cepas cercanas fueron las usadas para el análisis filogenético de máxima verosimilitud. Sin embargo, se recomienda el uso de otros marcadores como el gyrB y el XRE (Helgason et al., 2000; Punina et al, 2013) para identificar todas las cepas hasta el nivel taxonómico de especie.
A partir de los resultados de la identificación de las cepas estudiadas, se concluye que esta fue influenciada por el proceso selectivo realizado con las muestras de sedimento en el que se buscó favorecer el crecimiento de bacterias Gram positivas, en especial de actinobacterias con el propósito de identificar actividad antimicrobiana en estas y estudiar los procesos derivados de la comunicación celular quorum sensing como la formación de biopelículas. Aunque en este estudio se diseñó una metodología que consistió en emplear 20 g de sedimento marino para realizarles un tratamiento con calor a 50°C, durante 60 minutos, con el fin de eliminar la microbiota vegetativa acompañante en la muestra, se obtuvieron aislados pertenecientes al género Bacillus, bacterias que presentan estructuras de resistencia como las endosporas, lo que les permiten tolerar temperaturas hasta aproximadamente 60 °C (Kristjansson, 1992). De manera general, muchos estudios respaldan el uso de tratamientos mediante calentamiento, con el fin de inhibir el crecimiento de bacterias Gram negativas de crecimiento rápido (Leiva et al., 2015; Matobole et al., 2017), descontaminación de muestras de sedimentos (Kim, 2013) o tratamientos selectivos para favorecer el crecimiento de grupos microbianos (Bredholt et al., 2008).
Formación de biopelículas
Para evaluar la capacidad de formar biopelículas de las cepas aisladas del sedimento marino, se realizaron los ensayos de inducción y cuantificación usando diferentes métodos. El primero, cristal violeta, permitió medir de forma indirecta a cada una de las cepas y así clasificarlas según la capacidad de adherencia, partiendo de la comparación de cada cepa con el control negativo S. aureus, el cual, según Martínez y Arévalo (2010), muestra disminución en la formación de la biopelícula a temperaturas por debajo de 37 °C. Por lo tanto, las cepas cuya absorbancia fue menor al control negativo fueron consideradas no adherentes (Tabla 2). También este método permitió evaluar, en función de la biomasa, diferentes condiciones de crecimiento. El segundo método, cuantificación por reducción del XTT, proporcionó información sobre la actividad metabólica de las células en la biopelícula.
La formación de la biopelícula se evaluó a 24 y 48 h para todos los 11 aislados, entre estos las especies que mostraron más biomasa y actividad metabólica fueron Bacillus sp 64097, B. safensis 64181 y Bacillus sp 64186. Las dos últimas fueron seleccionadas por producir la mayor cantidad de biomasa, éstas durante los ensayos a diferentes tiempos (24, 48, 72 h), mostraron que eran más activas metabólicamente a las 24 y 48 h; sin embargo, las biopelículas fueron más densas a las 48 h.
Esto se explica porque cada microorganismo produce diferentes cantidades de exopolisacáridos, que además tienden a aumentar a medida que transcurre el tiempo, desde que se inició la fase de adhesión irreversible de las bacterias a la superficie hasta el biofilm maduro (Leriche et al., 2000). Según Flemming y Wingender (2010), los microorganismos representan menos de 10 % de la masa seca, mientras que la matriz puede representar más de 90 % en la mayoría de los casos. No obstante, la formación de éstas depende de la capacidad de las bacterias para detectar señales ambientales; por ejemplo, la disponibilidad de nutrientes (Costerton et al., 1995; Adams y McLean, 1999; Costerton et al., 1999), los cuales se van agotando con el transcurso del tiempo, dificultado su difusión a través de la matriz en la biopelícula, lo que genera un gradiente que afecta la actividad metabólica de las células.
Para la actividad metabólica, los resultados respecto al tiempo evidencian que existen diferencias entre el ensayo de cada una y juntas (mixto). La biopelícula mixta a través del tiempo no mostró variación, posiblemente porque la formación de un microconsorcio permite a las células estar en una matriz más dinámica que les favorezca para alcanzar una homeostasis, debido a la heterogeneidad de sustancias que esta presenta, y así adaptarse a los cambios ambientales o al agotamiento de nutrientes (Sutherland, 2001; Flemming y Wingender, 2010).
Las diferencias entre la cantidad de biomasa y la actividad metabólica de las bacterias en relación con la temperatura para los ensayos monoespecie indican que a 28°C se evidencia la biopelícula más densa, mientras que a las otras temperaturas sucede lo contrario, a pesar de estar activas metabólicamente, sugiriendo que la temperatura puede afectar otras etapas del desarrollo de las biopelículas como la adhesión irreversible y así tardar más tiempo para generar una matriz más densa. El ensayo mixto muestra una biopelícula menos densa que las producidas por las especies individuales para cada temperatura, posiblemente la alta síntesis de enzimas de β-galactosidasas por parte de la cepa 64181, identificada como B. safensis, afecta a los polisacáridos que conforman la matriz mixta (Nath et al., 2012). En estudios realizados con biopelículas mixtas en ambientes naturales marinos, se ha demostrado que a medida que se aumenta la temperatura, la actividad metabólica de la biopelícula incrementa (Rao, 2010), al igual que ocurrió con el ensayo de la biopelícula mixta a 30 °C, el cual incrementó su actividad metabólica respecto a las otras temperaturas (Figura 9). Además, Yang et al (2013) demostraron que cepas de Bacillus en altas temperaturas tenían una mayor tasa de crecimiento planctónico que de la biopelícula.
Complementario a los ensayos de biopelículas, la viabilidad celular observada a las dos mediante la tinción LIVE/DEAD (Figura 10), facilitó visualizar algunas estructuras de las biopelículas y características particulares de su formación; por ejemplo, la especie B. safensis 64181 no se adhirió al vidrio y formó la biopelícula en la interfase líquido- aire. En muchas investigaciones se ha afirmado que la adherencia de las bacterias a las superficies es directamente proporcional a la rugosidad de la misma (Characklis y Marchall, 1990). Sin embargo, la adherencia irreversible de las bacterias depende de otros factores como la interacción entre la naturaleza del material y célula; por ejemplo, el tipo de interacción hidrofóbica que se produce entre la bacteria y la superficie podría ayudarla a superar las fuerzas repulsivas y unirse irreversiblemente a ésta (Donlan, 2002).
A pesar de ello, estas afirmaciones son contradictorias en la literatura, debido a que no hay metodologías estandarizadas para medir la naturaleza de los materiales, pese a que se sugiere que los microorganismos se adhieren más rápidamente a superficies hidrófobas y no polares como el teflón, poliestireno y otros plásticos, que a materiales hidrófilos como el vidrio o los metales (Soler, 2018). En muchas bacterias Gram positivas, como las del género Bacillus, la superficie bacteriana se caracteriza por ser hidrófoba y tener afinidad a estos materiales.
En cuanto, a las biopelículas de Bacillus sp 64186 y la mixta, se observó una matriz densa que presentaba en algunas zonas cúmulos de bacterias y en algunos casos la tinción se visualizaba roja. Esto se debe a que la composición de la matriz está formada por ADN extracelular (eADN), que comúnmente ha sido considerado un remanente de las células lisadas, aunque se produce en algunos casos en cantidades muy altas (Whitchurch et al, 2002; Flemming et al, 2007). Allesen-Holm et al. (2006) descubrieron que en Pseudomonas aeruginosa el eADN que formaba parte de la matriz se deriva de ADN genómico, y que este se organizaba de tal forma que servía como una estructura de soporte en forma de rejilla para la biopelícula; por lo tanto, se le atribuye un papel estructural.











 text in
text in