Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Boletín de Investigaciones Marinas y Costeras - INVEMAR
Print version ISSN 0122-9761
Bol. Invest. Mar. Cost. vol.50 supl.1 Santa Marta Dec. 2021 Epub Dec 13, 2021
https://doi.org/10.25268/bimc.invemar.2021.50.suplesp.933
Artículos de Investigación
Conexión migratoria entre zonas de cría del Pacífico Este y áreas de alimentación en la Península Antártica basado en coincidencias de genotipos individuales
1Laboratorio de Ecología Molecular de Vertebrados Acuáticos (LEMVA), Departamento de Ciencias Biológicas, Universidad de los Andes, carrera 1 No. 18A-10, Bogotá, Colombia. sj.caballero26@uniandes.edu.co
2Marine Mammal Institute and Department of Fisheries and Wildlife, Oregon State University, 2030 SE Marine Science Drive, Newport, OR 97365, USA
3Department of Ecology and Evolutionary Biology, University of California Santa Cruz, Coastal Biology Building, 130 McAllister Way, Santa Cruz, CA 95060, USA
4Fundación Macuaticos Colombia, Calle 27 No. 79-167, Medellin, Colombia
5Museo de Ballenas, Salinas, Ecuador/PO Box 09-06-2370, Guayaquil, Ecuador.
6Centro de Estudios Avanzados en Zonas Aridas (CEAZA), Raúl Bitrán 1305, La Serena, Chile.
7Programa Marino, Conservation International, Carrera 13 No. 71-41, Bogotá, Colombia.
8Fundación Malpelo y otros Ecosistemas Marinos, Carrera 11 No. 87-51, Bogotá, Colombia.
9Marine Mammal Institute and Department of Fisheries and Wildlife, Oregon State University, 2030 SE Marine Science Drive, Newport, OR 97365, USA Department of Ecology and Evolutionary Biology, University of California Santa Cruz, Coastal Biology Building, 130 McAllister Way, Santa Cruz, CA 95060, USA
10Marine Mammal Institute and Department of Fisheries and Wildlife, Oregon State University, 2030 SE Marine Science Drive, Newport, OR 97365, USA
We analysed 114 DNA samples collected in different locations within the stock G breeding grounds, including Ecuador and the northern and southern Pacific of Colombia. We genotyped 15 microsatellite loci, sexed all samples, and performed genotype comparisons. Genotype comparisons were done using a DNA register of previously genotyped individuals from the Colombian breeding ground and the Antarctic Peninsula feeding Area. We confirmed connectivity between feeding grounds in Antarctic Peninsula and breeding grounds in Colombia and Ecuador using microsatellite loci. We found recaptures within each sampling location in the same year, but we also found a few recaptures in the same locations between years both in breeding and feeding grounds. Interestingly, we found recaptures between breeding and feeding grounds using samples collected 20 years apart, providing some information about longevity in this stock. Further comparisons with other data sets (Brazil, North Pacific) are needed to understand the migratory connectivity of this stock.
KEYWORDS: Megaptera novaeangliae; Southeastern Pacific; Genotyping; Microsatellite; Migratory connectivity.
En este estudio, se analizaron 114 muestras de ADN colectadas en diferentes localidades en la zona de reproducción de la población G (stock G), incluyendo la costa de Ecuador y localidades en el norte y sur del Pacífico colombiano, así como muestras obtenidas en la primera expedición científica colombiana a la Antártica en el estrecho de Gerlache. Se genotipificaron 15 loci microsatellites y se realizaron comparaciones de los genotipos obtenidos. Para estas comparaciones se utilizó una base de datos genéticos de individuos tanto en zonas de reproducción de Colombia y la zona de alimentación alrededor de la península Antártica. Se confirmó la conectividad existente entre las zonas de alimentación alrededor de la península Antártica y las zonas de reproducción en Colombia y Ecuador utilizando marcadores microsatélites. Se encontraron recapturas entre localidades en un mismo año, pero también se obtuvieron algunas recapturas en una misma localidad entre años diferentes, tanto en zonas de reproducción como de alimentación. Se encontraron recapturas de individuos en muestras colectadas con 20 años de diferencia, dando información preliminar sobre la longevidad de los animales de esta población.
PALABRAS CLAVE: Megaptera novaeangliae; Pacífico sudeste; Genotipificación; Microsatélite; Conexión migratoria.
INTRODUCCIÓN
Las ballenas jorobadas tienen una distribución cosmopolita, con poblaciones distribuidas en las tres cuencas oceánicas: Atlántico Norte, Pacífico Norte y el hemisferio sur. Para el hemisferio sur, la Comisión Ballenera Internacional (International Whaling Comission, IWC) reconoce seis sub poblaciones o stocks (IWC, 1998). La identidad de cada una de estas sub poblaciones está definida de acuerdo con su distribución en zonas de alimentación en altas latitudes y sus áreas de cría en zonas del trópico. Las conexiones migratorias existentes entre estas zonas de cría y alimentación han sido sugeridas según evidencia obtenida por estudios comparativos basados en foto identificación (v. g., Acevedo et al. 2017, Flórez-González et al. 1998, Garrigue et al. 2002, Stevick et al. 2004), análisis de ADN mitocondrial (v. g., Baker et al. 1998, Olavarría et al. 2007, Félix et al. 2012) y, recientemente, por comparación de genotipos a nivel individual (Steel et al. 2017).
Se describió una conexión migratoria entre lugares de reproducción y cría en la costa Pacífica de Colombia y las zonas de alimentación alrededor de la Península Antártica (sub población o stock G) a partir de análisis genéticos (Caballero et al. 2000, 2001; Olavarría et al. 2007). Cuando se compara la sub población G con otras sub poblaciones del Hemisferio Sur, se encuentra que la población que visita y se reproduce anualmente en el Pacífico Colombiano presenta una diferenciación genética significativa con respecto a las demás de esta región (Olavarría et al. 2007). Adicionalmente se ha reportado una diversidad haplotípica (h) y nucleotídica (π) relativamente altas para esta zona de reproducción (Caballero et al. 2001, Olavarría et al. 2007). La zona de reproducción del Pacífico Colombiano puede tener una importancia particular ya que se ha sugerido como un posible corredor para flujo genético en el pasado y actualmente, permitiendo el intercambio genético entre los grupos del Pacífico Norte y el Hemisferio Sur. Esto ha sido sugerido con base en la identificación de haplotipos de la región control del ADN mitocondrial que se encuentran compartidos entre la zona de reproducción del Pacífico Colombiano y las zonas de alimentación del Pacífico Norte (Caballero et al. 2001, Baker y Medrano-González 2002)
Otras áreas del Pacífico Sudeste han sido reconocidas también como zonas de reproducción de la sub población G. Estas incluyen la costa de Ecuador y las Islas Galápagos en el sur y la frontera entre Panamá y Colombia en el norte (Félix et al. 2011, Félix et al. 2012). Parece existir también sub estructuración o estratificación poblacional entre las localidades que constituyen esta zona de reproducción, ya que algunos haplotipos encontrados en muestras de Ecuador solo han sido previamente descritos en el Estrecho de Magallanes y no alrededor de la Península Antártica (Félix et al. 2012). Estudios moleculares en estas zonas adicionales podrán ayudar a clarificar si esta posible sub estructuración existe entre localidades de esta zona de reproducción (Félix et al. 2012), ya que estudios de foto identificación (Acevedo et al., 2017) y marcaje satelital también la sugieren (Guzmán and Félix, 2017).
En este trabajo presentamos resultados de comparaciones a nivel individual a partir de genotipos realizados a partir de muestras colectadas en diferentes localidades de alimentación y reproducción del Pacífico sudeste, incluyendo la costa de Ecuador, el Pacífico Colombiano y la Península Antártica.
ZONA DE ESTUDIO
Se colectaron muestras de piel de ballenas jorobadas durante la temporada de reproducción en Salinas, Ecuador, entre 2006 y 2010 (N=55), cerca de Nuquí, Golfo de Tribugá, Departamento del Choco, en la zona norte del Pacífico Colombiano entre 2015 y 2016 (N=43), en Bahía Málaga y la Isla Gorgona, Departamento del Valle del Cauca en el sur del Pacífico Colombiano (N=2), y en el Estrecho de Gerlache, en la Península Antártica, como parte de la primera expedición Colombiana a la Antártica en 2014 (N=14) (Tabla 1 y Figura 1).
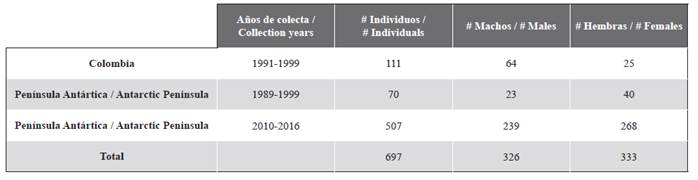
Tabla 1 Resumen de las muestras y de los perfiles de ADN de las zonas de reproducción y las zonas de alimentación de la subpoblación o stock G, que incluyen el sexo de las muestras. QC hace referencia a las muestras que cumplieron con los estándares de control de calidad y fueron incluidas en los análisis finales.
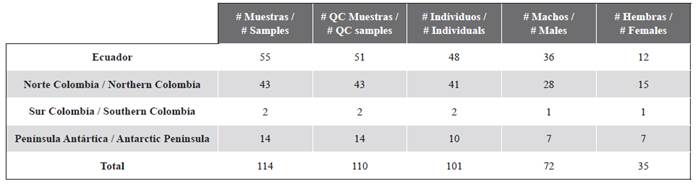
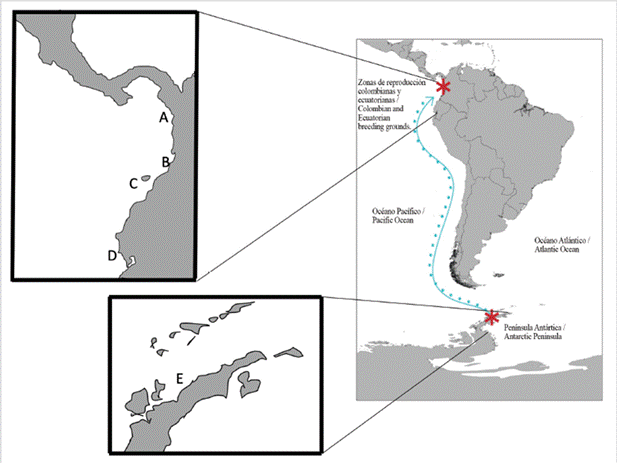
Figura 1 Localidades de muestreo en las zonas de alimentación y de reproducción para la subpoblación G: A. Nuquí, Departamento del Chocó, Colombia (Norte de Colombia), B. Bahía Málaga y C. Isla Gorgona Departamento del Valle del Cauca, Colombia, D. Salinas, Ecuador, y E. Estrecho de Gerlache, Península Antártica. En azul, ruta migratoria.
MATERIALES Y MÉTODOS
Se colectaron biopsias de piel utilizando el rifle modificado Paxarms (Krützen et al. 2002) y se colectó piel desprendida, flotando en el agua, utilizando una malla para atraparla (ver Félix et al., 2012). Las muestras fueron preservadas en etanol al 95 % y mantenidas 4 ºC hasta su análisis en el laboratorio. Se extrajo ADN total utilizando proteinasa K para digerir el tejido, seguido por un protocolo clásico de fenol-cloroformo (Sambrook et al. 1989). Se genotipificaron 15 loci microsatélites siguiendo los protocolos de Constantine et al. (2012) y de Steel et al. (2017) utilizando los primers o cebadores previamente publicados: Ev1, Ev14, Ev21, Ev37, Ev94, Ev96, Ev104 (Valsecchi y Amos 1996) GATA417, GATA28 (Palsboll et al. 1997) rw31, rw 4-10, rw48 (Waldick et al. 1999) and GT211, GT575, GT23 (Bérubé et al. 2000). La identificación individual fue confirmada con sexaje molecular y secuenciación de la región control del ADN mitocondrial (mtDNA) siguiendo los protocolos de Olavarría et al. (2007). Cada loci microsatélite fue amplificado por PCR de manera independiente y luego todos los productos de amplificación fueron combinados (multiplex) para su montaje en el secuenciador ABI 3730 xl DNA (Applied Biosystems), utilizando el marcador de peso 500LIZ™. La identificación de los alelos se realizó utilizando el programa ABI PRISM GeneMapper 4.0 (Life Technologies). Las muestras amplificadas para 10 loci o menos se consideraron de baja calidad y se eliminaron de la base de datos. Los genotipos de alta calidad fueron resueltos utilizando el programa CERVUS (Marshall et al. 1998) requiriendo una coincidencia exacta de al menos ocho loci, soportado por la información del sexo y el haplotipo mitocondrial. Los valores de la probabilidad (PI) de identidad se encontraron en el rango entre 2.1 x10-7 y 5.7 x10-12 para el criterio de mínimo de ocho loci coincidentes. Se realizaron comparaciones de los genotipos generados utilizando dos “registros” de ADN (definiendo como “registros” una base de datos electrónica de perfiles de ADN) para individuos de ballenas jorobadas previamente identificados para la zona de reproducción de la costa Pacífica de Colombia (N = 111; 1991-1999) y la zona de alimentación alrededor de la Península Antártica (N = 70; 1989-1999) mantenidas por el South Pacific Whale Research Consortium (Steel et al. 2017) y, por un acuerdo de colaboración, también se compararon con un archivo de 508 muestras individuales colectadas en la Península Antártica en años recientes (2010-2016; Pallin 2018). Adicionalmente, y con el fin de confirmar los patrones de estructura poblacional previamente descritos para la subpoblación del Pacífico sudeste, dicha estructura poblacional fue evaluada con el programa STRUCTURE® v. 2.3 (Pritchard, et al. 2000) que asigna individuos a grupos utilizando un modelo Bayesiano que minimiza el desequilibrio de ligamiento y las desviaciones del equilibrio de Hardy-Weinberg. Se corrió el modelo de panmixia con correlación de frecuencias alélicas para estimar el número más probable de grupos genéticos (K) en la muestra (length burn-in period: 10 000; MCMC: 100 000). Este programa evalúa diferentes valores de K utilizando diversos modelos de ancestría. Se corrieron diez réplicas en STRUCTURE y los resultados fueron visualizados en el programa Structure Harvester (Earl and vonHoldt, 2012). En este análisis se incluyeron 254 muestras de la Península Antártica, de Colombia y de Ecuador y para realizar comparaciones se incluyó un grupo de muestras de Brasil (n=50) (Cypriano-Sousa et al., 2016) y del Pacífico Sur (Nueva Caledonia, n = 50; Polinesia Francesa y Samoa Americana, n = 100; Tonga, n= 50) (Steel et al. 2017)
RESULTADOS
Ciento catorce muestras fueron obtenidas a partir de biopsias remotas o de piel desprendida en tres localidades del Pacífico sudeste y de la Península Antártica. Se buscó obtener perfiles genéticos de estas muestras para realizar comparaciones con los genotipos de los registros de ADN del South Pacific Whale Research Consortium, que incluyen 16 loci microsatélite, haplotipos de ADN mitocondrial y sexo (Steel et al. 2017). Para todas las muestras se logró determinar el sexo y el haplotipo mitocondrial, información utilizada para poder aclarar recapturas potenciales (Tabla 1). De los 15 microsatélites utilizados, uno (EV21) no amplificó apropiadamente y no fue incluido en los análisis. Cuatro muestras, todas de Ecuador, no amplificaron para lograr el mínimo de calidad requerida de 10 loci para análisis y no fueron incluidas en la base de datos.
Las comparaciones realizadas con el resto de los 110 genotipos revelaron nueve coincidencias (matches) dentro de las localidades, tres en Ecuador, dos en Colombia y cuatro en la Península Antártica. (Table 1). Todas estas recapturas se dieron dentro de una misma temporada de campo con la excepción de un individuo, que fue muestreado en Ecuador en 2009 y nuevamente en 2010. Luego de eliminar estas recapturas dentro de cada localidad, los 101 genotipos restantes fueron comparados con individuos muestreados en 181 Colombia (N = 111) y la Península Antártica (N = 70) 20 años antes, y con un catálogo genético contemporáneo de la Península Antártica (Pallin et al. 2017, N = 510; 2010 - 2016) (Tabla 2). Considerando el sobrelapamiento especial y temporal entre 10 individuos muestreados en la Península Antártica incluidos en este estudio y en el catálogo de Pallin et al. (2018), estas dos bases de datos fueron comparadas primero y luego se unieron para realizar comparaciones posteriores con otras colectas temporales y espaciales. Se identificaron dos recapturas; un individuo muestreado en 2013 y nuevamente en 2015 y una muestra obtenida en 2015 y nuevamente en 2016. Luego de quitar dichas recapturas, un total de 518 individuos se definieron en la colección contemporánea de genotipos de la Península Antártica. Al comparar estos genotipos con los perfiles de los 70 individuos colectados 20 años antes, se encontraron dos recapturas en la misma región con un espacio temporal de 18 años, una hembra muestreada en 1997 y nuevamente en el 2015 (Tabla 3). Por el contrario, no se encontró ninguna recaptura al comparar las bases de datos de genotipos obtenidos en años recientes en las localidades de la zona de reproducción (Colombia 2015-2017 y Ecuador 2006-2010) ni con el catálogo de genotipos de años anteriores (Colombia 1991-1999) (Tabla 3). Una comparación de genotipos entre las localidades de la zona de reproducción y las de alimentación reveló cinco recapturas (Tabla 3), una previamente reportada por et al. (2017), tres entre la base de datos de Colombia 1991-1999 y la base de datos de la Península Antártica 2010-2016 y una entre Ecuador y la Península Antártica 2010-2016 (Tabla 3).
Tabla 3 Recapturas individuales a partir de genotipos dentro y entre muestreos en la región. El número sobre la diagonal representa las recapturas entre años, dentro de la misma localidad. El número debajo de la diagonal representa recapturas entre las zonas de alimentación y reproducción. * representa una recaptura previamente descrita.

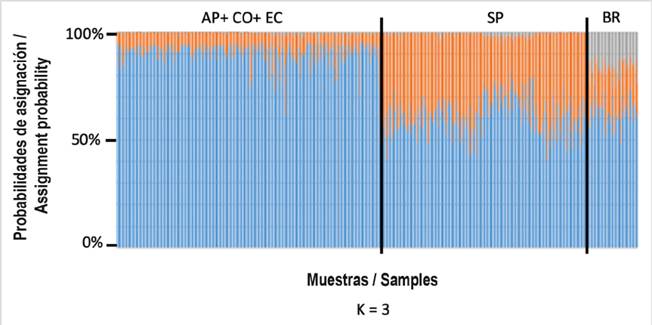
El análisis de estructura genética confirmó que todas las muestras analizadas pertenecían a una misma población (K=3) (Figura 2), confirmando la conectividad genética existente entre las zonas de alimentación alrededor de la Península Antártica y las zonas de reproducción, y mostrando diferenciación genética al realizar comparaciones con grupos de otras áreas del Hemisferio Sur (Pacífico Sur y Brasil), soportando lo previamente establecido por otros autores (Cypriano-Sousa et al. 2016; Steel et al. 2017).
DISCUSION
En este trabajo presentamos resultados de genotipificación individual y recapturas en muestras de ballenas jorobadas de diversas localidades de las zonas de reproducción del Pacífico Este y sus zonas de alimentación alrededor de la Península Antártica. Este es el primer trabajo presentando resultados sobre el uso de marcadores moleculares nucleares (microsatélites) para esta subpoblación o stock, ya que los trabajos anteriores sobre genética poblacional de estos grupos se habían enfocado en análisis de secuencias de la región control del ADN mitocondrial (Caballero et al. 2002, Olavarría et al. 2007). Estos resultados confirman conocimiento previo sobre la conectividad genética existente entre los grupos que se alimentan en la zona del estrecho de Gerlache en la Península Antártica y sus zonas de reproducción en la costa de Ecuador y el Pacífico Colombiano.
En este estudio se presenta información sobre recapturas genéticas individuales encontradas entre estas dos regiones. La mayoría de las recapturas encontradas en nuestros análisis fueron recapturas dentro de cada una las localidades de muestreo, pero hubo algunas interesantes para la misma localidad en años diferentes, tanto en zonas de alimentación como en zonas de reproducción, apoyando los datos presentados por Steel et al., (2017).
Adicionalmente, el encontrar recapturas entre zonas de reproducción y alimentación para muestras tomadas con una diferencia temporal de 20 años proporciona información inicial sobre la longevidad de estos animales y confirma la filopatría y la fuerte conexión migratoria existente entre estas áreas para el stock G. Así mismo, el no haber encontrado recapturas para muestras colectadas en diferentes localidades dentro de la zona de reproducción (Ecuador y el norte del Pacífico Colombiano vs sur del Pacífico Colombiano), parece soportar la hipótesis sobre estratificación poblacional dentro de las áreas de esta zona de reproducción (Félix et al., 2012, Guzmán and Félix, 2016). Estudios futuros deberán incluir muestras adicionales de estas áreas, así como muestras de animales que se alimentan en el Estrecho de Magallanes y que se reproducen potencialmente en Panamá y Costa Rica (Acevedo et al. 2017).
Análisis adicionales permitirán realizar la identificación de recapturas potenciales cuando estos datos sean comparados con registros de ADN para el Pacífico Sur y subpoblaciones adyacentes (e.g., Brasil, Cypriano-Souza et al. 2016) y ayudarán a informar sobre potencial migración entre Hemisferios cuando se puedan hacer comparaciones con muestras obtenidas de grupos del Pacífico Norte.
AGRADECIMIENTOS
Queremos agradecer al South Pacific Whale Research Consortium por permitirnos tener acceso a las bases de datos genéticas utilizadas para realizar estas comparaciones. Por el apoyo logístico para realizar la toma de muestras en el Estrecho de Gerlache, agradecemos al programa Antártico Colombiano y a D. Mojica. El apoyo financiero para realizar esta investigación fue otorgado por la Facultad de Ciencias, Universidad de los Andes (Proyecto Semilla Profesor, a S. Caballero) y de Oregon State University (a C. S. Baker)
REFERENCIAS
Acevedo, J., A. Aguayo-Lobo, J. Allen, N. Botero-Acosta, J. Capella, C. Castro, L. Dalla Rosa, J. Denkinger, F. Félix, L. Flórez-González, F. Garita, H.M. Guzmán, B. Haase, G. Kaufman, M. Llano, C. Olavarría, A.S. Pacheco, J. Plana, K. Rasmussen, M. Scheidat and E.R. Secchi. 2017. Migratory preferences of humpback whales between feeding and breeding grounds in the eastern South Pacific. Mar. Mam. Sci., 33: 1035-1052. [ Links ]
Baker, C. S. and L. Medrano-González. 2002. Worldwide distribution and diversity of humpback whale mitochondrial DNA lineages. In: Pfeiffer, C.J. (Ed.). Molecular and Cell Biology of Marine Mammals. Krieger Publishing, Malabar, Florida. [ Links ]
Baker, C. S., L. Flórez-González, R. B. Abernethy, H. C. Rosenbaum, R. W. Slade, J. Capella and J. L. Bannister. 1998. Mitochondrial DNA variation and maternal gene flow among humpback whales of the Southern Hemisphere. Mar. Mam. Sci., 14:721-737. [ Links ]
Bérubé, M., H. Jorgensen, R. McEwing and P. J. Palsboll. 2000. Polymorphic di-nucleotide microsattelite loci isolated from the humpback whale, Megaptera novaenagliae. Mol. Ecol., 9:2181-2183. [ Links ]
Caballero, S., H. Hamilton, L. Fórez-González, J. Capella, C. Olavarría, H. Rosenbaum and C. S. Baker. 2000. Stock identity and diversity of humpback whale mitochondrial DNA lineages on the Colombian winter breeding grounds. Internat. Whaling Com., report SC/52/IA14. 1-7 pp. [ Links ]
Caballero, S., Hamilton, H. Jaramillo, C., Capella, J., Flórez-González, L., Olavarría, C. Rosenbaum, H., Guhl, F. and C. S. Baker. 2001. Genetic characterization of the Colombian Pacific Coast humpback whale population using RAPD and mitochondrial DNA sequences. Mem. Queensland Mus., 47: 459-464. [ Links ]
Constantine, R., Jackson, J. A. , Steel, D., Baker, C. S., Brooks, L., Burns, D., Clapham, P., Hauser, N., Madon, B., Matilla, D., Oremus, M., Poole, M., Robbins, J., Thompson, K. and C. Garrigue. 2012. Abundance of humpback whales in Oceania using photo-identification and microsatellite genotyping. Mar. Ecol. Progr. Ser., 453: 249-261. [ Links ]
Cypriano-Souza, A. L., Engel, M. H., Caballero, S., Olavarría, C., Flórez-González, L., Capella, J., Steel, D., Sremba, A., Aguayo, A., Thiele, D., Baker, C. S. and S. L. Bonato. 2016. Genetic differentiation between humpback whales (Megaptera novaeangliae) from Atlantic and Pacific breeding grounds of South America. Mar. Mam. Sci., 33:457-479. [ Links ]
Earl, D. A. and B. M Vonholdt. 2012. Structure Harvester: a website and program for visualizing structure output and implementing the Evanno method. Cons. Gen. Res., 4: 359-361. [ Links ]
Félix, F., D. M. Palacios, S. K. Salazar, S. Caballero, B. Haase and J. Falconi. 2011. The 2005 Galápagos humpback whale expedition: a first attempt to assess and characterise the population in the Archipelago. J. Cet. Manag., 3:291-299. [ Links ]
Félix, F., S. Caballero and C. Olavarría. 2012. Genetic diversity and population structure of humpback whales (Megaptera novaeangliae) from Ecuador based on mitochondrial DNA analyses. J. Cet. Manag., 12:71-77. [ Links ]
Flórez-González, L., J. Capella, B. Hasse, G. Bravo, F. Félix and T. Gerrodette. 1998. Changes in winter destinations and the northernmost record of southeastern Pacific humpback whales. Mar. Mam. Sci., 14:189-196. [ Links ]
Garrigue, C., A. Aguayo, V. L. U. Amante-Helweg, et al. 2002. Movements of humpback whales in Oceania, South Pacific. J. Cet. Manag., 4:255-260. [ Links ]
Guzmán, H. M. and F Félix. 2017. Movements and habitat use by Southeast Pacific humpback whales (Megaptera novaeangliae) stellite tracked at two breeding sites. Aq. Mam., 43: 139-155. [ Links ]
International Whaling Commission. 1998. Annex G - Report of the sub-committee on the comprehensive assessment of Southern Hemisphere humpback whales. Report to the International Whaling Commission 48:170-182 [ Links ]
Krützen, M., L. M. Barré, L. M. Möller, M. R. Heithaus, C. Simms and W. B. Sherwin. 2002. A biopsy system for small cetaceans: darting success and wound healing in Tursiops spp. Mar. Mam. Sci., 18: 863-878. [ Links ]
Marshall, T. C., J. Slate, L. E. B. Kruuk and J. M. Pemberton. 1998. Statistical confidence for likelihood-based paternity inference in natural populations. Mol. Ecol., 7: 639-655. [ Links ]
Olavarría, C., C. S. Baker, C. Garrigue, et al. 2007. Population structure of South Pacific humpack whales and the origin of the eastern Polynesian breeding grounds. Mar. Ecol. Progr. Ser., 330: 257-268. [ Links ]
Pallin, L. J., C. S Baker, D. Steel, et al. 2018. High pregnancy rates in humpback whales (Megaptera novaeangliae) around the Western Antarctic Peninsula, evidence of a rapidly growing population. Roy. Soc. Open Sci., 5: 180017. [ Links ]
Palsboll, P. J., M. Bérubé, A. H. Larsen and H. Jorgensen. 1997. Primers for the amplification of tri-and tetramer microsatellite loci in baleen whales. Mol. Ecol., 6: 893-895. [ Links ]
Pritchard, J. K., M. Stephens and P. Donnely. 2000. Inferences of population structure using multilocus genotype data. Genetics, 155: 945-959. [ Links ]
Sambrook, J., E. F. Fritsch and T. Maniatis. 1989. Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory Press, New York, U.S.A. 1546 pp [ Links ]
Steel, D., M. Anderson, C. Garrigue, C. Olavarría, S. Caballero, S. Childerhouse, P. Clapham, R. Constantine, S. Dawson, M. Donoghue, L. Flórez-González, N. Gibbs, N. Hauser, M. Oremus, D. Paton, M. M. Poole, J. Robbins, L. Slooten, D. Thiele, J. Ward and C. S. Baker. 2017. Migratory interchange of humpback whales (Megaptera novaeangliae) among breeding grounds of Oceania and connections to Antarctic feeding areas based on genotype matching. Polar Biol. https://doi.org/10.1007/s00300-017-2226-9 [ Links ]
Stevick, P. T., A. Aguayo, J. Allen, et al. 2004. Migrations of individually identified humpback whales between the Antarctic Peninsula and South America. J. Cet. Res. Manag., 6: 109-113. [ Links ]
Valsecchi, E. and W. Amos. 1996. Microsatellite markers for the study of cetacean populations. Mol. Ecol., 5: 151-156. [ Links ]
Waldick, R. C., M. W. Brown and B. N. White. 1999. Characterization and isolation of microsatellite loci from the endangered North Atlantic right whale. Mol. Ecol., 8: 1763-1765. [ Links ]
Recibido: 04 de Septiembre de 2019; Aprobado: 21 de Abril de 2020











 text in
text in