Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Boletín de Investigaciones Marinas y Costeras - INVEMAR
Print version ISSN 0122-9761
Bol. Invest. Mar. Cost. vol.51 no.1 Santa Marta Jan./June 2022 Epub Aug 01, 2022
https://doi.org/10.25268/bimc.invemar.2022.51.1.1166
NOTA
GEN CITOCROMO OXIDASA I CONFIRMA LA PRESENCIA DEL NEMERTINO CARCINONEMERTES CONANOBRIENI, PARÁSITO DE LA LANGOSTA ESPINOSA DEL CARIBE (PANULIRUS ARGUS) EN COLOMBIA
1Grupo de Investigación MIKU, Universidad del Magdalena, Carrera 32 # 22-08, Santa Marta DTHC, Colombia. amyberbenhenriquez23@gmail.com
2Grupo de Investigación MIKU, Universidad del Magdalena, Carrera 32 # 22-08, Santa Marta DTHC, Colombia.
3Grupo de Investigación MIKU, Universidad del Magdalena, Carrera 32 # 22-08, Santa Marta DTHC, Colombia.
4Facultad de Ciencias Básicas, Programa de Biología, Universidad del Magdalena, carrera 32 # 22-08, Santa Marta DTHC, Colombia. sigmerquiroga@unimagdalena.edu.co
Morphological identifications complemented with molecular analysis are important for the delimitation of cryptic species. This document contains annotations on the molecular identification of the parasitic nemertine Carcinonemertes conanobrieni in the Caribbean spiny lobsters Panulirus argus. The collected nemerteans were found parasitizing ovigerous lobsters from the Gulf of Salamanca (Puebloviejo), Colombia. DNA was extracted from a specimen and a fragment of the mitochondrial gene Cytochrome Oxidase I (COI) was amplified using Folmer’s universal primers, the sequence was edited with ProSeq 3.5 and aligned with all the C. conanobrieni sequences available in GenBank. A sequence of 488 base pairs corresponding to the section of the genetic barcode of the COI gene was obtained, the matrix of genetic distances showed little variation when comparing the obtained sequence with those available in the GenBank database. These results confirm the presence of this nemertean in the Panulirus argus lobsters in Colombia providing valuable information for the correct identification and detection of this parasitic species in the country, thus facilitating its monitoring.
Keywords: Carcinonemertidae; COI; Puebloviejo.
Las identificaciones morfológicas, complementadas con análisis moleculares son importantes para la delimitación de especies crípticas. Este documento contiene anotaciones sobre la identificación molecular del nemertino parásito Carcinonemertes conanobrieni en langostas espinosas del Caribe Panulirus argus. Los nemertinos recolectados se encontraron parasitando langostas ovadas del golfo de Salamanca (Puebloviejo), Colombia. Se extrajo ADN de un individuo y se amplificó un fragmento del gen mitocondrial Citocromo Oxidasa I (COI) utilizando los primers universales de Folmer, la secuencia obtenida se editó con ProSeq 3,5 y alineó con todas las secuencias de C. conanobrieni disponibles en GenBank. Se obtuvo una secuencia de 488 pares de bases correspondientes a la sección del código de barras genético del gen COI, la matriz de distancias genéticas mostró poca variación al comparar la secuencia obtenida con las disponibles en la base de datos GenBank. Estos resultados confirman la presencia de este nemertino en las langostas Panulirus argus en Colombia, y aportan valiosa información para la correcta identificación y detección de esta especie parasita en el país, facilitando así su monitoreo.
Palabras claves: Carcinonemertidae; COI; Puebloviejo.
La langosta espinosa del Caribe Panulirus argus (Latreille, 1804) es un crustáceo decápodo distribuido en Colombia en los departamentos costeros de la Región Caribe, incluyendo el área insular de San Andrés, Providencia y Santa Catalina (Gracia y Díaz, 2002). Esta langosta es considerada uno de los recursos pesqueros más importante para las poblaciones de pescadores artesanales marinos ya que posee el mayor valor económico dentro de los crustáceos que se pescan en el gran Caribe y el Golfo de México (Seijo, 2007; FAO, 2015, 2019).
En departamentos como Magdalena, La Guajira y Archipiélago de San Andrés, Providencia y Santa Catalina, esta langosta es importante social y económicamente, ya que su extracción es esencial para el sustento de las comunidades de pescadores artesanales. Sin embargo, es considerada como una especie vulnerable en Colombia (Gracia y Díaz, 2002; Minambiente, 2017), debido a la sobreexplotación de su stock pesquero y a la destrucción sistemática de su hábitat. Adicionalmente, existen factores naturales que han contribuido a la disminución de las poblaciones de P. argus, tales como enfermedades (v. g. Panulirus argus virus 1-PaV1) (Shields y Behringer, 2004) y parásitos (v. g. nemertinos) (Atherley et al., 2020a). Específicamente el parásito Carcinonemertes conanobrieni (Simpson, Ambrosio y Baeza, 2017) es un nemertino que se alimenta de los embriones que las hembras ovadas cargan en sus pleópodos, ocasionando efectos negativos sobre el rendimiento reproductivo de la langosta (Baeza et al., 2016; Simpson et al., 2017).
En 2018 se registró por primera vez C. conanobrieni en las costas del Caribe de Colombia (González-Cueto y Quiroga, 2018), con base en el análisis morfológico de cuatro individuos encontrados en una langosta ovada del golfo de Salamanca (Magdalena). Sin embargo, era necesario complementar esta información con datos de secuencias genéticas, imprescindibles para delimitar especies crípticas dentro del filo (Sundberg et al., 2016). Además, en el caso de especies de nemertinos asociadas a P. argus, las identificaciones moleculares son necesarias ya que en esta langosta se encuentran al menos dos especies del género Carcinonemertes (Atherley et al., 2020b).
Se obtuvieron langostas ovadas de la especie P. argus capturadas por pescadores artesanales del golfo de Salamanca (10,97982 N; -74,32758 O), municipio de Puebloviejo (Magdalena) (Figura 1). Al examinarlas se aislaron nemertinos en diferentes etapas de desarrollo; algunos de los cuales se fijaron en ETOH al 100 %.
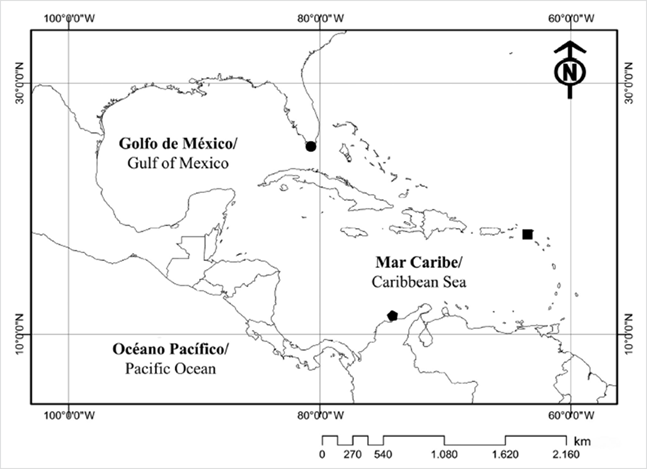
Figura 1 Registros de Carcinonemertes conanobrieni en el Golfo de México y el Mar Caribe. El círculo indica la localidad tipo en los Cayos de la Florida (24.7707 N; -80.7615 O - 24.7325 N; -80.9121 O); el pentágono corresponde al registro para la costa Caribe de Colombia (10.97982 N; -74.32758 O); y el cuadrado es el registro para Saint Kitts, West Indies (see Atherley et al., 2020b).
El ADN de uno de los individuos identificados como C. conanobrieni se extrajo utilizando el kit QuickExtract ™, siguiendo el protocolo del fabricante (Lucigen). La región “Folmer” se amplificó con primers universales (Folmer et al., 1994). Las extracciones de DNA se amplificaron usando 2 μL de ADN en un mix de PCR con un volumen final de 23 μL que contenía 1,25 μL MgCl2, 5 μL Buffer 5X (BIOLINE), 1 μL de cada primer, 0,625 μL de dNTP,s, 13,625 μL de H2O y 0,5 μL de Taq polimerasa (BIOLASETM, BIOLINE). El programa del termociclador fue: 1 min a 95 °C, seguido de 35 ciclos de 15 s 95 °C, 1 min a 40 °C, 1,5 min a 72 °C y un período de extensión final de 5 min a 72 °C. La secuencia obtenida fue editada con ProSeq 3,5 y alineada con todas las secuencias de C. conanobrieni disponibles en GenBank utilizando el algoritmo ClustalW en MEGA (Tamura et al., 2011). Otras secuencias de Carcinonemertes sp. registrados en P. argus por Atherley et al. (2020b) se consideraron en el análisis. Se construyó una matriz de distancias genéticas evolutivas intraespecíficas e interespecíficas (Hebert et al., 2003), usando el modelo K2P (Kimura, 1980).
Se obtuvo una secuencia de 488 pares de bases correspondientes a la sección del código de barras genético del gen COI, este se depositó en GenBank con el código de acceso MW205898. La secuencia obtenida tiene una distancia genética que varía entre 0 % y 0,2 % cuando se compara con otros individuos de C. conanobrieni y 22,5 % con otras especies de este género encontradas en P. argus (Tabla 1).
Tabla 1 Matriz de distancias genéticas interespecíficas e intraespecíficas construida con base en secuencias de 488 pb del gen Citocromo Oxidasa I (COI) utilizando el modelo de evolución de sustitución de nucleótidos K2P (Kimura, 1980). La secuencia generada en este estudio está resaltada en negrita; el resto de secuencias fueron extraídas de la base de datos GenBank (https://www.ncbi.nlm.nih.gov/).
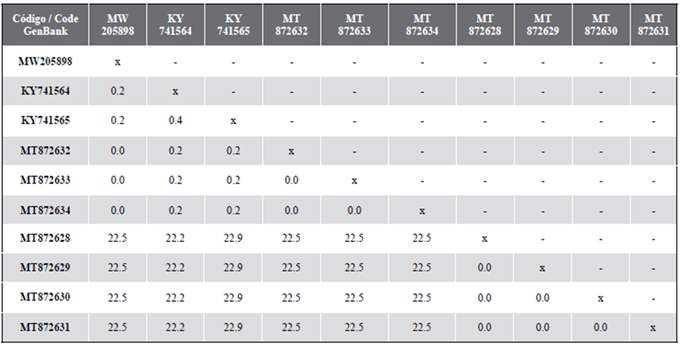
Estos resultados permiten la confirmación de la presencia de C. conanobrieni en Colombia, ya que la distancia genética hallada fue menor a 0,5 % si se compara con otros individuos de C. conanobrieni secuenciados por Simpson et al. (2017) en la descripción original de la especie. Dentro del grupo Hoplonemertea, el cual contiene al género Carcinonemertes, se estima que las distancias interespecíficas son superiores a 5 % (Sundberg et al., 2016).
La información presentada provee herramientas que permiten tener una correcta identificación de las especies de nemertinos que se pueden encontrar parasitando a la langosta. Los resultados de este estudio indican que se deben establecer programas de monitoreo para evaluar el efecto de C. conanobrieni en las poblaciones de langosta de Colombia. Para mantener los valores actuales y futuros de pesca y de mercado de P. argus, se deben considerar los patógenos y parásitos emergentes, particularmente a la luz de un entorno cambiante.
AGRADECIMIENTOS
Este trabajo derivó del proyecto “Efecto del parásito de crustáceos Carcinonemertes conanobrieni sobre el desempeño reproductivo de la langosta espinosa del Caribe, en un contexto de manejo pesquero participativo en el departamento del Magdalena, Colombia” financiado por Fonciencias 2017 de la Universidad del Magdalena, con resolución de permiso número 0080 del 17 de enero del 2019 por parte de la Corporación Autónoma Regional del Magdalena, Corpamag. Especiales agradecimientos a los pescadores artesanales del golfo de Salamanca, quienes ayudaron en la obtención de las langostas especímenes
REFERENCIAS
Atherley, N.A.M., M.A. Freeman and M.M. Dennis. 2020a. Post-mortem examination of the Caribbean spiny lobster (Panulirus argus, Latreille 1804) and pathology in a fishery of the Lesser Antilles. J. Invertebr. Pathol., 175:107453. https://doi.org/10.1016/j.jip.2020.107453 [ Links ]
Atherley, N.A.M., M.M. Dennis and M.A. Freeman. 2020b. Two species of Carcinonemertes Coe, 1902 (Nemertea: Carcinonemertidae) infesting the Caribbean spiny lobster, Panulirus argus (Latreille, 1804) (Decapoda: Achelata: Palinuridae), in Saint Kitts, West Indies. J. Crust. Biol., 40(6): 933-942. https://doi.org/10.1093/jcbiol/ruaa060 [ Links ]
Baeza, J.A., L. Simpson, L.J. Ambrosio, N. Mora, R. Guéron and M.J. Childress. 2016. Active parental care, reproductive performance, and a novel egg predator affecting reproductive investment in the Caribbean spiny lobster Panulirus argus. BMC Zool., 1(6). https://bmczool.biomedcentral.com/articles/10.1186/s40850-016-0006-6 [ Links ]
FAO. 2015. Report of the first meeting of the OSPESCA/WECAFC/ CRFM/CFMC Working group on Caribbean spiny lobster. FAO Fisheries and Aquaculture Report SLC/FIPS/ SLM/R1095 (Bi), Panama City, Panama. 112 p. [ Links ]
FAO. 2019. Report of the second meeting of the OSPESCA/WECAFC/CRFM/CFMC Working Group on Caribbean Spiny Lobster. FAO Fisheries and Aquaculture Report, Santo Domingo. 68 p. [ Links ]
Folmer. O., M. Black, W. Hoeh, R. Lutz and R. Vrijenhoek. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Mol. Mar. Biol. Biotechnol., 3(5): 294-299. [ Links ]
González-CuetoJ.A. and S. Quiroga. 2018. First record of Carcinonemertes conanobrieni Simpson, Ambrosio & Baeza, 2017 (Nemertea, Carcinonemertidae), an egg predator of the Caribbean spiny lobster Panulirus argus (Latreille, 1804), on the Caribbean coast of Colombia. Check List, 14(2): 425-429. https://doi.org/10.15560/14.2.425 [ Links ]
Gracia, A. y J. M. Díaz. 2002. Panulirus argus: 113-115. En Ardila, N., G. R. Navas y J. Reyes (Eds.). Libro rojo de los invertebrados marinos de Colombia. Invemar. Ministerio del Medio Ambiente. La serie Libros rojos de especies amenazadas de Colombia. Bogotá. 177 p. [ Links ]
Hebert, P. D. N., S. Ratnasingham and J. R. de Waard. 2003. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species. Proc. R. Soc. Lond. B. Biol Sci., 270 (Suppl 1): S96-S99. https://doi.org/10.1098/rsbl.2003.0025 [ Links ]
Kimura, M. 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. J. Mol. Evol., 16(2):111-120. https://doi.org/10.1007/BF01731581 [ Links ]
Minambiente. 2017. Resolución 1912 de 2017. Por la cual se establece el listado de las especies silvestres amenazadas de la diversidad biológica colombiana continental y marino costera que se encuentran en el territorio nacional, y se dictan otras disposiciones. Ministerio de Ambiente y Desarrollo Sostenible, fecha de expedición: 15/09/2017_ Diario Oficial No. 50364 del 22 de septiembre de 2017. [ Links ]
Seijo, J.C. 2007. Considerations for management of metapopulations in small-scale fisheries of the Mesoamerican barrier reef ecosystem. Fish. Res., 87(1): 86-91. https://doi.org/10.1016/j.fishres.2007.06.016 [ Links ]
Shields, J. D. and Behringer, D. C. 2004. A new pathogenic virus in the Caribbean spiny lobster Panulirus argus from the Florida Keys. Dis. Aquat. Organ., 59(2):109-118. https://doi.org/10.3354/dao059109 [ Links ]
Simpson, L.A., L.J. Ambrosio and J.A. Baeza. 2017. A new species of Carcinonemertes, Carcinonemertes conanobrieni sp. nov. (Nemertea: Carcinonemertidae), an egg predator of the Caribbean spiny lobster, Panulirus argus. PLoS One, 12(5): e0177021. https://doi.org/10.1371/journal.pone.0177021 https://doi.org/10.1371/journal.pone.0155541 [ Links ]
Tamura, K., D. Peterson, N. Peterson, G. Stecher, M. Nei and S. Kumar. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol. Biol. Evol., 28(10): 2731-2739. https://doi.org/10.1093/molbev/msr121 [ Links ]
Recibido: 23 de Marzo de 2020; Aprobado: 28 de Octubre de 2021











 text in
text in 



